基于高通量测序数据的插入/删除新突变检测方法
邢文昊, 刘永壮, 王亚东
(哈尔滨工业大学 计算机科学与技术学院, 哈尔滨 150001)
0 引 言
新突变是指并非遗传自亲代,而是在子代中首次出现的基因组变异,这种变异既可能来自于生殖细胞,也可能来源于胚胎发育早期的受精卵。大量研究表明新突变与人类疾病的发生密切相关,尤其是自闭症、癫痫等精神类疾病[1]。随着高通量测序技术的迅猛发展,基于高通量测序数据进行新突变检测已经成为常规手段。目前的基于高通量测序数据的新突变检测方法大概可以分为2类。一类是在亲代和子代样本中独立地进行基因组变异检测,然后通过比较子代和亲代的基因型发现新突变;另一类是对亲代和子代基因组数据建立联合检测模型直接检测新突变[2]。例如PolyMutt[3]和Triodenovo[4]均为基于似然度的新突变检测方法,该方法分别计算无孟德尔遗传约束和有孟德尔遗传约束下的最大基因型似然度,然后根据似然比确定候选位点是否是新突变,似然比越大,候选位点是新突变的可能性越高;DeNovoGear[5]是一种基于贝叶斯的新突变检测方法,该方法利用贝叶斯公式计算候选位点是新突变的后验概率,并根据后验概率的大小来判断候选位点是否是新突变;DNMFilter[2]是一种基于Gradient Boosting[6]的新突变检测方法,该方法利用Gradient Boosting作为分类器,从子代和亲代序列比对上下文选择序列特征,该方法相比其它方法能够显著降低新突变的错误发现率。在上述4种方法中,PolyMutt、Tridenovo均能够检测单核苷酸新突变和插入/删除新突变,但插入/删除新突变的检测准确率明显低于单核苷酸新突变的检测准确率;而DNMFilter只能够检测单核苷酸新突变而不能检测插入/删除新突变。
本文提出一种基于Adaboost的插入/删除新突变检测方法。该方法利用Adaboost作为分类模型;该方法对read比对之后的家系基因组数据进行局部重新拼接,并从局部拼接后的read比对上下文中提取序列特征;该方法利用已经验证的插入/删除新突变和随机选取的错误插入/删除新突变构建训练集。最后本文将训练的Adaboost模型应用于千人基因组计划CEU家系基因组测序数据,并同常用的新突变检测方法进行比较。
1 Adaboost算法
Adaboost算法[7]是一种经典的boosting算法,其主要思想是将一系列较弱的分类器结合起来,形成具有较强分类能力的组合分类器(强分类器)。
假设N个被标记的样本数据集合{(X1,Y1), (X2,Y2), (X3,Y3),…,(XN,YN)}为训练集,Xi为特征向量,Yi为类别标记,定义Wk(i)为第K次循环样本i的权重(K的取值范围为1,2,3,…,Kmax,其中Kmax为最大循环次数)。Adaboost算法流程可表述如下。
首先,初始化W1(i)=1/N(其中i=1,2,3,…,N)。
然后,在每一轮循环中根据样本权重Wk(i),在训练集中进行样本重采样生成子集Dk。
最后,在子集Dk上训练弱分类器Ck,计算Ck在训练集上的训练误差Ek,并且根据本轮的分类结果计算下一轮训练的权重Wk+1(i),研究推得其数学运算公式如下:

(1)
其中,Zk是归一化系数;hk(xi)是分类器Ck对特征向量Xi分类的标记;αk的数学求值则可写作如下形式:
(2)
使用更新后的权重Wk+1(i)重复上述过程,当循环次数达到最大循环次数Kmax或者是误差率小于某一阈值时停止,样本被划分类别可使用以下公式做出判断:
(3)
Adaboost算法可以有效降低分类中的偏差和方差[8],而与其它boosting算法相比,Adaboost对过拟合有着更强的抗性[9]。选择Adaboost算法可以有效降低假阳性错误,进而减少过滤结果中的假阳性位点的数量。
2 基于Adaboost的插入/删除新突变过滤方法
基于Adaboost的插入/删除新突变检测方法总体流程如图1所示,使用千人基因组计划的家系基因组数据作为实验数据。首先使用已有的新突变检测工具检测出候选的插入/删除新突变位点,并对候选位点进行局部从头拼接(local de novo assembly);然后结合已经存在的真实突变位点,创建带标签的候选位点集合,根据候选位点从重组的家系基因组数据中提取特征构建训练集;最后使用训练集对模型进行调整优化,并且用测试集来验证模型的效果。

图1 插入/删除新突变检测流程图
2.1 数据集
本文使用千人基因组计划CEU家系全基因组测序数据进行模型构建与实验结果分析,该数据集的BAM文件可以从此处:ftp://ftp-trace.ncbi.nih.gov/1000genomes/ftp/technical/working/20120117_ceu_trio_b37_decoy/下载得到。此外,该数据集包含了经过生物实验验证的53个插入/删除新突变(6个插入突变,47个删除突变)[5],这些突变位点的位置信息可以在http://www.nature.com/nmeth/journal/v10/n10/extref/nmeth.2611-S2.xls获得。
2.2 训练集构建
本文构造训练集应该满足的条件是真实的新突变尽可能集中分布,假的新突变在空间分布上应该与新突变尽可能分离,并且在空间中尽可能分布广泛。
本文构造正例集的具体方法是:首先将2.1节中经生物实验验证的53个真实新突变加入到正例集;由于正例集规模较小,无法满足模型构建的需求,本文模拟部分插入/删除新突变并将其加入到正例集中,此后接续的模拟过程就是选择父本(或母本)和子代是野生型且母本(或父本)是杂合型的位点,将母本(或父本)与子代进行交换,即可模拟出满足亲代是野生型、子代是杂合型的新突变位点。
本文构造反例集的具体方法是:从除去经过生物实验验证的53个真实新突变的候选新突变位点集合中,随机选取一定量的位点加入到反例集;从胚系基因组变异(Germline Variation)位点集合,随机选取一定量的位点加入到反例集中。
2.3 序列特征选择
研究中选定了55个特征来训练模型,这些特征分为4个部分。就整体而言,第一部分是父本read的特征,第二部分是母本read的特征,第三部分是子代read特征,第四部分是公共特征。其中,前三部分特征都是相同的,所选用的特征见表1。

表1 插入/删除新突变检测选择的序列特征描述信息
3 实验结果与分析
3.1 Adaboost与Gradient boosting性能比较
Gradient boosting和Adaboost一样,也是一种常用的boosting方法,两者的主要区别是损失函数的不同,直观上看前者是由梯度下降来决定,后者是由高权重的点来决定。Adaboost可以看作是Gradient boosting的特殊情况[10]。
为了测试比较两者的运行效果,在同一组数据上分别使用Gradient boosting和Adaboost进行了实验,如图2所示,纵轴表示百分比,横轴表示cutoff(cutoff是分类时使用的值,小于此值被分为假的插入/删除突变,大于此值被划分为真的插入/删除突变,实验中需要对其进行设置),FP表示cutoff-假阳性率曲线,TP表示cutoff-真阳性率曲线。
在图2所示的曲线中,GBFP在0~0.5下降比较快,ADATP在0.5以后下降比较快,但是所设定的cutoff都大于0.5(只有2个类别: 1个是真的插入/删除突变,1个是假的插入/删除突变),在0.5以后的区域Adaboost的表现比较好,可以筛选掉大部分的假阳性的插入/删除新突变。在cutoff-TP曲线上,尽管GBTP表现比较好,在0.97左右才出现了突然下降,即大量的真实插入/删除新突变开始被错分为假的插入删除突变,而ADAFP在0.84左右就已经开始下降,但是考虑到设置cutoff时,为了防止原本数量就很少的真实的插入/删除新突变被错判为假的插入删除突变,所以这个值不会过高,尽管Adaboost在cutoff-TP曲线上下降较早,但是和Gradient boosting一样满足精度要求,可以选择设置在0.6~0.8之间。
3.2 基于Adaboost的插入/删除新突变检测模型评估
为了考察这些特征对于分类的重要性,本文选择部分正例和反例,使用R语言中的adabag包对其进行训练,然后提取各个特征对于分类的相对重要性,设计结果如图3所示,在本组数据中,父本等位基因平衡性、父本和子代PValue值、母本等位基因平衡性、Homopolymer、父本平均mapping质量等因素对于分类的影响比较大,而子代链方向等因素影响较小。
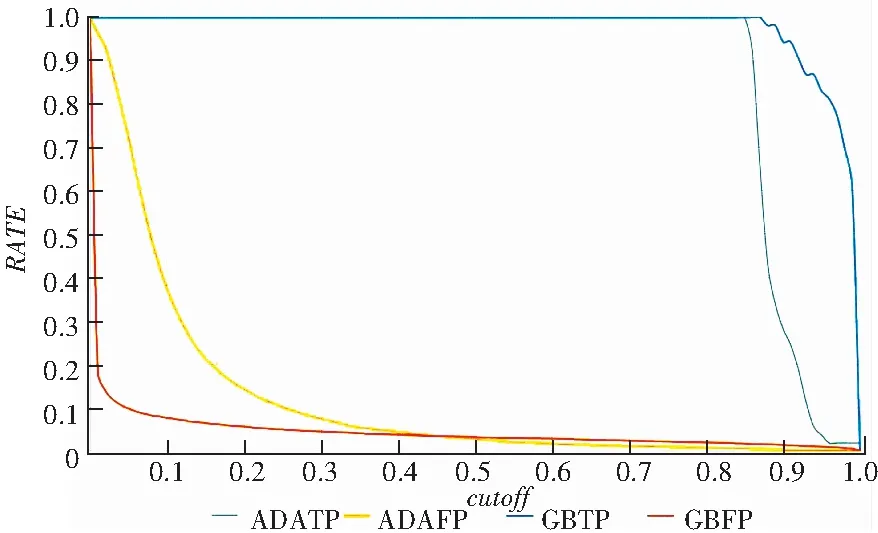
图2 基于Adaboost与基于Gradient boosting构建模型对比
Fig. 2 Comparison of model based on Adaboost and Gradient boosting

图3 用于插入/删除新突变检测的各特征相对重要性
为了评价构造的训练集的分类性能,对所选择的训练集进行主成分分析,最终绘制后则如图4所示,前三个主成分之和达到了75%以上。其中,红色的点代表了正例集的数据,蓝色代表反例集的数据。可以看出正例样本与反例样本能够明显区分开,表明了构建的训练集和选择的分类特征能够较好区分真实的插入/删除新突变和假的插入/删除新突变。
3.3 千人基因组计划家系数据的实验结果与分析
将本模型与其它常用的模型进行了对比,研究得到的对比后结果详见表2。其中,Triodenovo是基于似然模型对新突变检测的工具,相较于Polymutt的运行,性能要更胜一筹。DeNovoGear 是基于似然模型对新突变进行检测的工具,GATK PhaseByTransmission用于矫正家系中的模糊的位点。使用4种方法对同一个家系基因组数据进行处理,各个方法使用默认的参数,检出总数目中本文设计实现的DNINDELFilter具有最小的数目,而且DNINDELFilter检测出了数据中所有的53个真实的插入/删除新突变。从而达到了保留所有的真实插入/删除新突变,保证真阳性率,同时尽可能删除较多的假的插入/删除新突变,降低假阳性率的预期目标。

图4 训练集数据主成分分析
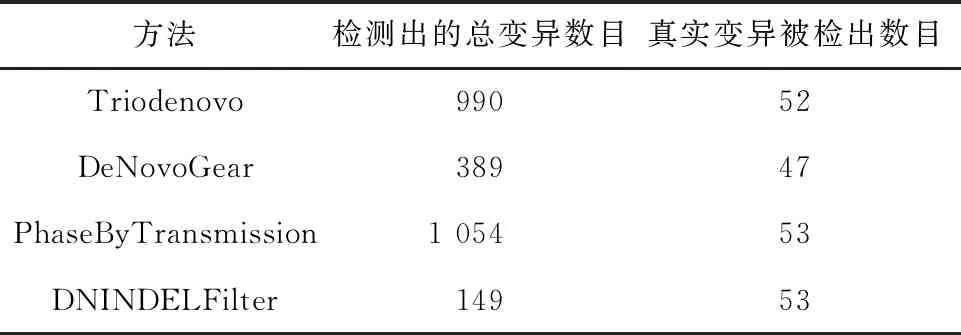
表2 新突变检测方法比较
4 结束语
本文将Adaboost算法应用在插入/删除新突变筛选中,并且应用Adaboost算法,实现了DNINDELFilter。在此研究过程中,选择了与插入/删除新突变相关的55个特征作为训练特征,对高通量测序数据进行了局部重新组装,通过构建训练集、继而训练、测试该模型、及对模型参数辅以一定调整,最后使得模型能够在保留大部分的真实插入/删除新突变同时,筛选掉绝大多数的假的突变,并且本文提出的模型具有一定的抗过拟合能力,筛选结果表现出较高的可信度。在本文的研究基础之上,未来的工作可以在如下方面展开:应深入探讨、并进一步优化模型中的特征,使得模型对插入/删除新突变具有更好的筛选能力;目前该模型在插入/删除新突变检测中取得了良好的效果,后续可以尝试将本文的模型做出研究改进,并应用到结构新突变筛选中。

