PSO_BFA优化词袋模型及蛋白质亚细胞定位预测
胡雪娇,陈行健,赵 南,薛 卫
南京农业大学 信息科学技术学院,南京210095
1 引言
词袋模型(Bag of Words model,BOW)源自于文本处理领域,研究人员不断发掘其潜能,在物体识别领域中[1-2]也有广泛应用。BOW 模型一般由特征提取、聚类分析构建字典、直方图构建三部分组成,将目标对象转换为特征向量送入分类器完成分类,最终将目标对象转换为特征向量送入分类器完成分类[3]。为提高BOW模型的性能,提出了许多优化算法。如Irfan等人[4]通过对字典降维并使用TF_IDF(Term Frequency_Inverse Document Frequency)算法赋予相应词权重,解决了文本错误匹配问题;Li 等人[5]使用空间金字塔匹配技术(Spatial Pyramid Matching,SPM),在图像表示阶段加入局部特征的空间位置信息,从而提高了分类性能。Xie 等人[6]通过分析邻域像素和局部图像,引入LQP(Local Quantized Pattern)构建字典,取得了较好效果;Zhu等人[7]利用模糊均值(Fuzzy C-Means)代替K 均值(K-Means)优化字典,使初始聚类中心的选择更加合理;汪荣贵等人[8]引入显著区域提取,结合三角剖分方法融入图像全局信息,得到了较好的预测效果。
当前众多词袋模型的算法改进虽然取得了良好的效果,但是忽略了对词袋模型的参数的优化,其中参数大多根据经验值选取。在使用BOW 模型完成特征提取的过程中,存在窗口大小d 和字典大小k 组成的一组参数,对BOW 的性能影响非常大。本文结合群体智能化的粒子群算法(Particle Swarm Optimization,PSO)和全局寻优能力强的细菌觅食算法(Bacterial Foraging Algorithm,BFA),在PSO进行局部搜索时,加入BFA的复制和迁移行为,求得混合算法PSO_BFA 的最优解为窗口大小d 和字典大小k 的最佳组合。将优化后的BOW 模型应用到蛋白质亚细胞定位预测中,结合蛋白质序列的氨基酸组成和伪氨基酸组成获得蛋白质序列的词袋特征[9],最后送入支持向量机多类分类器进行定位预测,实验证明PSO_BFA 优化的词袋模型能有效提高蛋白质亚细胞定位预测的精度。
2 PSO_BFA优化词袋模型
词袋模型最初用于文本建模,是一种利用机器学习算法对文本数据进行特征表达的方法。其基本原理是将目标对象用若干无序单词来表示,这些单词组成一个集合。通过统计每个单词在文档中出现的次数对目标对象进行向量化描述。应用词袋模型进行某项具体研究通常分为以下几个步骤:首先对目标对象进行局部区域分析,并提取局部特征作为特征单词训练字典。此时存在一个窗口值d,即特征单词的长度。在获得目标对象的局部特征后,需要对这些特征单词进行聚类,获取一定数量的聚类中心作为字典,通常使用K-means 聚类[10],字典大小k 的取值决定了最终的聚类效果。利用字典将目标对象的各个局部特征映射到与之距离最近的聚类中心,然后统计目标对象属于各个聚类中心的特征单词个数,获得单词直方图,计算目标对象中每个特征单词出现的频率,即为最终的词袋特征。在传统词袋模型中,窗口值d 和字典大小k 值的选取对词袋模型的性能影响较大,针对这一问题,提出一种基于PSO_BFA优化的改进词袋模型,寻找窗口大小d 和字典大小k 的最佳组合。
2.1 粒子群算法
粒子群算法是由文献[11]提出的一种新型群体智能进化算法,具有记忆最佳位置、共享信息等特点。在PSO 优化问题中,每一个解即为一个粒子,每个粒子在N 维搜索空间中按照一定的速度运动,通过适应度函数来评价粒子的优劣,粒子根据自己的运动经验以及其他粒子的运动经验,来动态调整运动速度和位置,向搜索空间中最优的位置运动,从而得到优化问题的最优解。粒子通过跟踪两个“极值”来更新状态[12]:第一个是整个种群当前时刻找到的最优解,叫作全局极值Gbest ;第二个是粒子自身所找到的最优解,叫作个体极值Pbest。在N 维搜索空间中第i 个粒子的位置和速度分别表示为Xi=(xi1,xi2,…,xiN)和Vi=(vi1,vi2,…,viN)T。通过评价各粒子的目标函数,确定t 时刻每个粒子的最佳位置(Pbest)Pi=(pi1,pi2,…,piN)T以及群体所发现的最佳位置(Gbest)Pg=(pg1,pg2,…,pgN)T。
2.2 细菌觅食算法
细菌觅食算法是由Passino等人[13]于2002年基于细菌觅食行为过程而提出的一种随机搜索的智能仿生优化算法。细菌的觅食过程主要描述为趋向行为、复制(繁殖)行为和迁移(驱散)行为。其中复制行为使细菌觅食算法拥有优胜略汰的特点。淘汰较差的半数细菌,保留较好的半数菌体并复制产生新细菌。假设细菌种群规模为M ,群体中要淘汰的细菌数为Mr=M/2。在淘汰过程中首先将群体中的细菌个体按照自身的适应值好坏进行排序,将适应值较差的Mr 个细菌淘汰,剩下的Mr 细菌个体进行自身复制,即复制出与原来的个体一样的新个体,从而保持群体总数保持不变[14]。
BFA算法中的细菌驱散过程,能够加强细菌觅食算法的全局寻优能力,避免细菌陷入局部最优。细菌的驱散操作是在菌体经过数次复制行为之后发生的。驱散操作会根据一定的迁移概率Ped来决定是否驱散。当迁移概率Ped满足驱散条件时,则将随机地把细菌驱散到其所在搜索空间中去。由以上过程可看出BFA 算法有群体智能算法的全局寻优、易计算等优点[15]。
2.3 PSO_BFA优化词袋模型优化实现
PSO进行群体、协同搜索,记忆个体和群体信息[16],具有相当快的逼近最优解的速度,原理简单,通用性强,不依赖问题信息,易实现[17]。缺点是在局部的搜索准确性不高,且易陷入局部最优[18]。BFA在沿某一方向进行搜索的过程中,可根据适应值的变化调整运动方向,具有变方向搜索能力[19],使细菌个体易找到所在邻域内的最优值,不易错过更优解所在的区域,大大提高了对全局的搜索精度和搜索能力[20]。主要缺点是没有对菌群最优信息的记忆功能,全局搜索能力弱且收敛速度慢[21]。混合粒子群和细菌觅食算法弥补了两种算法的缺陷,将细菌觅食算法的复制和迁移行为应用到粒子群算法中提高算法的全局搜索能力和收敛速度。
在运用BOW 模型提取特征的过程中,存在两个重要参数:窗口大小d 和字典大小k。为优化窗口大小d和字典大小k 的取值过程,将d 的取值范围作为横向坐标,k 的取值范围作为纵向坐标构建参数搜索空间,每一组(d,k)值对应一组特征向量,每一组特征向量具有相应的分类准确率。将求得最高分类准确率的过程抽象为粒子寻求最高适应度值的过程,(d,k)为粒子在搜索空间的位置。因此,d 、k 值的选取过程转化为粒子的移动过程,可以利用PSO 和BFA 的群体智能寻优方法实现BOW 模型的参数寻优,从而获得识别精度较高的特征向量。
鉴于以上分析,在PSO 进行局部搜索时,加入BFA的复制和迁移行为,如果粒子的适应度值有增长趋势,则按原始方式更新速度和位置。如果大多粒子的适应度值没有明显提高,保留适应度值较高的半数粒子,去除适应度值较低的另半数粒子,留下的半数粒子复制产生新粒子,保持粒子总数不变。然后将粒子随机驱散到搜索空间的其他位置,进行下一阶段的搜索。解空间由窗口大小d 和字典大小k 构成,粒子的适应度值即为该组(d,k)计算出的分类准确率。
PSO-BFA混合优化算法流程如图1所示。

图1 PSO_BFA混合优化算法流程图
设计实现的算法思想:在一个两维的目标搜索空间中,由T(T >10)个粒子构成一个群体,其中第i 个粒子的位置表示为Xi=(d,k),d 为窗口大小,k 为字典大小。每个粒子的位置就是一个潜在的解。首先初始化一群随机粒子,通过迭代找到适应度值最高的解,在迭代过程中,粒子通过跟踪自身最优解(Pbest)Pi=(pi1,pi2,…,piN)T和整个种群的最优解pg2,…,pgN)T更新自己的位置[13]。用公式表示粒子的速度和位置更新如下:
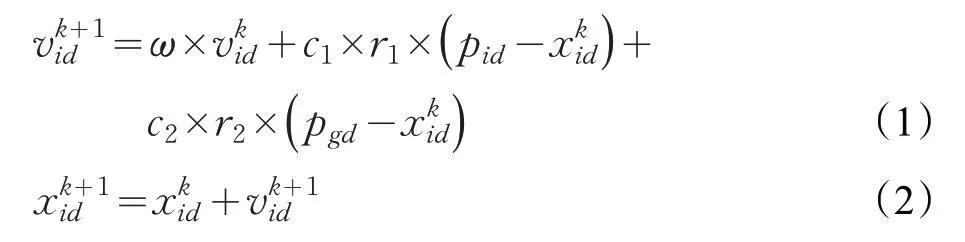
式中,i=1,2,…,M,M 为粒子总数;d=1,2,…,N,N 为解空间维数;ω 为惯性因子;是粒子的速度向量;c1、c2为加速因子;r1和r2是均匀分布在[0,1]上的随机数,互相独立;是当前粒子的位置;pid表示当前粒子本身最优解的位置;pgd表示当前整个种群最优解的位置[14]。
输入:惯性因子c1、c2,最大迭代次数Tmax,粒子个数M ,每代复制粒子数和淘汰粒子数为M/2,窗口大小d 和字典大小k 的取值范围,粒子速度范围[-vdmax,vdmax],迁移概率Ped=0,取值根据不同情况的经验值进行选取。
输出:种群最优值Gbest 及位置Pg。
算法步骤如下:
(1)设置初始种群,初始化各参数,根据窗口大小d和字典大小k 的取值范围设置粒子位置范围[xmin,xmax],在位置范围内随机初始化粒子的位置Xi以及速度Vi,用Pi记录粒子当前位置为粒子初始最优位置,设置迁移概率Ped=0。
(2)循环操作:for i=1,2,…,Tmax,如果i ≤Tmax循环继续,否则循环结束。
(3)根据SVM 分类预测结果评价各粒子的适应度值,用Gbest 记录当前种群最优的适应度值,相应粒子位置为当前最优位置记做Pg,用Pbest 记录个体最优适应度值,然后根据公式(1)来更新粒子的速度,通过公式(2)更新粒子的位置。
(4)更新粒子速度位置后,则根据分类预测结果重新评价粒子的适应度值,若优于更新前的粒子,则更新该粒子的Pi为粒子当前位置。若某粒子位置更新后成为种群适应度值最高的粒子且优于已有的Gbest ,则更新Gbest ,种群最优Pg更新为该粒子的位置。若更新后最高适应度值低于上一代Gbest,则Ped=Ped+0.1。Ped值按一定粒度值增加,是为了防止粒子陷入局部最优发生早熟的现象,将SVM 分类预测结果终分类结果作为粒子适应度值,根据适应度值来动态更新迁移概率值,经多次实验得出粒度设为0.1效果最佳。
(5)如果Ped<0.5,转向下一步,否则,保留适应度值较高的M/2 个粒子,淘汰适应度值较低的另M/2 个粒子,留下的M/2 个粒子复制产生新粒子,新粒子属性与原粒子相同,粒子总数不变,然后将粒子随机驱散到搜索空间的其他位置。迁徙概率Ped的取值范围在0到1之间,设置值过大会导致算法在搜索空间中变成随机穷举搜索。因此,根据经验值将Ped的最大值设为0.5。
(6)i=i+1,转向步骤(2)。
(7)循环结束,输出种群最优解Gbest ,以及种群最优位置Pg。
为检验优化词袋模型的性能,将其应用到蛋白质亚细胞定位预测研究中,运用优化词袋模型提取蛋白质序列特征,然后将特征值送入支持向量机进行分类预测,并将分类准确率作为适用度值,对每一组(d,k)值对应的词袋特征值进行评价。
3 实验与分析
3.1 数据集
采用两个凋亡蛋白数据集ZD98和CH317,ZD98数据集由Zhou 和Doctor[22]构建,分为4 个亚细胞定位类别,共有98 条蛋白质序列,分别是线粒体蛋白(Mitochondrial proteins,mi)13 条、细胞质蛋白(Cytoplasmic proteins,cy)43 条、膜蛋白(Membrane proteins,me)30条和其他类蛋白(other)12条。CH317数据集是由Chen和Li[23]构建,分为6个亚细胞定位类别,共有317条蛋白质序列,分别是分泌蛋白(Secreted proteins,se)17 条、细胞核蛋白(Nuclear proteins,nu)52 条、细胞质蛋白(Cytoplasmic proteins,cy)112 条、内质网蛋白(Endoplasmic reticulum proteins,en)47 条、膜蛋白(Membrane proteins,me)55 条和线粒体蛋白(Mitochondrial proteins,mi)34条。
3.2 参数选择
对实验的PSO_BFA 优化算法参数进行设置,惯性因子c1=c2=2,最大迭代次数Tmax=1 000,粒子个数M =50,每代复制粒子数和淘汰粒子数为M/2,窗口大小d 取值范围设为[L/5,L],其中L 为数据集中最短蛋白质序列的长度,字典大小k 取值范围设为[20,500],k,d 值均取整数,粒子速度范围[-vdmax,vdmax] ,其中vdmax=k ⋅xdmax,0.1 ≤k ≤0.2,xdmax为d 维空间上限,迁移概率Ped初始化为0。
3.3 检验方法与评价指标
首先根据优化后的词袋模型提取蛋白质序列的词袋特征。实验借助Lin等人[24]设计开发的LIBSVM工具箱构造SVM 多类分类器,采用一对一的构造方法为任意两类样本构造一个SVM分类器。对一个未知样本进行分类时,根据当前类别投票数来判别该样本属于哪一类别,采用Jackknife进行假设检验。Jackknife是亚细胞定位预测中应用最多的检验方法,基本原理为:先从数据集中取出一条蛋白质序列作为测试序列,其余蛋白质序列作为训练集,一次测试结束后将这条测试序列放回数据集。第二轮检验,取出下一条蛋白质序列作为测试序列,剩下的做训练集,以此类推,直到数据集中所有序列都测试完毕[25]。测试次数等于数据集的大小,这种检验方法具有最小的任意性,是一种客观有效的交叉验证方法[26]。
为了便于比较蛋白质亚细胞区间预测的结果,对实验方法进行有效评估,引入敏感性Sn、特异性Sp和相关系数MMCi这3个指标进行评价,并统计总的准确率A,定义如下:
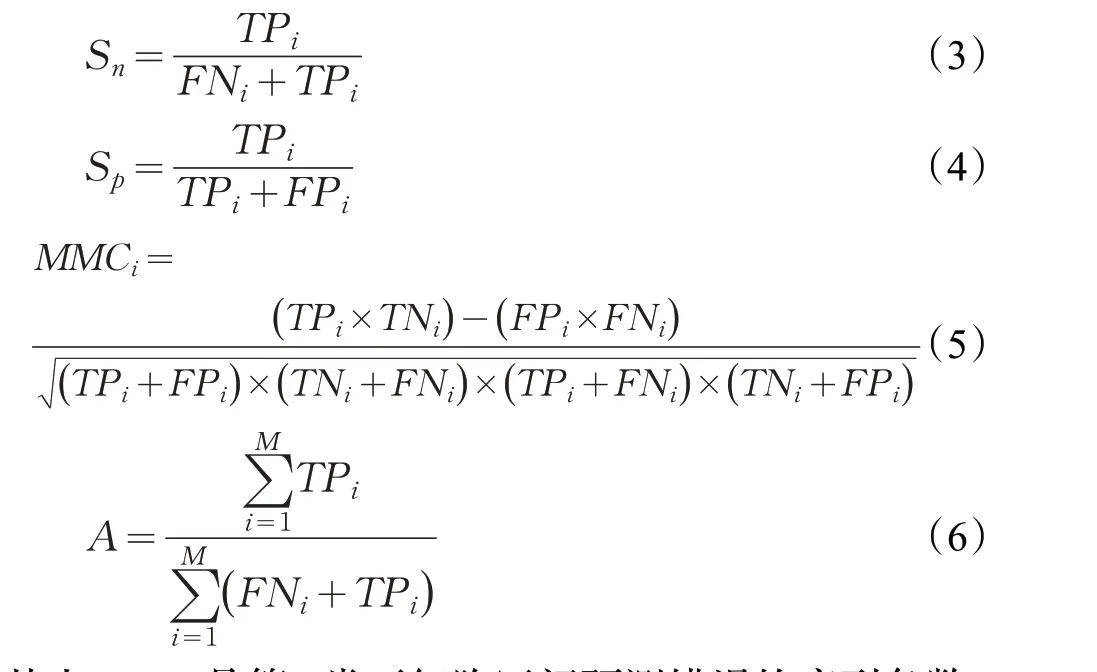
其中,FNi是第i 类亚细胞区间预测错误的序列条数,TPi是第i 类亚细胞区间预测正确的序列条数,TNi是被正确预测的非第i 类亚细胞区间的序列条数,FPi是非第i 类亚细胞区间但被预测为第i 类区间的序列条数,M 为亚细胞类别总数。
3.4 实验结果与分析
运用PSOBFA优化前后的BOW模型对数据集ZD98和CH317进行蛋白质序列特征提取,并将各特征值送入SVM 分类器进行亚细胞定位预测,实验结果分别列于表1和表2中。在表中,用PSO算法优化的BOW_AAC和BOW_PseAAC特征提取算法表示为pso1和pso2,用BFA算法优化的BOW_AAC 和BOW_PseAAC 特征提取算法表示为bfa1 和bfa2,经PSO_BFA 优化的特征提取算法表示为b_f1 和b_f2。根据实验结果绘制优化前后的预测结果图,如图2~5所示。
3.4.1 ZD98实验结果与分析
表1、图2 和图3 结果显示,在数据集ZD98 上,经PSO_BFA 优化的BOW 模型得到的词袋特征综合性能提升明显,并能进一步提高识别准确率。经过PSO_BFA优化后的BOW_AAC 和BOW_PseAAC 的预测准确率比单PSO 优化方法分别提高了1.1%和1.0%,比单BFA优化方法均提高了2.1%。在other 亚细胞类上,经优化的BOW_PseAAC特征的特异性Sp达到了100%。
3.4.2 CH317实验结果与分析
由表2、图4 和图5 结果可以看出,在数据集CH317上,PSO_BFA 优化后的BOW 模型比PSO 优化的BOW准确率分别提高了1.0%和0.6%,与BFA_BOW_AAC相比,预测准确率分别提高了1.9%和1.2%,PSO_BFA_BOW_AAC的综合性能均有明显提升。与PSO_BOW_AAC 相比,PSO_BFA_BOW_AAC 在se 和mi 亚细胞类上,敏感性Sn提升了5.9%,在en亚细胞类上,特异性Sp提升3.4%,在mi亚细胞类上,相关系数MMCi提升3.8%。PSO_BFA_BOW_PseAAC与PSO_BOW_PseAAC、BFA_BOW_PseAAC相比在各亚细胞区间均有不同程度的性能提升。

表1 ZD98数据集预测结果 %
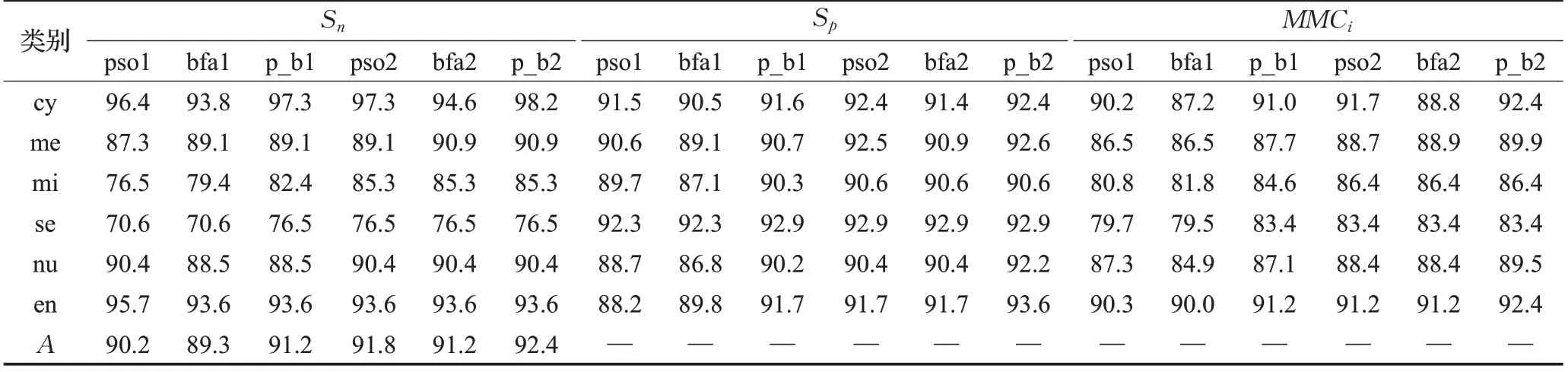
表2 CH317数据集预测结果 %
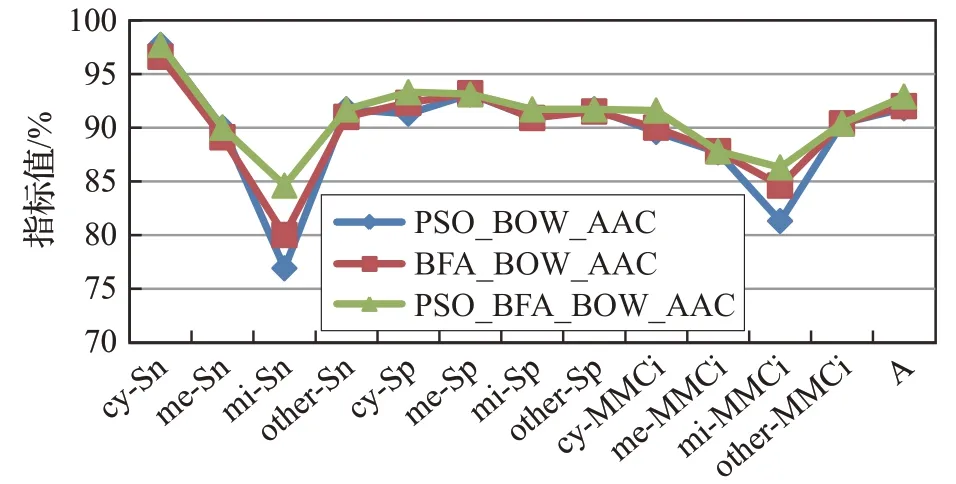
图2 优化前后的BOW_AAC在ZD98上的预测结果

图3 优化前后的BOW_PseAAC在ZD98上的预测结果

图4 优化前后的BOW_AAC在CH317上的预测结果
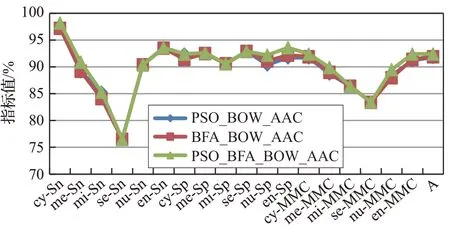
图5 优化前后的BOW_PseAAC在CH317上的预测结果
对比以上结果可以看出,在两个数据集上经加入细菌觅食优化的粒子群算法优化的BOW 模型都获得了更高的识别准确率。不管是在特异性Sp和相关系数MMCi还是在总的准确率A 的评价上,结果均优于粒子群算法和细菌觅食算法优化的BOW模型。这说明在同一的目标搜索空间下,PSO_BFA 优化算法的收敛概率大于细菌觅食算法,且同时大于粒子群优化算法。这印证了粒子群算法与细菌觅食算法的不同特点:粒子群算法的搜索速度快、较粗略,能相对快速确定全局极值的邻域,但局部搜索准确率不高;而细菌觅食算法的搜索比较细致,易在局部取得最优值,但全局寻优能力差且收敛速度慢。混合粒子群和细菌觅食算法结合两者的优点,将细菌觅食算法的复制和迁移行为应用到粒子群算法中提高算法的全局搜索能力和收敛速度。因此,无论在准确率还是在搜索速度上,均显著优于单一的粒子群算法和细菌觅食算法,上述实验成功证明了优化算法的有效性。
3.4.3 与其他算法对比
为了进一步说明优化算法在蛋白质亚细胞区间定位预测的有效性,将本文方法在ZD98和CH317数据集上的预测结果列于表3、4中,同时将选取在蛋白质亚细胞定位预测领域中具有代表性的传统蛋白质序列特征提取算法AAC、PseAAC、PSSM 和组合特征向量(Combined Feature Vector)等算法进行特征提取,并送入SVM分类器得到的预测准确率一并列出;表中也列出了赵南等人[9]将词袋模型结合AAC 算法对蛋白质序列进行特征提取,采用Jackknife进行检验的实验结果。表格的前两行,根据文献[27]和[28]中提到的AAC、PseAAC方法作为特征提取的方法,提取20 维向量作为一条蛋白质序列特征,然后放入SVM 中进行分类得出预测结果。实验使用libsvm中的库函数,主要参数为最佳惩罚参数c 和核函数参数g,文中通过交叉验证方法训练得到一组最佳参数。文献[29]中采用位置特异性评分矩阵(PSSM)提取蛋白质序列特征,主要参数PSI-BLAST设为0.001。文献[9]中,词袋模型中窗口大小d 和字典大小k 根据经验值选取。

表3 ZD98数据集预测结果比较%

表4 CH317数据集预测结果比较 %
从表3 可以看出,在ZD98 数据集上本文算法相比传统蛋白质序列特征提取算法AAC、PseAAC等在总体预测精度上最大提升了约10 个百分点,在cyto 亚细胞类上的预测准确率达到了100%,实验证明本文方法能有效增加蛋白质亚细胞区间定位预测的准确率。将本文方法与基于特征融合和词袋模型的实验结果进行对比,在相同数据集上的准确率也都提高了约3 到4 个百分点,实验表明本文算法较基于传统蛋白质序列特征提取的改进算法也具有显著优势。通过表4 的比较可以看出,在CH317 数据集上,本文算法在cyto 这一亚细胞类上的预测准确率最高达到了98.2%,相比其他算法提升了约3.6个百分点,在Nucl这一亚细胞类上的准确率最高提升了19.8个百分点,总的准确率较优化前的词袋模型提升了4.4个百分点。
词袋模型是一种十分有效的文本特征提取方法,经过本文的优化效果得到进一步提升。词袋模型中d 值和k 值对特征提取效果的影响很大。对比算法[9]中,词袋模型中窗口大小d 和字典大小k 均是根据经验值选取的,然而针对不同的研究对象以及样本空间最优参数值都是不同的,随机选取或根据经验值选取参数均会影响最终预测结果。本文使用PSO_BFA 混合优化算法,将最终分类结果作为粒子适应度值,动态求得d 值和k值的最佳组合,从而改进词袋模型的预测分类效果。同时,随着数据规模的增大,发挥粒子群和细菌觅食结合算法的群体智能优化性能,在参数空间中对窗口大小d和字典大小k 的选取更加合理,能够找到一组或多组(d,k)使提取的词袋特征值具有较高的识别精度。
4 结束语
BOW模型应用广泛,但针对传统BOW模型的改进工作很少涉及参数优化。本文利用BOW模型的主要参数窗口大小d 和字典大小k 构建参数搜索空间,将BOW 模型求得最高分类准确率的过程,抽象为粒子寻求最高适应度值的过程。充分发挥PSO_BFA算法的群体智能优化的性能,对(d,k)的选取过程进行优化,并将其应用到蛋白质亚细胞定位预测研究中。经PSO_BFA优化的BOW模型得到的词袋特征能在更短的时间内找到相应的亚细胞区间,数据集规模越大,性能提升越明显。实验证明基于PSO_BFA优化的词袋模型应用于蛋白质亚细胞定位预测中能有效提高识别精度。本文在词袋模型参数优化方面的研究取得了一些成果,然而词袋模型忽略了数据的位置信息,接下来将对位置特征的提取融合进行研究,并尝试在预测模型构建方面做一些工作,集成学习、深度学习等方法将是关注的重点。

