磷脂囊泡阵列的制备及其在生物分析领域中的应用
王雪靖 穆韡 韩晓军


摘要:磷脂囊泡的组分与真实细胞十分接近,它不仅可作为人造细胞模型,模拟细胞的结构与功能,还在生物分析方面有着广泛应用。磷脂囊泡阵列是进行高通量膜蛋白筛选和生物分析的良好平台。本文详细综述了匀质小磷脂囊泡阵列、异质小磷脂囊泡阵列、巨型磷脂囊泡阵列的制备方法,以及这些阵列在抗生素检测、酶反应、膜蛋白与膜的相互作用等生物分析方面的应用,评价了现有制备方法的优缺点,并提出了本领域未来的发展方向。
关键词:匀质小磷脂囊泡阵列;异质小磷脂囊泡阵列;巨型磷脂囊泡阵列;人造细胞;生物分析;评述
1引言
细胞作为生命活动的基本单位,由细胞膜将其内外环境分开。细胞膜是一个具有特殊功能的半透膜,它具有能量传递、物质传递、信息识别与传递等重要功能,由磷脂双分子层和嵌在其上的膜蛋白所构成。膜蛋白是细胞膜行使这些功能的重要组分。由于细胞内部及细胞膜本身结构复杂,而且其結构和成分的提取常涉及繁杂的纯化步骤,因此,研究者多采用与真实细胞膜成分最为接近的磷脂囊泡[1]或磷脂膜[2~10]模拟细胞及细胞膜的结构、性质与功能。根据组成囊泡的磷脂双层膜的层数,磷脂囊泡可分为单层磷脂囊泡(Unilamellarvesicles)、双层磷脂囊泡(Oligolamellarvesicles)、多层磷脂囊泡(Multilamellarvesicles)及多室囊泡(Multivesicularvesicles)。由于单层磷脂囊泡的结构更接近真实细胞,因此其研究和应用也更为广泛。根据尺寸,单层磷脂囊泡可分为小磷脂囊泡(Smallunilamellarvesicle,SUV,0.02~0.2μm)、大磷脂囊泡(Largeunilamellarvesicle,LUV,0.2~1μm)和巨型磷脂囊泡(Giantunilamellarvesicle,GUV,>1μm)[11]。SUV和LUV在载药[4,12~15]和生物医学领域已有广泛的应用。GUV[16~18]的尺寸更接近真实细胞,且易于在显微镜下观察,因此可用于研究细胞膜的机械性质[19~21]、相分离和脂筏行为[22,23]、细胞的粘附[23]、磷脂组分的动态变化[24]、蛋白与磷脂的相互作用[24]、细胞的形变行为[25](生长[26]、出芽[27-29]、分裂[27]、融合[30])等;此外,GUV还可作为人造细胞模型模拟细胞内的代谢反应[31~34]、基因表达调控的蛋白质合成[35,36]、遗传物质的自我复制[37~39]等功能。
在生物医学领域,DNA微阵列[40]、细胞阵列[41]、蛋白质阵列[42]均含有大量的生物信息,能够高效、高通量地实现生物信息的分析。DNA微阵列中含有多种DNA序列,能够实现多种基因功能的分析[40,43];细胞阵列中含有多种细胞,能够高通量地检测细胞代谢产物,分析细胞间的相互作用;蛋白质阵列中的抗体阵列可作为疾病分析诊断的工具[44]。上述的DNA阵列、蛋白质阵列及细胞阵列均为细胞中真实物种或真实细胞的阵列,囊泡作为一种人造细胞模型,结构和功能确定,以期其阵列能像上述阵列一样在生物分析领域具有应用,结合微阵列的高通量特性和囊泡在生物分析领域的应用,研究者致力于囊泡微阵列的构建及应用研究。与单个囊泡相比,囊泡阵列能够同时展示更多的生物化学信息,在蛋白质的高通量筛选、研究蛋白与磷脂膜的相互作用、医学诊断等方面具有良好的应用前景。目前,关于磷脂囊泡的制备和应用类的综述较多[23,45,46],但磷脂囊泡阵列相关的综述仍然较少,因此本文将着重阐述磷脂囊泡阵列的制备及相关应用。从组成成分上分,囊泡阵列可分为匀质囊泡阵列和异质囊泡阵列。匀质囊泡阵列是指只含有一种囊泡的阵列,这种囊泡阵列容易制备,但它能够展示的信息较为单一,在高通量应用方面具有一定的局限性。异质囊泡阵列是指同时含有不同种类囊泡的阵列,该种囊泡阵列的制备技术要求较高,但它在高通量的生物分析方面具有很好的应用前景。本文从不同尺寸的囊泡阵列出发,详细阐述匀质囊泡阵列和异质囊泡阵列的制备及其在生物分析方面应用的研究进展。
2小磷脂囊泡(SUV)阵列
2.1SUV阵列的制备
小磷脂囊泡(SUV)阵列可用于膜蛋白的筛选和高通量的生物分析。SUV阵列的制备一般是将预先制备好的SUV固定在图案化的基底上。在固定过程中,基底的钝化和用于连接囊泡和基底间连接物的选择十分重要。惰性的基底表面不仅能够降低背景噪声,还可避免囊泡和蛋白的非特异性吸附,有助于提高传感器的选择性。惰性基底的制备一般采用修饰的方法,如蛋白质修饰[47,48]、自组装膜修饰[49]、聚合物修饰[50~52]、磷脂双层膜修饰[53~56]等。将SUV固定在基底上的方法一般有两种:一种是将具有特异结合特性的物质修饰在SUV的磷脂膜上,具有这种特性的物质有生物素-亲和素[47,49,57]、抗原-抗体[58,59]、互补的寡核苷酸对等;另一种是利用化学连接物(如组氨酸(His)[60]、二硫化物[61]等)将SUV与基底相连。尽管采用生物素-亲和素、抗原-抗体、化学键合等连接方式能够将SUV稳定的链接在基底表面,但却不能选择性地将不同类型的SUV同时固定在特定的区域,构建异质的SUV阵列,而互补的寡核苷酸对恰好能够弥补这一不足。互补的寡核苷酸对的概念由Niemeyer等[62,63]提出后,利用其固定SUV很快成为研究者们青睐的固定方法[64,65]。
2.1.1匀质SUV阵列的制备匀质SUV阵列中每个结构单元上的SUV种类相同。化学修饰图案化基底法是用于制备SUV阵列的一种常用方法。Svedhem等[48]在Au/SiO2复合基底的Au表面修饰了链有生物素的牛血清蛋白(BSA),在SiO2表面修饰了磷脂双层膜,通过亲和素,单链DNA修饰的生物素可链接到生物素-BSA修饰的Au表面,这样修饰有互补DNA链的SUV可选择性地聚集在生物素修饰的金表面,从而形成匀质的SUV阵列。除了Au/SiO2复合表面外,也有研究者用TiO2/SiO2复合表面[49]制备SUV匀质阵列。他们分别在TiO2和SiO2表面自组装疏水的十二烷基磷酸盐(DDP)单层膜和聚赖氨酸-g-聚乙二醇(PLL-g-PEG)膜,然后将荧光标记的亲和素吸附在DDP自组装膜表面,生物素修饰的SUV即可与亲和素结合,形成SUV阵列。利用类似的聚合物自组装方法,Stadler等[51]在生物素修饰的PLL-g-PEG表面利用互补的DNA链制备了匀质的方形SUV阵列(图1A)。Voskuhl等[66]合成了两种具有α-螺旋结构的互补多肽(多肽E和多肽K),将多肽E和多肽K分别修饰在图案化基底和SUV上,利用它们的配对特性和微接触印刷技术得到了匀质的圆形囊泡阵列(图1B)。Zhang等[67]将带负电的纳米粒子(聚苯乙烯小球)包裹在SUV表面,通过静电吸引的方法将负电的SUV固定在正电聚合物(聚乙烯吡咯烷酮PVP)修饰的基底表面(图1C1),得到了匀质的六边形SUV阵列(图1C2)。Roling等[68]报道了一种新型三维多层SUV阵列结构,他们分别利用甘露糖-伴刀豆球蛋白A和生物素-亲和素的特异性连接作用,在图案化的基底表面将环糊精修饰的囊泡层层组装起来,形成了多层磷脂囊泡阵列(图1D)。
2.1.2异质SUV阵列的制备异质SUV阵列是指在同一阵列中含有两种或两种以上的SUV,和匀质阵列相比,异质SUV阵列能够展示更多的生物信息,为生物分析研究提供更好的平台。利用互补的DNA链修饰SUV和基底是常用于制备异质SUV阵列的方法。Pfeiffer等[69]通过将不同种类DNA修饰的胆固醇分子嵌入磷脂膜中,在复合Au/SiO2表面上得到了同时含有红色荧光和绿色荧光的SUV阵列(图2A)。Dusseiller等[64]利用一种新型交叉式微流控装置,在聚合物修饰的基底表面制备了两种囊泡交替存在的SUV阵列(图2B)。除了蛋白质和聚合物修饰的基底外,磷脂双层膜也可用来修饰基底。Yoshina-Ishii等[55]将链接有不同DNA序列的磷脂双层膜修饰在玻璃基底的不同區域,利用DNA序列的互补特性将两种不同种类的SUV链接在同一阵列中(图2C)。基于DNA寡核苷酸链的互补特性已经发展出了一种生物分子打印技术,可用于制备囊泡阵列。Stdler等[52]进一步发展了这一技术,他们将两种不同的单链DNA修饰在聚合物基底的不同区域,形成斑点状的图案化基底,然后加入分别链有上述DNA互补链的两种囊泡混合溶液,制备出同时含有绿色膜联蛋白A5标记的囊泡和包有BSA蛋白的红色荧光囊泡的SUV阵列(图2D),该方法的优点是能够控制异质SUV阵列的图案。

在SUV阵列的制备过程中,图案化基底的质量直接影响SUV阵列的质量,因此,图案化基底的制备十分关键。像Au/SiO2这种复合物基底多采用模板溅射法制备,这种方法需要精密的溅射设备及高质量的掩膜。此外,图案化基底表面的修饰对SUV阵列的制备也十分重要,无论是蛋白质修饰、聚合物或者磷脂膜修饰,修饰的时间及修饰物的浓度将直接影响基底的修饰质量,并影响SUV在基底表面的固定。在SUV的固定过程中,具有特异性结合性质的物质在SUV和基底表面的含量要适当,以确保SUV在特定区域的分布均匀。总之,制备SUV阵列的每一步都非常关键,需要多次反复制备优化条件,以得到高质量的SUV阵列。SUV阵列的密度取决于图案化基底的密度,目前所获得的SUV阵列的密度约为106~109/m2(每平方米约有106~109个阵列结构单元)。在应用时,阵列密度越大,越有利于物质高通量检测。
2.2SUV阵列的应用
2.2.1SUV阵列在生物传感方面的应用(1)生长激素释放因子和生长激素抑制素的定量检测Shoji等[70]利用可裂解的二硫化物将囊泡固定在硅烷化的基底上,通过TritonX-100使囊泡破裂,并从硅烷化的基底表面脱落,再在还原剂三(2-羧乙基)磷盐酸盐(TCEP)的作用下将烷基化修饰的表面巯基化,并可再次链接囊泡。该方法形成的基底能够重复制备磷脂囊泡阵列,他们利用该阵列实现了生长激素释放因子和生长激素抑制素的同时定量检测,二者的检出限分别为2×10Symbolm@@10g/mL和3×10Symbolm@@10g/mL。(2)流感病毒M1抗体和H1N2型流感病毒的检测聚乙二炔(PDA)在外界刺激(如温度、pH值、压力)下会发生颜色的转变[71~73],利用这一特性可制备用于生物传感的囊泡阵列。Seo等[74]设计了一种PDA传感囊泡阵列检测流感病毒M1抗体和H1N2型流感病毒,他们将修饰有探针分子的PDA囊泡链接在氨基修饰的图案化基底表面,然后通过紫外照射使PDA发生光聚合,聚合后的分子探针可与目标物结合。他们发现,将M1多肽作为探针修饰在PDA囊泡表面,可检测M1抗体;以M1抗体作为分子探针修饰在PDA囊泡表面,该分子探针能够选择性的与含有M1多肽的流感病毒H1N2目标物结合,实现流感病毒的检测。该传感器对流感病毒的检出限为2Symbolm@@2HAU,与市售的流感病毒试剂盒的检出限相当。(3)氨基糖苷类抗生素的检测除了流感病毒抗体外,PDA囊泡阵列还可用于检测氨基糖苷类抗生素。Kang等[75]将10,12-二十五碳二炔酸(PCDA)磷脂与1,2-豆蔻酸二甘油酯-sn-甘油-3-磷酸盐(DMPA)及磷脂酰肌醇-4,5-二磷酸(PIP2)以摩尔比7∶2∶1混合,制备了PDA-PIP2复合囊泡,经过紫外照射聚合后(图3A1),分别将四种氨基糖苷类抗生素庆大霉素(Gentamicin)、妥布霉素(Tobramycin)、链霉素(Streptomycin)、新霉素(Neomycin)加入囊泡阵列中,其中新霉素的荧光强度最强(图3A2),这是由于新霉素能够与PIP2磷脂特发生异性结合。该PDA-PIP2传感体系对新霉素的检出限为61ppb。为了提高传感器的灵敏度,Lee等[76]在硅柱阵列上形成碳纳米管网络,再将PDA囊泡加入该网络中,形成了三维的PDA囊泡阵列(图3B1),环糊精能与PDA作用生成红色荧光物质,与二维的PDA囊泡阵列相比,三维的PDA囊泡网络阵列对环糊精具有更高的检测灵敏度(图3B2),检出限为2μmol/L。
2.2.2SUV阵列在生物分析领域的潜在应用目前,SUV阵列已被用于激素检测、病毒检测和抗生素检测,这些SUV阵列多为匀质阵列,异质SUV阵列的应用还存在空白。组成SUV阵列的磷脂囊泡还可用于制备其它类型的生物传感器。Guan等[77]将乙酰胆碱酯酶包入磷脂囊泡中,用于农药检测。Lin等[78]将淀粉转葡萄糖苷酶包入磷脂囊泡中,用于检测凝血酶和C反应蛋白(CRP)。利用荧光标记物的荧光共振能量转移(FRET)性质,磷脂囊泡传感器还可用于检测内分泌干扰物[79]、磷脂酶A2[80]、腺苷[81]、氯霉素[82]、霉菌毒素和黄曲霉毒素[83]等物质。以上这些应用在磷脂囊泡阵列中还未有报道,可利用匀质SUV阵列实现其中某种物质的高通量检测,利用异质SUV阵列可实现其中多种物质的同时高通量检测。
3巨型磷脂囊泡(GUV)阵列
单囊泡阵列是指每个节点上只含有单个囊泡的阵列。单囊泡阵列不仅能够提高生物分析的通量,还便于在单分子水平上理解膜蛋白的识别特性。Stamou等[47]结合微接触印刷技术,利用生物素与亲和素的作用在BSA修饰的基底上制备了随机分布的匀质LUV单囊泡阵列和异质单囊泡阵列,他们所用的囊泡尺寸为800nm。Kalyankar等[50]同样利用生物素与亲和素的特异性连接作用将预先制备好的囊泡填充在经过刻蚀的微井阵列中,得到匀质的LUV单囊泡阵列,囊泡尺寸为1μm。但是,该方法未能得到异质的LUV阵列。目前LUV阵列方面的研究较少,下面将着重阐述GUV阵列的制备及应用。

3.1巨型磷脂囊泡陣列的制备
3.1.1微接触印刷法微接触印刷法是一种制备GUV阵列较为普遍的方法。Taylor等[84,85]率先利用微接触印刷技术与电形成法相结合得到了GUV阵列。该方法将蘸有磷脂溶液的图案化PDMS印章印在ITO基底上,形成磷脂膜阵列,再施加电场,诱导形成GUV阵列(图4A)[85]。由于有磷脂聚集体吸附在囊泡表面,因此该法制备的GUV阵列并不理想。利用类似的微接触剥离方法,本课题组[86]以ITO为基底,使用钨丝-ITO点面电极制备了边界清晰且尺寸均匀的GUV阵列(图4B1),不仅得到大范围且尺寸均匀的GUV阵列,还可利用混合的磷脂组分制备具有相分离现象的GUV阵列(图4B2),相分离的磷脂膜结构更加接近真实的细胞。除PDMS外,琼脂糖水凝胶印章[87]也可用于GUV阵列的制备,与PDMS相比,琼脂糖水凝胶的生物相容性较好,有利于GUV磷脂膜中蛋白质的嵌入。除电形成方法外,微接触印刷法还能与水合法相结合制备GUV阵列[88,89]。

微接触印刷技术与电形成法相结合能够制备匀质的GUV阵列,制备GUV阵列的成功率主要取决于印章的质量和微接触印刷过程的操作控制,而且GUV的尺寸可以通过改变印章中结构单元的尺寸或改变电形成过程的时间控制。
3.1.2微流控法微流控技术是一种新兴的流体控制技术,该方法不仅能够精确的控制流体的速度,还能够利用限域性的图案达到捕获目标物质的目的,因此也有研究者利用微流控法制备匀质GUV阵列。Paterson等[90]将电形成装置与囊泡捕获装置连接在一起,发明了一种新型集成式微流控装置(图5A1),得到了匀质GUV阵列(图5A2)。Kazayama等[91]将囊泡筛选区域与囊泡捕获区域集成在同一块微流控芯片中(图5B1),得到了大范围的GUV阵列(图5B2),并且通过改变捕获区域限域单元的尺寸可得到不同尺寸的GUV阵列。
目前,用微流控法制备GUV阵列的报道仍然较少,虽然微流控法已能够分选不同尺寸的GUV,并得到尺寸可控的GUV阵列,但微流控芯片的制备工艺较为复杂,限制了该方法的应用。
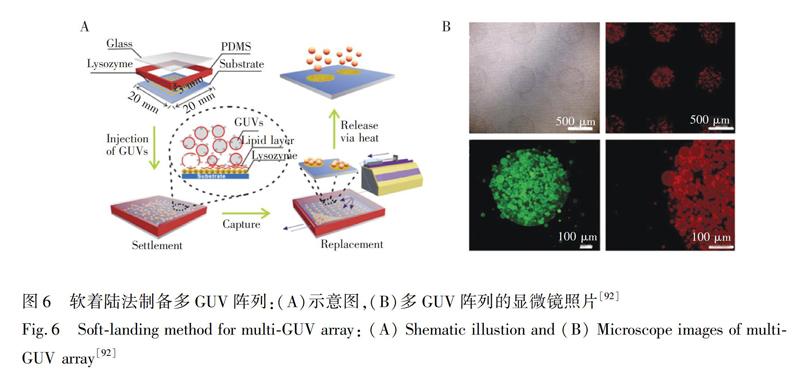
3.1.3软着陆法以上所述方法制备的GUV阵列为单囊泡阵列(每个结构单元含有一个囊泡),Wang等[92]利用软着陆法制备了多囊泡阵列(每个结构单元含有多个囊泡)。他们首先在基底上修饰图案化的正电溶酶酵素(Lysozymes)膜,然后将带负电的GUV加入该基底上,预先与溶酶酵素膜接触的GUV会在静电引力的作用下发生破裂,形成一层磷脂双层膜,后到达基底的GUV由于有磷脂膜的缓冲作用不会破裂,且会吸附在磷脂膜上,未修饰溶酶酵素的区域被GUV占据后,由于没有静电吸附作用,在流体的作用下可被冲走,由此形成了多囊泡阵列。该方法的示意图如图6A所示,所制备的多囊泡阵列如图6B所示。该方法形成的GUV阵列在加热后还能重新脱离基底。
目前,多GUV阵列的制备方法仍然较少。软着陆法方法巧妙,虽然可制备多GUV阵列,但每个结构单元上GUV的个数不可控制。已报道的GUV阵列的密度约为107~108/m2。
3.2巨型磷脂囊泡阵列的应用
最初,制备GUV阵列是为了获得单分散且尺寸均匀的GUV,后来,研究者们拓展了GUV阵列的应用,利用GUV阵列作为微反应器研究简单的酶反应,或进行膜蛋白的研究。
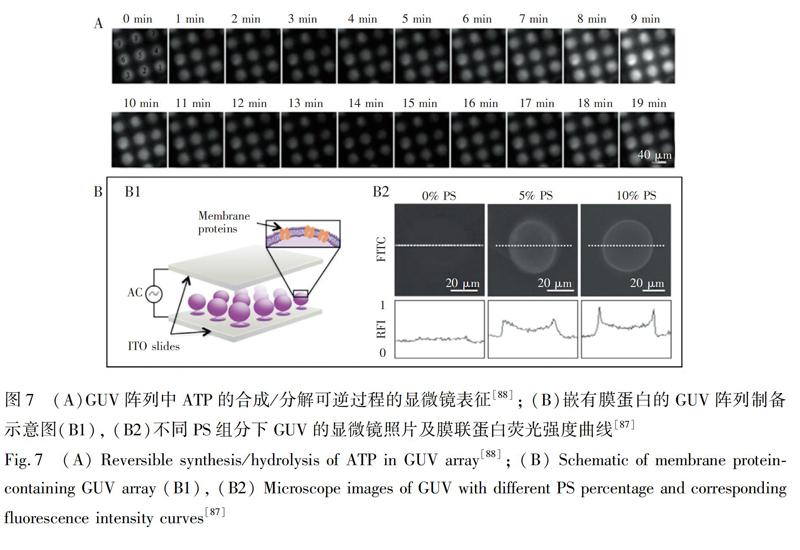
本课题组[86]在单室GUV阵列中实现了辣根过氧化物酶(HRP)催化邻苯二胺(oPD)的反应,得到了GUV微反应器阵列。将HRP包裹进GUV阵列中,然后向阵列中加入H2O2和oPD的混合溶液,H2O2和oPD分子可通过GUV与基底连接的部位进入GUV中,在GUV内部HRP的催化作用下生成具有绿色荧光的2,3-二氨基吩嗪(2,3-DAP)。Liu等[88]利用GUV阵列观察到了ATP在F0F1-ATP酶催化作用下合成/水解的可逆过程。他们首先利用微接触印刷结合水合法制备包有色素体(Chromatophore)的GUV阵列,其中色素体表面利用静电吸附作用修饰一层对pH值敏感的量子点。当F1-ATP酶顺时针旋转,ADP与磷酸合成ATP,产生的质子被泵出色素体外部,使外部pH值降低;相反的,当ATP水解时,质子会被泵入色素体内部,使外部环境的pH值升高。色素体表面吸附的量子点荧光强度随pH值的降低而增强,因此可指示pH值的变化,从而间接证明色素体内部ATP的合成及分解过程。图7A为GUV阵列中ATP的合成/分解可逆过程的显微镜表征,随时间的变化,GUV阵列的荧光强度由弱变强再变弱多次循环,证实了GUV内部ATP的合成-分解-合成-分解的可逆循环过程。
GUV阵列还能够用于研究膜蛋白与磷脂膜的相互作用。蜂毒素又称蜂毒肽,是由26个氨基酸残基组成的阳离子型溶血蛋白[93,94],其具有的两亲性质使蜂毒素成为研究磷脂膜与蛋白质相互作用的典型蛋白[95],蜂毒素可诱导生物膜表面产生孔道结构从而使膜内物质穿过孔道释放出来。本课题组[86]将小分子荧光素包入GUV阵列中,研究了不同蜂毒素浓度对荧光素释放速度的影响,得到了不同浓度蜂毒素下荧光素的扩散系数,定量研究了蜂毒素与磷脂膜的相互作用。Paterson等[90]也利用微流控法制备的GUV阵列研究了蜂毒素嵌入磷脂膜后GUV内荧光素的释放情况。


除蜂毒素外,还有研究者将其它种类的膜蛋白嵌入GUV阵列中。Kang等[87]将嵌有水通道蛋白Z(AqpZ)的小囊泡(SUV)溶于琼脂糖凝胶印章中,在电形成的过程中将其嵌入GUV阵列中(图7B1),并利用免疫荧光方法证明了AqpZ的成功嵌入。此外,他们还将膜联蛋白-V嵌入含有磷脂酰丝氨酸(PS)的GUV表面,研究了其与磷脂膜的相互作用。膜联蛋白-V与磷脂膜相连,参与细胞膜的凝结破坏过程,在细胞凋亡的过程中有着十分重要的作用[96]。这种蛋白在Ca2+存在时对带负电的PS具有很强的亲和作用[97],据此制备了不同PS含量的GUV阵列,观察到PS含量越高,荧光标记的膜联蛋白-V的荧光强度越强(图7B2),证实了膜联蛋白-V与磷脂膜中PS组分存在相互作用。
组成磷脂囊泡的磷脂膜具有渗透性,当囊泡内外存在渗透压差时,囊泡的形貌会发生变化。基于微流控法制备的GUV阵列平台,Kazayama等[91]研究了连续渗透压刺激下的GUV形貌的动态变化过程。他们研究了两种尺寸(12和20μm)GUV在高渗和低渗刺激下的响应行为,在高渗条件下,囊泡尺寸逐渐减小,并且还观察到了内凹现象;在低渗透条件下,12μmGUV在30min内逐渐膨胀,绝大多数20μmGUV在80min内胀破。他们还测得了两种尺寸下水分子的透过率。
表1中详细对比了SUV阵列和GUV阵列的制备方法、应用及未来的发展方向。匀质SUV阵列制备方法较为成熟,并且在蛋白质检测方面已初具应用。异质SUV阵列的制备已有报道,但应用仍然欠缺。匀质GUV阵列的制备方法较多,而且在酶反应、膜蛋白方面已有应用,但阵列类型和应用仍然较为简单。异质GUV阵列的制備和应用仍然存在较大空白,具有很大的开发空间。
4总结与展望
囊泡阵列不仅可作为高通量的生物分析平台检测抗生素等,研究膜蛋白与磷脂膜的相互作用,还可研究细胞内部的代谢反应。匀质小囊泡阵列的制备方法已较为成熟,利用互补的DNA寡核苷酸链可制备出异质SUV阵列,在生物检测方面表现出良好的应用前景。匀质GUV阵列的制备方法也已较为成熟,但多囊泡阵列和异质GUV阵列的制备还未见报道,但已报道的酶反应和膜蛋白与膜的相互作用体系少且简单,还没有商品化的SUV或GUV阵列产品及阵列制备仪器系统。拓展囊泡阵列的制备方法及其在生物分析方面的高通量应用,制定囊泡阵列的制备标准,使其进一步商品化,将成为其未来的发展方向。
References
1GhellabSE,LQC,FuhsT,BiHM,HanXJ.ColloidSurfaceB,2017,160:697-703
2BiHM,WangXJ,HanXJ,VoitchovskyK.Langmuir,2018,34(32):9561-9571
3WangXJ,ZhangY,BiHM,HanXJ.RSCAdv.,2016,6(76):72821-72826
4BiHM,MaSH,LiQC,HanXJ.J.Mater.Chem.B,2016,4(19):3269-3277
5BiHM,FuDG,WangL,HanXJ.ACSNano,2014,8(4):3961-3969
6ZhangY,WangXJ,MaSH,JiangKP,HanXJ.RSCAdv.,2016,6(14):11325-11328
7ZhangY,WangL,WangXJ,QiGD,HanXJ.Chem.Eur.J.,2013,19(27):9059-9063
8ZhangY,WangXJ,WangL,YuM,HanXJ.Bioelectrochemistry,2014,95:29-33
9LiQC,HanXJ.iScience,2018,8:138-147
10LiQC,HanXJ.Adv.Mater.,2018,30(25):1707482
11JesorkaA,OrwarO.Annu.Rev.Anal.Chem,2008,1:801-832
12BiHM,HanXJ.Chem.Phys.Lett.,2018,706:455-460
13ZhangXN,ZongW,BiHM,ZhaoKM,FuhsT,HuY,ChengWL,HanXJ.Eur.J.Pharm.Biopharm.,2018,127:177-182
14ZhangXN,ZongW,WangJL,DongMD,ChengWL,SunTM,HanXJ.NewJ.Chem.,2019,43(12):4895-4899
15ZhangXN,ZongW,ChengWL,HanXJ.J.Mater.Chem.B,2018,6(32):5243-5247
16BiHM,YangB,WangL,CaoWW,HanXJ.J.Mater.Chem.A,2016,1(24):7125-7130
17LiQC,WangXJ,MaSH,ZhangY,HanXJ.ColloidSurfaces.B,2016,147:368-375
18ZhuCT,ZhangY,WangYN,LiQC,MuW,HanXJ.Chem.Eur.J,2016,22(9):2906-2909
19SchaferE,KlieschTT,JanshoffA.Langmuir,2013,29(33):10463-10474
20LuL,DoakWJ,SchertzerJW,ChiarotPR.SoftMatter,2016,12(36):7521-7528
21SchaferE,VacheM,KlieschTT,JanshoffA.SoftMatter,2015,11(22):4487-4495
22WesolowskaO,MichalakK,ManiewskaJ,HendrichAB.ActaBiochim.Pol.,2009,56(1):33-39
23FenzSF,SenguptaK.Integr.Biol.-UK,2012,4(9):982-995
24Garcia-SaezAJ,SchwilleP.Methods,2008,46(2):116-122
25ZongW,LiQC,ZhangXN,HanXJ.ColloidSurfaceB,2018,172:459-463
26PeterlinP,ArriglerV,KogejK,SvetinaS,WaldeP.Chem.Phys.Lipids,2009,159(2):67-76
27AllainJM,BenAmarM.SoftMatter,2006,20(4):409-420
28TsudaS,SakakuraT,FujiiS,SuzukiH,YomoT.PloSOne,2015,10(7):e0132963
29BaumgartT,HessST,WebbWW.Nature,2003,425(6960):821-824
30TanakaT,YamazakiM.Langmuir,2004,20(13):5160-5164
31KentaroSKM,KazuoY,TadashiS.Chem.Phys.Lipids,2018,210:70-75
32NutiN,VerboketPE,DittrichPS.LabChip,2017,17(18):3112-3119
33ElaniY,LawRV,CesO.Nat.Commun.,2014,5:5305
34ZongW,ZhangXN,LiQC,HanXJ.ACSSynth.Biol.,2018,7(3):945-951
35vanNiesP,WesterlakenI,BlankenD,SalasM,MenciaM,DanelonC.Nat.Commun.,2018,9(1):1583
36ElaniY,LawRV,CesO.Phys.Chem.Chem.Phys.,2015,17(24):15534-15537
37ZongW,MaSH,ZhangXN,WangXJ,LiQC,HanXJ.J.Am.Chem.Soc.,2017,139(29):9955-9960
38TsuganeM,SuzukiH.Sci.Rep.,2018,8(1):9214
39KuriharaK,TamuraM,ShohdaK,ToyotaT,SuzukiK,SugawaraT.Nat.Chem.,2011,3(10):775-781
40DeRisiJ,PenlandL,BrownPO,BittnerML,MeltzerPS,RayM,ChenYD,SuYA,TrentJM.Nat.Genet.,1996,14(4):457-460
41ErfleH,PepperkokR.Method.Enzymol.,2005,404:1-8
42DixitCK,AguirreGR.Microarrays,2014,3(3):180-202
43DouglasES,ChandraRA,BertozziCR,MathiesRA,FrancisMB.LabChip,2007,7(11):1442-1448
44SutandyFX,QianJ,ChenCS,ZhuH.Curr.Protoc.ProteinSci.,2013,72:27.1
45DaraeeH,EtemadiA,KouhiM,AlimirzaluS,AkbarzadehA.Artif.CellNanomed.B,2016,44(1):381-391
46WesolowskaO,MichalakK,ManiewskaJ,HendrichAB.ActaBiochim.Pol.,2009,56(1):33-39
47StamouD,DuschlC,DelamarcheE,VogelH.Angew.Chem.Int.Edit.,2003,42(45):5580-5583
48SvedhemS,PfeifferI,LarssonC,WingrenC,BorrebaeckC,HookF.Chembiochem,2003,4(4):339-343
49MichelR,ReviakineI,SutherlandD,FokasC,CsucsG,DanuserG,SpencerND,TextorM.Langmuir,2002,18(22):8580-8586
50KalyankarND,SharmaMK,VaidyaSV,CalhounD,MaldarelliC,CouzisA,GilchristL.Langmuir,2006,22(12):5403-5411
51StadlerB,FalconnetD,PfeifferI,HookF,VorosJ.Langmuir,2004,20(26):11348-11354
52StadlerB,BallyM,GrieshaberD,VorosJ,BrissonA,GrandinHM.Biointerphases,2006,1(4):142-145
53SottK,KarlssonM,PihlJ,HurtigJ,LobovkinaT,OrwarO.Langmuir,2003,19(9):3904-3910
54RossettiFF,BallyM,MichelR,TextorM,ReviakineI.Langmuir,2005,21(14):6443-6450
55Yoshina-IshiiC,BoxerSG.J.Am.Chem.Soc.,2003,125(13):3696-3697
56Yoshina-IshiiC,MillerGP,KraftML,KoolET,BoxerSG.J.Am.Chem.Soc.,2005,127(5):1356-1357
57JungLS,Shumaker-ParryJS,CampbellCT,YeeSS,GelbMH.J.Am.Chem.Soc.,2000,122(17):4177-4184
58MacKenzieCR,HiramaT,LeeKK,AltmanE,YoungNM.J.Biol.Chem.,1997,272(9):5533-5538
59ShibataSekiT,MasaiJ,TagawaT,SorinTKondoS.ThinSolidFilms,1996,273(1-2):297-303
60StoraT,DienesZ,VogelH,DuschlC.Langmuir,2000,16(12):5471-5478
61StanishI,SantosJP,SinghA.J.Am.Chem.Soc.,2001,123(5):1008-1009
62NiemeyerCM,SanoT,SmithCL,CantorCR.NucleicAcidsRes.,1994,22(25):5530-5539
63NiemeyerCM.NanoToday,2007,2(2):42-52
64DusseillerMR,NiederbergerB,StadlerB,FalconnetD,TextorM,VorosJ.LabChip,2005,5(12):1387-1392
65Yoshina-IshiiC,ChanYHM,JohnsonJM,KungLA,LenzP,BoxerSG.Langmuir,2006,22(13):5682-5689
66VoskuhlJ,WendelnC,VersluisF,FritzEC,RolingO,ZopeH,SchulzC,RinnenS,ArlinghausHF,RavooBJ,KrosA.Angew.Chem.Int.Edit.,2012,51(50):12616-12620
67ZhangLF,HongL,YuY,BaeSC,GranickS.J.Am.Chem.Soc.,2006,128(28):9026-9027
68RolingO,WendelnC,KauscherU,SeelheimP,GallaHJ,RavooBJ.Langmuir,2013,29(32):10174-10182
69PfeifferI,HookF.J.Am.Chem.Soc.,2004,126(33):10224-10225
70ShojiA,SugimotoE,OritaS,NozawaK,YanagidaA,ShibusawaY,SugawaraM.Anal.Bioanal.Chem.,2010,397(3):1377-1381
71LiY,WangLH,YinX,DingB,SunG,KeT,ChenJY,YuJY.J.Mater.Chem.A,2014,2(43):18304-18312
72LeeKM,MoonJH,JeonH,ChenXQ,KimHJ,KimS,KimSJ,LeeJY,YoonJ.J.Mater.Chem.,2011,21(43):17160-17166
73SunXM,ChenT,HuangSQ,LiL,PengHS.Chem.Soc.Rev.,2010,39(11):4244-4257
74SeoS,LeeJ,ChoiEJ,KimEJ,SongJY,KimJ.Macromol.RapidCommun.,2013,34(9):743-748
75KangDH,JungHS,AhnN,LeeJ,SeoS,SuhKY,KimJ,KimK.Chem.Commun.,2012,48(43):5313-5315
76LeeS,LeeJ,LeeDW,KimJM,LeeH.Chem.Commun.,2016,52(5):926-929
77GuanHN,ZhangFL,YuJ,ChiDF.FoodRes.Int.,2012,49(1):15-21
78LinBQ,LiuD,YanJM,QiaoZ,ZhongYX,YanJW,ZhuZ,JiTH,YangCJ.ACSAppl.Mater.Interfaces,2016,8(11):6890-6897
79NakaneY,KuboI.Materials,2014,7(1):170-179
80AiliD,MagerM,RocheD,StevensMM.NanoLett.,2011,11(4):1401-1405
81DaveN,LiuJW.Chem.Commun.,2012,48(31):3718-3720
82MiaoYB,RenHX,GanN,CaoYT,LiTH,ChenYJ.Biosens.Bioelectron.,2016,81:454-459
83BeloglazovaNV,GoryachevaOA,SperanskayaES,AubertT,ShmelinPS,KurbangaleevVR,GoryachevaIY,deSaegerS.Talanta,2015,134:120-125
84TaylorP,XuC,FletcherPDI,PaunovVN.Phys.Chem.Chem.Phys.,2003,5(21):4918-4922
85TaylorP,XuC,FletcherPDI,PaunovVN.Chem.Commun.,2003,(14):1732-1733
86ZhuCT,LiQC,DongMD,HanXJ.Anal.Chem.,2018,90(24):14363-14367
87KangYJ,WosteinHS,MajdS.Adv.Mater.,2013,25(47):6834-6838
88LiuXL,ZhaoR,ZhangY,JiangXY,YueJC,JiangPD,ZhangZX.BBA-Gen.Subjects,2007,1770(12):1620-1626
89SalibaAE,VonkovaI,CeschiaS,FindlayGM,MaedaK,TischerC,DeghouS,vanNoortV,BorkP,PawsonT,EllenbergJ,GavinAC.Nat.Methods,2014,11(1):47-50
90PatersonDJ,ReboudJ,WilsonR,TassieriM,CooperJM.LabChip,2014,14(11):1806-1810
91KazayamaY,TeshimaT,OsakiT,TakeuchiS,ToyotaT.Anal.Chem.,2016,88(2):1111-1116
92WangDH,WuZF,GaoAT,ZhangWH,KangCY,TaoQ,YangP.SoftMatter,2015,11(16):3094-3099
93DempseyCE.Biochim.Biophys.Acta,1990,1031(2):143-161
94HabermannE.Science,1972,177(4046):314-322
95RaghuramanH,ChattopadhyayA.Biosci.Rep.,2007,27(4-5):189-223
96FadokVA,deCathelineauA,DalekeDL,HensonPM,BrattonDL.J.Biol.Chem.,2001,276(2):1071-1077
97TaitJF,GibsonD.Arch.Biochem.Biophys.,1992,298(1):187-191

