广西右江流域野生罗非鱼种群及其杂合性研究
郑雄 赵立朝 吴志强 张洁 黄亮亮 汪开成 张曼





摘要:【目的】明確广西右江流域野生罗非鱼的种群类别及其杂合情况,为广西右江流域罗非鱼的入侵防控及本土珍稀物种保护提供科学依据。【方法】从广西右江流域隆安、平果、田东、田阳和百色5个采样点采集野生罗非鱼样本共136尾,提取各采样点罗非鱼样本DNA,设计特异性引物对线粒体COⅠ基因及核基因(enc1、ptr和sh3px3)进行群体扩增,通过DNASTAR 7.1的Megalign程序对所有样本的COⅠ基因序列进行同源性比对分析,以确定罗非鱼样本的母本;采用MEGA 6.0对enc1、ptr和sh3px3基因进行序列比对和校正,并构建各采样点罗非鱼样本的系统发育进化树。【结果】广西右江流域5个采样点的罗非鱼母本种类共有4种,分别为尼罗罗非鱼(Oreochromis niloticus)、齐氏罗非鱼(Coptodon zillii)、莫桑比克罗非鱼(O. mossambicus)和奥利亚罗非鱼(O. aureus)。在26尾隆安罗非鱼样本中无杂合个体;在36尾平果罗非鱼样本中有3个杂合个体,杂合率为8.3%;在20尾田东罗非鱼样本中有2个杂合个体,杂合率为10.0%;在30尾田阳罗非鱼样本中有2个杂合个体,杂合率为6.7%;在24尾百色罗非鱼样本中有4个杂合个体,杂合率为16.7%。【结论】广西右江流域野生罗非鱼的种间杂交率并不高,但其杂交优势更有利于罗非鱼种群的扩散,进一步侵占土著鱼种的生存空间,严重威胁广西右江流域鱼类资源的多样性,有关部门应加强监管力度以防止野生罗非鱼种群迅速扩散。
关键词: 罗非鱼;DNA条形码;杂合性;广西右江流域
中图分类号: S965.125 文献标志码: A 文章编号:2095-1191(2019)12-2797-09
Wild tilapia population and its hybridization in
Youjiang River, Guangxi
ZHENG Xiong1, ZHAO Li-chao2, WU Zhi-qiang1,3*, ZHANG Jie4,
HUANG Liang-liang1,5, WANG Kai-cheng2, ZHANG Man2
(1College of Environmental Science and Engineering, Guilin University of Technology, Guilin, Guangxi 541006, China; 2College of Animal Science and Technology, Guangxi University,Nanning 530004, China; 3Collaborative Innovation Center for Water Pollution Control and Water Safety in Karst Area, Guilin University of Technology, Guilin, Guangxi 541004, China; 4Institute of Zoology, Chinese Academy of Sciences, Beijing 100101, China; 5Guangxi Key Laboratory of Environmental Pollution Control Theory and Technology, Guilin, Guangxi 541004, China)
Abstract:【Objective】Researching species and heterozygosity among the population of wild tilapias in Youjiang Ri-ver basin of Guangxi to provide scientific base for prevention of tilapia invasion and conservation of indigenous species.【Method】A total of 136 tilapia individuals from Long’an, Pingguo, Tiandong, Tianyang and Baise in Youjiang River basin were studied. The DNA extraction, PCR amplification and gene sequencing based on COⅠ from mitochondria,and enc1,ptr, sh3px3 from ribosome with specific primers for each individual were performed. Through the megalign program of DNASTAR 7.1, the homologies of COⅠ gene sequences of all samples were compared and analyzed to determine the female parents of tilapia samples. The sequences of enc1,ptr,sh3px3 gene were compared and corrected by MEGA 6.0, and the phylogenetic trees of all samples were constructed. 【Result】The results showed that there were four female parents species in the five sampling sites, they were Oreochromis niloticus, Coptodon zillii, O. mossambicus and O. aureus. The hybrid rates of samples in Longan(26 individuals, 0 hybrid individual), Pingguo(36 individuals, 3 hybrid individuals), Tiandong(20 individuals, 2 hybrid individuals), Tianyang(30 individuals, 2 hybrid individuals) and Baise(24 individuals, 4 hybrid individuals) were respectively 0, 8.3%, 10.0%, 6.7% and 16.7%. 【Conclusion】Although the interspecific hybridization rate is not too high, the situation will be more conducive to the spread of wild tilapia population and further encroachment on the living space of indigenous species,and severely threaten diversity of local fishery resources in Youjiang River basin due to heterosis. The government department concerned ought to enhance supervision,and prevent further proliferation of population of wild tilapia.
Key words: tilapia; DNA barcode; hybridization; Youjiang River of Guangxi
0 引言
【研究意义】罗非鱼是我国主要淡水养殖水产品之一,也是广西的主要养殖鱼类(Bergstrom and Mensinger,2009;张红燕等,2015)。罗非鱼对环境的适应能力极强,具有发病少、耐低氧、食性杂、繁殖快及生长快的特性,其肉质鲜美、无骨间刺、富含营养,在促进渔民收入增长及渔业经济发展方面发挥着重要作用(刘孝华,2007)。广西属于典型的热带季风和亚热带季风气候区域,非常适宜罗非鱼生长繁殖(Chen et al.,2012);但罗非鱼也是广西江河流域的主要入侵鱼类之一(汪开成等,2018)。罗非鱼从养殖环境逃逸并成功入侵到自然水体后具有较强的表型适应性和种间竞争力(Arbačiauskas et al.,2013;管嘉俊等,2017),会对当地生物多样性与生态环境造成严重威胁(赵立朝等,2019)。因此,明确广西野生罗非鱼的主要品种及入侵种群,研究其杂合情况,可为广西内陆水域生态安全保护及罗非鱼入侵预警和防治提供理论依据。【前人研究进展】DNA条形码是利用基因组中通用短序列片段对物种进行鉴别的技术,具有自动化高、操作简易及鉴定准确的优点(Hebert et al.,2003;Kress et al.,2005;Hollingsworth et al.,2011;Zhang and Hanner,2011)。其中,线粒体细胞色素C氧化酶Ⅰ基因(COⅠ)因具有应用广泛、数据库信息完整等优点,是目前鱼类DNA条形码研究中最常用的基因之一(武宇鹏等,2011;单云晶等,2013)。但由于线粒体DNA源自于母本,且罗非鱼种间杂交频繁(郭奕惠等,2009),若只鉴别线粒体DNA则无法确定其父本,而需对其细胞核基因进行二次鉴定,才能确认种群类别(高连明,2015)。李建林等(2011)筛选出7个微卫星位点(GM066、UNH995、UNH948、GM017、UNH162、GM440和GM166)作为罗非鱼鉴定的分子标记,且每个位点均能将尼罗罗非鱼、奥利亚罗非鱼和杂交罗非鱼区分开来。Falade等(2016)对尼日利亚西南部淡水鲶鱼和两种罗非鱼的16S rRNA和COⅠ基因进行比较分析,结果发现基于COⅠ基因能精确地区分尖齿胡鲶、萨罗罗非鱼和齐氏罗非鱼。Ordoñez等(2017)对菲律宾养殖场内Oreochromis属10个品系罗非鱼、3个不同地点的红罗非鱼和2种纯系罗非鱼进行COⅠ基因比对分析,结果发现养殖罗非鱼母本可被明确区分为4种,但COⅠ基因无法区分同种不同品系的罗非鱼。Zhu等(2017)利用14个微卫星标记对3个不同地区180份红罗非鱼样本进行鉴定,发现台湾红罗非鱼与以色列红罗非鱼和马来西亚红罗非鱼均存在明显遗传差异。管嘉俊等(2018)对广西6个不同地点的尼罗罗非鱼D-loop序列进行比对分析,发现钦州尼罗罗非鱼种群个体间的遗传距离比玉林、南宁、贵港、防城港和北海群体的要远,因而具有更明显的遗传分化现象。总体而言,目前国内外已有大量针对罗非鱼种群遗传多样性的相关研究,但主要是对线粒体内1~2个基因位点进行研究,或利用微卫星标记对罗非鱼进行种群鉴定,而鲜见基于DNA条形码对罗非鱼细胞核基因的探索,或结合线粒体基因与细胞核基因进行研究。【本研究切入点】至今,有关广西罗非鱼的相关研究已有较多报道(强竣等,2012;Giery et al.,2015;沈夏霜等,2018),但尚无基于分子生物学对广西右江流域野生罗非鱼入侵种群进行研究的报道。【拟解决的关键问题】基于DNA分子鉴定对广西隆安、平果、田东、田阳及百色5个采样点的野生罗非鱼样本进行比对分析,通过设计引物对群体线粒体COⅠ基因及细胞核enc1、ptr和sh3px3基因进行PCR扩增测序(Li et al.,2007),旨在明确广西右江流域野生罗非鱼的种群类别及其杂合情况,为广西右江流域罗非鱼的入侵防控及本土珍稀物种保护提供科学依据。
1 材料与方法
1. 1 试验材料
2017年7月,在广西隆安、平果、田东、田阳和百色5个采样点进行罗非鱼样本采集,采集方式为垂钓和网捞,采集样本数量分别为26、36、20、30和24尾。采集后现场解剖并剪取鱼类肌肉组织,分别装入2.0 mL的离心管中,按照采样点粗略分类。罗非鱼肌肉组织10000 r/min离心3 min,采用移液枪吸取水分后,以无水乙醇浸泡换洗2次,每次换洗后倒掉溶液并置于通风橱风干(刘军等,2016),最后将肌肉组织浸泡在无水乙醇中,对每份组织样本进行无序随机标号,号码前面标记采样点名字的两个拼音首字母,如隆安写作LA。
1. 2 DNA提取
取出浸泡在无水乙醇中的罗非鱼肌肉组织,剪取小块肌肉并以双蒸水冲洗2次以上,将其剪碎后放入2.0 mL的离心管内,10000 r/min离心5 min,弃上清液(王娜泠等,2017)。使用DP324海洋动物基因组提取试剂盒[天根生化科技(北京)有限公司]进行DNA提取,以2.0%琼脂糖凝胶进行电泳检测,然后在凝胶成像系统上观察拍照(Rognon and Guyomard,1997;Ward et al.,2008),保留條带明亮或稍暗的样本进行后续试验,未扩增出条带的样本则进行DNA提取重复试验。
1. 3 PCR扩增及产物检测
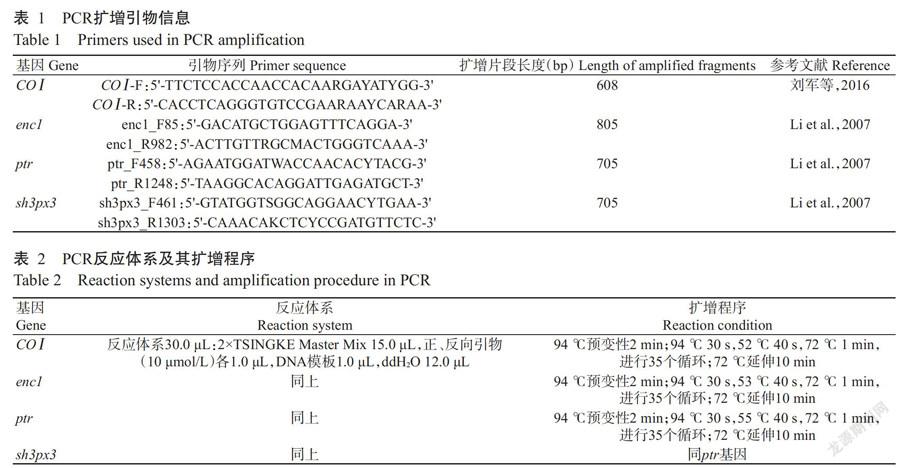
以引物COⅠ-F和COⅠ-R对线粒体COⅠ基因进行PCR扩增(郭新红等,2004),同时采用enc1_F85和enc1_R982、ptr_F458和ptr_R1248、sh3px3_F461和sh3px3_R1303分别对细胞核基因enc1、ptr和sh3px3进行PCR扩增(李晶等,2013)。所有引物均由北京擎科新业生物技术有限公司合成,引物信息及其反应体系分别见表1和表2。
1. 4 统计分析
对PCR扩增产物进行凝胶电泳检测,在凝胶成像系统上观察拍照,将扩增效果良好的条带送至北京擎科新业生物技术有限公司测序。通过DNASTAR 7.1的Megalign程序,将所有样本的COⅠ基因序列与GenBank中不同种罗非鱼相应位置序列进行同源性比对分析,以确定罗非鱼样本的母本。enc1、ptr和sh3px3基因测序结果均采用MEGA 6.0进行序列比对和校正(Tamura et al.,2013),并通过邻位相连法(Neighbor-joining,NJ)构建各采样点样本的系统发育进化树(李晶等,2013;Tamura et al.,2013),Bootstrap重复次数设为1000。
2 结果与分析
2. 1 PCR扩增结果
PCR扩增效果良好。以百色罗非鱼样本的ptr和sh3px3基因为例,2.0%琼脂糖凝胶电泳检测结果(图1)显示,扩增获得的条带大小约700 bp,清晰明亮,且无特异性扩增条带,也没有引物二聚体产生,可用于后续研究。
2. 2 COⅠ基因序列分析结果
以百色罗非鱼样本为例进行同源性比对分析,图2最右侧纵列KU565867.1、HM882909.1、EU751879.1和KJ553787.1分别对应NCBI中尼罗罗非鱼、齐氏罗非鱼、莫桑比克罗非鱼和奥利亚罗非鱼的COⅠ基因编号及其序列;从第5行起为百色罗非鱼样本的COⅠ序列编号,编号最前面的两个字母为地名缩写,数字为样本序号,如BS-C1_CF即为百色罗非鱼1号样本。同源性比对分析结果显示,尼罗罗非鱼、齐氏罗非鱼、莫桑比克罗非鱼和奥利亚罗非鱼的COⅠ基因序列分别与本研究中5个采样点罗非鱼样本的相应序列匹配度很高,同源性均在98.0%以上;尼罗罗非鱼、莫桑比克罗非鱼和奥利亚罗非鱼COⅠ基因的同源性也较高(92.0%~96.0%),与其同为Oreochromis属的实际情况相符,说明同属异种罗非鱼具有极高的基因相似度;齐氏罗非鱼与其他3种罗非鱼的同源性相对较低(85.0%~89.0%),与齐氏罗非鱼和其他3种罗非鱼不同属的情况也相符。
罗非鱼样本的母本种类可确定为4种:尼罗罗非鱼、齐氏罗非鱼、莫桑比克罗非鱼和奥利亚罗非鱼。基于罗非鱼COⅠ基因序列鉴定结果(表3)可知,所有罗非鱼样本中母本为尼罗罗非鱼的数量最多,齐氏罗非鱼次之,莫桑比克罗非鱼和奥利亚罗非鱼数量相对较少;各采样点的罗非鱼样本中,母本为尼罗罗非鱼的比例最高;除田东之外,其他采样点罗非鱼样本的母本均以齐氏罗非鱼的比例次之,而莫桑比克和奥利亚罗非鱼的比例相对较低,其中,百色和隆安两个采样点未检出以奥利亚罗非鱼作为母本的样本,而在田东采样点未检出以齐氏罗非鱼和莫桑比克罗非鱼作为母本的样本。
2. 3 核基因的系统发育进化分析结果
基于enc1、ptr和sh3px3基因分别构建获得3种系统发育进化树,每个分支后的字母为地点名缩写,中间数字为罗非鱼样本序号,后面字母为其COⅠ基因对母本的鉴定结果。以N代替尼罗罗非鱼、Z代替齐氏罗非鱼、M代替莫桑比克罗非鱼、A代替奥利亚罗非鱼,特殊序号0 N和0 Z分别代表从NCBI中检索到的纯系尼罗罗非鱼和齐氏罗非鱼各核基因序列(表4)。
3種核基因的系统发育进化树(图3~图5)显示,每种系统发育进化树中所有样本均聚类成两大分支,分别代表两个不同的属,即齐氏罗非鱼的Coptodon属和其他3种罗非鱼的Oreochromis属。齐氏罗非鱼个体间的遗传距离较小,系统发育进化树分支整齐,且节点支持率高,大多都在60.0%以上;奥利亚罗非鱼和尼罗罗非鱼同属不同种罗非鱼分为两支,其遗传距离较大;而莫桑比克罗非鱼几乎完全和尼罗罗非鱼聚为一支,其遗传距离非常小,难以进行区分辨别。
基于enc1基因的系统发育进化树(图3)显示,田阳45号(TY45A)、平果2号(PG2A)和15号(PG15A)、田东3号(TD3A)和19号(TD19A)样本的母本为奥利亚罗非鱼,但未与奥利亚罗非鱼聚成一支,反而处于尼罗罗非鱼分支内,可认定其为杂合个体;百色7号(BS7Z)和19号(BS19Z)样本的母本为齐氏罗非鱼,但处于不同属的尼罗罗非鱼分支内,也认定其为杂合个体。基于ptr基因的系统发育进化树(图4)显示,田阳40号(TY40Z)样本的母本为齐氏罗非鱼,却分类在不同属的奥利亚罗非鱼分支内,可认定其为杂合个体。基于sh3px3基因的系统发育进化树(图5)显示,平果1号(PG1Z)、百色20号(BS20Z)和21号(BS21Z)样本的母本为齐氏罗非鱼,虽然其在齐氏罗非鱼属分支内,但与其他大多数齐氏罗非鱼并不在同种的分支内,且距离稍远,节点支持低,故认为其与纯合齐氏罗非鱼具有一定的遗传距离,即为杂合个体。
综上所述,在26尾隆安罗非鱼样本中无杂合个体;在36尾平果罗非鱼样本中有3个杂合个体,杂合个体数占该采样点罗非鱼样本总数的8.3%;在20尾田东罗非鱼样本中有2个杂合个体,杂合个体数占该采样点罗非鱼样本总数的10.0%;在30尾田阳罗非鱼样本中有2个杂合个体,杂合个体数占该采样点罗非鱼样本总数的6.7%;在24尾百色罗非鱼样本中有4个杂合个体,杂合个体数占该采样点罗非鱼样本总数的16.7%。
3 讨论
本研究的COⅠ基因同源性比对及系统发育进化分析结果显示,26尾隆安罗非鱼样本的母本种类有尼罗罗非鱼、齐氏罗非鱼和莫桑比克罗非鱼,杂合率为0;36尾平果罗非鱼样本的母本种类有尼罗罗非鱼、齐氏罗非鱼、莫桑比克罗非鱼和奥利亚罗非鱼,杂合率为8.3%;20尾田东罗非鱼样本的母本种类有尼罗罗非鱼和奥利亚罗非鱼,杂合率为10.0%;30尾田阳罗非鱼样本的母本种类有尼罗罗非鱼、齐氏罗非鱼、莫桑比克罗非鱼和奥利亚罗非鱼,杂合率为6.7%;24尾百色罗非鱼样本的母本种类有尼罗罗非鱼、齐氏罗非鱼和莫桑比克罗非鱼,杂合率为16.7%。除了隆安罗非鱼没有种间杂交情况外,广西右江其他河段罗非鱼均存在种间杂交情况。说明在广西右江流域,野生罗非鱼的确存在着杂合情况,但杂合现象不明显,且杂合率并不高。
本研究发现近乎一半的杂合个体,其母本是奥利亚罗非鱼,另一半则为齐氏罗非鱼。以奥利亚罗非鱼和尼罗罗非鱼可杂交出尼奥罗非鱼(奥利亚♀+尼罗♂),且奥利亚罗非鱼(♀)与尼罗罗非鱼(♂)的杂交后代表现出高雄性率和杂交双重优势(万松良等,2018)。由系统发育进化分析结果可知,所有母本为奥利亚罗非鱼的杂合个体均处于尼罗罗非鱼分支内,COⅠ基因在线粒体内继承自母本,核基因则继承自父本和母本,因此可确定是奥利亚罗非鱼(♀)与尼罗罗非鱼(♂)杂交。杂交产生的子代具有杂交优势,尽管杂合情况并不严重,但仍会对当地生态环境和生物多样性造成威胁。李莉好等(2008)研究表明,尼罗♀+奥利亚♂杂交产生的子代较奥利亚♀+尼罗♂杂交产生的子代具有更强的遗传多样性和多态性,因而拥有更强的杂交优势。本研究中并未发现以尼罗罗非鱼为母本的杂合个体,只出现奥利亚♀+尼罗♂的杂交方式,可能是不同罗非鱼种群的性别比例不同所致。此外,系统发育进化分析结果显示齐氏罗非鱼与其他罗非鱼虽不在同一属内,但其杂交数仍占罗非鱼样本杂交总数的一半,说明罗非鱼可进行属间杂交,且较频繁,也表明齐氏罗非鱼具有很强的杂交能力。王金龙等(2007)也曾研究证实,奥利亚罗非鱼同样拥有强大的杂交能力,其作为母本可与作为父本的鳜(Siniperca chuatsi)进行科间杂交,并产生可育后代。
在罗非鱼种群杂合率表现最低的隆安罗非鱼样本中,发现其母本仅有尼罗罗非鱼、齐氏罗非鱼和莫桑比克罗非鱼,而未检出奥利亚罗非鱼,进一步说明奥利亚罗非鱼杂交占据野生罗非鱼杂交的比例很高。在罗非鱼种群杂合率最高的百色罗非鱼样本中,同样未检出以奥利亚罗非鱼作为母本的杂合个体,也说明齐氏罗非鱼杂交占据野生罗非鱼杂交的比例很高。在平果罗非鱼样本中同时发现有4种罗非鱼作为母本,理论上应具有较高的杂合率,但其杂合率仅8.3%;在田阳罗非鱼样本中,其罗非鱼种类构成与平果罗非鱼样本相似,杂合率同样不高,仅6.7%。究其原因可能与各罗非鱼种群的数量、种群性比构成及外部温度等因素不同有关。田东罗非鱼样本的母本种类只有尼罗罗非鱼和奥利亚罗非鱼,其杂合率却达10.0%,表明尼罗罗非鱼与奥利亚罗非鱼在自然条件下可发生杂交,且具有较高的杂合率。
在广西右江流域完成入侵的罗非鱼主要以尼罗罗非鱼和齐氏罗非鱼为主,同时存在一些其他种类的罗非鱼,如莫桑比克罗非鱼和奥利亚罗非鱼等。丰富的入侵种类为罗非鱼种间几乎不存在的生殖隔离提供了良好杂交基础,同时由于外界环境压力的作用,致使广西右江上游罗非鱼杂合情况极其普遍。在本研究的5个采样点中,罗非鱼杂合率最高的百色采样点处于广西右江最上游,渔业资源丰富,水流较缓,有利于鱼类繁殖;而没有种间杂交的隆安采样点处于广西右江最下游,地势低,水流较湍急,不利于鱼类产卵繁殖,说明环境因素可能是影响罗非鱼种间杂合的重要因素之一。这与高天扬等(2018)对北江鱼类群落进行调查的结果相似,上游鱼类多样性多于下游,即上游鱼类拥有更多的捕食与繁衍机会。
罗非鱼具有适应性强、发病少、耐低氧、食性杂、繁殖快及生长快的特性,是联合国粮食及农业组织(FAO)向全世界大力推广的优良养殖品种(谭细畅等,2012),但这些特性也促使罗非鱼成为世界性生物入侵鱼类(Peterson et al.,2006)。罗非鱼种间杂交表现出一定的杂交优势,如奥利亚罗非鱼(♀)与尼羅罗非鱼(♂)的杂交后代表现出高雄性率和杂交双重优势(万松良等,2018),莫桑比克罗非鱼与红罗非鱼或尼罗罗非鱼的杂交后代均具有更强的耐高盐性(Urmaza and Aguilar,2005)。这种杂交优势进一步提升了罗非鱼对环境的适应能力,使其能更快地适应新的生境,提高存活率。杂合罗非鱼对环境适应能力的增强加快了罗非鱼对本土鱼类生态位的侵占,而导致本土鱼类资源多样性受到严重威胁(周辉明等,2011)。广西右江流域野生罗非鱼的杂合率虽然不高,但仍然建议有关管理部门加强对野生罗非鱼的监管力度,采取有针对性的措施降低罗非鱼种群的分布和扩散,建立完善的本土鱼类保护机制,以保护广西右江流域鱼类资源多样性和生物完整性。
4 结论
广西右江流域野生罗非鱼的种间杂合率并不高,但其杂交优势更有利于罗非鱼种群的扩散,进一步侵占土著鱼种的生存空间,严重威胁广西右江流域鱼类资源的多样性,有关部门应加强监管力度以防止野生罗非鱼种群迅速扩散。
参考文献:
高连明. 2015. DNA条形码在生物多样性编目与评价中的应用[J]. 生物多样性,23(3):286-287. [Gao L M. 2015. Applications of DNA barcoding in biodiversity inventory and assessment[J]. Biodiversity Science,23(3):286-287.]
高天扬,谢迪,彭宁东,张少平,李朝,罗锦桢,王春晖,赵俊. 2018. 北江鱼类群落结构多样性及其演替趋势[J]. 水生态学杂志,39(4):54-62. [Gao T Y,Xie D,Peng N D,Zhang S P,Li C,Luo J Z,Wang C H,Zhao J. 2018. Fish community structure diversity and succession in the Bei-jiang River[J]. Journal of Hydroecology,39(4):54-62.]
管嘉俊,韩卓君,郑慧芳,李文红,郭忠宝. 2018. 广西主要养殖区内逃逸尼罗罗非鱼线粒体D-loop控制区序列分析[J]. 激光生物学报,27(5):451-459. [Guan J J,Han Z J,Zheng H F,Li W H,Guo Z B. 2018. The analysis of the mitochondrial D-loop control region sequences from escape Oreochromis niloticus in the main aquaculture area of Guangxi[J]. Acta Laser Biology Sinica,27(5):451-459.]
管嘉俊,郑惠芳,乔庆,周毅,郭忠宝,陈日钊,李文红. 2017. 广西主要养殖区内逃逸尼罗罗非鱼形态差异分析[J]. 南方农业学报,48(11):2071-2077. [Guan J J,Zheng H F,Qiao Q,Zhou Y,Guo Z Z,Chen R Z,Li W H. 2017. Morphological variation analysis on escaped Nile tilapia from main aquaculture areas in Guangxi[J]. Journal of Southern Agriculture,48(11):2071-2077.]
郭新红,刘少军,刘巧,刘筠. 2004. 鱼类线粒体 DNA 研究新进展[J]. 遗传学报,31(9):983-1000. [Guo X H,Liu S J,Liu Q,Liu Y. 2004. New progresses on mitochondrial DNA in fish[J]. Acta Genetica Sinica,31(9):983-1000.]
郭奕惠,喻達辉,黄桂菊,李莉好,符云,叶卫. 2009. 中国主要养殖罗非鱼亲缘关系的COI序列分析[J]. 华中农业大学学报,28(1):75-79. [Guo Y H,Yu D H,Huang G J,Li L H,Fu Y,Ye W. 2009. Phylogenetic relationship of Oreochromis tilapias cultured in mainland China based on COI sequences[J]. Journal of Huazhong Agricultural University,28(1):75-79.]
李建林,唐永凯,李红霞,俞菊华,徐跑. 2011. 尼罗罗非鱼、奥利亚罗非鱼及其杂交后代微卫星标记鉴别和种质纯度分析[J]. 上海海洋大学学报,20(1):27-33. [Li J L,Tang Y K,Li H X,Yu J H,Xu P. 2011. Identification and pureness of germplasm analysis of Oreochromis niloticus,Oreochromis aureus,and their hybrids by microsatellite markers[J]. Journal of Shanghai Ocean University,20(1):27-33.]
李晶,刘雪涛,郭华威,岳碧松,李静. 2013. 动物DNA条形码之分析方法研究进展[J]. 四川动物,32(6):950-954. [Li J,Liu X T,Guo H W,Yue B S,Li J. 2013. Progress of analytic methods in animal DNA barcoding[J]. Si-chuan Journal of Zoology,32(6):950-954.]
李莉好,喻达辉,黄桂菊,叶卫,杜博,符云,童馨,郭奕惠. 2008. 尼罗罗非鱼、奥利亚罗非鱼及其正、反杂交群体的遗传多样性[J]. 中国水产科学,15(4):585-592. [Li L H,Yu D H,Huang G J,Ye W,Du B,Fu Y,Tong X,Guo Y H. 2008. Genetic diversity of Oreochromis niloticus,O. aureus and their reciprocally-crossed hybrid stocks[J]. Journal of Fishery Sciences of China,15(4):585-592.]
刘军,赵良杰,凡迎春,张新磊,刘其根. 2016. 鱼类环境 DNA 研究中通用引物的筛选验证[J]. 淡水渔业,46(1):9-17. [Liu J,Zhao L J,Fan Y C,Zhang X L,Liu Q G. 2016. Universal primer screening and verification for fish environment DNA research[J]. Freshwater Fisheries,46(1):9-17.]
刘孝华. 2007. 罗非鱼的生物学特性及养殖技术[J]. 湖北农业科学,46(1):115-116. [Liu X H. 2007. Biological cha-racteristics and cultivation cechnology of the tilapia mossambica[J]. Hubei Agricultural Sciences,46(1):115-116.]
强竣,杨弘,王辉,徐跑,柒壮林,何杰. 2012. 海豚链球菌感染对不同品系罗非鱼血液生化指标和肝脏HSP70 mRNA表达的影响[J]. 水产学报,36(6):958-968. [Qiang J,Yang H,Wang H,Xu P,Qi Z L,He J. 2012. Studies on blood biochemical indices and expression of hepatic HSP70 mRNA of different tilapia strains artificially cha-llenged with Streptococcus iniae[J]. Journal of Fisheries of China,36(6):958-968.]
单云晶,鲁翠云,李超,张明昭,顾颖,孙效文. 2013. 基于线粒体COI基因序列的5种鲤养殖品种遗传多样性研究[J]. 中国水产科学,20(5):931-938. [Shan Y J,Lu C Y,Li C,Zhang M Z,Gu Y,Sun X W. 2013. Study of DNA barcoding and genetic variation based on the mitochondrial COI genesequences in five carp varieties[J]. Journal of Fishery Sciences of China,20(5):931-938.]
沈夏霜,敖秋桅,甘西,谭芸,罗永巨,梁军能,朱佳杰. 2018. 吉富罗非鱼抗病品系F5代抗病性能和生长性能的评估[J]. 南方水产科学,14(3):83-90. [Shen X S,Ao Q G,Gan X,Tan Y,Luo Y J,Liang J N,Zhu J J. 2018. Estimation of disease resistance and growth in F5 generation families of GIFT tilapia[J]. South China Fisheries Scien-ce,14(3):83-90.]
谭细畅,李新辉,李跃飞,李捷. 2012. 尼罗罗非鱼早期发育形态及其在珠江水系的空间分布[J]. 生物安全学报,21(4):295-299. [Tan X C,Li X H,Li Y F,Li J. 2012. Early development and spatial distribution of the Nile tilapia (Oreochromis niloticus) in the Pearl River[J]. Journal of Biosafety, 21(4):295-299.]
萬松良,筴金华,任炳琛,魏志宇,张艳红,万珊. 2018. 奥利亚罗非鱼(WZ♀)×尼罗罗非鱼(XY♂、YY♂)杂交子代的性比分析[J]. 水产学杂志,31(3):14-19. [Wan S L,Ce J H,Ren B C,Wei Z Y,Zhang Y H,Wan S. 2018. Sex ratio analysis of hybrid F1 from female blue tilapia Oreochromis aureus(WZ ♀)×male Nile tilapia O. niloticus (XY ♂,YY ♂) crosses[J]. Chinese Journal of Fisheries,31(3):14-19.]
汪开成,吴志强,武琳,谭虹雨,赵立朝,刘博文,赵雪倩,高胜男,程光平,张曼,黄钧. 2018. 广西红水河外来红腹罗非鱼形态差异分析[J]. 西南农业学报,31(10):2208-2216.[Wang K C,Wu Z Q,Wu L,Tan H Y,Zhao L C,Liu B W,Zhao X Q,Gao S N,Cheng G P,Zhang M,Huang J. 2018. Morphological variation analysis on the redbelly tilapia(Coptodon zillii) of Hongshui River in Guangxi[J]. Southwest China Journal of Agricultural Sciences,31(10):2208-2216.]
王金龙,杨弘,吴婷婷,张志伟. 2007. 奥利亚罗非鱼(♀)×鳜(♂)远缘杂交子代的遗传结构[J]. 中国水产科学,14(1):32-38. [Wang J L,Yang H,Wu T T,Zhang Z W. 2007. Genetic constitutions of distant hybridization offsprings between Oreochromis aurea (♀) ×Siniperca chuatsi(♂)[J]. Journal of Fishery Sciences of China,14(1):32-38.]
王娜泠,胡则辉,卞光明,柴学军. 2017. DNA条形码技术及其在水生动物中的应用[J]. 浙江海洋大学学报(自然科学版),36(5):432-438. [Wang N L,Hu Z H,Bian G M,Chai X J. 2017. DNA barcoding and its application in aquatic animals[J]. Journal of Zhejiang Ocean University(Natural Science),36(5):432-438.]
武宇鹏,丁亮,李捷,武春生,范仁俊,朱朝东. 2011. DNA条形码的应用进展及讨论[J]. 环境昆虫学报,33(1):99-106.[Wu Y P,Ding L,Li J,Wu C S,Fan R J,Zhu C D. 2011. DNA barcoding:Current progresses and discussions[J]. Journal of Environmental Entomology,33(1):99-106.]
张红燕,袁永明,贺艳辉,王红卫,代云云. 2015. 中国罗非鱼生产与贸易现状分析及建议[J]. 中国渔业经济,33(3):95-100. [Zhang H Y,Yuan Y M,He Y H,Wang H W,Dai Y Y. 2015. Analysis and suggestions on production and trade of tilapia in China[J]. Chinese Fisheries Economics,33(3):95-100.]
赵立朝,吴志强,张曼,郑雄,李德越,刘博文. 2019. 广西南部主要水系野生罗非鱼建群状况调查分析[J]. 南方农业学报,50(2):397-404. [Zhao L C,Wu Z Q,Zhang M,Zheng X,Li D Y,Liu B W. 2019. Establishment of wild tilapia population community in mainrivers of southern Guangxi[J]. Journal of Southern Agriculture,50(2):397-404.]
周輝明,刘引兰,李飞. 2011. 广西右江鱼类资源研究[J]. 江西水产科技,(3):22-25. [Zhou H M,Liu Y L,Li F. 2011. Study on fish resources in Youjiang River,Guangxi[J]. Jiangxi Fishery Science and Technology,(3):22-25.]
Arbačiauskas K,Lesutiene J,Gasiūnait Z R. 2013. Feeding strategies and elemental composition in Ponto-Caspian peracaridans from contrasting environments:Can stoichiometric plasticity promote invasion success?[J]. Freshwater Biology,58(5):1052-1068.
Bergstrom M A,Mensinger A F. 2009. Interspecific resource competition between the invasive round goby and three native species: Logperch,slimy sculpin,and spoonhead sculpin[J]. Transactions of the American Fisheries Socie-ty,138(5):1009-1017.
Chen M,Li L P,Wang R,Liang W W,Huang Y,Li J,Lei A Y,Huang W Y,Gan X. 2012. PCR detection and PFGE genotype analyses of streptococcal clinical isolates from tilapia in China[J]. Veterinary Microbiology,159(3-4):526-530.
Falade M O,Opene A J,Benson O. 2016. DNA barcoding of Clarias gariepinus,Coptodon zillii and Sarotherodon me-lanotheron from Southwestern Nigeria[J]. F1000 Research,5:1268.
Giery S T,Layman C A,Langerhans R B. 2015. Anthropogenic ecosystem fragmentation drives shared and unique pa-tterns of sexual signal divergence among three species of Bahamian mosquitofish[J]. Evolutionary Applications,8(7):679-691.
Hebert P D N,Cywinska A,Ball S L,Dewaard J R. 2003. Bio-logical identifications through DNA barcodes[J]. Procee-dings of the Royal Society B-biological Science,270(1512):313-321.
Hollingsworth P M,Graham S W,Little D P. 2011. Choosing and using a plant DNA barcode[J]. PLoS One,6(5):e19254.
Kress W J,Wurdack K J,Zimmer E A,Weigt L A,Janzen D H. 2005. Use of DNA barcodes to identify flowering plants[J]. Proceedings of the National Academy of Sciences of the United States of America,102(23):8369-8374.
Li C H,Orti G,Zhang G,Lu G Q. 2007. A practical approach to phylogenomics:The phylogeny of ray-finned fish(Actinopterygii) as a case study[J]. BMC Evolutionary Biology,7:44.
Ordoñez J F F,Ventolero M F H,Santos M D. 2017. Maternal mismatches in farmed tilapia strains(Oreochromis spp.) in the Philippines as revealed by mitochondrial COI gene[J]. Mitochondrial DNA (Part A),28(4):526-535.
Peterson M S,Slack W T,Waggy G L,Finley J,Woodley C M,Partyka M L. 2006. Foraging in non-native environments:Comparison of Nile tilapia and three co-occurring native centrarchids in invaded coastal Mississippi watersheds[J]. Environmental Biology of Fishes,76:283-301.
Rognon X,Guyomard R. 1997. Mitochondrial DNA differentia-tion among east and west African nile tilapia populations[J]. Journal of Fish Biology,51(1):204-207.
Tamura K,Stecher G,Peterson D,Filipski A,Kumar S. 2013. MEGA 6:Molecular evolutionary genetics analysis version 6.0[J]. Molecular Biology and Evolution,30(12):2725-2729.
Urmaza E B,Aguilar R O. 2005. Growth performance of saline-tolerant tilapia produced from cross combinations of various tilapia spieces[J].Journal of Aquaculture in the Tropics,20(1):11-27.
Ward R D,Costa F O,Holmes B H,Steinke D. 2008. DNA barcoding of shared fish species from the North Atlantic and Australasia: Minimal divergence for most taxa,but Zeus faber and Lepidopus caudatus each probably constitute two species[J]. Aquatic Biology,3(1):71-78.
Zhang J B,Hanner R. 2011. DNA barcoding is a useful tool for the identification of marine fishes from Japan[J]. Bioche-mical Systematics and Ecology,39(1):31-42.
Zhu W B,Yang H,Yuan X H,Dong Z J,Fu J J,Wang L M,Su S Y,Liu N,Song F B,Chen X T. 2017. High genetic diversity and differentiation in three red tilapia stocks revealed by microsatellite DNA marker analysis[J]. Aquaculture International,25(6):1997-2006.
(責任编辑 兰宗宝)

