基于Sequenom MassARRAY SNP技术检测嗅觉受体基因与工作犬嗅探能力的关联性研究
高一龙, 贺星亮, 强京宁, 宋珍华, 温 海, 陶晓冉, 孙 勇, 秦海斌, 包喜军, 孟 戈, 朱 骞
(公安部南京警犬研究所, 警犬技术公安部重点实验室, 南京 210012)
鉴于犬的嗅觉能力突出, 目前各国警方广泛应用训练有素的工作犬搜查诸如爆炸物、毒品等隐匿的违禁品,打击和预防犯罪。警犬的嗅探能力是受犬嗅觉受体(OR)基因调控的,因此如果能够运用分子生物学技术开展警犬嗅探能力与OR 基因之间的关联性研究工作,发现主要的分子标记来挑选嗅觉灵敏的受训犬,可以显著提高警犬的训成率。
OR 是由嗅觉细胞表达的一种蛋白质,OR 基因属于G 蛋白偶联受体超家族[1]。1991 年, 首先在褐家鼠中发现OR 基因超家族[2],阐明了嗅觉系统的结构组成和工作原理。OR 基因很小, 每一OR 由大约1 kb 单一编码的外显子组成,编码区没有内含子。Olender 等[3]研究识别了犬基因组中1300 个OR 基因,约占犬OR 基因组的80%。虽然目前科研人员对犬OR 基因的组成结构、分类分布和多态性等方面展开了研究,但很少有人对犬OR 基因多态性与其嗅探能力之间的关联性进行研究。
在本研究工作中,以警用工作犬作为模式动物,采用Sequenom MassARRAY SNP 分型技术,对OR 基因的68 个SNP 位点进行分型检测,并分析不同SNP 基因型与工作犬嗅探能力之间的关联性,来探索验证犬主效OR 基因SNP 位点,寻找与犬嗅探能力相关的分子标记。
1 材料与方法
1.1 动物
本研究受试对象是由113只犬组成的群体,为来自公安部南京警犬研究所实验动物中心生产的工作犬以及各市局警犬队的1~3 岁的史宾格犬44 只(雄24,雌20)、拉布拉多犬40 只(雄25,雌15)和德国牧羊犬29 只(雄16,雌13)。这些犬大多是经过实战检验,并且通过年度复核的嗅探犬,或者是在某个特定专业方向进行训练,处于训练最后一个阶段即实战验证之前的嗅探犬。所有工作犬全年饲养于户外,一犬一舍(4 m×4 m),犬舍屋顶有隔热层,并有独立的户外运动场(15 m×6 m)。采用颗粒饲料定时定量饲喂,自由饮水,每日适度散放、运动,定时训练。全程兽医监控健康保障。带犬民警和饲养管理人员,按照犬训练使用保障条例以人道的方式对待所有受试工作犬。
1.2 主要仪器及试剂
2720 型PCR 仪购自美国ABI 公司;System electrophoresis Mupid (Mupid 2 plus)电泳槽购自日本TaKaRa公司; image system(EUV-LDUV)凝胶成像系统购自韩国 Biotech公司; Centrifuge (MICRO 17TR)离心机购自韩国Hanil公司; Agena Mass ARRAY购自美国Agena 公司。血液基因组DNA 提取试剂盒(DP348)和蛋白酶K 购自天根生化科技有限公司;PCR Enzyme 购自瑞士Roche 公司; PCR Accessory Set、SpectroCHIP Resin Kit 和IPLEX Gold Reagent Kit(Large)均购自美国Agena 公司; 滤膜(0.22 μm)购自南京天为生物科技有限公司; 引物由英潍捷基(上海)有限公司合成。
1.3 方法
1.3.1 嗅探能力测试以及评分 本实验研究参考文献[4]测试方法,结合公安机关使用的警犬专业考核方法,通过警犬训导员专业考核评分(professional test,PT)和生物本能中性测试(instinct test,IT)两种方式,综合考核受试犬只的嗅探能力。PT 法(百分制)是由具有丰富经验的专业警犬训导员,按照公安部颁发的缉毒犬、搜爆犬和血迹搜索犬考核方法和评分标准,对受试犬只的嗅探能力按百分制进行打分。IT 法(五分制)是设置与专业方向无关的食物气味的中性测试方法,利用动物觅食的本能,考核工作犬嗅觉的灵敏性[4]。
最终结果以这2 种测试方法的加权成绩得分(grading score,GS),确定嗅探能力等级,GS 越高,则意味着该犬的嗅探能力等级越高。
加权公式: GS= PT×0.1×0.5 + IT
1.3.2 血样采集 由具备资格兽医,对试验犬使用一次性真空抗凝管颈静脉采血,每只2~5 mL,送至实验室-20 ℃冰箱保存。
1.3.3 犬基因组DNA提取与检测 按照试剂盒说明书提取基因组DNA,琼脂糖凝胶电泳质检,并测定浓度和吸光度(A)值, 合格的DNA样品, A260/A280在1.6~2.0。质检合格的DNA 将浓度调整到50 ng/μL。
1.3.4 OR 基因SNP 的筛选 本研究根据前期预研实验和参阅相关文献[5,6], 从犬OR 基因超级家族的SNP 数据库中, 挑选出68 个SNP 位点。挑选原则:(1)具有较高多态性, 最小等位基因频率(MAF)>0.1;(2)考虑到OR基因SNP在犬种分布上的异质性,例如某些OR基因在某一犬种中的SNP位点数目特别多,而在另外一个犬种的SNP 位点相对较少,因此重点将文献[5,6]中所发现的史宾格犬和拉布拉多犬这2 个犬种的OR 基因SNP 位点纳入范围(表1)。

表1 挑选的SNP 信息(部分)
1.3.5 引物设计 根据SNP 的ID 从NCBI 数据库(http://www.ncbi.nlm.nih.gov/),查找所在OR 基因SNP 位点上下游100 bp 参考序列,使用Sequenom公司的引物设计软件Assay design3.1,设计PCR反应和单碱基扩展引物并合成,引物具体序列信息见表2。
1.3.6 PCR 扩增 采用多重PCR 技术, 在384孔板中进行, 每个反应体系总体积为5 μL, 在兼容384孔板的PCR 仪上,进行扩增反应。 PCR 反应条件:95 ℃预变性2 min; 95 ℃ 20 s,56 ℃ 30 s,72 ℃60 s,共45 个循环; 72 ℃再延伸3 min。
1.3.7 PCR产物碱性磷酸酶处理 在PCR反应结束后,将配制好的SAP mix 溶液加入384 孔PCR 反应板,进行碱性磷酸酶处理反应。反应体系总体积为7 μL (PCR 产物5 μL,SAP 混合液2 μL)。碱性磷酸酶处理反应条件: 37 ℃: 40 min; 85 ℃ 5 min,4 ℃维持。
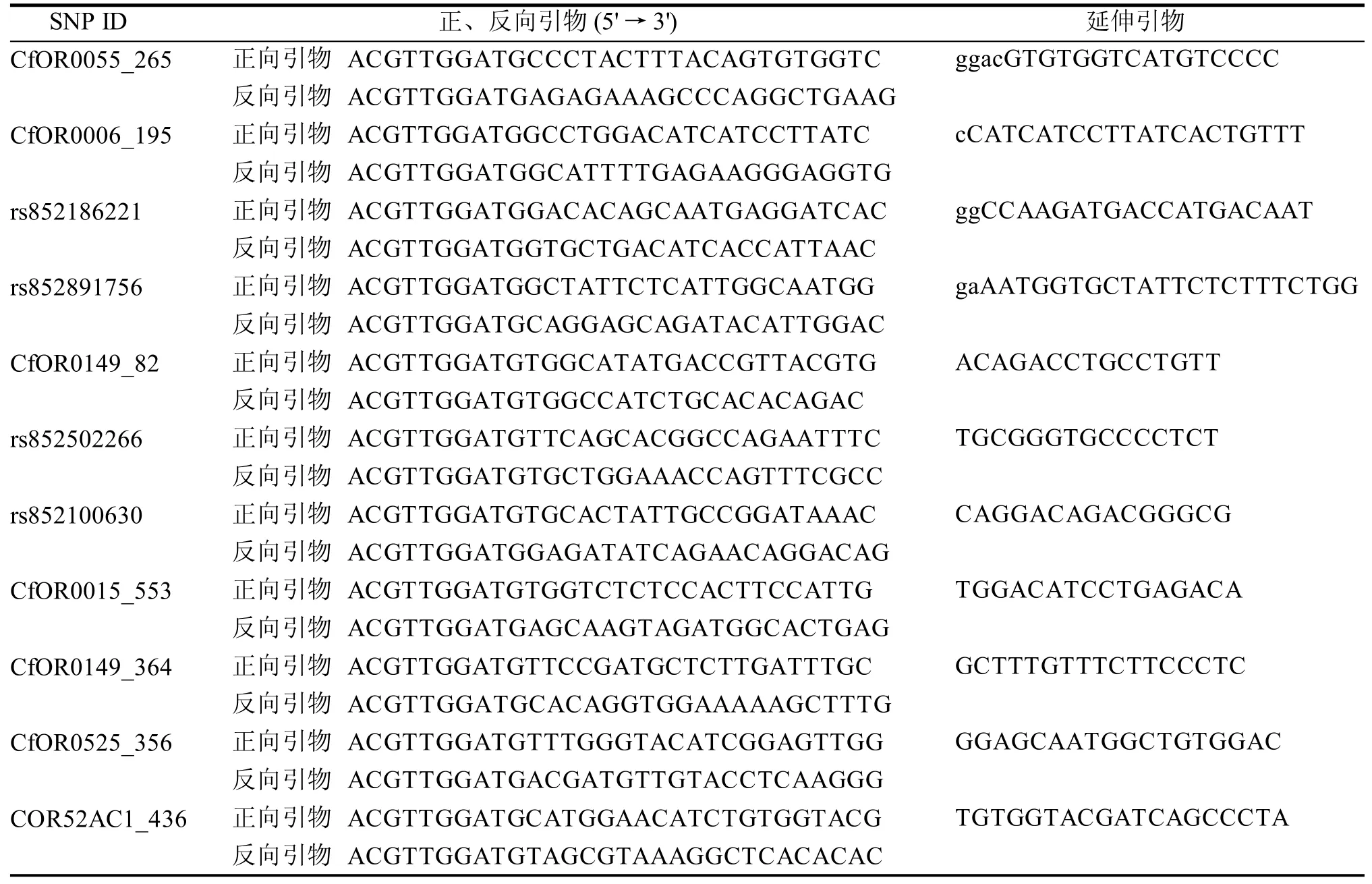
表2 SNP 引物序列信息(部分)
1.3.8 单碱基延伸 取出反应产物,将配制好的Extend mix 对应加入384 孔反应板。对于每个反应孔,单碱基延伸反应体系包含SAP 处理后PCR 产物7 μL 及 Extend Mix 液2 μL,总体系9 μL。启动PCR 仪进行单碱基延伸反应。单碱基延伸PCR反应条件: 94 ℃预变性30 s;94 ℃ 5 s,(52 ℃ 5 s,80 ℃ 5 s,5 个内循环),再一起40 个外循环;72 ℃再延伸3 min。
1.3.9 树脂纯化 将反应产物加16 μL水进行稀释,稀释后使用树脂进行脱盐。
1.3.10 芯片点样 沉淀树脂后使用MassARRAY Nanodispenser RS1000 点样仪转移至384 孔SpectroCHIP(Sequenom)芯片上,自然结晶。
1.3.11 质谱检测 上机进行MALDI-TOF 质谱检测,并收集数据,采用TYPER4.0 软件(Sequenom)进行基因型判读。
1.4 关联分析
关联分析中, 质控主要是评价样本及 SNP 位点性能的操作, 通过质控剔除不符合条件的样本或 SNP位点可有效降低关联假阳性的概率。样本的质控主要包括: 样本群体结构、杂合度等; SNP 位点的质控主要包括: call rate(检出率)、哈迪温伯格(H-W)平衡检验、最小等位基因频率等。
采用 plink 软件进行统计分析, 用线性回归模型, 对所有合格样品SNP与对应的工作犬考核评分,进行嗅探能力关联分析: 分析嗅觉灵敏个体(GS 高分)和嗅觉迟钝个体(GS 低分)在每个SNP 位点上基因型和等位基因的差异,寻找与工作犬嗅探能力显著相关的SNP 位点。
多重检验校正一般采用bonferroni (BONF)校正法,校正的方法是P*位点数。比如我们一共做了20 个位点,校正后的BONF 值为P*20,结果仍然<0.05,这是可以判定为显著,<0.01 则为极显著。BONF 校正方法检验过于严格,导致找不到显著差异的基因位点(假阴性)。而FDR_BH 法,是用一种简单好用且比较温和的方法校正P值,可以在假阳性和假阴性间达到平衡,将假/真阳性比例控制到一定范围之内。FDR(假阳性率)错误控制法基本原理是通过控制FDR值来决定P值的值域。那么怎么从P 值来估算FDR 呢,人们设计了几种不同的估算模型,其中使用最多的是Benjamini and Hochberg 方法,简称BH 法。
因此,本研究对关联分析结果的P 值,采取了BONF 法和BH 法进行多重检验校正。
2 结果
2.1 工作犬嗅探能力考核评分
嗅探能力综合考核评分结果见表3。
2.2 SNP 位点检测分型
2.2.1 SNP 位点检出率 68 个SNP 位点检出率,数据结果见表4。

表3 工作犬嗅探能力考评得分情况(部分)

表4 SNP 位点检出率(部分)
2.2.2 SNP位点基因分型结果 受试的113只工作犬, 在各个OR 基因的SNP位点上,均能有效分型。
2.3 关联分析结果
2.3.1 最小等位基因频率及H-W平衡检验 对所研究的群体进行卡方检验,P>0.05,说明接受零假设, 即群体满足H-W 平衡, 反之, 如果P<0.05, 说明群体不满足H-W 平衡。68 个SNP 位点经质控后合格的有52 个(P>0.05),说明采样的犬群满足H-W平衡,可以进行关联性分析研究(表5)。
2.3.2 关联分析结果 有23 个SNP 位点与工作犬的嗅探能力有统计学意义上的显著相关(P<0.05),其余29 个SNP 位点的基因型与工作犬嗅探能力显著不相关(P>0.05),工作犬嗅探能力关联分析情况详见表6。
经过多重检验BONF法校正后, COR52AC1_436、rs850523383、CfOR0149_364 和CfOR0055_265 这4个SNP位点与嗅探能力显著相关(P<0.05); 经过多重检验FDR_BH 法校正后,COR52AC1_436、rs850523383、CfOR0149_364 和CfOR0055_265 这4 个SNP 位点与嗅探能力极显著相关(P<0.01)。
3 讨论
3.1 检测方法的建立
本实验研究通过应用设计软件Assay design3.1,设计PCR 反应和单碱基扩展引物,对引物浓度、碱性磷酸酶处理反应液(SAP Mix)、单碱基延伸反应液(EXTEND Mix)退火温度、模板量等进行优化,成功筛选出68 组特异性好、灵敏度更高的引物, 结果表明, 建立的Sequenom MassARRAY SNP检测OR 基因分型技术方法, 68 个SNP 位点在在史宾格犬、拉布拉多犬和德国牧羊犬3 个犬种中检出率在90%以上,说明该方法可以有效应用于这3个犬种的OR 基因SNP 分型。
3.2 关联分析结果校正
本研究对关联分析结果的P值,采取了BONF法和BH 法进行了多重检验校正,均发现有同样的4 个SNP位点与嗅探能力相关,只是显著程度有所区别,说明COR52AC1_436、rs850523383、CfOR0149_364 和CfOR0055_265 这4 个SNP位点,可以作为建立嗅探能力分子检测方法的首选位点。
3.3 OR 基因与犬嗅探能力的研究
Anna 等[4]曾挑选了5 个OR 基因, 试图探究OR基因与警犬嗅探能力之间的关系, 结果检测到18个SNPs, 其中cOR52N9_176 和cOR9S13_592 这2 个基因显著影响嗅探犬的检测能力。 Yang等[7]曾应用Beckman Genome-Lab SNP stream 技术,选择了OR 基因的22 个SNP 位点,试图探究其与德国牧羊犬嗅探能力之间的关系, 结果显示,OR10H1-like 632C>T、OR10H1-like 770A>T、OR2K2-like 518G>A、 OR4C11-like 511T>G 和OR4C11-like 692G>A 这5 个SNP 位点可能极显著影响德国牧羊犬嗅探能力。

表5 基因频率及H-W 平衡检验情况(部分)

表6 工作犬嗅探能力关联分析情况(部分)
而本研究筛选了OR基因的68个SNP位点,应用Sequenom MassARRAY SNP 技术对史宾格犬、拉布拉多犬和德国牧羊犬这3个品种工作犬OR基因的SNP 位点进行基因分型检测,发现与工作犬的嗅探能力呈一般统计学意义上的显著相关(P<0.05)的SNP位点有23个, 其中经过多重检验校正后,发现COR52AC1_436、rs850523383、CfOR0149_364和CfOR0055_265 这4 个SNP 位点与这3 个品种工作犬嗅探能力显著相关,这4 个SNP位点是国内外首次发现。本研究较Anna 等[4]和Yang 等[7]研究筛选的SNP 数量更多,发现的与工作犬嗅探能力显著相关的SNP 位点适应的品种更为丰富,为工作犬嗅探能力相关基因的辅助育种提供了候选标记。
综上所述, 本研究筛选出与工作犬嗅探气味能力相关联的主效OR 基因SNP 位点4 个, 初步建立了一种能适用于鉴定史宾格犬、拉布拉多犬和德国牧羊犬等3个工作犬种嗅探能力的分子遗传学检测方法。该方法可应用于选种选育, 促进精准育种; 也可以用于选择嗅探能力优秀的合格受训犬, 节省人力物力和训练时间, 提升警犬训练和实战使用的效果。

