基于Cre/loxP系统的无筛选标记转耐低磷转录因子GmPTF1大豆种质创制与分析
张小芳 董秋平 乔 潇 乔亚科 王冰冰 张 锴 李桂兰
基于Cre/系统的无筛选标记转耐低磷转录因子GmPTF1大豆种质创制与分析
张小芳1,**董秋平1,**乔 潇2乔亚科1,*王冰冰1张 锴1李桂兰1,*
1河北科技师范学院农学与生物科技学院, 河北昌黎 066600;2河北科技师范学院物理系, 河北秦皇岛 066004
筛选标记基因在转基因植物应用中存在一定潜在安全风险, 在转基因植物改良中如何合理消除该基因非常必要。转录因子PTF1具有改善植物在低磷胁迫下吸收磷效率的作用。本研究用根癌农杆菌介导法将Cre/-导入大豆品种豫豆22, 利用β-雌二醇诱导Cre/系统删除筛选标记基因, 获得了无筛选标记的转基因大豆。用PCR法扩增删除标记基因后的重组序列并测序显示, 筛选标记基因在大豆基因组中已经被完全删除, 重组序列中目的基因序列正确并保持了正确开放读码框;位点重组出现一种新的拼接类型, 重组后2个序列全部缺失, 新重组拼接位点长38 bp, 与NCBI数据库的其他序列均无同源性, 并且重组涉及2个位点的外侧翼序列, 造成Cre/盒上游和下游外侧翼序列部分缺失。经过RT-PCR和Western杂交验证显示无筛选标记转基因大豆植株中能够正常转录和翻译, 在根系、叶片及茎中的GmPTF1蛋白表达量均高于野生型对照, 而在种子中与对照无显著差异。沙培试验表明, 在低磷条件下无筛选标记转基因大豆苗期根系指标、生物干重、叶绿素含量和磷含量均显著高于野生型对照, 而丙二醛含量低于对照。利用Cre/重组系统可以有效删除转基因大豆中的筛选标记基因。
Cre/; GmPTF1; 无筛选标记; 转基因大豆
自Hinchee等[1]在1988年用农杆菌浸染法获得了转基因大豆植株以来, 转基因技术已作为一种高科技成功应用于大豆新种质的培育[2]。抗草甘膦转基因大豆于1994年首次被美国批准商业化种植, 目前转基因大豆已成为种植面积较大的转基因作物之一[3]。随着转基因植物商业化推广, 转基因植物的生物安全问题一直是人们争议的焦点[4-7], 其中筛选标记基因是转基因作物潜在安全风险主要来源之一。筛选标记基因主要有抗生素抗性基因和除草剂抗性基因。常用的抗生素抗性基因有(潮霉素磷酸转移酶)和基因(新霉素磷酸转移酶), 除草剂抗性基因有抗草丁膦和双丙氨膦的和、抗草甘膦的基因等。转基因植物中筛选标记基因转移至环境中会造成环境安全问题, 例如带有一些抗除草剂基因的植物中其抗性基因可能漂流到其野生近缘种上, 出现超级杂草; 转基因植物中的抗生素抗性基因是否会通过食物在肠道中水平转移至微生物来影响人的健康等[7-8]。这些不可预测的风险引起人们对转基因植物安全问题的广泛关注[8], 也影响了转基因作物的应用及发展。因此作物转基因后删除选择标记基因, 对推动转基因作物的商业化具有重要的价值和意义。
目前位点特异性重组是删除选择标记基因的一种有效途径, 已经广泛应用于不同植物的转基因体系。其中来自噬菌体P1的Cre/系统是在所有位点特异性重组系统中应用最广、可控性较高的转化系统[8]。Cre/位点特异性重组系统中的Cre重组酶专一性识别由34对碱基对组成的特异序列(位点), 使2个位点片段发生重组, 2个位点之间的基因或DNA序列从而被剔除[9]。目前在烟草[10]、番茄[11]等获得筛选标记删除植株。Zuo等[12]构建了一种化学诱导与特异性位点重组相结合的高效DNA切除载体PX6-GFP, 应用β-雌二醇诱导Cre酶表达, 删除了拟南芥中的标记基因。
大豆是一种需要磷较多的作物, 利用现代生物技术与常规育种相结合的手段, 获得磷高效利用品种, 是解决大豆对磷需求的一个有效途径。目前已发现并克隆出多个与磷代谢相关的基因[13]。研究较多的是转录因子类的基因,基因是bHLH转录因子家族的成员之一[14], 在植物逆境胁迫应答中起重要的作用[15]。在水稻中过表达基因其植株磷含量和干物质重高出野生型30%, 大豆中基因转入拟南芥中超表达, 增强了拟南芥的耐低磷能力[16]。本研究旨在通过Cre/系统获得无筛选标记基因的转耐低磷基因大豆, 探讨Cre/系统删除转基因大豆中筛选标记基因的效果, 同时为大豆耐低磷育种创制新种质资源。
1 材料与方法
1.1 材料
植物表达载体为px-(图1-A), 其为c DNA; 转化受体材料为大豆栽培品种“豫豆22”。
1.2 方法
1.2.1 转基因大豆的获得 用农杆菌介导大豆子叶节遗传转化法, 按照李桂兰等[17-18]方法进行。
1.2.2 筛选标记基因删除 转Cre/-的大豆种子, 消毒后接种在含4 μmol L–1β-雌二醇的MS培养基中。培养25~30 d。将大豆苗移栽至营养土中, 提取基因组DNA, 进行检测。
1.2.3 转基因植株的PCR检测 转基因植株筛选标记基因删除前及删除后插入序列示意图见图1, 设计PCR引物检测转化及删除效果, 根据PX6载体序列设计载体骨架引物, P1: 5¢-CCATCTCCACTGA CGTAAGGGAT-3¢, P2: 5¢-CTCGTCAATTCCAAGG GCATCGGT-3¢, P1在启动子内, P2在下游的内, 预计扩增产物长度为653 bp。根据基因序列设计扩增引物, P3: 5¢-AGACAATCGGCT GCTCTGA-3¢, P4: 5¢-TCATTTCGAACCCCAGAGT C-3¢, 预计扩增产物长度为740 bp。根据目的基因序列设计特异性引物对, P5: 5¢-ATGGACC AAGCTCCGGGAGG-3¢, P6: 5¢-CCGTCCCTGATAT CTTGTCG-3¢, 预计扩增产物长度为727 bp。筛选标记基因删除后重组序列使用P1/P6引物对扩增(图1-B), 预计产物长度为901 bp。PCR扩增体系为20 μL, 包含10×buffer (缓冲液) 2 μL、dNTP (2.5 mmol L–1) 1.6 μL、上下游引物(10 μmol L–1)各0.8 μL、酶(5 U μL–1) 0.2 μL、模板DNA 2 μL、灭菌双蒸水12.6 μL。扩增程序为95℃预变性10 min; 95℃变性30 s, 55℃退火1 min, 72℃延伸1 min, 共31个循环; 最后72℃延伸10 min。PCR产物经0.8%琼脂糖凝胶电泳检测并拍照。分别对引物P5/P6、P1/P6所扩增的产物进行切胶回收, 送北京华大基因有限公司测序。

图1 标记基因删除前(A)和删除后(B)转基因植株插入序列及PCR鉴定示意图
1.2.4 目的基因的Southern杂交检测 用CTAB法提取大豆基因组DNA, 取100 μg左右DNA样品, 分别选用I和I酶进行酶切, 野生型材料的总DNA作为阴性对照, 杂交程序按照罗氏Southern杂交试剂盒提供的说明书进行。以转入的基因序列为模板, 应用特异引物P5/P6, PCR法合成Dig标记的探针。
1.2.5 荧光定量PCR 使用Bio-Rad iQ5荧光定量PCR仪, 内参基因为看家基因, 采用2–DDCt法对目的基因表达量进行相对定量分析。设计目的基因与内参基因的实时定量PCR引物(PTF-F: 5'-GTCCCAGGCTGCGACAAG-3', PTF-R: 5'-ACTGCTGCCAGTTTCATTGATAAG-3';n-F: 5'-GAGCTATGAATTGCCTGATGG-3',- R: 5'-CGTTTCATGAATTCCAGTAGC-3')。使用TaKaRa Mini BEST Plant RNA Extraction Kit提取删除筛选标记基因PCR阳性植株叶片总RNA。取部分RNA根据Prime Script RT reagent Kit步骤进行反转录。将各样品cDNA稀释10倍, 取2 μL作模板, 分别用目的基因引物和内参基因引物进行扩增。扩增程序为95℃预变性10 min; 95℃变性15 s, 60℃延伸60 s, 循环40次。荧光定量PCR体系为20 μL, 包含2.5×Real master Mix 8 μL、20×SYBR Green solution 1 μL、上下游引物(10 μmol)各0.4 μL、cDNA模板2 μL、水8.2 μL。利用荧光定量PCR分析系统对样品进行相对表达量分析, 计算出相对表达量, 同时在60~95℃进行溶解曲线分析。
1.2.6 转基因植株的Western blot 提取无筛选标记转基因大豆的根、茎、叶及成熟种子的蛋白, 测定蛋白质浓度, 上样量为50 μg, 进行Western blot分析, 参考王关林等[19]的操作流程。以β-actin蛋白为内参, WB杂交图像用Quantity one图像分析系统进行各条带平均灰度值的分析, 以目的条带的灰度值与内参β-actin灰度值的比值表示GmPTF1蛋白的相对表达量。
转基因材料中GmPTF1蛋白相对表达量= (转基因材料目的条带的灰度值/转基因材料内参β-actin灰度值)/(对照目的条带的灰度值/对照内参β-actin灰度值)
1.2.7 转基因植株的测定项目与方法 对转基因植株进行沙培, 定期浇灌霍格兰氏营养液, 设营养液含低磷(0.05mmol L–1)和正常磷(1 mmol L–1) 2个处理, 3次重复, 每重复10株, 在处理30 d后, 测定各指标。用EPSON的PERFECTION V700型扫描仪测定根系各指标, 测量植株高度及上部第一完全展开三出复叶的中间小叶的长和宽。用丙酮法测定光合色素含量, 硫代巴比妥酸法测定丙二醛(MDA)含量。冲洗干净植株根系上的沙子, 将茎、叶和根系分离, 分别装入纸袋, 于105℃杀青20 min, 60℃烘干至恒重, 经硫酸-过氧化氢消煮, 钒钼黄比色测定样品含磷量。
1.3 数据分析
采用Microsoft Excel进行数据处理, DPSv7.05统计分析软件进行分析。
2 结果与分析
2.1 无筛选标记转GmPTF1基因大豆的获得
应用农杆菌介导法转化大豆品种“豫豆22”子叶节, 获得目的基因PCR检测为阳性的植株(图2)。选择纯合株系后代进行雌二醇处理诱导筛选标记基因删除, 筛选标记基因删除后P1/P2和P3/P4则不能扩增出产物(图1)。PCR扩增检测删除处理后的转基因大豆植株显示, P1/P2和P3/P4分别表现阴性, 而P5/P6和P1/P6表现阳性, 获得符合预期大小的片段(图3)。回收P1/P6扩增段, 以此为模板利用引物P5/P6进行PCR扩增并测序验证, 测序结果显示, 扩增长度为727 bp完全符合预期大小, 与目的基因序列比对完全相同, 进一步确定P1/P6扩增片段包含转入的基因。说明标记基因已经被删除并且保留了目的基因。

图2 转基因植株删除标记之前的PCR检测图
1: 100 bp DNA ladder marker; 2: 水对照; 3: 野生型对照; 4, 5: P1/P2 PCR产物; 6, 7: P3/P4PCR产物; 8, 9: P5/P6基因PCR产物。
1: 100 bp DNA marker ladder; 2: blank control; 3: wild type control; 4, 5: PCR products of P1/P2; 6, 7:PCR products of P3/P4; 8, 9:gene PCR products of P5/P6.

图3 转基因植株删除标记之后的PCR检测图
1, 12: 100 bp DNA ladder marker; 2: 水对照; 3: 野生型对照; 4, 5: P1/P2载体骨架片段PCR产物; 6, 7: P3/P4PCR产物; 8, 9: P5/P6基因PCR产物; 10, 11: P1/P6 PCR产物, 删除标记后重组DNA片段。
1, 12: 100 bp DNA marker ladder; 2: blank control; 3: wild type control; 4, 5: PCR products of P1/P2; 6, 7:PCR products of P3/P4; 8, 9:gene PCR products of P5/P6; 10, 11: PCR product of P1/P6, recombinant DNA fragment after deletion of markers.
2.2 筛选标记基因删除后的重组序列分析
对重组后的P1/P6扩增产物测序显示(图4), 扩增长度为859 bp, 阴影标出部分为P1和P6引物序列, 黑线框出的为筛选标记基因删除后启动子与基因之间的拼接位点。比对分析表明, P1/P6重组后的序列中包含目的基因序列(P5/P6之间序列)。重组序列中第1~94 bp与所用转化载体中的G10-90启动子序列一致, 而G10-90启动子3¢末端的12 bp缺失, 其位于Cre/系统上游位点的外侧; 第133~854 bp与转入的基因的序列一致, 转化载体下游位点与基因起始密码ATG之间的30 bp的序列缺失。说明Cre/系统的重组可以涉及位点外的侧翼序列。在第95~132 bp之间为拼接位点的重组序列, 长38 bp, 该序列经过比对发现与所用载体中的位点序列及NCBI数据库的其他序列均无同源性, 并且拼接位点序列中出现了一个起始密码子。分析开放阅读框发现, 新出现的起始密码子不改变原有基因的读码框, 从新出现的起始密码子开始, 翻译出的氨基酸序列比目的序列增加8个氨基酸(图4)。
通过3个对比实验,并综合3个评价指标来看,IOCAP算法在整体上较K-Means算法、Spectral Clustering算法和AP算法有一定的优势.该算法在一定程度保证聚类效率的同时,也较好地提高了准确度.因此,综合考虑聚类的时间、精确度及通信开销等因素,IOCAP算法的优势较为明显,也更适合大数据集的高效精准聚类.

图4 标记基因删除后重组序列P1/P6 PCR产物测序结果分析
阴影部分为P1和P6引物序列, 线框内为重组拼接位点。
The shaded parts were sequences of P1 and P6 primer, and therecombination splice sites were in the frame.
2.3 转基因植株目标基因遗传稳定性分析
对上述P1/P6 PCR阳性的植株按单株逐年种植, 对T5代纯合株系进行Southern blot检测, 提取全基因组DNA, 分别用限制性内切酶I和I进行酶切, 用地高辛标记的探针杂交。结果表明(图5), 野生型对照材料没有出现杂交信号, 两种酶切的转基因材料均出现单一的杂交信号条带, 确认目的基因以单拷贝插入大豆基因组并且可以稳定遗传。
2.4 无筛选标记转基因大豆的GmPTF1表达分析
2.4.1基因的转录水平分析 对标记基因删除后的转基因大豆根据基因设计引物进行逆转录, 扩增出了符合预期大小的cDNA片段(图6)。由于转基因植株转入的基因为大豆来源, 野生型对照内具有内源基因, 野生型出现一定的本底信号。
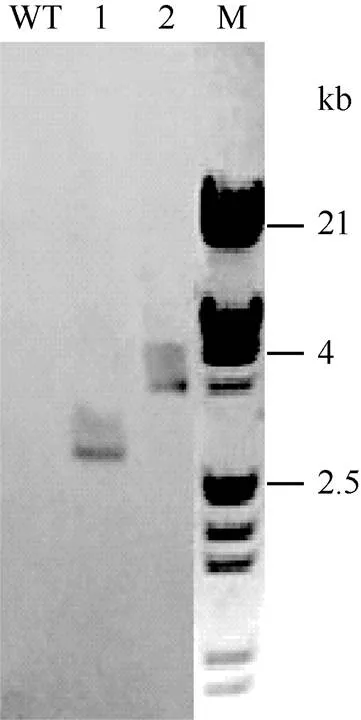
图5 无筛选标记转GmPTF1基因大豆T5代Southern blot检测图
1: 转基因材料I酶切Southern blot; 2: 转基因材料I酶切Southern blot; WT: 野生型对照I酶切Southern blot; M: λDNA/d III marker。
1: Southern blot of transgenic soybean digested byI; 2: Southern blot of transgenic soybean digested byI; WT: Southern blot of wild type controls digested byI; M: λDNA/d III marker.

图6 无筛选标记转基因大豆GmPTF1基因RT-PCR
H: 空白对照; WT: 野生型对照; 1~7: 转基因植株; P: 质粒阳性对照; M: 100 bp ladder DNA marker。
H: blank control; WT: wild type control; 1-7: transgenic soybean; P: plasmid positive control; M: 100 bp DNA marker ladder.
转基因株系植株中的目的基因均能够正常转录表达, 表达量显著高于野生型, 是野生型对照的5.74倍(图7)。
2.4.2 无筛选标记转基因大豆不同器官中GmPTF1蛋白的Western blot分析 对转基因大豆不同器官中目的基因表达GmPTF1蛋白进行Western blot分析(图8), 在转基因株系的根系、叶片和茎中GmPTF1蛋白水平的相对表达量均增加, 分别是野生型对照的3.17、2.16和1.19倍, 并且差异显著; 而成熟种子中的GmPTF1蛋白相对表达量在转基因株系和对照之间差异不显著(图9)。

图7 无筛选标记转基因大豆GmPTF1转录水平分析
1: 野生型; 2: 转基因大豆。
1: wild type controls; 2: transgenic soybean plants.

图8 无筛选标记转基因大豆不同器官中GmPTF1蛋白的Western杂交结果
1: 野生型对照; 2: 转基因材料; A: GmPTF基因表达蛋白; B: Actin基因表达蛋白。
1: wild type controls; 2: transgenic plants; A: GmPTF1 protein; B: Actin protein.
2.5 低磷胁迫下无筛选标记转GmPTF1基因大豆苗期形态表现
结果表明, 正常磷条件下, 转基因株系的根系指标与野生型对照的指标差异不显著; 低磷条件下, 转基因株系的根总长、根总表面积、根平均直径和根体积均极显著高于对照(表1)。说明转基因植株在低磷条件下根系受低磷影响较小。
正常磷条件下, 转基因大豆与野生型对照的单株干物质重差异不显著(表3)。低磷胁迫下, 大豆幼苗干物质重均下降, 转基因株系下降幅度显著低于对照; 并且转基因株的干物重显著高于对照, 根和茎叶干物重分别比对照高60.5%和85.9%。表明基因过表达提高了低磷胁迫下大豆干物质合成与积累。

图9 无筛选标记转基因大豆不同器官中GmPTF1蛋白相对表达量
1: 野生型对照; 2: 转基因大豆; A: 根; B: 叶; C: 茎; D: 种子。
1: wild type controls; 2: transgenic soybean plants; A: root; B: leaf, C: stem; D: seed.
2.6 无筛选标记转GmPTF1大豆苗期对低磷胁迫的生理响应
丙二醛含量上升和光合色素含量下降是植物在低磷条件下的生理生化响应。正常磷处理条件下, 转基因株系与野生型对照的丙二醛和光合色素无显著差异。在低磷条件下, 转基因株系的丙二醛含量及增幅显著低于野生型对照, 而光合色素含量显著高于对照, 降幅低于野生型对照(图10和表4)。表明转基因株系增加了对磷的利用能力, 进而减轻了低磷胁迫的不利影响。
2.7 无筛选标记转GmPTF1大豆磷含量分析
正常磷处理下, 野生型与转基因株系叶片磷含量无显著差异。低磷处理下, 野生型磷含量显著降低, 比正常磷处理下降了20.3%; 而转基因株系叶片磷含量与正常磷处理无显著差异, 与野生型对照相比提高了46.17% (图11)。表明转大豆增强了对磷的利用能力。
3 讨论
Cre/重组系统可以定位定向地删除转基因作物中位于2个位点中的选择标记基因, 在基因工程领域中有巨大的应用前景[20]。Cre重组系统是通过诱导重组酶启动, 使两侧的位点重组, 删除中间的标记基因, 重组后保留1个位点。在重组序列的研究中, Cheng等[21]构建利用Cre/重组系统在伴放线杆菌()中实现了标记基因的删除,重组序列拼接位点只剩下1个loxp位点序列, 没有出现其他序列变化[21]; Sreekala等[22]利用该系统成功获得了无选择标记基因的水稻, 重组序列拼接位点只剩下1个位点序列; Togawa等[23]利用Cre/系统对嗜热菌()进行筛选标记基因敲出, 虽然筛选标记基因完全敲除, 但同时观察到多个位点之间的染色体区域的不理想的缺失和反转[23]。Zhang等[24]利用该系统删除了转基因番茄的标记基因, 重组序列拼接位点同样只剩下1个位点序列; Li等[25]利用胚胎特异启动子构建Cre/自删除系统, 转化后发育到胚状体形成阶段启动Cre删除转基因大豆标记基因后, 重组序列中拼接位点长83 bp, 包括1个34 bp的位点以及其上游的22 bp下游27 bp的载体序列。在本研究中, 删除标记基因后重组序列在启动子与目的基因之间出现38 bp拼接位点序列, 该拼接位点序列与载体、和大豆基因组均无同源性, 为1个新位点重组的拼接类型; 并且重组涉及位点外的侧翼序列, 位于Cre/系统中2个位点外侧的部分载体序列均缺失, 这与上述报道不同, 说明Cre/系统重组后拼接位点序列的多样性; 新的拼接序列中出现了一个起始密码子, 但未改变原有目的基因的读码框; Real-time PCR和Western Blot结果显示, 转基因植株由于的拷贝数增加致使相应的表达量提高, 说明重组后G10-90启动子3¢末端的12 bp缺失没有影响启动子的功能, 同时验证了Cre/重组系统定位定向删除的可靠性。鉴于重组可能涉及位点外的侧翼序列, 构建载体时在目的基因及调控元件与连接处应增加相应保护序列。

表1 不同磷浓度下不同大豆植株的根系性状

表2 低磷胁迫对不同大豆幼苗地上部的影响

表3 低磷对不同转基因大豆干物质积累的影响

图10 大豆叶片中光合色素与丙二醛含量

表4 大豆叶片丙二醛与光合色素变幅

图11 不同磷浓度处理下大豆叶片磷含量
1: 野生型对照; 2: 转基因大豆。
1: wild-type control; 2: transgenic line.
有研究表明低磷胁迫条件下, 根总长、根总表面积、根宽、根干重、地上干重、地上磷含量可以作为综合评价大豆苗期磷效率筛选的重要指标[26-27]。本研究低磷胁迫下无筛选标记转基因大豆苗期的根总长、根总表面积、根宽、根总体积、根干重及地上干重均极显著高于野生型对照豫豆22植株, 证明转入的基因在植株体内成功表达, 提高了磷的利用效率, 从而促进了转基因大豆植株的根系的生长发育, 使植株生长更加健壮。张彦丽等[26]研究认为大豆苗期相对株高受低磷胁迫的影响较小, 变异系数较小, 不适合作为大豆苗期磷效率的筛选指标。本研究结果亦得到相似结果, 表现为低磷胁迫下苗期不同材料株高无差异, 苗期株高对低磷不敏感。植物在低磷胁迫后发生一系列的生理生化反应, 从而调节植物的生长发育[28]。光合色素含量的下降是植物受到胁迫所表现的关键生理特征, 在逆境条件下, 植物体内叶绿素受到破坏, 光合作用受到抑制, 最终导致植物不能正常生长。丙二醛是膜脂过氧化分解的终产物, 在逆境胁迫下, 超氧自由基不断累积并提高了植物组织脂膜氧化的程度, 使其最终产物丙二醛含量增加[29-30]。本研究无筛选标记转基因大豆材料在低磷条件下叶片光合色素含量显著高于野生型对照, 而MDA含量低于野生型对照。说明在低磷条件下基因的过量表达调控相关磷代谢基因表达, 减少了低磷环境给大豆植株带来的伤害。本研究中基于Cre/系统删除选择标记的转大豆在苗期低磷条件下的磷利用率提高, 根系、生物干重、植株含磷量显著高于野生型对照, 与钟磊等[31]获得的带筛选标记转大豆表现一致, 证明Cre/系统删除选择标记的可靠性及有效性, 并再次证明转录因子在大豆磷代谢中的作用。
4 结论
利用Cre/系统有效删除筛选标记基因, 获得无筛选标记的转大豆。位点重组出现一种新的拼接类型, 38 bp的拼接位点重组序列与其他序列均无同源性, 重组后2个序列全部缺失;位点重组涉及位点外的侧翼序列, Cre/重组系统之外的侧翼序列部分缺失, 构建载体时在目的基因及启动子调控元件与之间应增加保护序列。无筛选标记的转大豆植株提高了基因在根系及叶片中的表达, 提高大豆苗期低磷条件下的磷利用率, 根系、生物干重、植株含磷量显著高于野生型对照。
[1] Hinchee M A W, Connorward D V, Newell C A, McDonnell R E, Sato S J, Gasser C S, Fischhoff D A, Re D B, Fraley R T, Horsch R B. Production of transgenic soybean plants usingmediated DNA transfer., 1988, 6: 915–922.
[2] 杨乐坤. 中国转基因农产品的标识规制策略. 南京大学硕士学位论文, 江苏南京, 2016. Yang L K. China’s Strategic Labeling of GM Food Based on Two-stage Dynamic Game Model. MS Thesis of Nanjing University, Nanjing, Jiangsu, China, 2016 (in Chinese with English abstract).
[3] 崔宁波, 宋秀娟. 国外转基因大豆种植与育种研究进展国外转基因大豆种植与育种研究进展. 东北农业大学学报, 2015, 46(8): 103–108. Cui N B, Song X J. Research progress on development and planting of foreign genetically., 2015, 46(8): 103–108 (in Chinese with English abstract).
[4] 陈晓亚, 杨长青, 贾鹤鹏, 凌飞. 中国转基因作物面临的问题. 华中农业大学学报, 2014, 33(6): 115–117. Chen X Y, Yang C Q, Jia H P, Ling F. Issues confronting GMO crops in China., 2014, 33(6): 115–117 (in Chinese with English abstract).
[5] 石红璆, 查代明. 转基因食品的道德风险及其解决对策. 安徽农业科学, 2018, 46(8): 7–9.Shi H Q, Zha D M. Moral risks of genetically modified foods and their countermeasures., 2018, 46(8): 7–9 (in Chinese with English abstract).
[6] 黄大昉. 我国转基因作物育种发展回顾与思考. 生物工程学报, 2015, 31: 892–900. Huang D F. Review of transgenic crop breeding in China., 2015, 31: 892–900 (in Chinese with English abstract).
[7] 逄金辉, 马彩云, 封勇丽, 胡瑞法. 转基因作物生物安全:科学证据. 中国生物工程杂志, 2016, 36(1): 122–138. Peng J H, Ma C Y, Feng Y L, Hu R F. Biosafety of genetically modified crops: scientific evidence., 2016, 36(1): 122–138 (in Chinese with English abstract).
[8] 祁永斌, 刘庆龙, 陆艳婷, 金庆生. 转基因植物中删除选择标记基因的研究进展. 浙江农业学报, 2014, 26: 1387–1393.Qi Y B, Liu Q L, Lu Y T, Jin Q S. Research progress of the selectable marker genes eliminated in the transgenic plants., 2014, 26: 1387–1393 (in Chinese with English abstract).
[9] Guo F, Gopaul D N, Van Duyne G D. Structure of Cer recombinase complexes with DNA in a site-specific recombination synapse., 1997, 389: 40–46.
[10] Dale E C, Ow D W. Intra- and intermolecular site-specific recombination in plant cells mediated by bacteriophage P1 recombinase., 1990, 91: 79–85.
[11] Ma B G, Duan X Y, Niu J X, Ma C, Hao Q N, Zhang L X, Zhang H P. Expression of stilbene synthase gene in transgenic tomato using salicylic acid-inducible Cre/recombination system with self-excision of selectable marker., 2009, 31: 163–169.
[12] Zuo J, Niu Q, Moller S, Chua N H. Chemical-regulated, site-specific DNA excision in transgenic plants., 2001, 19: 157–161
[13] 李喜焕, 常文锁, 张彩英. 提高植物磷营养效率(候选)基因研究进展. 植物遗传资源学报, 2012, 13: 83–97. Li X H, Chang W S, Zhang C Y. Research progress of candidate genes for improving plant phosphorus-effiency., 2012, 13: 83–97 (in Chinese with English abstract).
[14] 张全琪, 朱家红, 倪燕妹, 张治礼. 植物bHLH转录因子的结构特点及其生物学功能. 热带亚热带植物学报, 2011, 19: 84–90. Zhang Q Q, Zhu J H, Ni Y M, Zhang Z L. The structure and function of plant bHLH transcription factors., 2011, 19: 84–90 (in Chinese with English abstract).
[15] 杨致荣, 毛雪, 李润植. 植物次生代谢基因工程研究进展. 植物生理与分子生物学学报, 2005, 31: 11–18. Yang Z R, Mao X, Li R Z. Progress in studies on genetic engineering of secondary metabolites in plant., 2005, 31: 11–18 (in Chinese with English abstract).
[16] 吴冰. 大豆耐低磷转录因子与功能分析. 河北农业大学硕士学位论文, 河北保定, 2013. Wu B. Functional Analysis of Transcription FactorandResponsed to Low Phosphorus in Soybean. MS Thesis of Agricultural University of Hebei, Baoding, Hebei, China, 2013 (in Chinese with English abstract).
[17] 李桂兰, 刘晨光, 乔潇, 杨晓倩, 王迪, 乔亚科.共培养条件对农杆菌转化大豆子叶节的影响.核农学报, 2014, 28: 1567–1575. Li G L, Liu C G, Qiao X, Yang X Q, Wang D, Qiao Y K. Conditions of co-culture affecting on the efficiency ofmediated transformation of cotyledonary node of soybean., 2014, 28: 1567–1575 (in Chinese with English abstract).
[18] 李桂兰, 乔亚科, 杨少辉, 靳朝霞, 李明刚.农杆菌介导大豆子叶节遗传转化的研究. 作物学报, 2005, 31: 170–176. Li G L, Qiao Y K, Yang S H, Jin Z X, Li M G. Study of the-mediated transformation systems of soybean cotyledonary node., 2005, 31: 170–176 (in Chinese with English abstract).
[19] 王关林, 方宏筠. 植物基因工程原理与技术. 北京: 科学出版社, 1998. pp 634–636.Wang G L, Fang H J. Plant Gene Engineering Principle and Technique. Beijing: Science Press, 1998. pp 634–636 (in Chinese).
[20] 龙定沛, 谭兵, 赵爱春, 许龙霞, 向仲怀. Cre位点特异性重组系统在高等真核生物中的研究进展. 遗传, 2012, 34: 177–189. Long D P, Tan B, Zhao A C, Xu L X, Xiang Z H. Progress in Cre/site-specific recombination system in higher eu-karyotes., 2012, 34: 177–189 (in Chinese with English abstract).
[21] Cheng Y A, Jee J, Hsu G, Huang Y Y, Chen C, Lin C P. A markerless protocol for genetic analysis of., 2014, 113: 114–123.
[22] Sreekala C, Wu L, Gu K, Wang D, Tian D, Yin Z. Excision of a selectable marker in transgenic rice (L.) using a chemically regulated Cre/system., 2005, 24: 86–94.
[23] Togawa Y, Nunoshiba T, Hiratsu K. Cre/-based multiple markerless gene disruption in the genome of the extreme thermophile., 2018, 293: 277–291.
[24] Zhang Y, Li H, Ouyang B, Ye Z. Chemical-induced autoexcision of selectable markers in elite tomato plants transformed with a gene conferring resistance to lepidopteran insects., 2006, 28: 1247–1253.
[25] Li Z, Xing A, Moon B P, Burgoyne S A, Guida A D, Liang H, Lee C, Caster C S, Barton J E, Klein T M, Falco S C. A Cre/-mediated self-activating gene excision system to produce marker gene free transgenic soybean plants., 2007, 65: 329–341.
[26] 张彦丽, 贾健辉, 赵月琪, 谷思玉, 许景钢. 大豆苗期耐低磷筛选指标的研究. 安徽农业科学, 2010, 38: 5506–5507. Zhang Y L, Jia J H, Zhao Y Q, Gu S Y, Xu J G. Screening index of soybean for low phosphorus tolerance at seedling stage., 2010, 38: 5506–5507 (in Chinese with English abstract).
[27] 梁泉, 尹元萍, 严小龙, 廖红. 不同磷水平下大豆根系性状的遗传特性分析. 分子植物育种, 2009, 7: 321–329. Liang Q, Yin Y P, Yan X L, Liao H. Genetic analysis of root characters in soybean using a recombinant inbred line population at two phosphorus levels., 2009, 7: 321–329 (in Chinese with English abstract).
[28] 敖雪, 谢甫绨, 刘婧琦, 张惠君. 不同磷效率大豆品种光合特性的比较. 作物学报, 2009, 35: 522–529. Ao X, Xie F T, Liu J Q, Zhang H J. Comparison of photosynthetic characteristics in soybean cultivars with different phosphorus efficiencies., 2009, 35: 522–529 (in Chinese with English abstract).
[29] 李俊, 张春雷, 秦岭, 马霓, 李峰. 不同磷效率基因型油菜对低磷胁迫的生理响应. 中国油料作物学报, 2010, 32: 222–228. Li J, Zhang C L, Qin L, Ma N, Li F. Physiological response to low phosphorus stress for different P-efficiency genotype of rapeseed., 2010, 32: 222–228 (in Chinese with English abstract).
[30] 王晶, 韩晓日, 站秀梅, 侯玉慧, 赵伟力. 低磷胁迫对番茄叶片脂膜过氧化及保护酶活性的影响. 植物营养与肥料学报, 2005, 11: 851–854. Wang J, Han X R, Zhan X M, Hou Y H, Zhao W L. Influence of low-phosphorus stress on membrane lipid peroxidation and protective enzyme activities in tomato leaves., 2005, 11: 851–854 (in Chinese with English abstract).
[31] 钟磊, 乔亚科, 乔潇, 李桂兰, 王林红, 刘晨光. 转基因大豆在低磷胁迫下的表现. 核农学报, 2013, 27: 1041–1047. Zhong L, Qiao Y K, Qiao X, Li G L, Wang L H, Liu C G. Performance of transgenic soybean withgene under low phosphorus stress., 2013, 27: 1041–1047 (in Chinese with English abstract).
Creation and analysis of marker free transgenic soybean germplasm with low phosphate tolerance transcription factor GmPTF1based on Cre/system
ZHANG Xiao-Fang1,**, DONG Qiu-Ping1,**, QIAO Xiao2, QIAO Ya-Ke1,*,WANG Bing-Bing1, ZHANG Kai1, and LI Gui-Lan1,*
1College of Agronomy and Biotechnology, Hebei Normal University of Science and Technology, Changli 066600, Hebei, China;2Department of Physics, Hebei Normal University of Science and Technology, Qinhuangdao 066004, Hebei, China
The screening marker genes in transgenic crops have potential safety risks, which are necessarily eliminated in the improvement of transgenic plants. The transcription factorPTF1 has the effect improving phosphorus uptake by plants under low phosphorus stress. So Cre/-GmPTF1 was transferred into soybean cultivar Yudou 22 [(L.) Merr] via-mediated cotyledonary node transformation method. And then, the marker-free transgenic soybean withwas obtained by means of induced Cre/site specific recombination with β-estradiol. After the marker gene was deleted, the recombinant sequences were amplified by PCR and then sequenced. The sequencing results indicate that the marker genes were completely deleted out of the transgenic soybean genome. The recombinant maintained with correct ORF correct target gene sequence, and a new splicing type appeared inrecombination. Twosequences were missing in the recombination. The length of the new recombinant in splicing site was 38 bp which was no homology with other sequences in NCBI database. And the recombination was involved in the flank sequence outside the twosites, which lead partially deleted in the outer flank sequences of Cre/cassette. The results of RT-PCR and Western blot showed thatcould be transcribed and translated normally in marker-free transgenic soybean plants.The expression level ofGmPTF1 in roots, stem and leaves was higher than that in wild type, which was not significantly different in seeds. in the sand culture experiment under low phosphorus condition, the root indexes, dry biomass, chlorophyll content and phosphorus content in marker-free transgenic soybean were significantly higher than there in the wild type, while the MDA content was lower than that in the control. We conclude that screening marker genes in transgenic soybean could be effectively deleted by Cre/loxP recombinant system.
Cre/; GmPTF1; marker-free; transgenic soybean
2018-08-27;
2018-12-24;
2019-01-16.
10.3724/SP.J.1006.2019.84118
李桂兰, E-mail: lgl63@126.com; 乔亚科, E-mail: qiaoyake@126.com
**同等贡献(Contributed to equally this work)
张小芳, E-mail: zxf13214@163.com; 董秋平, E-mail: qiuping.dong@kejidalong.com
本研究由国家转基因生物新品种培育科技重大专项(2014ZX0800404B)和现代农业产业技术体系河北省创新团队建设项目(HBCT2018090203)资助。
This study was supported by the National Transgenic Major Project of China (2014ZX0800404B) and Modern Agricultural Industrial Technology System Hebei Province Innovation Team Construction Project (HBCT2018090203).
URL:http://kns.cnki.net/kcms/detail/11.1809.S.20190115.1603.004.html

