基于StarBase数据库分析lncRNA SNHG6在消化系统肿瘤中的表达及作用机制
林思敏,蓝智贤,姚翔,王馨珂
消化系统肿瘤(Digestive system neoplasms, DSNs)是全世界癌症相关死亡的主要原因。2018年因消化系统肿瘤死亡人数达到3,056,412例死亡,占总死亡人数的32%[1]。其中包括结直肠癌、胃癌、肝癌、食管癌和胰腺癌,均为常见的肿瘤。虽然近年来消化系统肿瘤诊断和治疗的方法有所改善,但是预后仍然很差。因此,寻找有效的诊断、治疗及预后预测靶点仍是目前的研究重点。
非编码RNA(non-coding RNA, ncRNA)约占98%的基因转录产物,包括微小RNA(micro RNA, miRNA)和长链非编码RNA(long non-coding RNA, lncRNA),其中约有93%的转录本为lncRNA[2]。LncRNA是一类转录本长度超过200 nt、不编码蛋白的RNA,通常位于细胞核和细胞质中。lncRNA与小分子RNA相比,序列更长、空间结构也较为复杂,参与表达调控的机制也更具有多样性和复杂性。尽管目前只有一小部分lncRNA的功能有相关报道,但可以明确的是lncRNA可以通过多种途径调控肿瘤的发生、发展过程[3]。
LncRNA SNHG6(small nucleolar RNA host gene 6, U87HG)属于5’基因家族,编码两个非编码RNA:由内含子编码的SNORD87[4]和外显子编码的SNHG6 RNA。人类SNHG6 RNA最长的ORF起始于第二个AUG密码子(第136位点),包括一个很弱的翻译码(GGUGUAUGA),包括其他起始密码子为GUG,UUG,CUG和ACG的ORF,均不编码任何可以生成蛋白质的短肽。因此SNHG6 RNA不编码任何蛋白质,是一个长链非编码RNA[5]。研究发现SNHG6在多种消化系统肿瘤中表达上调,促进肿瘤增殖、侵袭和转移[6-14]。本文旨在通过StarBase数据库分析lncRNA SNHG6在消化系统肿瘤中的表达及可能的下游调控机制,为消化系统肿瘤的治疗及预后分析提供新的理论依据。
1 材料与方法
1.1 SNHG6在消化系统肿瘤中的表达
从StarBase数据库(http://starbase.sysu.edu.cn)中的Pan-Cancer模块分析中选择“Gene Differential Expression”,以“SNHG6”为关键词进行检索,得到SNHG6在食管癌、结肠癌、胆管癌、肝癌、胰腺癌、胃癌中的表达数据。
1.2 SNHG6相关机制分子筛选
从StarBase数据库RNA-RNA模块分析中选择“lncRNA-RNA”,在Query Gene中以“SNHG6”关键词进行检索,得到SNHG6可能直接结合的下游RNA数据;同时在RBP-Target模块分析中选择“RBP-lncRNA”,在Target Gene中以“SNHG6”为关键词进行检索,得到SNHG6可能结合的上游基因。
1.3 SNHG6相关靶基因生物学功能及通路分析
将两种方法得到的相关靶基因去重,分别删除无对应名称的基因片段后,得到274个SNHG6相关靶基因。利用生物学信息注释数据库DAVID(https://david.ncifcrf.gov)对所有靶基因进行功能富集分析(GO, gene ontology)和通路富集分析(KEGG,KEGG pathway analysis),以此找出最显著的可能作用机制。
1.4 SNHG6及其下游靶基因EIF4A3的相关性分析
通过功能富集分析发现SNHG6可能与EIF4A3基因相互作用。利用StarBase数据库Pan-Cancer模块分析中选择“RNA-RNA CoExpression”分析SNHG6和EIF4A3基因在不同消化系统肿瘤中的相互关系。
1.5 EIF4A3基因在消化系统肿瘤中的表达及预后分析
通过StarBase数据库分析EIF4A3在不同消化系统肿瘤中的表达,同时利用Pan-Cancer分析模块中“Gene Survival Analysis”分析EIF4A3在不同消化系统肿瘤中的预后。
1.6 差异基因计算方法
StarBase数据Pan-Cancer模块中,通过TCGA数据库(https://portal.gdc.cancer.gov/)获得各种肿瘤的表达数据,每个基因的表达值由RNA-seq数据通过log2(FPKM+0.01)获得。
2 结果
2.1 SNHG6在结肠癌中高表达
StarBase数据库中“Gene Differential Expression”分析表明6在除了胰腺癌以外的消化系统肿瘤中均为高表达(图1)。
2.2 靶基因功能富集分析
通过“lncRNA-RNA”及“RBP-lncRNA”两种分析,我们得到和SNHG6相关靶基因共274个,GO TERM功能富集分析发现其中结合蛋白(Binding Protein)主要集中在mRNA的相关过程,包括剪切、转运或转录的调控(图2);KEGG信号通路富集分析发现主要和剪切体、RNA转运、核糖体以及mRNA监测相关通路有关(图3)。说明SNHG6主要调控转录及转录前相关机制。同时我们发现EIF4A3基因在其多种KEGG信号通路中均有参与,因此我们对EIF4A3基因进行进一步的研究。
2.3 SNHG6与EIF4A3基因在不同消化系统肿瘤中的相关性
通过“RNA-RNA CoExpression”分析,我们发现在肝癌中SNHG6与EIF4A3明显正相关(图4,n=374,r=0.257,P<0.001),而在其他消化系统肿瘤中未见明显相关性(P>0.05)。因此我们推测在肝癌中SNHG6通过与EIF4A3直接作用,调控相关下游通路。
2.4 EIF4A3基因在肝癌中高表达并且预后较差
通过“Gene Differential Expression”分析发现EIF4A3基因在肝癌中高表达(图5),并且提示预后较差(图6,P=0.0054)。
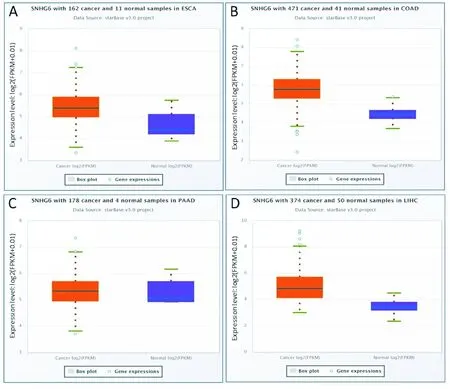
A:SNHG6在食管癌中高表达;B:SNHG6在结肠癌中高表达;C:SNHG6在胰腺癌中无明显差异;D:SNHG6在肝癌中高表达

图2 SNHG6靶基因功能富集分析

图3 SNHG6靶基因KEGG通路富集分析
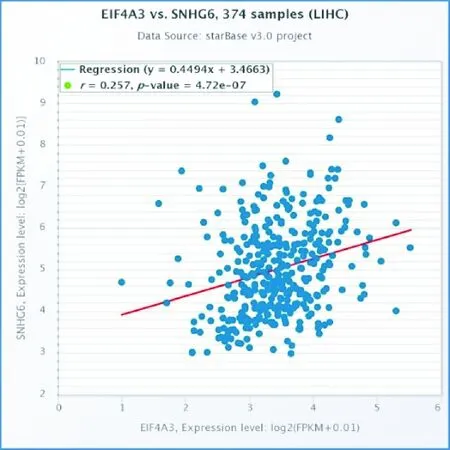
图4 肝癌中SNHG6与EIF4A3基因正相关
3 讨论
消化系统肿瘤是一组最常见的恶性肿瘤,指在消化系统多个部位发展的肿瘤,包括胃、结直肠、肝脏及胰腺。近年来虽然分子生物学的进步已经很大程度上提高了消化系统肿瘤的诊断、治疗以及预防,但是因为它们的恶性程度高、转移速度快,而导致较高的死亡率。因此,研发新的分子靶标尤其是预后相关分子,对制定更有效的治疗策略至关重要[15]。

图5 EIF4A3基因在肝癌中高表达
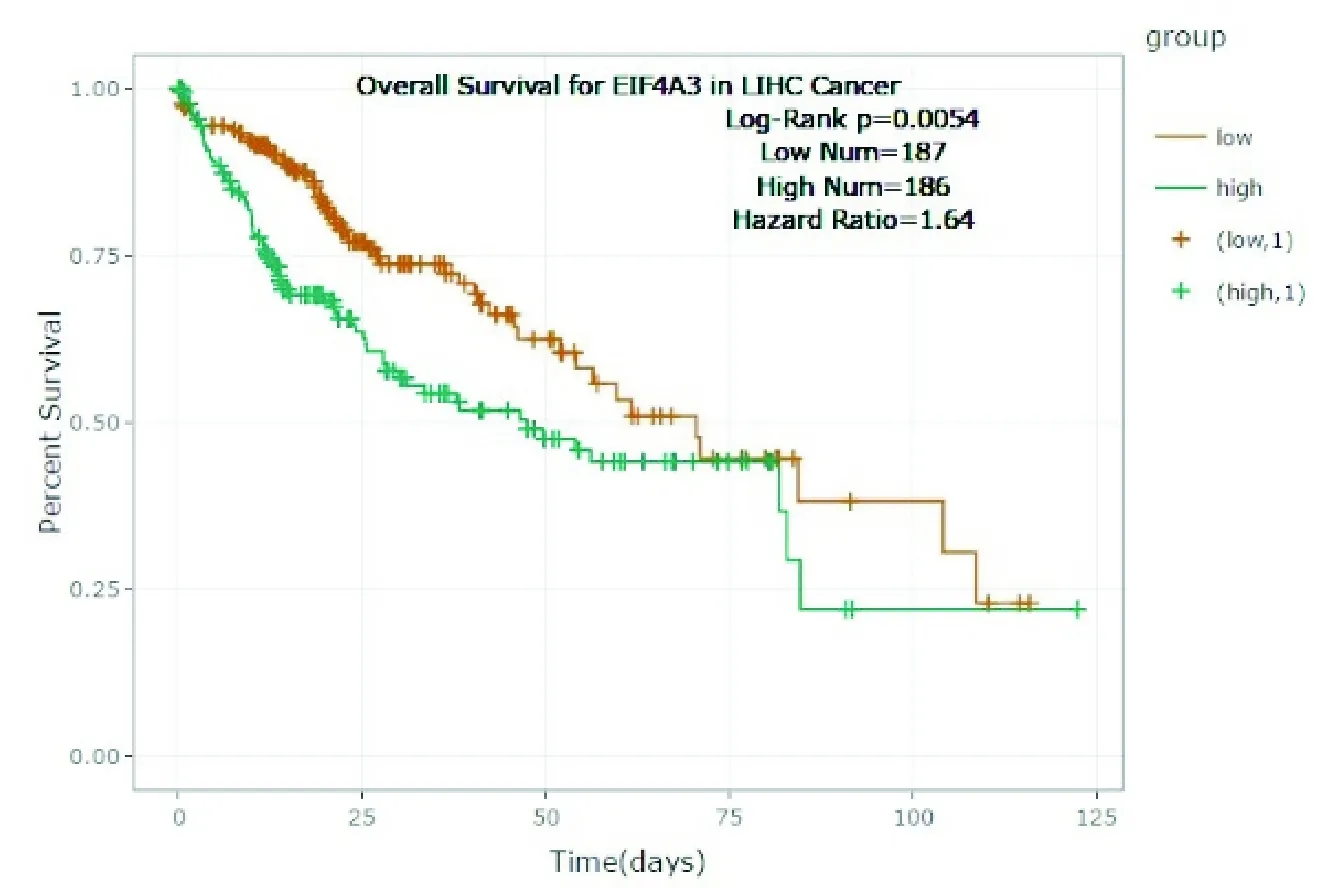
图6 EIF4A3基因在肝癌中提示较差预后(P=0.0054)
人类RNA可分为编码RNA及无编码蛋白能力的非编码RNA,前者在人体庞大的RNA体系中仅占约1%,这意味着非编码RNA占据主要部分[2]。非编码RNA包括管家基因(housekeeping noncoding RNAs)及调控基因(regulatory noncoding RNAs)两大类。管家基因有核糖体RNA(rRNAs)、转录RNA(tRNAs)、小核RNA(small nuclear RNAs)、核仁小RNA(small nucleolar RNAs, snoRNAs);调控基因则分为miRNA、小干扰RNA(siRNAs)、Piwi相关RNA(piRNAs)[16-17]。
其中长链非编码RNA是指RNA大小超过200个核苷酸且无蛋白编码能力的RNA,曾一度被认为是基因组转绿的“噪音”,并不具有生物学功能。随着对长链非编码RNA的深入研究,人们发现长链非编码RNA可以通过与其他RNA、蛋白质、染色体等相互作用参与转录调控、转录后调控、表观遗传、剪接等调控肿瘤细胞增殖分化、迁移侵袭、EMT过程等。
小核仁RNA(snoRNA)是一类小的非编码RNA分子,它们集中在核仁中并具有稳定的代谢。作为真核细胞核仁中的小的非编码RNA,snoRNA的长度为60~300个核苷酸(nt)。它们的主要功能是参与细胞质中rRNA和其他RNA的转录后修饰,例如假尿苷酸化和2’-O-甲基化。通过snoRNA加工后与核仁中的核糖体蛋白结合,然后通过进一步复杂的成熟和转运过程离开细胞核,最终在细胞质中形成成熟的功能性核糖体。核糖体作为蛋白质合成的位点,几乎控制细胞中的所有蛋白质合成。因此很明显,snoRNA对细胞生长甚至其他重要活动极为重要。
大多数snoRNA由宿主基因编码并从中加工前mRNA的内含子。宿主基因内含子snoRNA属于编码50个末端的一类基因寡嘧啶(50TOP)mRNA。50TOP mRNAs很大程度上编码参与翻译的技术,如核糖体蛋白和反式累积数据[14]。研究表明50TOP基因,包括snoRNA宿主基因,在控制细胞存活和发挥作用中发挥作用细胞凋亡和编码必需的蛋白质或转录本核糖体生物发生或功能。活动过度的核糖体生物发生在癌症中被广泛观察到;这种观察已部分发生归因于Pol I增加的rDNA转录。除了snoRNA参与癌症发展外,有研究表明snoRNA宿主基因(SNHG)也可能导致肿瘤发生。
已知的SNHGs有20余种[18-22],如: SNHG1在可以通过内源性竞争相应microRNA在食管癌、肝癌及鼻咽癌中促进肿瘤进展[19, 23-24];SNHG5在胃癌组织中被发现可与MTA2相互作用抑制MTA2从胞质到胞核的过程,使得MTA2留滞于细胞质中,从而干预核苷酸重构及组蛋白H3去乙酰化,促进肿瘤细胞迁徙转移;同时SNHG5过表达的肿瘤细胞中与侵袭相关的蛋白如P21、和E-钙粘蛋白显著升高,而MMP9、MMP1、EGRF表达显著下降[18, 25];高表达的SNHG15在胃癌细胞中也可作为肿瘤细胞增殖与侵袭的分子标志之一[26-27]。
已有研究发现SNHG6在多种消化系统肿瘤中高表达,发挥了促癌基因的作用[6-14,28-29]。为了探讨SNHG6在消化系统肿瘤中的可能作用机制,我们通过StarBase数据库对SNHG6在消化系统肿瘤中的表达进行分析,发现SNHG6在除了胰腺癌以外的消化系统肿瘤中均高表达。同时对其下游作用机制做了初步的探讨,发现SNHG6可能的下游通路主要集中于mRNA的相关过程,包括剪切、转运或转录的调控。其中EIF4A3与其有密切关系。因此我们推测SNHG6可能通过与EIF4A3直接作用,调控相关下游通路。
EIF4A3编码DEAD盒蛋白家族的成员。以保守基序Asp-Glu-Ala-Asp(DEAD)为特征的DEAD盒蛋白是推定的RNA解旋酶。它们涉及许多涉及RNA二级结构改变的细胞过程,例如翻译起始,核和线粒体剪接,以及核糖体和剪接体组装。基于它们的分布模式,该家族的一些成员被认为参与胚胎发生,精子发生和细胞生长和分裂。由该基因编码的蛋白质是核基质蛋白质。其氨基酸序列与翻译起始因子DEAD盒蛋白家族的另外两个成员eIF4AI和eIF4AII的氨基酸序列高度相似[30]。
通过StarBase数据库,我们发现EIF4A3在肝癌中高表达,并且与SNHG6表达呈正相关性,同时提示了较差预后。因此我们推测在肝癌中,SNHG6可能通过与EIF4A3结合,调控肝癌发生发展。这可能为肝癌的诊断及预后评估,提供一定的理论依据。
综上所述,长链非编码RNA SNHG6在多种消化系统肿瘤中高表达,发挥促癌基因的作用,可能通过与EIF4A3基因相互作用,促进肝癌发生发展,提示不良预后。

