恩施市3种泡辣椒样品中细菌多样性研究
尚雪娇,折米娜,邓长阳,廖 华,赵 楠,郭 壮,4*
(1.湖北文理学院 食品科学技术学院 鄂西北传统发酵食品研究所,湖北 襄阳441053;2.恩施市农业局,湖北 恩施445000;3.四川省农业科学院 农产品加工研究所,四川 成都610066;4.恩施市公共检验检测中心,湖北 恩施445000)
泡辣椒是一种流行于四川、湖南和贵州等地区的传统发酵辣椒产品[1],其主要利用辣椒表面附着的微生物自然发酵而成[2]。由于传统发酵食品制作环境开放,使得各类产品中的微生物具有多样性[3]。钟燕青[4]研究发现,湖南地区自然发酵剁辣椒中的乳酸菌主要为植物乳杆菌(Lactobacillus plantarum)、戊糖片球菌(Pediococcus pentosaceus)和短乳杆菌(Lactobacillus breris),同时沙漠等[5]从自然发酵的辣椒酱中分离出两株产酸量高、生长良好且适用于发酵辣椒试验的菌株,分别为L.plantarum和肠膜串球菌(Streptococcus faecalis)。周俊良[6]在贵州地区市售泡辣椒和农家自制酱辣椒产品中筛选出4株适用于发酵辣椒制品制作的乳酸菌,分别为双发酵乳杆菌(Lactobacillus bifermentans)、发酵乳杆菌(Lactobacillus fermentum)、食品乳杆菌(Lactobacillus alimentarius)和食果糖乳杆菌(Lactobacillus fructivorans),然而关于湖北恩施地区泡辣椒中微生物多样性的研究报道尚少。
相对于传统微生物培养方式,Illumina MiSeq第二代高通量测序技术可以获得不同环境条件下微生物的群落结构,不仅能够检测到低丰度的微生物,而且具有很高的可信度和准确度[7]。同时聚合酶链式反应结合变性梯度凝胶电泳(polymerase chain reaction-denaturing gradient gel electrophoresis,PCR-DGGE)是一种新型且不依赖于培养的分子生物学技术,其可以直接提取微生物的宏基因组,通过电泳图来检测和鉴定自然环境或人工环境中微生物的种类[8]。Illumina MiSeq技术和PCR-DGGE技术改善了传统微生物培养方法中耗时长和工作量大等缺点,同时具有快速和简便且重复性好的特点,被广泛应用于发酵食品[9-10]、肉制品[11-12]、肠道[13-14]和土壤[15-16]微生态研究等领域。
因此,本研究采用MiSeq高通量测序技术与PCR-DGGE技术相结合的方法对恩施地区3种泡辣椒中微生物的多样性进行解析,同时利用传统微生物纯培养方法及分子生物学技术对其中所含的乳酸菌进行分离鉴定。以期全面解析恩施市传统发酵辣椒中细菌的多样性,同时对恩施地区发酵食品中乳酸菌的开发利用奠定基础。
1 材料与方法
1.1 材料与试剂
1.1.1 材料
3种泡辣椒样品(编号分别为LJS01、LJS02和LJS03,泡制时间均在30 d左右):采购于恩施市土桥坝菜市场。
1.1.2 试剂
三羟甲基氨基甲烷、乙酸、乙二胺四乙酸、聚丙烯酰胺、尿素、去离子甲酰胺、N,N-亚甲基二丙烯酰胺(均为分析纯):国药集团化学试剂有限公司;MRS培养基:青岛海博生物技术有限公司;D5625-01脱氧核糖核酸(deoxyribonucleic acid,DNA)提取试剂盒、DNA marker、PCR清洁试剂盒:京科博汇智生物科技发展有限公司;2PCR×mix:南京诺唯赞生物科技有限公司;rTaq酶(5 U/μL)、脱氧核糖核苷三磷酸(deoxy-ribonucleoside triphosphate,dNTP)Mix、pMD18-T vector:大连宝生物技术有限公司;大肠杆菌(Escherichia coli)Top10感受态细胞:鄂西北传统发酵食品研究所自制;PCR引物合成由武汉天一辉远生物科技有限公司完成,引物信息如表1所示。
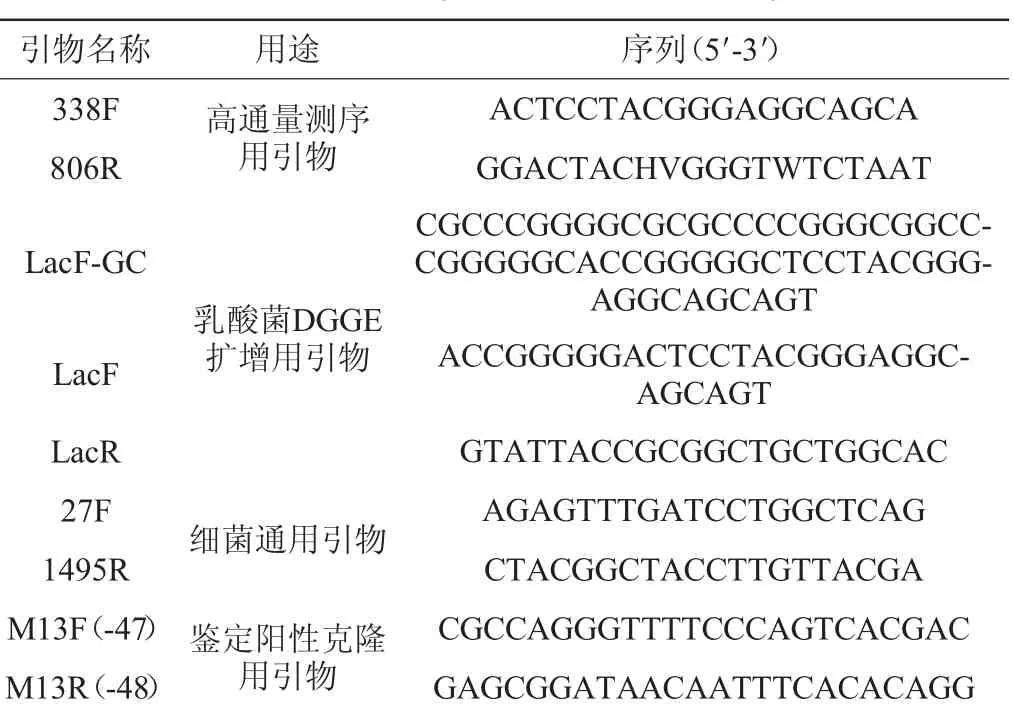
表1 本实验采用的引物信息Table 1 Information of primers used in the experiment
1.2 仪器与设备
MiSeq PE300高通量测序平台:美国Illumina公司;R930机架式服务器:美国DELL公司;CT15RE冷冻离心机:日本HITACHI公司;VeritiTM96-well thermal cycler PCR仪:美国AB公司;NanoDrop 2000/2000c超微量分光光度计:美国Thermo Fisher公司;DCodeTMSystem:美国Bio-Rad公司;DYY-12电泳仪:北京六一仪器厂;Bio-5000 plus扫描仪:上海中晶科技有限公司;DG250厌氧工作站:英国DWS公司。
1.3 试验方法
1.3.1 泡辣椒样品宏基因组提取与检测
采用OMEGA D5625-01试剂盒提取3种泡辣椒样品的宏基因组,采用0.8%琼脂糖凝胶进行电泳检测并用NanoDrop超微量分光光度计测定其DNA的浓度。
1.3.2 泡辣椒样品细菌16S rRNA PCR扩增及MiSeq高通量测序
参照沈馨等[17]的方法对泡辣椒样品中细菌的16S rRNA基因进行PCR扩增及MiSeq高通量测序。PCR扩增体系:5×PCR缓冲液4.0 μL,dNTP mix 2.0 μL,正反向引物338F/806
R各0.8 μL,rTaq酶0.4 μL,DNA模板10 ng,无菌超纯水补充至20 μL。PCR扩增条件:95 ℃预变性3 min;95 ℃变性30 s,55℃退火30s,72℃延伸45s,共30次循环;72℃再延伸10min。其扩增产物用1.0%琼脂糖凝胶电泳检测合格后将浓度稀释至100 nmol/L,寄至上海美吉生物医药科技有限公司进行MiSeq高通量测序。
1.3.3 序列拼接及质量控制
参照周书楠等[18-19]的方法,根据成对序列之间的重叠关系,将双端序列拼接成一条序列;其次根据barcode将所有序列划分到各个样品并对序列方向进行校正,最后切除序列的barcode和引物。使用QIIME v1.7.0分析平台对拼接及质控成功的序列进行操作分类单元(operational taxonomic units,OTU)划分和物种鉴定(包括界、门、纲、目、科和属水平),并计算各分类单元的相对含量。
1.3.4 泡辣椒中乳酸杆菌多样性解析
对乳酸杆菌的16S rRNA基因进行PCR扩增。PCR扩增体系:10×PCR Buffer(含Mg2+)2.5 μL,dNTP 2.0 μL,正向引物(LacF-GC)、反向引物(LacR)和rTaq各0.5 μL,DNA模板1.0 μL,无菌超纯水补充至25 μL。PCR扩增条件:95 ℃预变性4 min;95 ℃变性30 s,55 ℃退火30 s,72 ℃延伸30 s,共循环30次;72 ℃再延伸10 min。PCR扩增产物用2%的琼脂糖凝胶电泳检测。
使用8%聚丙烯酰胺凝胶、变性剂梯度为35%~52%的变性剂(尿素和去离子甲酰胺)进行DGGE。电泳温度为60 ℃,电泳缓冲液为0.5×TAE,上样量为10 μL,120 V条件下运行80 min后,80 V条件下运行13 h。电泳结束后,对凝胶进行硝酸银染色,对DGGE图进行观察、扫描、拍照并找出优势条带切胶回收。回收的PCR扩增产物用不含GC夹子的引物LacF和LacR进行PCR扩增,PCR扩增体系及条件与上述方法相同。将清洁的PCR扩增产物与pMD18-T载体连接后转化到E.coli Top10感受态细胞中,经单克隆鉴定合格后送往武汉天一辉远生物科技有限公司进行测序。将测序结果除去两端载体的序列,然后使用BioEdit软件进行拼接,拼接后在美国国立生物技术信息中心(national center for biotechnology information,NCBI)进行同源性比对,选取同源性较高的模式菌株,采用MEGA 5.0软件中的邻接(neighbor joining,NJ)法构建系统发育树。
1.3.5 泡辣椒中乳酸菌的分离与鉴定
将泡辣椒样品进行倍比稀释并涂布于含有1.0%碳酸钙的MRS固体培养基上,置于厌氧工作站37 ℃恒温培养48 h。挑取有透明圈的单菌落进行纯化、革兰氏染色和保藏。采用十六烷基三甲基溴化铵(cetyl trimethyl ammonium bromide,CTAB)法[20]提取各菌株的DNA,以其为模板进行16S rRNA PCR扩增。在张鲁冀等[21]的方法上稍作修改,PCR扩增体系:10×PCR Buffer(含Mg2+)2.5 μL,dNTP 2.0 μL,正向引物(27F)、反向引物(1495R)、rTaq酶和DNA模板各0.5 μL,无菌超纯水补充至25 μL。PCR扩增条件:95 ℃预变性4 min;95 ℃变性1 min,55 ℃退火1 min,72 ℃延伸90 s,共30个循环;72 ℃再延伸10 min。PCR扩增产物经1.0%的琼脂糖凝胶电泳检测。将清洁的PCR扩增产物按照方法1.3.4处理并对测定结果进行拼接和比对。
1.3.6 数据处理
通过Origin 8.5软件对平均相对含量>1.0%的细菌属进行柱状图的绘制,维恩(Venn)图由在线网站(http://bioinfogp.cnb.csic.es/tools/venny/index.html)进行绘制。利用BioEdit软件和MEGA 5.0软件绘制系统发育树。
2 结果与分析
2.1 基于MiSeq高通量测序技术泡辣椒细菌多样性的研究
3种泡辣椒样品中共产生了131 480条16S rRNA高质量序列,平均每种样品产生43 826 条16S rRNA高质量序列。根据100%的相似性进行序列划分共得到78 050条代表性序列,根据序列的97%相似性进行OTU划分后,共得到8 879个OTU,每种样品平均得到2 959个OTU。使用Ribosomal Database Projec(tRDP)和Greengenes数据库对序列进行同源性比对,将所有的序列鉴定为10个门、18个纲、42个目、82个科和177个属。
在门水平上,3种泡辣椒样品中平均相对含量>1.0%的细菌门分别为硬壁菌门(Firmicutes)和变形菌门(Proteobacteria),其平均相对含量分别为85.70%和12.59%。在属水平上,有10.88%的序列不能被鉴定,其中平均相对含量>1.0%的属如图1所示。
由图1可知,泡辣椒样品中平均相对含量>1.0%的属为乳酸杆菌属(Lactobacillus)、片球菌属(Pediococcus)、克雷伯菌属(Klebsiella)、魏斯氏菌属(Weissella)、葡萄球菌属(Staphylococcus)和明串球菌属(Leuconostoc),其平均相对含量分别为67.37%、7.69%、1.99%、1.16%、1.08%和1.02%。由此可知,隶属于Firmicutes的Lactobacillus为泡辣椒中的优势属。利用MiSeq高通量测序方法,韩俊燕等[20]对发酵辣椒中微生物多样性进行分析,结果发现Firmicutes为所有样品的优势细菌门,其含量>50%,进一步研究得知,Lactobacillus和Weissella为优势细菌属。JUNG J Y等[22]使用454 GS FLX Titanium测序系统发现韩国泡菜中主要的细菌属为Leuconostoc、Lactobacillus和Weissella。

图1 泡辣椒样品中平均相对含量>1.0%的细菌属Fig. 1 Bacterial genera with average relative abundance more than 1.0% in the pickled chili samples
在OTU水平上,进一步统计OTU在3种泡辣椒样品中出现的频率,结果如图2所示。
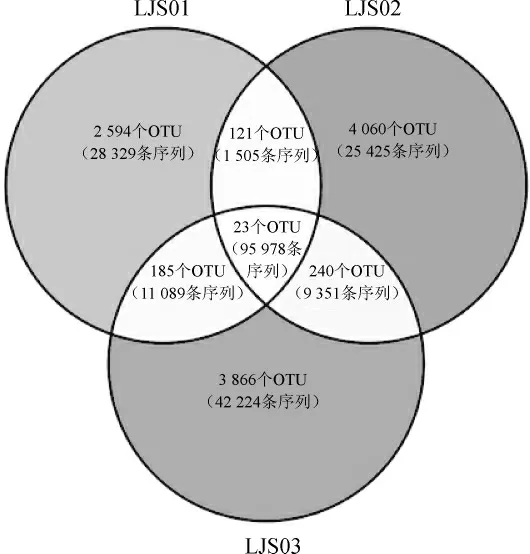
图2 基于OTU水平的Venn图Fig. 2 Venn diagram based on OTU level
由图2可知,3种泡辣椒样品中共有23个核心OTU,仅占OTU总数的0.2%,但包含了95 978条序列,占总序列数的73.0%。在23个核心OUT中,8个核心OTU在3种泡辣椒样品中的平均相对含量>1.0%,分别为OTU1140、OTU2282、OTU3351、OTU7763、OTU326、OTU227、OTU5266和OTU 7746。进一步构建了8个核心OTU的系统发育树,结果如图3所示。
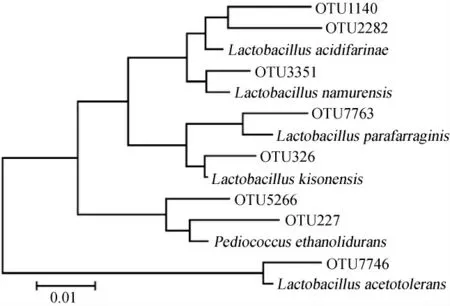
图3 泡辣椒样品中平均相对含量>1.0%核心OTU的系统发育树Fig. 3 Phylogenetic tree of core OTU with average relative abundance more than 1.0% in the pickled chili samples
由 图3 可 知,OTU1140 和OTU2282 与Lactobacillus acidifarinae聚为一类,OTU3351与Lactobacillus namurensis聚为一类,OTU7763和Lactobacillusparafarraginis聚为一类,OTU326与Lactobacillus kisonensis聚为一类,OTU5266和OTU227与Pediococcus ethanolidurans聚为一类,OTU7746与耐酸乳杆菌(Lactobacillus acetotolerans)聚为一类。由此可见,上述8个核心OTU中2个鉴定为Pediococcus,6个鉴定为Lactobacillus,因此,采集自恩施市的3种泡辣椒样品中平均相对含量>1.0%的核心菌群均为乳酸杆菌。
2.2 基于PCR-DGGE测序技术泡辣椒乳酸杆菌多样性的研究

图中L1~L5代表特异性条带编号。图4 泡辣椒样品中乳酸杆菌PCR-DGGE结果Fig. 4 PCR-DGGE results of Lactobacillus in the pickled chili samples
由于本研究中MiSeq高通量测序的靶点主要针对16S rRNA的V3~V4区,所以其引物通用性较强,在种属鉴定时通常仅将其鉴定到属水平。本研究进一步使用乳酸杆菌专用引物对样品中的乳酸杆菌属进行了PCR扩增,并结合PCR-DGGE技术对其多样性进行解析。泡辣椒样品中乳酸杆菌PCR-DGGE电泳结果如图4所示。
由图4可知,在电泳图中共找到5条特异性条带,其中条带L1、L2、L4是3种泡辣椒样品的共有条带,条带L5存在于泡辣椒样品LJS01和LJS03中,但是各条带的亮度不一致,说明同种微生物在不同样品中的含量存在差异,而条带L3只存在于泡辣椒样品LJS03中。由此可见,不同泡辣椒样品间乳酸杆菌多样性存在一定差异,且泡辣椒样品LJS03中乳酸杆菌多样性较高。进一步对各优势条带进行切胶回收和序列分析,结果如表2所示。

表2 DGGE中优势乳酸杆菌序列比对结果Table 2 Sequence alignment results of dominant Lactobacillus in DGGE
由表2可知,5个特异性条带的基因序列与NCBI中模式菌株的基因序列具有较高的相似度(99%~100%)。条带L1为植物乳杆菌(L.plantarum),条带L2为L.namurensis,条带L3为发酵乳杆菌(L.fermentum),条带L4为耐酸乳杆菌(L.acetotolerans),条带L5为短乳杆菌(L.breris)。由此可见,恩施市泡辣椒中乳酸菌多样性较高,采用纯培养技术对其中蕴含的乳酸菌菌种资源进行挖掘显得尤为必要。
2.3 泡辣椒中乳酸菌分离鉴定结果及系统发育分析
使用传统微生物培养方法对3种泡辣椒样品中的乳酸菌进行分离鉴定。从3种泡辣椒样品中共分离得到14株乳酸菌,编号分别为LJS01-1、LJS01-2、LJS01-3、LJS01-4、LJS01-6、LJS01-7、LJS02-1、LJS02-2、LJS02-4、LJS03-1、LJS03-2、LJS03-3、LJS03-5和LJS03-6。将各菌株与NCBI中同源性较高的模式菌构建系统发育树,结果如图5所示。
由图5可知,菌株LJS01-1、LJS01-3、LJS01-6、LJS01-7、LJS03-1、LJS03-5和LJS03-6与L.plantarum聚于一支,菌株LJS01-4、LJS02-2与L.fermentum聚于一支,菌株LJS02-1、LJS01-2、LJS02-4与屎肠球菌(Enterococcus faecium)聚于一支,菌株LJS03-2和LJS03-3与香肠乳杆菌(Lactobacillus farciminis)聚于一支。因此,14株乳酸菌经16S rRNA基因序列鉴定,7株为L. plantarum,2株为L. fermentum,3株为E.faecium,2株为L.farciminis。
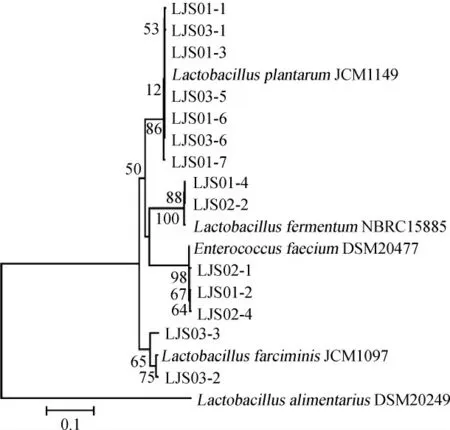
图5 14株菌株基于16S rRNA基因序列的系统发育树Fig. 5 Phylogenetic tree of 14 strains based on 16S rRNA gene sequences
通过纯培养和分子生物学相结合的方法,本研究从泡辣椒样品中分离出的乳酸菌主要为L.plantarum。利用传统分子生物学及16S rRNA测序方法,王修俊等[23]在贵阳发酵辣椒中发现一株产酸能力较强、生长良好的优势菌株,经鉴定为L.plantarum;叶陵等[24]从自然发酵剁辣椒中共鉴定出5株乳酸菌,其中3株为L.plantarum、2株为L.breris。
3 结论
恩施市3种泡辣椒样品中优势细菌门为硬壁菌门(Firmicutes)和变形菌门(Proteobacteria),其平均相对含量分别为85.70%和12.59%;优势细菌属为乳酸杆菌属(Lactobacillus),其平均相对含量高达67.37%。通过传统微生物培养方法共分离鉴定出14株乳酸菌,其中7株为植物乳杆菌(Lactobacillus plantarum)、2株为发酵乳杆菌(Lactobacillusfermentum)、3株为屎肠球菌(Enterococcus faecium)、2株为香肠乳杆菌(Lactobacillus farciminis),表明优势乳酸菌为植物乳杆菌(Lactobacillus plantarum)。

