推导IBS评分在无关个体对人群中概率分布的计算公式
赵焕东 ,赵书民 ,陈玉祥 ,李成涛
(1.中南大学湘雅医院 国家卫生部纳米生物技术重点实验室,湖南 长沙 410008;2.中南大学药学院,湖南长沙 410013;3.江苏东南证据科学研究院有限公司,江苏 南京 210042;4.司法鉴定科学研究院 上海市法医学重点实验室 上海市司法鉴定专业技术服务平台,上海 200063)
2014年,司法鉴定科学研究院与国内其他机构共同研制的《生物学全同胞关系鉴定实施规范》(SF/Z JD0105002—2014)作为部颁规范正式发布实施(以下简称《规范》)。《规范》发布以来,对国内各鉴定机构相关业务的规范实施发挥了积极作用。在该《规范》中,作为判断标准的核心参数并未采用传统的全同胞指数,而是采用了两个个体间常染色体多重STR分型系统的状态一致性(identity by state,IBS)评分。后者计算简单,避免了实际应用中的复杂计算。由于技术规范在内容和篇幅上有特殊要求,该《规范》对完成三种不同数量STR基因座时对应的IBS评分阈值以及相应的系统效能进行了简要说明,但并未对其基本判断原理过多阐述。近年来已有学者对如何更为灵活地使用《规范》进行了一些有益的探索[1]。本研究结合《规范》的研制过程,对IBS评分在生物学全同胞关系鉴定的原假设(H0:被检验个体间为无关个体)中概率分布的计算公式进行了推导与验证。
1 方法与结果
1.1 参数定义
假设两名无关个体(A与B)某一常染色体STR基因座可能的分型结果如表1所示,比较A与B在该STR基因座分型结果有且仅有三种互相排斥的结果:有2个相同的等位基因、仅有1个等位基因相同、无相同等位基因,分别以a2=1、a1=1、a0=1表示。A与B在该基因座相同的等位基因个数即为两者在该基因座上的IBS评分,记作ibs。对特定个体对而言,在某个常染色体STR基因座有:a2+a1+a0=1,且ibs=2a2+a1。表1是对《规范》中单个常染色体STR基因座IBS评分表的扩展。
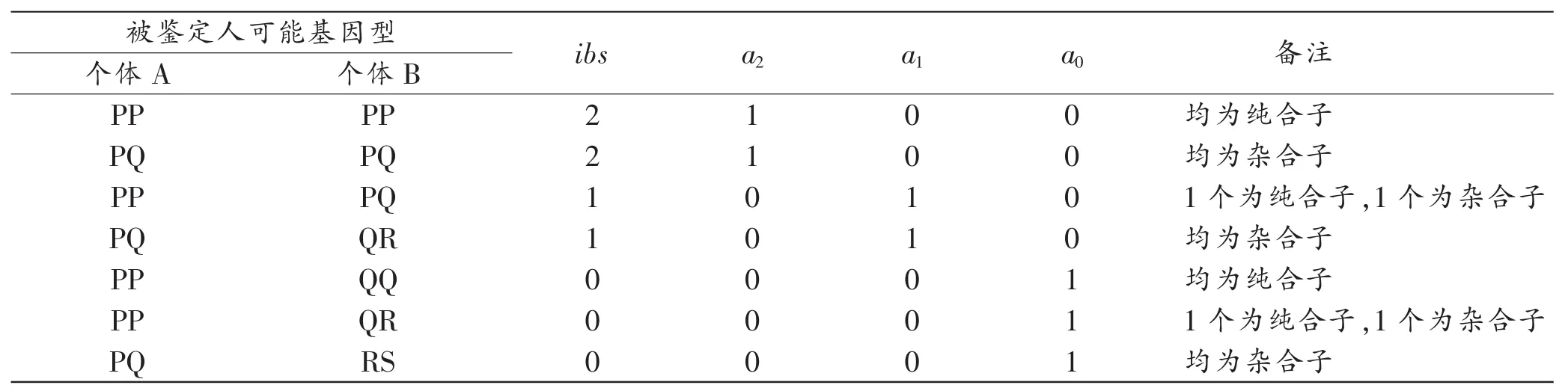
表1 单个常染色体STR基因座分型结果对应的IBS评分表
若对A与B完成n个相互独立的常染色体STR基因座分型,A与B间基因型相同的基因座总个数A2、仅有1个等位基因相同的基因座总个数A1、无相同等位基因的基因座总个数A0则分别为各个STR基因座上 a2、a1、a0值的总和,且有:

依据A2、A1、A0可以计算得到无关个体对在所检测的n个常染色体STR基因座上总相同等位基因个数,即IBS评分,记作IBS,其计算公式为:
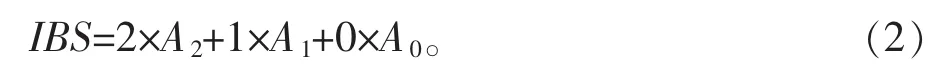
这一计算公式与《规范》中给出的计算公式是等效的。
1.2 无关个体对间不同参数对应概率的分解
若以 p2、p1、p0分别表示 a2、a1、a0取值为 1 的概率,由于A与B在同一个STR基因座上a2、a1、a0三者中必有且仅有其一能取值为1,则有:

依据表1备注信息,以Ho代表纯合子,以He代表杂合子,可将 p2、p1、p0分解为:
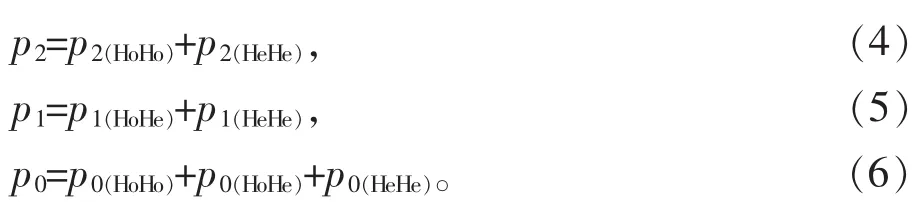
1.3 无关个体对间不同参数概率计算公式的推导
设某STR基因座有 m个等位基因,并以fi(i=1,2,3,…,m)表示该基因座上第 i个等位基因的频率,则有:

1.3.1 推导p2计算公式
1.3.1.1 推导 p2(HoHo)计算公式
依据表1 备注信息,式(4)中 p2(HoHo)指 A 与 B 基因型相同且均为纯合子的概率,依据这一定义,p2(HoHo)可以写作:

1.3.1.2 p2(HeHe)计算公式的推导
依据表1 备注信息,式(4)中 p2(HeHe)指 A 与 B 基因型相同且均为杂合子的概率,依据这一定义,p2(HeHe)可以写作:
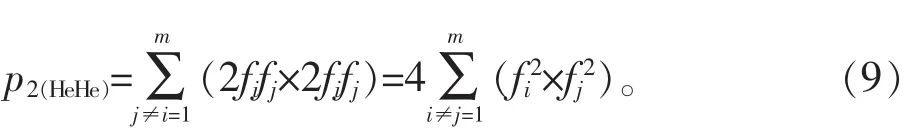
展开后得到:
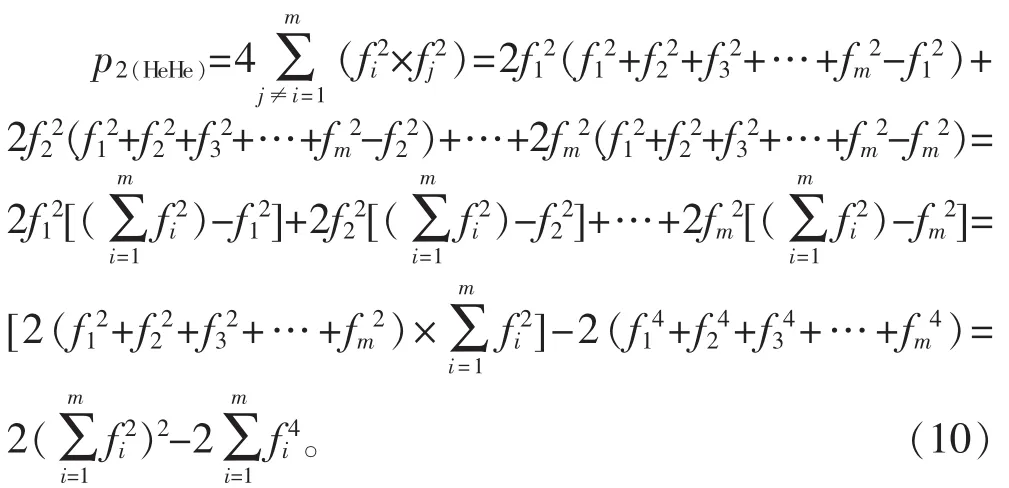
依据式(4)可以得到:
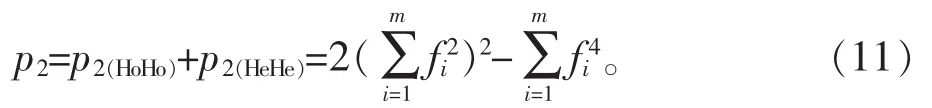
1.3.2 推导p1计算公式
1.3.2.1 推导 p1(HoHe)计算公式
依据表1 备注信息,式(5)中 p1(HoHe)指 A 与 B 在该基因座只有1个相同的等位基因且二者1个为纯合子、1个为杂合子的概率。依据这一定义,p1(HoHe)可以写作:
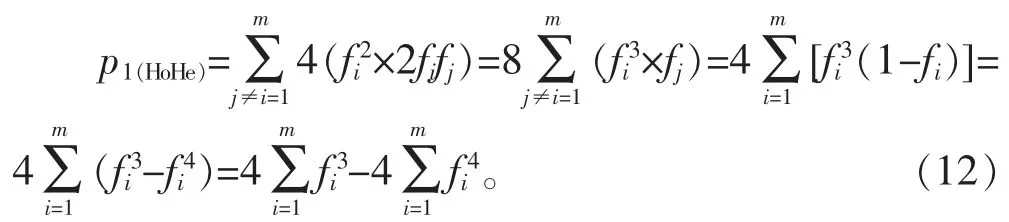
1.3.2.2 p1(HeHe)计算公式的推导
依据表1 备注信息,式(5)中 p1(HeHe)指 A 与 B 在该基因座只有1个相同的等位基因且二者均为杂合子的概率。 依据这一定义,p1(HeHe)可以写作:
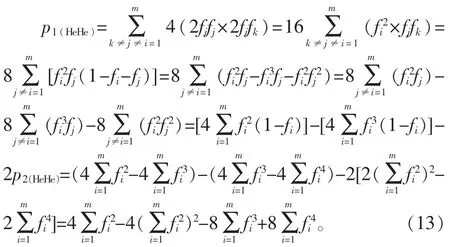
依据 p1(HoHe)、p1(HeHe)的计算公式和式(5),可以得到p1的计算公式为:
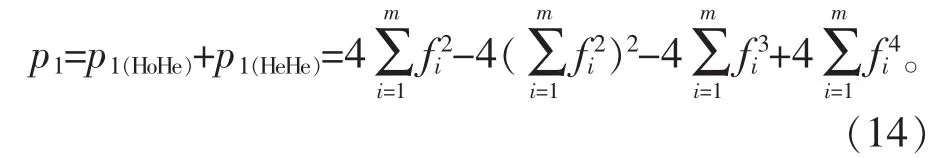
1.3.3 推导p0计算公式
依据式(3)和 p2、p1的计算公式,可以得到 p0的计算公式为:
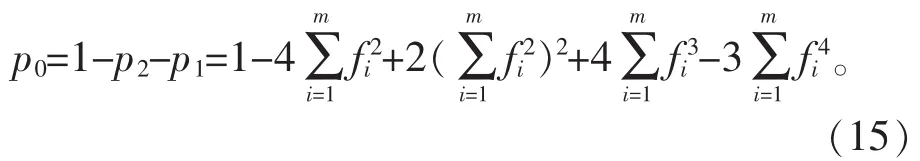
1.3.3.1 推导 p0(HoHo)计算公式
依据表1 备注信息,式(6)中 p0(HoHo)指 A 与 B 无相同等位基因且二者均为纯合子的概率。依据这一定义,p0(HoHo)可以写作:
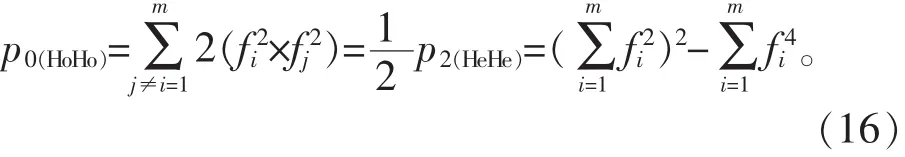
1.3.3.2 推导 p0(HoHe)计算公式
依据表1 备注信息,式(6)中 p0(HoHe)指 A 与 B 无相同等位基因且二者1个为纯合子、1个为杂合子的概率。 依据这一定义,p0(HoHe)可以写作:
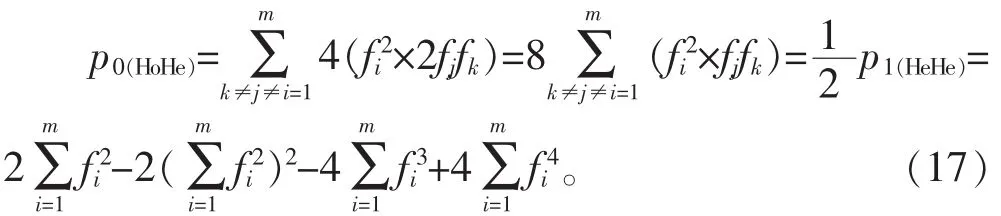
1.3.3.3 p0(HeHe)计算公式的推导
依据表1 备注信息,式(6)中 p0(HeHe)指 A 与 B 均为杂合子且无相同的等位基因。依据这一定义,p0(HeHe)可以写作:
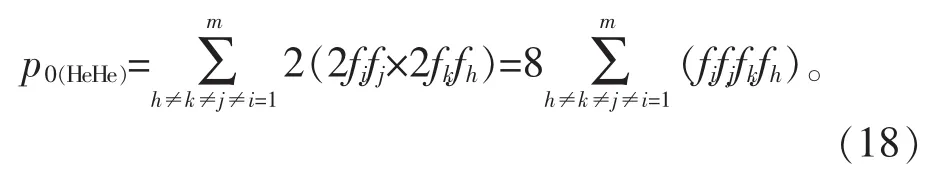
依据式(6)及上述推导得到的 p0、p0(HoHo)和 p0(HeHo)计算公式,即可换算得到的 p0(HeHe)展开式为:
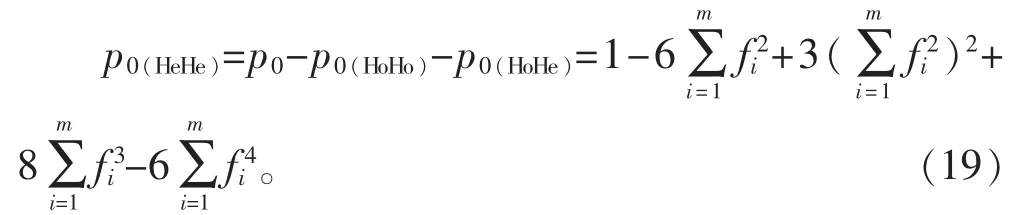
1.4 推导无关个体对人群中IBS二项分布的总体率
依据1.1节中关于a2、a1、a0的定义可知:对无关个体对A与B采用包含n个相互独立的STR基因座进行分型检测,a2=1这样的事件所发生的次数即A2,服从总体率为 P2的二项分布,可记作 A2~B(n,P2)[2]。 同样的,A1服从总体率为P1的二项分布,记作A1~B(n,P1)。其中 P2为 1.3.1 节推导得到的各个 p2l(l=1,2,…,n)的算术平均数,P1为1.3.2节推导得到的各个p1l(l=1,2,…,n)的算术平均数,即有:

依据式(2)和二项分布的可加性[3],IBS亦为服从二项分布的变量。对于包含n个相互独立的STR基因座的分型系统而言,IBS的最大值为2n,则IBS服从总体率为 π 的二项分布:IBS~B(2n,π)。
IBS 的期望值 E(IBS)为:
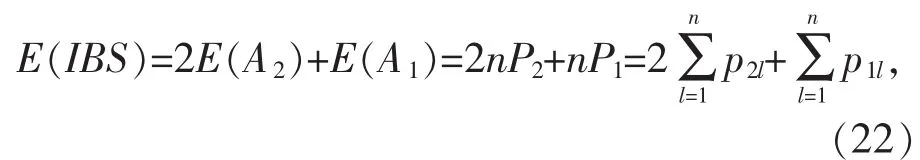
IBS的总体率π为:
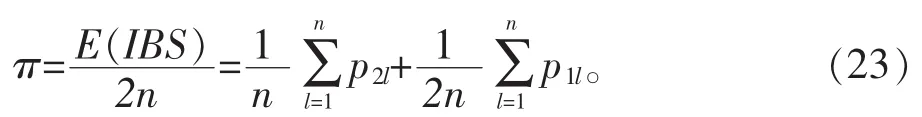
1.5 应用示例
采用参考文献[3-4]中FGA等19个STR基因座在华东汉族人群中的等位基因频率,依据每一个STR基因座各自的等位基因频率和1.3节推导的公式,可以分别计算出各个STR基因座对应的p2、p1、p0值,进而得到对于这19个STR基因座所组成的分型系统各参数在无关个体对人群中的二项分布的总体率(表2),并得到相应参数在无关个体对人群中的二项分布(图1),同时也可以计算得到不同参数、不同取值时对应的全同胞鉴定原假设(H0)对应的概率值。例如,当无关个体对A与B经FGA等19个必检STR基因座分型后,IBS=10,依据IBS二项分布的总体率,通过EXCEL的二项分布函数BINOM.DIST,输入BINOM.DIST(10,38,0.3110,FALSE)即可得到二者为无关个体的概率为0.1181。在得到生物学全同胞对人群中IBS分布特征参数后,即可直接计算IBS=10时二者为生物学全同胞(检验假设中的备择假设H1)的概率。备择假设H1对应概率与原假设H0对应概率的比值,意义即类似于全同胞指数,表示了A与B为生物学全同胞的可能性是二者为无关个体的可能性的倍数。

表2 FGA等19个STR基因座分型系统对应的各参数二项分布总体率
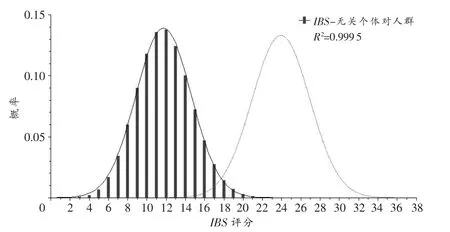
图1 19个STR基因座分型系统IBS评分在无关个体对人群中的二项分布
2 讨 论
在进行生物学全同胞等亲缘关系检验时,通常会有两种相互对立的检验假设,原假设(H0):被检验的两名个体间无任何亲缘关系,即为一对无关个体;备择假设(H1):被检验的两名个体间存在其所声称的亲缘关系(如生物学全同胞、生物学父子等)。依据得到的遗传标记分型结果(遗传学证据),计算某种参数并依据该参数的概率分布来推断两种假设成立的可能性(遗传学证据评价)。当H0成立的概率低于所规定的检验水准时(如 α=0.01),则拒绝 H0、接受 H1。 当 H0的发生概率大于检验水准时,则依据现有的遗传学证据不能拒绝H0,即还不能排除二者是无关个体。这一逻辑判断过程是统计学中的一种基本思想。
前期研究[5-8]已发现,IBS评分是一种理想的用于生物学全同胞的遗传学统计参数。在这些研究中,均尝试划定一个IBS评分阈值来区分全同胞与无关个体,并比较了IBS评分法与传统的亲权指数或似然比方法的一致性,其出发点均与《规范》一致。但在这些研究中均面临同样的问题:受制于特定的STR分型系统。每出现一种新的STR分型系统,或所使用的STR基因座个数发生变化时,均应对其判定全同胞的IBS评分阈值或有效性进行重新评估[1,7]。这无论是对实际案件中的应用还是对新的常染色体STR基因座分型系统的研究都是不利的。因此,需要一种可对任意STR基因座组合的生物学全同胞鉴定效能进行评估的数学模型。
如果能够仅依据STR基因座的等位基因频率这一基础数据,即可了解IBS评分在不同人群(H0对应的无关个体对人群和H1对应的生物学全同胞人群)中的概率分布,将可解决对任意STR分型系统全同胞鉴定效能的有效性评估问题。本研究依据两个个体同一STR基因座可能的基因型组合,通过对有2个相同的等位基因、有1个相同的等位基因和无相同的等位基因这三种情形的概率的分解,依据概率的基本原理,推导出了依据STR等位基因频率计算上述三种情形出现概率的通用公式,而上述三种情形又分别对应了单一STR基因座IBS评分分别为2、1、0的三种情形。随后依据二项分布的可加性得到多重分型系统IBS评分在相应人群中二项分布的关键参数(总体率)。基于所完成分型的STR基因座个数n以及依据这n个STR基因座的等位基因频率计算得到的IBS评分的总体率,即可对任意STR基因座组合的、任意IBS分值对应的H0概率进行计算。如计算检验26个STR基因座、IBS评分为21时的H0概率等。
如何验证这些推导结果的可靠性呢?作为一个可普遍应用的计算公式,可以用一些简单的特例来进行验证。如假定一个STR基因座只有3个等位基因,其等位基因频率分别为0.3、0.3和0.4。由于等位基因个数有限,可以很方便地通过穷举法列举出无关个体对基于这一STR基因座的基因型组合,从而对本研究所推导的公式进行验证。事实上,在既往的一些应用场景中,已有一些计算公式可以用来计算特定情形下STR基因座的概率p2,如在个体识别中个体识别率(discrimination power,DP)的计算公式[9]。某 STR 基因座的DP是指该STR基因座上任意两个无关个体基因型不同的概率,则1-DP即该基因座上任意两个体基因型相同的概率,即1.3.1节推导所得的p2,1-DP的结果与本研究从头推导结果一致,这也说明了本研究推导过程的正确性。另外,两个体间无相同的等位基因,在不考虑突变的情形下,类似于二联体亲权鉴定中的排除亲权的情形,亦即该STR基因座的二联体非父排除率[10]。而1.3.3节所示的从头推导结果与文献报道[10]的二联体非父排除率公式是一致的,这也证明了推导过程的正确性。1.3.3.3节中对p0(HeHe)定义式采用了“已知和求部分”这种简便的加减法计算方式。但如果对1.3.3.3节中p0(HeHe)定义式从头展开,也可以反向通过“已知部分求和”的方法进行验证,因为p2、p1、p0三部分的总和一定为 1。
在进行任一亲权关系鉴定时,原假设H0与备择假设H1都是一对有机整体。本研究仅完成了生物学全同胞关系鉴定中IBS评分在原假设H0对应人群中的概率分布的推导。IBS评分在备择假设(H1)对应人群中的概率分布仍需进一步研究。

