用45个SNP位点组合对国家啮齿类实验动物种子中心3个封闭群小鼠群体的遗传状况分析*
李银银 王 洪 李长龙 郭 萌 魏 杰张 钰 黄 韧 陈振文 杜小燕
(1.首都医科大学基础医学院,北京 100069)(2.中国食品药品检定院实验动物研究所,北京 100050)(3.广东省实验动物监测所,广州 510633)
封闭群小鼠在生物医学研究各个领域中广泛应用[1]。国家啮齿类实验动物种子中心负责对国内各实验动物生产单位提供种源,其动物遗传质量关系到国家实验动物质量安全,影响巨大。定期对群体遗传质量实施监测可为维持群体符合封闭群要求提供可靠信息,为群体保种和繁殖方式提供指导。单核苷酸多态性(single nucleotide polymorphisms,SNP)是近年来出现的第三代遗传标记,由于其具有数目多、分布广、遍布整个基因组及稳定遗传的特点,适于对小鼠遗传状况进行描述[2]。但在国内报道中,大多数研究者选用微卫星DNA技术对封闭群小鼠进行遗传质量分析,而SNP则多用于近交系小鼠的遗传检测[2-4]。本实验选取45个SNP位点对来自国家啮齿类实验动物种子中心北京分中心的ICR(BICR)和上海分中心的ICR(SICR)和KM(SKM)小鼠3个群体进行遗传分析,以期探讨单核苷酸多态性分析在封闭群小鼠遗传质量检测中的应用,并与我们前期完成的微卫星DNA(microsatellite DNA or short tandem repeat, STR)和生化位点方法进行了比较。
1 材料与方法
1.1 动物样本
选取国内较常用的2个封闭群小鼠品系的3个群体,分别是来自国家啮齿类实验动物种子中心北京分中心的ICR群体(BICR)和上海分中心的ICR群体(SICR)及KM种群(SKM),均为SPF级。每个群体随机选择30只小鼠,取0.1 g肾组织于无菌EP管中,-20 ℃冻存备用。本实验已经通过首都医科大学动物实验及实验动物福利委员会审核(AEEI-2016-187)。
1.2 小鼠基因组DNA的制备
对EP管中的组织加入2 mL DNA裂解抽提缓冲液,20 μL 20 mg/mL蛋白酶K,10 μL RNA酶,55 ℃消化过夜,酚氯仿法提取基因组DNA,加入TE缓冲液进行溶解。经Nanodrop 2000c微量分光光度计检测吸光度A值在1.8~2.0之间,并经1.25%琼脂糖凝胶电泳检测,DNA质检合格。取原液适量稀释为50~100ng/μL浓度作为DNA模板,于4 ℃保存备用。
1.3 SNP位点的选择
本研究所用45个SNP位点从相关文献中选取[3-5],位点的选择尽量均匀分布于小鼠的1~20号染色体上,较大范围覆盖小鼠基因组。
1.4 飞行时间质谱(MALDI-TOF)技术SNP位点基因分型[6]
1.4.1PCR扩增反应:根据SNP位点查询NCBI数据库中的序列信息,使用Sequenom公司的引物设计软件Assay design 3.1设计PCR反应和单碱基扩展引物,并进行引物合成。PCR体系包括模板DNA1μL,上下游引物(500 nmol/L)各1 μL,dNTP Mix(25 mmol/L)0.1 μL,HotStarTaq DNA 聚合酶(5U/μL)0.1 μL,MgCl2(25 mmol/L)0.325 μL,PCR Buffer(1.25×)0.625 μL和ddH2O 1.85 μL。扩增条件为94 ℃预变性5 min;94 ℃变性20 s,56 ℃退火30 s,72 ℃延伸1 min,循环45次;最后72 ℃持续3 min。
1.4.2产物碱性磷酸酶处理反应:反应体系包括0.3 μL虾碱性磷酸酶(SAP)(1 U/μL),10×的SAP Buffer 0.17 μL和ddH2O 1.53 μL,将反应混合物加入到上一步PCR产物中。设置反应程序为:37 ℃保温20 min,85 ℃保温5 min。
1.4.3单碱基延伸反应:将多重PCR反应混合物加入上一步SAP酶消化后的产物中。iPLEX反应体系为iPLEX酶0.041 μL,iPLEX Buffer Plus 0.2 μL,引物混合液0.94 μL,iPLEX终止混合液0.2 μL和ddH2O 0.619 μL。设置延伸反应程序:94 ℃预变性30 s;94 ℃变性5 s,52 ℃退火5 s,80 ℃延伸5 s内部循环5个,持续外部循环40个。最后72 ℃延伸3 min。
1.4.4MALDI-TOF-MS检测:PCR反应完成后向产物384孔板中加入16 μL三蒸水,翻转离心机中室温旋转混匀30 min,经树脂纯化。将树脂纯化后的延伸产物点在样品靶上,MALDI-TOF质谱仪分析,检测结果使用TYPER4.0软件获取,根据质谱峰图判读各样本各位点基因分型。
1.5 群体遗传分析
将所有样本的每个SNP位点的基因型以AA、BB和AB等形式输入Popgen 1.32软件中分析[7]。利用该软件计算不同个体在各SNP位点上的基因频率、平均有效等位基因数(Ne)、平均杂合度(Ha)及香隆信息指数等群体遗传参数;利用PIC_CALC软件计算各位点的多态性信息含量(PIC)。
2 结果
2.1 MALDI-TOF-MS检测
采用MALDI-TOF-MS技术对3个小鼠封闭群共90个样本进行45个位点的单核苷酸多态性等位基因分型,并计算了每个SNP位点在3个群体中的等位基因频率(表1)。从表中可以看出,大多数SNP位点(27个)在3个群体中的基因频率有所不同,但是有6个SNP位点(rs8271367、rs29492976、rs13466915、rs3089109、rs3023342和rs3091048)在3个群体中均为单态,有相同的等位基因型;有8个位点(rs29778277、rs32390670、rs3089984、rs3023381、rs3091174、rs3023436、rs3023442和rs3089349)在2个ICR群体中均为纯合而且单态,而在KM群中则存在两种等位基因型;相反有3个位点(rs13476538、rs29315008和rs3089205)在KM小鼠群体中呈单态而在ICR中呈现多态;另有1个SNP位点(rs3089604)在ICR和KM群中的等位基因型完全不同。
2.2 群体遗传结构分析
封闭群SKM小鼠在45个SNP位点中,15个位点表现为单态性位点,30个位点出现二等位基因型。该小鼠群体平均观测等位基因数为1.673 9,平均有效等位基因数为1.376 9,平均杂合度为0.215 2,平均多态性信息含量为0.166 6。高度多态性位点(PIC>0.5) 有0个,中度多态性位点(0.25 而封闭群SICR小鼠在45个SNP位点中,20个位点表现为单态性位点,25个位点出现二等位基因型。该小鼠群体平均观测等位基因数为1.555 6,平均有效等位基因数为1.220 9,平均杂合度为0.143 2,平均多态性信息含量为0.119 9。高度多态性位点(PIC>0.5) 有0个,中度多态性位点(0.25 表1 封闭群小鼠3个群体在45个SNP位点上的等位基因频率 注:“*”标注为筛选的15个用于群体遗传结构分析的位点;“#”标注为筛选的20个用于群体遗传结构分析的位点 Note:“*”means 15 loci used for population genetic structure analysis;“#”means 20 loci used for population structure analysis 表2 封闭群SKM小鼠在45个SNP位点上的等位基因数、平均杂合度和多态性信息含量Table 2 Number of allele, average heterozygosity and polymorphism information content (PIC)of 45 SNP loci in SKM mice 注:Na:等位基因数;Ne:有效等位基因数;Ho:观测杂合度;He:期望杂合度;Ha:平均杂合度;SI:香隆指数;PIC:多态性信息含量 Note: Na:Number of allele; Ne:Number of effective allele; Ho: Observed heterozygosity; He: Expected heterozygosity; Ha: Average heterozygosity; SI: Shannon’s index; PIC: Polymorphism information content 而封闭群BICR小鼠在45个SNP位点中,22个位点表现为单态性位点,23个位点出现二等位基因型。该小鼠群体平均观测等位基因数为1.5111,平均有效等位基因数为1.2851,平均杂合度为0.1699,平均多态性信息含量为0.1375。高度多态性位点(PIC>0.5)有0个,中度多态性位点(0.25 利用Popgen1.32,计算SKM小鼠45个SNP位点的Hardy-Weinberg遗传平衡检验的P值。结果有2个位点显著偏离Hardy-Weinberg遗传平衡,25个位点在群体遗传中处于Hardy-Weinberg遗传平衡,各位点的P值见表2中所示。同样计算SICR、BICR中Hardy-Weinberg遗传平衡检验的P值。显示分别有0和1个位点显著偏离Hardy-Weinberg遗传平衡,11和17个位点在群体遗传中处于Hardy-Weinberg遗传平衡。 我们将SNP位点的检测结果与前期STR和生化位点检测同一群体的结果进行了比较,结果发现(表3),无论是SNP还是STR位点进行群体遗传检测,用杂合度来评价三个群体的遗传多样性时,趋势相同,均为SKM >BICR >SICR;而如果以香隆信息指数(SI)来评价时,SNP的检测结果与杂合度评价的趋势相同,但STR的结果却不同,变为BICR >SICR >SKM。我们进行基因频率排序并去除在群体中单态的SNP 位点,即用基因频率相对高的20和15个SNP位点计算群体遗传参数时,各遗传参数的绝对值有所上升,尤其是SI值与STR的检测结果更趋接近(表3)。在对群体的杂合性和均一性评价时,仍然是SKM群体的多样性(He)和均一性(SI)均为最好。 表3 同一群体分别用SNP、STR和生化位点检测的遗传参数比较 注:Na:等位基因数;Ne:有效等位基因数;Ho:观测杂合度;He:期望杂合度;Ha:平均杂合度;SI:香隆信息指数;PIC:多态性信息含量 Note: Na: Number of allele; Ne: Number of effective allele; Ho: Observed heterozygosity; He: Expected heterozygosity; Ha: Average heterozygosity; SI: Shannon’s index; PIC: Polymorphism information content 昆明小鼠(KM)和ICR小鼠,广泛应用于药物实验、生物制品及化妆品的鉴定、评价和筛选实验中,是我国应用最广泛的封闭群小鼠[8]。为了保证实验数据的重复性和可靠性,需要定期对KM和ICR小鼠乃至所有封闭群实验动物实施遗传质量检测[9],来监测其遗传稳定性[10]。单核苷酸多态性作为第三代遗传标记物在小鼠近交系遗传研究中已经得到应用,国内有多位研究者采用SNP对常用近交系小鼠的遗传质量状况进行了分析,表明SNP可用于小鼠遗传质量检测[3, 11-12]。MALDI-TOF质谱技术具有分析速度快、准确、分辨率高、自动化、灵敏等特点,已经成为快速、准确检测蛋白质和核酸分子的技术,是一种理想的SNP检测方法。因此本研究选取45个SNP位点,运用MALDI-TOF质谱技术对来自上海、北京的3个封闭群小鼠遗传状况分析,来探索SNP在封闭群小鼠遗传质量状况分析中的应用。 本研究选取位于小鼠1~20号染色体上的45个SNP位点,保证标记物对基因组的代表性及研究结果的可靠性。从我们的结果可知(表1),在3个群体中45个位点的基因频率显示,仅在KM小鼠群体中呈单态的位点有3个,相反仅在ICR小鼠群体中呈单态的位点有8个,此外,虽然rs3089604位点在ICR和KM群体都呈现单态,但基因型则完全不同。所以我们认为可以参考以上位点在两种群体中的等位基因频率的不同对ICR和KM小鼠群体进行辨别,即这些位点有可能作为ICR和KM两个品系的鉴定依据,这对于同为白色小鼠的KM和ICR非常有意义。 有效等位基因数和期望杂合度是反映群体遗传变异大小的指标。但由于SNP是二等位基因型,所以其有效等位基因数偏低,但也在一定程度上反映群体中等位基因的分布情况。本实验中3个封闭群中平均观测等位基因数分别为SKM 1.6739、SICR 1.5556和BICR 1.5111,群体观测等位基因数与有效等位基因数有偏差,说明所检测的SNP位点的等位基因在群体基因组中的分布还不够均匀[7]。有多个遗传标准认为一个较好的封闭群其平均杂合度在0.5~0.7之间,而本实验中SKM、SICR和BICR小鼠群体平均杂合度绝对数值偏低,用15个SNP计算后虽然数值有所升高,也远远达不到0.5。究其原因一方面可能是我们选的部分位点在两个封闭群中为单态;另一方面也可能是由于遗传标记本身的性质决定,STR本身就是多等位基因的,有时候一个位点的等位基因数能达到十几甚至几十个[13],而SNP只有两个等位基因,这说明用不同的遗传标记检测封闭群时可能不应以杂合度的绝对值作为其杂合性和多样性的判定标准。尽管如此,进行3个群体平均杂合度比较时,SNP位点的检测结果与STR的检测结果趋势一致,即SKM>BICR>SICR,从等位基因数和平均杂合度角度可以认为SKM小鼠的遗传多样性高于北京和上海的ICR小鼠群体。但是我们发现,用香隆信息指数(SI)来评价时,SNP的检测结果与等位基因数和平均杂合度判断的趋势相同,而STR的结果却不尽相同。我们知道,SI代表了等位基因在群体内个体分配的均匀性,这可能说明所选SNP和STR位点在KM和ICR两个群体中的均匀性不同。 总之,本研究表明在封闭群小鼠遗传状况的分析中,SNP可以作为一项遗传标记来反应其遗传状况,而且有些SNP位点可能具有进行封闭群小鼠品系鉴定的潜在应用前景。另外本研究所选的位点中有部分位点属于单态性,在群体遗传中可以适当舍弃,有必要开发更多的SNP位点及群体分析扩展SNP在小鼠封闭群中的应用前景。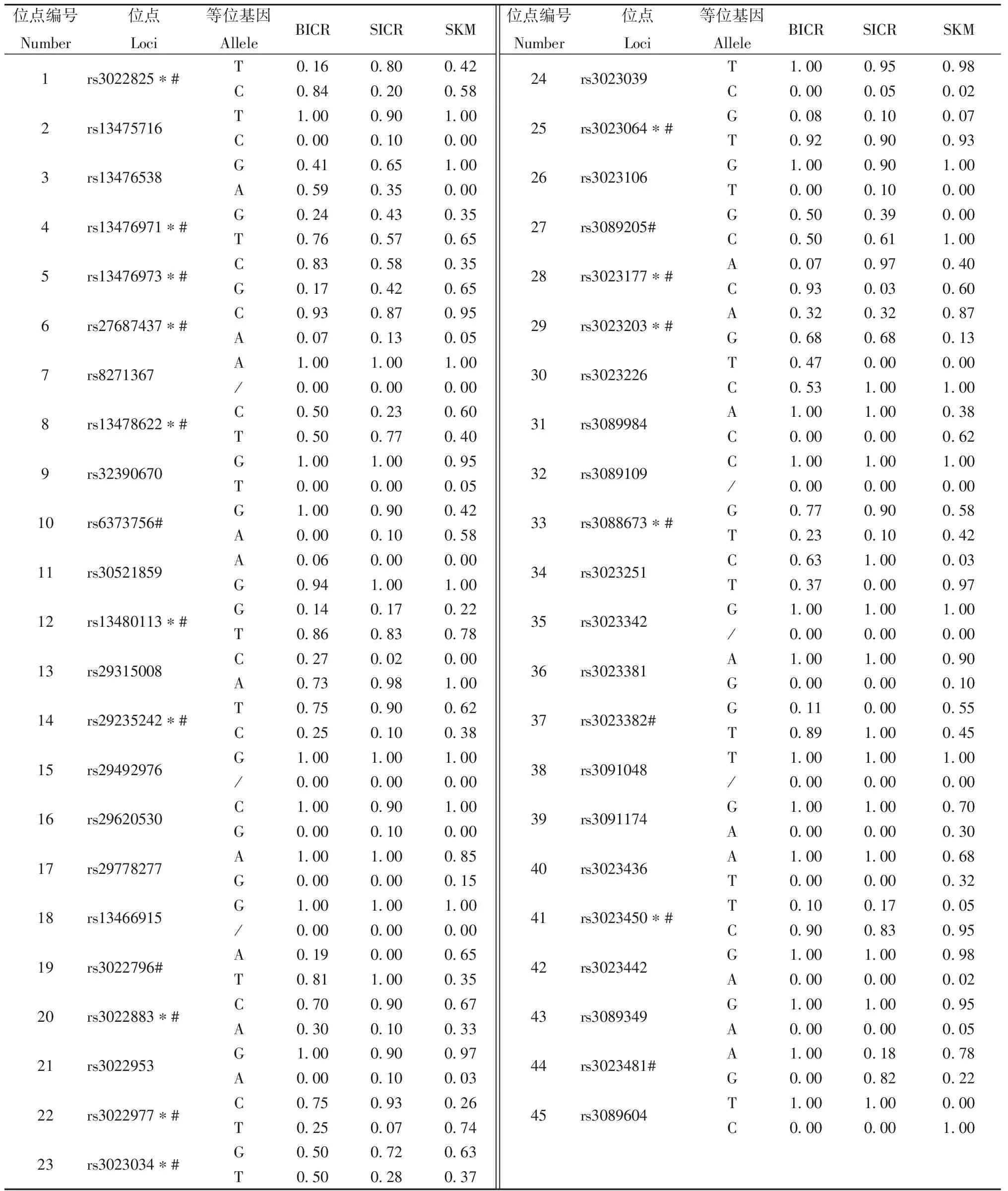

2.3 Hardy-Weinberg遗传平衡检验
2.4 SNP位点与微卫星DNA和生化位点方法的比较

3 讨论

