伤寒沙门菌非编码RNA AsfD的分子鉴定及表达特性
刘锐, 种晓丹, 赵昕, 陆仁飞, 孙嘉遥, 黄新祥
(1. 江苏大学医学院, 江苏 镇江 212013; 2. 海军军医大学转化医学研究院肿瘤研究所, 上海 200433)
伤寒沙门菌(Salmonellaentericaserovar Typhi,S. Typhi)主要通过污染的食物和水传播[1]。S.Typhi进入机体后,通过胃、十二指肠,最终侵入小肠的肠黏膜[2-3]。最近几年,在细菌中鉴定出大量的非编码RNA,其对细菌的表型起着重要的调节作用,比如动力、生物膜形成和毒力等。我们前期对伤寒沙门菌进行高通量测序和转录组分析,发现了许多非编码RNA,如flhDC基因对侧编码的疑似非编码RNA,并将其命名为AsfD(未发表)。在本研究中,我们先后通过RNA印迹,RT-PCR和cDNA 5′末端快速扩增实验(5′rapid amplification of cDNA ends,5′RACE)鉴定AsfD的长度,最后通过qRT-PCR检测AsfD在不同生长时相和环境应激条件下的表达量变化。
1 材料与方法
1.1 材料
S.Typhi野生株GIFU 10007由本实验室保存。NaCl、NaOH、HCl、无水乙醇、异丙醇均购于上海国药公司;反转录试剂盒、DL2000 Marker、rTaqDNA聚合酶、5′RACE试剂盒(大连TaKaRa公司);DIC Northern试剂盒(Roche公司);质粒提取试剂盒购于上海生工公司;RNA提取试剂Trizol(Invitrogen公司)。
PCR仪(ABI公司);荧光定量PCR仪(BIO-RAD公司);核酸紫外检测仪(Thermo公司);核酸杂交仪(Roller-Blot公司)。
1.2 引物
根据S.Typhi全基因组序列,设计特异性引物序列送苏州泓迅进行合成,引物序列及用途详见表1。
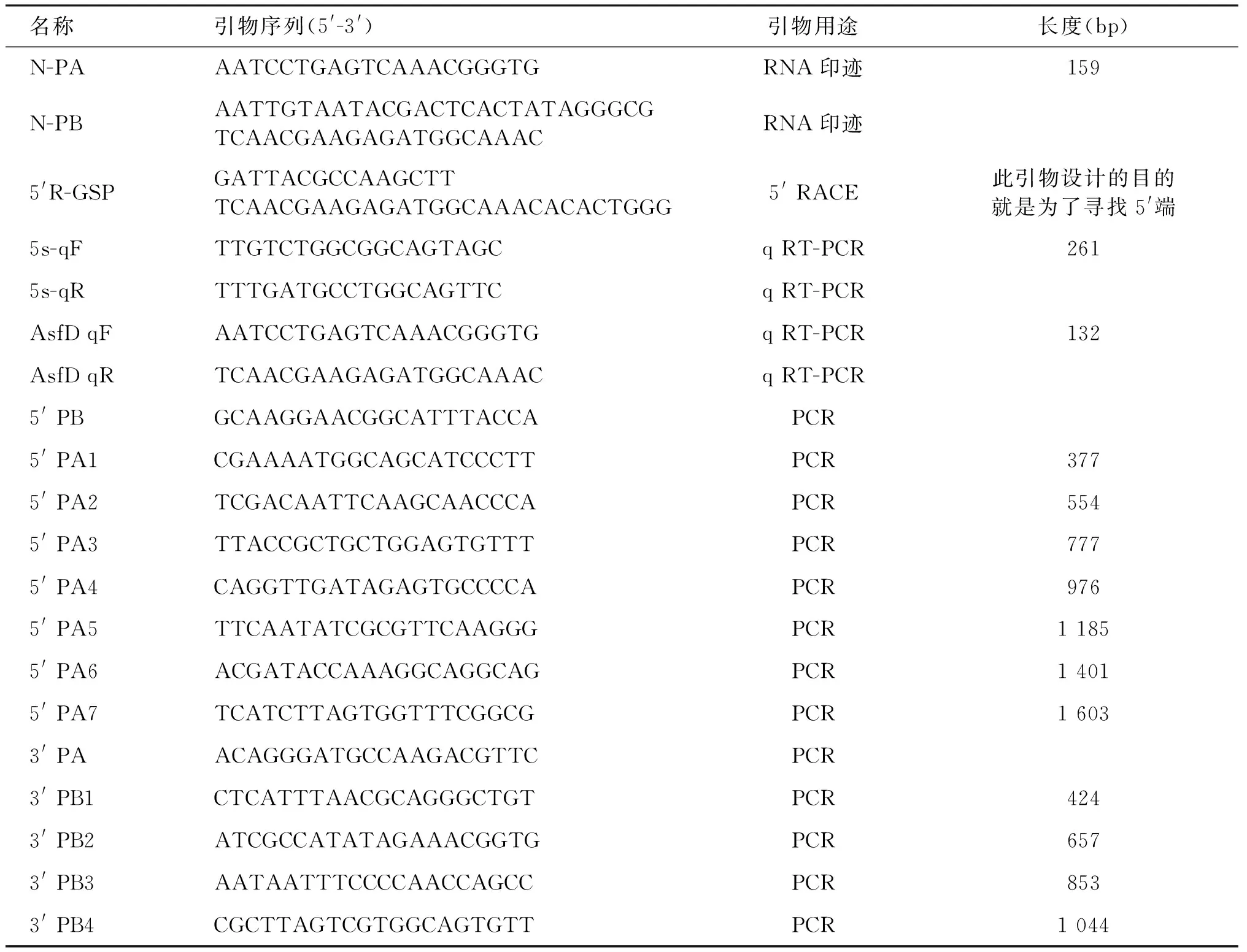
表1 引物名称及序列
1.3 细菌培养
过夜培养S. Typhi野生株,次日以1 ∶100的比例接种到LB培养液中,于37 ℃,250 r/min振荡培养,每培养一段时间检测细菌时相,直至生长到需要的时相,D(600nm)值分别为0.4、1.0、2.0。为模拟细菌环境应激,在D(600 nm)=0.4的菌液中分别加酸(HCl,pH=4.5),氧(H2O2, 4 mmol/L)和盐(NaCl,0.3mol/L),应激30 min。收集培养液中的细菌,用Trizol法提取细菌总RNA,用DNA酶Ⅰ去除残留DNA,通过核酸紫外检测仪检测RNA浓度。
1.4 RNA印迹检测AsfD表达
在已知AsfD序列区设计特异性引物(表1),以S.Typhi染色体为模板,进行PCR扩增获得探针模板。用QIA快速PCR纯化试剂盒(QIAGEN公司)纯化PCR产物,体外转录制备RNA探针。取RNA样品10 μg电泳4 h(200 V),转膜,56 ℃杂交12 h,洗膜并加地高辛抗体,曝光显影。
1.5 RT-PCR寻找AsfD转录起始和终止区域
寻找AsfD转录起始区域:在AsfD已知序列区设计1条逆向引物5′PB,已知序列区上游不同区域设计7条前向引物5′PA1~7,以S.Typhi野生株基因组DNA为模板和各种特异性引物进行PCR扩增的片段(377、554、777、976、1185、140、1 603 bp)作为阳性对照,以RNA样本和各种特异性引物进行RT-PCR扩增。
寻找AsfD转录终止区域:在AsfD已知序列区设计1条前向引物3′PA,已知序列区下游不同区域设计4条逆向引物3′PB1~4,以S.Typhi野生株基因组DNA为模板和各种特异性引物进行PCR扩增的片段(424、657、853、1 044 bp)作为阳性对照,以RNA样本和各种特异性引物进行RT-PCR扩增。
1.6 5′RACE
采用SMARTer RACE 5′/3′试剂盒进行AsfD 5′RACE。在AsfD近5′端1 216~1 443 bp设计特异性逆向引物5′R-GSP,取对数生长期的细菌总RNA 1 μg进行反转录,按说明书操作在cDNA5′端加SMARTer Ⅱ A寡核苷酸接头,UPM(前向随机引物)和5′R-GSP进行PCR扩增。将PCR产物克隆到PUC19载体。最后选出疑似阳性菌的质粒送泓迅进行DNA测序,检测AsfD的转录起始位点。
1.7 qRT-PCR检测AsfD的表达
取不同生长时相和环境应激条件下的细菌,提取总RNA各4 μg;用特异性逆向引物AsfD qR反转录合成cDNA,再以AsfD qF/qR进行qRT-PCR,以5S rRNA作为内参,用5S-R引物进行反转录,用5S-F/R进行qRT-PCR。实验分3批,每批重复3次。
1.8 统计学分析
运用GraphPad进行统计学分析,两组数据比较采用Student′st检验,P<0.05为差异有统计学意义。
2 结果
2.1 RNA印迹鉴定AsfD在伤寒沙门菌中的表达
结果如图1所示,电泳可见长为2 300 nt左右的特异性条带,说明伤寒沙门菌中确有AsfD表达,长约2 300 nt。

M: RNA标准参照物;1:RNA样本与特异性探针杂交条带
2.2 RT-PCR分析AsfD的转录起始和终止位区域
RT-PCR检测结果显示,片段长度分别为377、554、777、976和1 185 bp有阳性扩增,但长度为1 401 bp及1 603 bp的扩增为阴性。结合伤寒沙门菌基因组结构分析,结果表明AsfD转录起始区域位于flhC基因终止密码子下游434~660 bp之间。见图2。
结果如图3所示,片段长度分别为424 bp、657 bp、853 bp有阳性扩增,但长为1 044 bp的扩增为阴性。结合伤寒沙门菌基因组结构分析,表明AsfD转录终止区域位于flhD基因起始密码子上游558~788 bp之间。

M:DNA标准参照物;1、4、7、10、13、16、19:样本PCR产物;2、5、8、11、14、17、20:未加入反转录酶的阴性对照;3、6、9、12、15、18、21:阳性对照
图2RT-PCR分析AsfD的转录起始区域
M: DNA标准参照物;1、4、7、10:样本PCR产物;2、5、8、11:未加入反转录酶的阴性对照;3、6、9、12:阳性对照
图3RT-PCR分析AsfD的转录终止区域
2.3 5′RACE确定AsfD的转录起始位点
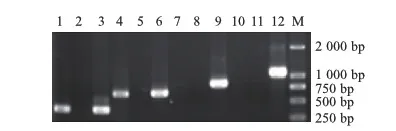
结果如图4所示,电泳中出现疑似阳性质粒条带;将疑似阳性质粒送泓迅进行DNA测序。结果显示AsfD转录起始位点位于flhC基因终止密码子下游590 nt处(图5)。并且序列分析整个AsfD片段没有发现明显的ORF序列。
2.4 伤寒沙门菌AsfD的表达特性分析
qRT-PCR结果表明,AsfD在伤寒沙门菌生长的稳态期表达较高,而在HCl,H2O2,NaCl应激后表达均明显下降。见图6。
M:DNA标准参照物;1:疑似阳性质粒;2:阴性对照
图45′RACE质粒电泳分析结果

+1:AsfD转录起始位点;方框内为flhC终止码
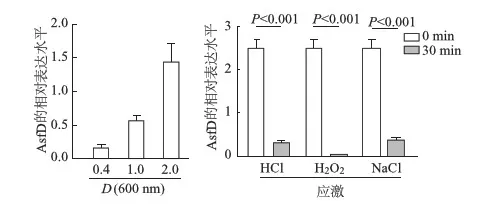
图6 不同时相和应激条件下AsfD的表达量
3 讨论
长链非编码RNA是指长度超过200 nt的非编码RNA,因缺少ORF系列而不能编码蛋白质[4-5]。本研究组前期通过RNAseq分析,发现伤寒沙门菌flhDC基因反义链编码一个疑似非编码RNA AsfD,其长度接近2 000 nt(未发表);为了进一步鉴定AsfD分子特性,进行RNA印迹,RT-PCR和5′RACE等实验,结果表明AsfD的长度在2 079 nt~2 310 nt之间,AsfD确定的2 079 nt序列能够完全覆盖flhDC基因;序列分析整个AsfD片段没有发现明显的ORF序列。以上结果表明新发现的疑似非编码RNA AsfD很可能为伤寒沙门菌中新的长链非编码RNA。
我们通过qRT-PCR分别检测S.Typhi在不同生长阶段和应激条件下AsfD的表达水平。结果表明,AsfD的转录水平随着S.Typhi生长而增加,并且在稳态期达到最高,这与之前的一些非编码RNA在稳态期能够高表达的研究结果相一致[6]。同时还发现,伤寒沙门菌AsfD的转录水平在酸、氧和高渗环境应激下均下降,说明其可能为细菌中的一个调节性长非编码RNA。
flhDC基因是由flhD和flhC基因构成的操纵子,能够编码鞭毛的主要调控因子FlhDC[7]。之前的研究表明,一些非编码RNA能够调控flhDC操纵子的表达以及动力:例如,sRNA ArcZ、OmrA和OmrB能够通过与flhDC操纵子mRNA 5′端碱基配对从而调控动力和flhDC的表达[8],McaS能够增强鞭毛合成的主要转录调控子FlhD表达,从而增强细菌动力[9]。由于本研究鉴定的AsfD能够完全覆盖flhDC操纵子。因此,我们猜测AsfD很可能影响flhDCmRNA的稳定性而调控其表达水平,从而影响细菌的动力。
综上所述,本研究针对在RNAseq分析基础上发现的一个新的疑似非编码RNA AsfD,通过分子鉴定分析,确定为长链非编码RNA,且其长度在2 079~2 310 nt之间。AsfD的转录水平随着生长而增加,在稳态期达最高,而在酸、氧和高渗环境应激下均下降;关于其具体功能作用机制尚有待进一步研究。
[ 1 ] Chiou CS, Lauderdale TL, Phung DC, et al. Antimicrobial resistance inSalmonellaentericaSerovar Typhi isolates from Bangladesh, Indonesia,Taiwan,and Vietnam[J]. Antimicrob Agents Chemother, 2014,58(11): 6501-6507.
[ 2 ] Ahirwar SK, Pratap CB,Patel SK,et al.Acid exposure induces multiplication ofSalmonellaentericaserovar Typhi[J]. J Clin Microbiol, 2014, 52(12): 4330-4333.
[ 3 ] Kaur J, Jain SK. Role of antigens and virulence factors ofSalmonellaentericaserovar Typhi in its pathogenesis[J]. Microbiol Res, 2012, 167(4): 199-210.
[ 4 ] Wang Y, Xu G, Chen W, et al. Detection of long-chain non-encoding RNA differential expression in non-small cell lung cancer by microarray analysis and preliminary verification[J]. Mol Med Rep, 2015, 11(3): 1925-1932.
[ 5 ] He K, Wang P. Unregulated long non-coding RNA-AK058003 promotes the proliferation, invasion and metastasis of breast cancer by regulating the expression levels of the gamma-synuclein gene[J]. Exp Ther Med, 2015, 9(5): 1727-1732.
[ 6 ] Vogel J, Bartels V,Tang TH,et al.RNomics inEscherichiacolidetects new sRNA species and indicates parallel transcriptional output in bacteria[J]. Nucleic Acids Res, 2003, 31(22): 6435-6443.
[ 7 ] Singer HM, Erhardt M, Hughes KT. RflM functions as a transcriptional repressor in the autogenous control of theSalmonellaFlagellar master operonflhDC[J]. J Bacteriol, 2013, 195(18): 4274-4282.
[ 8 ] De Lay N,Gottesman S.A complex network of small non-coding RNAs regulate motility inEscherichiacoli[J]. Mol Microbiol, 2012, 86(3): 524-538.
[ 9 ] Thomason MK, Fontaine F, De Lay, et al. A small RNA that regulates motility and biofilm formation in response to changes in nutrient availability inEscherichiacoli[J]. Mol Microbiol, 2012, 84(1): 17-35.

