基于蒙特卡洛法评定转基因玉米MIR604的测量不确定度
宋 君,常丽娟,陶 李,张富丽,王 东(四川省农业科学院分析测试中心,四川 成都 610066)
【研究意义】由于价格和消费者的接受程度等因素影响以及相关法律规定,人们需要区分转基因产品与非转基因产品以及知道产品中转基因成分的含量。目前,转基因成分的鉴定一般可以采用检测外源基因(核酸)和外源蛋白质的方法实现[1-2],而转基因成分的精准定量目前得到世界广泛认可的方法是采用实时荧光定量PCR方法。近年,转基因生物及产品的检测方法研究,普遍集中在转化事件的终点PCR法和实时PCR法[3-5]。此外,有转基因生物及产品的传感器方法、质谱法、近红外光谱法等报道,但因成本高、仪器设备配置要求高、可操作性低等原因未得到广泛推广应用。近20年来,人们对基于DNA的转基因成分实时荧光定量PCR检测技术研究非常重视[6-8],建立的部分转基因成分定量检测方法已经非常成熟且上升为国际、国内检测技术标准并在实践中大量应用,但却忽略了这些方法在应用中的质量保证研究。【前人研究进展】有报道指出转基因成分的定量检测结果与理论值的相对偏差不高于25 %就能被广泛接受[9],而且随着转基因成分含量的降低,检测结果偏差将会上升。是何原因导致转基因成分定量有较高的相对偏差,影响因素是有哪些,影响因素的贡献大小是多少,如何提高转基因成分的定量检测质量。截止目前,尚无针对上述问题的研究报道。转基因玉米MIR604是由美国先正达种子公司(Syngenta Seeds Inc.)开发的的一种抗虫转基因玉米,该品系包含由启动子MTL和终止子NOS调控的抗虫基因Cry3A和由启动子Ubi Zm1、终止子NOS调控的标记基因pmi。2006年该品系经过安全评价作为食品和饲料加工原材料首次在澳大利亚和新西兰市场投放。自2007年开始,包括美国、加拿大、日本、阿根廷和巴西在内共有5个国家进行商业化种植MIR604玉米品系(http://www.isaaa.org/gmapprovaldatabase/event/default.asp?EventID=131)。我国自2008年开始批准进口该转玉米品系用作食品和饲料的加工原材料,禁止商业化种植。【本研究切入点】常丽娟等针对转基因玉米MIR604品系外源基因的侧翼序列设计了一对特异引物和探针[10],建立了成熟的转基因玉米MIR604品系的实时荧光定量PCR检测方法并确认了该法对转基因玉米MIR604品系的特异性和灵敏度(达到5拷贝数)。测量不确定度是评估检测质量的重要参数。【拟解决的关键问题】通过对转基因成分的测量不确定评定研究,可发现影响检测质量的因素,并能将影响因素对不确定度的贡献进行数值化定量分析,找到影响检测质量最大的“干扰因子”,从而有针对性地改进实验环节和步骤,提高转基因成分的定量检测质量。本文首次采用蒙特卡洛方法(Monte Carlo Method, MCM)开展转基因玉米MIR604的测量不确定度评定研究,回答并解决长期困扰影响转基因成分定量检测质量的问题。
1 材料与方法
1.1 材料、试剂及仪器
校准定量PCR仪(拟合校准曲线)的校准子(Calibrator)为10 %的转基因玉米DAS-4-40278-9有证标准物质基因组DNA的5个梯度浓度稀释液;1 %含量转基因玉米DAS-4-40278-9的标准品作为测试样品;DNA分离和定量PCR试剂采用TIANGEN生化科技有限公司(北京)生产的高效植物基因组DNA 提取试剂盒和SuperReal PreMix (Probe)试剂盒;实验中使用到引物/探针序列(上海生工合成和标记)参照本实验室建立的方法[10];本研究用到的主要仪器有超微量分光光度计(Nanodrop-1000, Thermo, USA)和实时荧光定量PCR仪(7500,ABI, USA),分别测量分离、纯化到的DNA溶液质量(浓度、纯度和完整性)和扩增、测量玉米内源基因(zSSIIb)、转基因玉米MIR604片段及其绝对含量。
1.2 DNA提取
标准物质和试样DNA的分离、纯化以及PCR反应体系配置,按照试剂盒说明书操作。
1.3 校准曲线拟合和试样测量
把标准物质DNA的溶液分别稀释成105、21、4.2、0.84和0.17 ng/μl,然后用上述4 μl系列标准物质DNA溶液[按照玉米基因组大小(5×109base) pairs][11]计算,分别相当于1.9×106、4×105、8×104、1.6×104、3.2×103copies DNA分子)的对数值(校准子)及其PCR仪检测到的响应Ct值(校准子)作为拟合zSSIIb基因、MIR604片段校准曲线的输入量。试样DNA溶液稀释到12.5 ng/μl。PCR反应体系含4 μl标准物质DNA溶液或试样溶液、2×主混液10 μl,上、下游引物各0.5 μM,探针0.25 μM,灭菌水补至20 μl。每个DNA浓度点做3个平行实验;试样重复做24次测量。PCR反应程序为 95 ℃,10 min; 95 ℃,15 s;59 ℃,1 min(45 cycles)。
1.4 数学测量模型和不确定度评定
转基因玉米MIR604的测量分为3个阶段,包含3个数学模型。
第1阶段是按照(1)式(数学模型)拟合zSSIIb基因和MIR604片段的校准曲线:
Ct=m×lgA+k
(1)
式中,A为zSSIIb基因或MIR604片段的已知量,Ct为A的仪器响应值(threshold of cycles,Ct),二者作为拟合曲线的输入量;k和m分别为校准曲线的输出量截距和斜率。
第2阶段是把PCR仪检测到的试样zSSIIb基因和MIR604片段的Ct值作为输入量代入(2)式(校准曲线方程的变形式),计算获得试样zSSIIb基因和MIR604片段的绝对量:
A=10(Ct-k)/m
(2)
第3阶段是把试样zSSIIb基因和MIR604片段的绝对量作为输入量代入(3)式计算获得试样中转基因玉米MIR604的相对百分含量C输出量:
C=A外/A内×100 %
(3)
式中,A外和A内分别为试样中MIR604片段和zSSIIb基因的绝对量。
按照ISO-GUM Supp1[12],采用MCM进行概率分布传递来评定转基因玉米MIR604的测量不确定度。编写MATLAB代码实现评定过程中的抽样、计算。
2 结果与分析
2.1 输入量PDF的设定和抽样
本研究涉及到的输入量包括用于PCR仪校准的系列校准子(Cti)、校准曲线斜率(m)和截距(k)、试样内/外源基因Ct值、试样内/外源基因绝对含量(A内/A外),其中校准曲线斜率(m)和截距(k)、试样内/外源基因绝对含量(A内/A外)在不同的阶段既作为输入量同时也作为输出量。
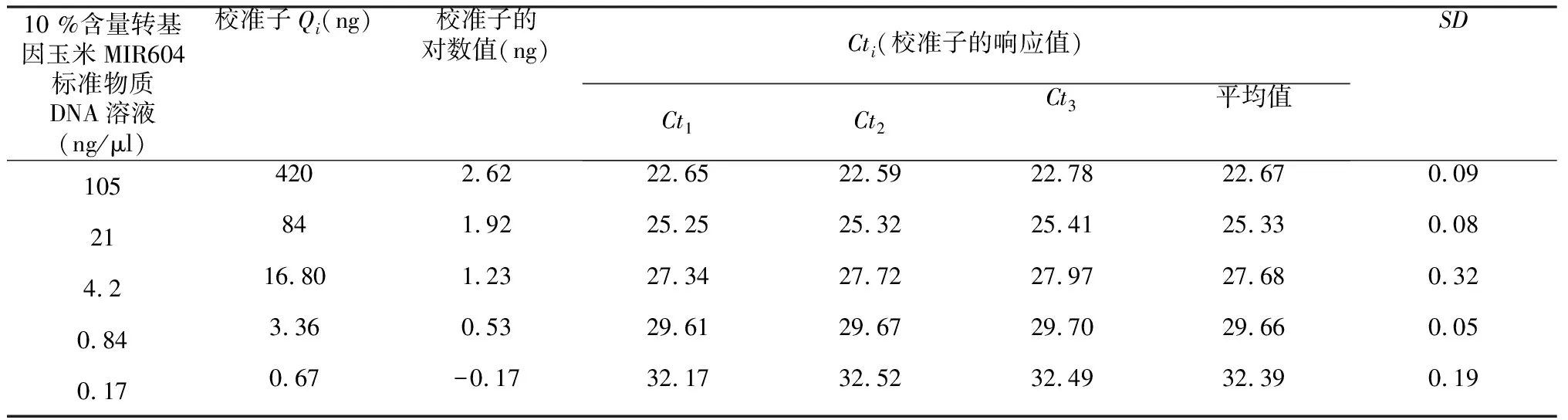
表1 zSSIIb内源基因校准曲线拟合Table 1 Fitting of calibration curve for endogenous gene zSSIIb
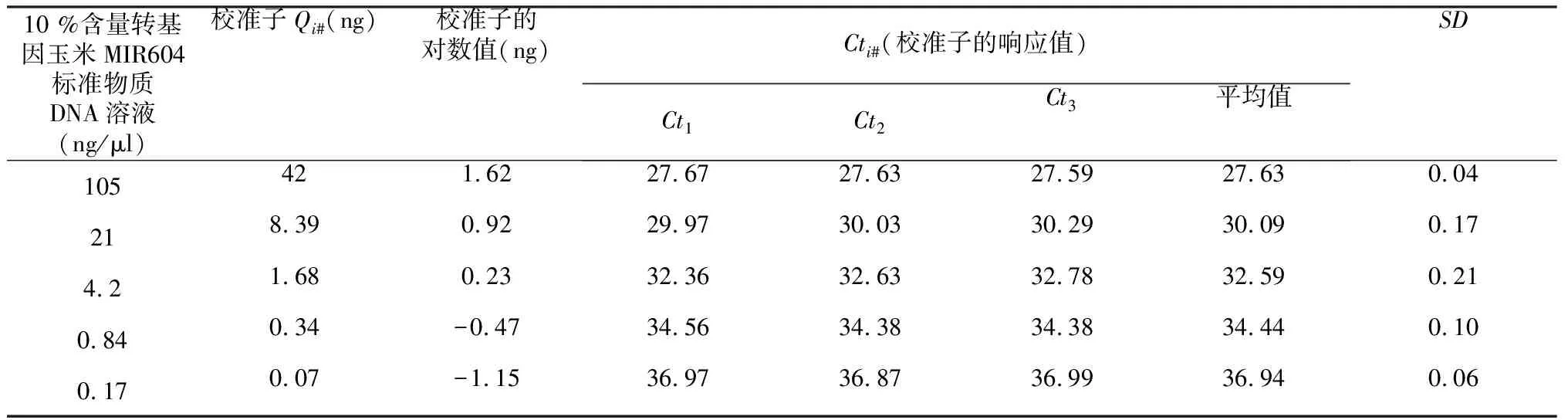
表2 MIR604片段校准曲线拟合Table 2 Fitting of calibration curve for MIR604 fragment
PCR仪校准的系列校准子(Cti)(表1~2)、试样内/外源基因Ct值(表3)的PDF遵守均匀分布R(a,b)(a为均匀分布的下限,b均匀分布的上限),从标准均匀分布R(0,1)中抽取随机数r,对任意值ξ,满足ξ=a+(b-a)r;试样内/外源基因绝对含量(A内/A外)的PDF设定为正态分布N[y,u2(y)] [y为最佳估计值,u(y)为标准不确定度],从标准正态分布N(0,1)中抽取随机数r,对任意值ξ,构造ξ=y+u(y)r;由于校准曲线的斜率m和截距k是2个相关联的量。因此,m和k的PDF设定为双元正态联合,按照JCGM 101:2008[12]、JCGM 102:2011[13]规定和Sega[14]报道的方法处理。各输入量的分布类型、均值和包含区间见表4。
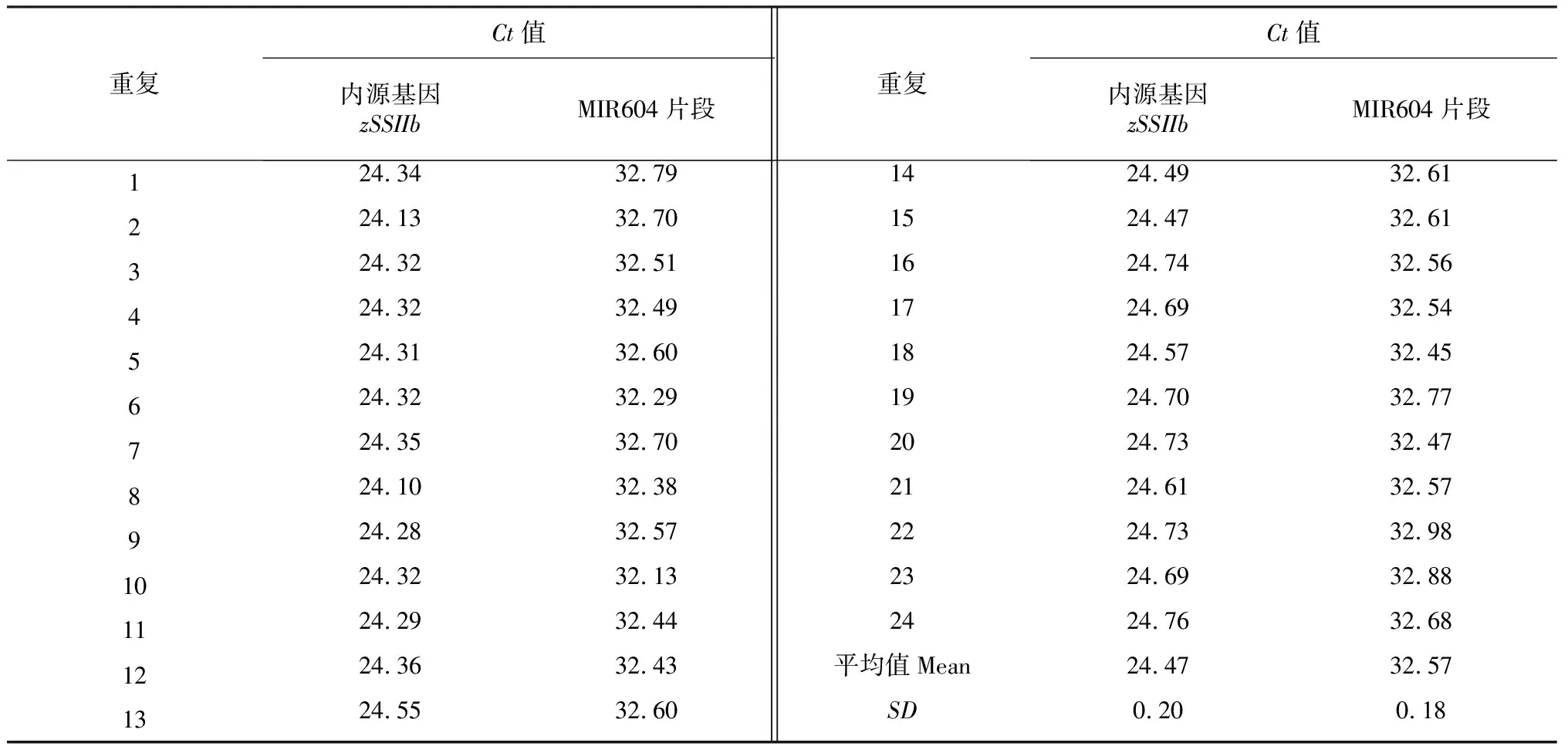
表3 试样中内源基因zSSIIb和MIR604片段的Ct值Table 3 Ct values of endogenous gene zSSIIb and MIR604 fragment in the sample

表4 各输入/输出量的分布及不确定度和95 %包含概率的包含区间Table 4 The probability distribution of all inputs and outputs and their 95 % coverage intervals
续表4 Continued table 4
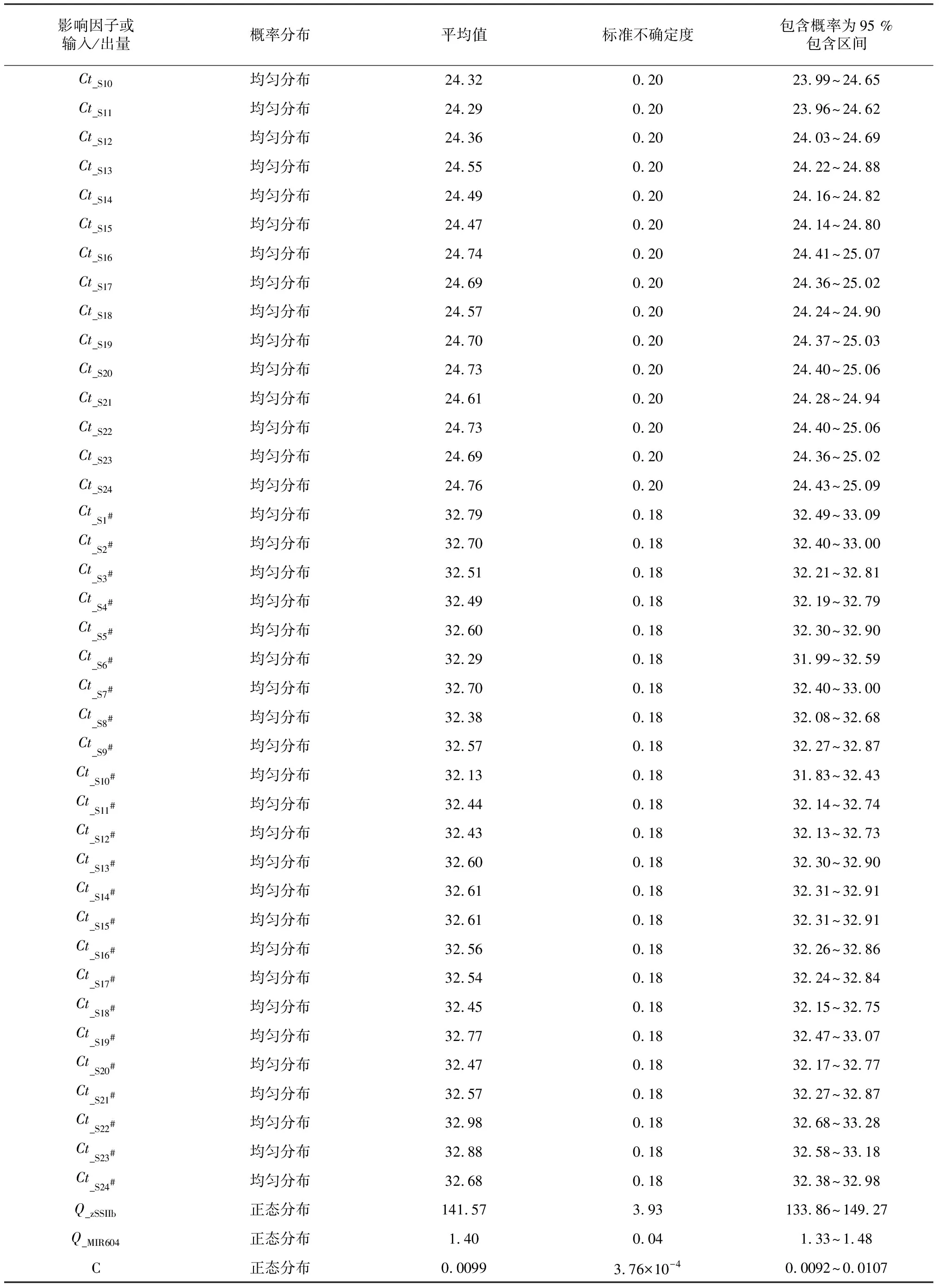
影响因子或输入/出量概率分布平均值标准不确定度包含概率为95 %包含区间Ct_S10均匀分布24.320.2023.99^24.65Ct_S11均匀分布24.290.2023.96^24.62Ct_S12均匀分布24.360.2024.03^24.69Ct_S13均匀分布24.550.2024.22^24.88Ct_S14均匀分布24.490.2024.16^24.82Ct_S15均匀分布24.470.2024.14^24.80Ct_S16均匀分布24.740.2024.41^25.07Ct_S17均匀分布24.690.2024.36^25.02Ct_S18均匀分布24.570.2024.24^24.90Ct_S19均匀分布24.700.2024.37^25.03Ct_S20均匀分布24.730.2024.40^25.06Ct_S21均匀分布24.610.2024.28^24.94Ct_S22均匀分布24.730.2024.40^25.06Ct_S23均匀分布24.690.2024.36^25.02Ct_S24均匀分布24.760.2024.43^25.09Ct_S1#均匀分布32.790.1832.49^33.09Ct_S2#均匀分布32.700.1832.40^33.00Ct_S3#均匀分布32.510.1832.21^32.81Ct_S4#均匀分布32.490.1832.19^32.79Ct_S5#均匀分布32.600.1832.30^32.90Ct_S6#均匀分布32.290.1831.99^32.59Ct_S7#均匀分布32.700.1832.40^33.00Ct_S8#均匀分布32.380.1832.08^32.68Ct_S9#均匀分布32.570.1832.27^32.87Ct_S10#均匀分布32.130.1831.83^32.43Ct_S11#均匀分布32.440.1832.14^32.74Ct_S12#均匀分布32.430.1832.13^32.73Ct_S13#均匀分布32.600.1832.30^32.90Ct_S14#均匀分布32.610.1832.31^32.91Ct_S15#均匀分布32.610.1832.31^32.91Ct_S16#均匀分布32.560.1832.26^32.86Ct_S17#均匀分布32.540.1832.24^32.84Ct_S18#均匀分布32.450.1832.15^32.75Ct_S19#均匀分布32.770.1832.47^33.07Ct_S20#均匀分布32.470.1832.17^32.77Ct_S21#均匀分布32.570.1832.27^32.87Ct_S22#均匀分布32.980.1832.68^33.28Ct_S23#均匀分布32.880.1832.58^33.18Ct_S24#均匀分布32.680.1832.38^32.98Q_zSSIIb正态分布141.573.93133.86^149.27Q_MIR604正态分布1.400.041.33^1.48C正态分布0.00993.76×10-40.0092^0.0107
注:a.Ct11~Ct53(统称Cti)表示zSSIIb基因测量中标准物质各浓度点的响应值;b.Ct11#~Ct53#(统称Cti#)表示59122测量中标准物质各浓度点的响应值;c.Ct_S1~Ct_S24(统称Ct_Si)表示试样24次重复测量zSSIIb基因获得的Ct值;d.Ct_S1#~Ct_S24#(统称Ct_Si#)表示试样24次重复测量59122片段获得的Ct值;e.Q_zSSIIb表示试样中zSSIIb的绝对含量;f.Q_MIR604表示试样中59122片段的绝对含量;g.C表示试样中59122片段的相对含量;h.mzSSIIb和mMIR604分别zSSIIb基因和MIR604片段的校准曲线的斜率;i.kzSSIIb和kMIR604分别zSSIIb基因和MIR604片段的校准曲线的截距。
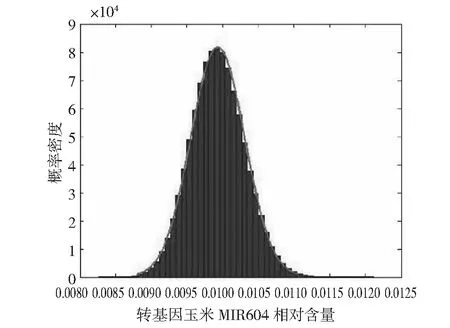
图1 MIR604玉米相对含量的概率分布Fig.1 The plot of probability distribution for relative content of MIR604 maize
2.2 PDF传递和输出
从2.1中各输入量的 PDF中抽取106个样本值,对每个样本向量计算相应的测量模型值。将106个模型值按照严格递增次序排序从而得到输出量的PDF、均值和包含区间。
3 讨 论
本研究首次使用MCM法实施各影响因素的“概率分布传播”来评定转基因成分的测量不确定度。本实验混合样品中转基因玉米MIR604的相对含量为0.99 %接近理论含量1 %,相对偏差为1 %,小于常丽娟等[10]报道结果(1.05 %)的相对偏差(5 %)。本次混合样品中转基因玉米MIR604的相对含量的标准不确定度为3.76×10-4,远远小于理化分析中不确定度的可接受水平(10 %)。程序计算获得的包含概率为95 %条件下的转基因玉米MIR604的相对含量包含区间为2.91 %~3.00 %,即转基因玉米MIR604的相对含量落在2.91 %~3.00 %非常窄的范围内的概率为95 %,充分显示本实验的测量质量非常高,测量数据非常可靠。
开展测量不确定度评定的目的是了解实验过程中影响测量结果的因素及其大小。转基因成分定量分析包括样品制备、DNA提取、PCR扩增、校准曲线拟合和转基因成分含量计算等步骤。每一个步骤都可能包含了一些不确定度成分,目前在样品制备、DNA提取阶段,由于尚未建立科学的测量模型(数学模型),所以很难对样品制备和DNA提取阶段的不确定度成分进行量化评定。本文仅对校准曲线制备、PCR扩增和含量计算等步骤产生的不确定度成分进行量化评估。从表4反应的各不确定度成分贡献大小依次(从大到小)为玉米内源基因zSSIIb绝对含量不确定度3.93,浓度点4.2 ng/μl稀释产生的不确定0.32(相同浓度点对应于外源片段Ct值的也有较大的不确定度0.21),测试样品内源基因Ct值不确定度0.20,测试样品外源片段Ct值不确定度0.18,浓度点21 ng/μl处外源片段Ct值的不确定0.17,其余不确定度都小于0.1,最终输出量转基因玉米MIR604的相对含量C的不确定度极小。本次评定的不确定大小反应出除最大的玉米内源基因zSSIIb绝对含量不确定度(因各种不可控因素影响一般不用绝对含量作为转基因含量)外,主要影响转基因成分检测质量的不确定度成分是校准曲线拟合过程中校准子制备产生的不确定度(0.32)。影响校准子制备的因素为液体转移量的准确性。因此,提高转基因成分定量分析质量的关键因素提高实验操作过程中的液体转移量的准确度。
4 结 论
经过基于MATLAB软件编写的程序代码模拟106次抽样、计算、传递各个变量的PDF,获得了混合样品中转基因玉米MIR604的相对含量C(最终输出量)的PDF和不确定度。测量过程的所有输入/出量的PDF、最佳估计值、标准不确定度和95 %包含概率的包含区间结果见表4。zSSIIb基因和MIR604片段的校准曲线分别为y=-3.38x+31.74和y=-3.30x+33.06。Q_zSSIIb和Q_MIR604绝对含量的平均值分别为141.57和1.40,标准不确定度分别为3.93和0.04,95 %包含概率的包含区间分别为133.86~149.27和1.33~1.48。MIR604片段相对含量C的平均值、标准不确定度和95 %包含概率的包含区间分别为0.99 %、3.76×10-4和0.92 %~1.07 %。图1为经MATLAB程序运行产生的呈正态分布的转基因玉米MIR604相对含量的概率密度。所有成分中,不确定度贡献最大的成分是是液体转移量的偏差。因此,液体转移量的准确度是提高转基因成分检测质量的关键。
参考文献:
[1]Zhang Fuli, Song Jun, Niu Bei, et al. An event-specific qualitative and real-time PCR detection of 98140 maize in mixed samples[J]. Food Control, 2015,57:1-8.
[2]常丽娟, 刘文娟, 张富丽, 等. 样品制备对转基因抗虫棉外源Bt蛋白定量检测的影响[J]. 棉花学报, 2015, 27(2):184-188.
[3]Rajesh K. Bhoge, Rashmi Chhabra, Gurinderjit Randhawa, et al. Event-specific analytical methods for six genetically modified maize events using visual and real-time loop-mediated isothermal amplification[J]. Food Control, 2015, 55:18-30.
[4]Jae-Hwan Kim, Saet-Byul Park, Hyo-Jeong Roh, et al. A simplified and accurate detection of the genetically modified wheat MON71800 with one calibrator plasmid[J]. Food Chemistry, 2015, 176:1-6.
[5]Holst-Jensen A, Berdal K G. The modular analytical procedure and validation approach and the units of measurement for genetically modified materials in foods and feeds[J]. J AOAC Int, 2004, 87(4):927-936.
[6]Charels D, Broeders S, Corbisier P, et al. Toward metrological traceability for DNA fragment ratios in GM quantification. 2. Systematic study of parameters influencing the quantitative determination of MON 810 corn by real-time PCR[J]. J Agric Food Chem, 2007, 55(9):3258-3267.
[7]Weighardt F. GMO quantification in processed food and feed[J]. Nat Biotecnol, 2007, 25:1213-1214.
[8]Murray S R, Butler R C, Timmerman-Vaughan G M. Quantitative real-time PCR assays to detect DNA degradation in soy-based food products[J]. J Sci Food Agric, 2009, 89(7):1137-1144.
[9]Zel J, Milavec M, Morisset D, et al. How to reliably test for GMOs [M]. America: Springer US, 2012. 95-95.
[10]常丽娟, 宋 君, 雷绍荣, 等. 转基因玉米MIR604 结构特异片段实时荧光定量检测方法的建立[J]. 江苏农业学报, 2015, 31(5):971-974.
[11]Arumuganathan K, Earle E. Nuclear DNA content of some important plant species[J]. Plant Molecular Biology Reporter, 1991, 9:208-218.
[12]JCGM. Evaluation of Measurement Data-Supplement 1 to the Guide to the Expression of Uncertainty in Measurement-Propagations of Distributions Using a Monte Carlo Method [M]. Switerland: JCGM, 2008.1-73.
[13]JCGM. Evaluation of Measurement Data-Supplement 2 to the Guide to the Expression of Uncertainty in Measurement-Extension to Any Number of Output Quantities [M]. Switerland: JCGM, 2011.1-68.
[14]Sega Michela, Pennecchi Francesca, Rinaldi Sarah, et al. Uncertainty evaluation for the quantification of low masses of benzo [a]pyrene: Comparison between the Law of Propagation of Uncertainty and the Monte Carlo method[J]. Analytica Chimica Acta, 2016, 920:10-17.

