利用de novo测序分析蚕豆瓣酱醅微生物多样性
周评平,李治华,董 玲,赵 驰,朱永清*(1. 四川省农业科学院,四川 成都 610066;.四川省农业科学院农产品加工研究所,四川 成都 610066)
【研究意义】郫县豆瓣酱是川菜重要的调味品,被誉为“川菜之魂”[1]。蚕豆是生产郫县豆瓣酱的重要原料之一,蚕豆瓣醅的质量好坏直接影响着郫县豆瓣的品质。【前人研究进展】目前国内外学者对黄豆酱醅的微生物多样性研究较多,但是对蚕豆酱醅的微生物多样性研究较少。【本研究切入点】长期以来,人们对发酵食品微生物菌落的研究主要采用传统的纯培养方法和免培养方法,免培养方法相对于传统的纯培养方法有很多优势,更能全面揭示微生物多样性和深入了解微生物群落及其功能[2]。随着测序费用的降低和生物信息学的发展,利用denovo测序方法研究微生物菌落多样性已成为一个重要手段。过去采用16S、ITS等扩增子测序分析的准确度有限,往往只能到属,而denovo测序可以弥补上述不足,可以准确到种甚至菌株水平[3]。【拟解决的关键问题】本研究以郫县豆瓣制作中蚕豆瓣醅为研究对象,采用denovo测序的结果分析其微生物多样性。不仅为后续深入研究其发酵过程微生物功能提供了技术支撑,而且对郫县豆瓣微生物安全性评价提供了有效的方法。

表1 质控统计结果Table 1 Statistic data for quality control
1 材料与方法
1.1 宏基因组DNA提取材料
传统方法生产的蚕豆瓣酱醅。样品采集后放在冰上立即运回实验室分析。
1.2 试验方法
1.2.1 基因组DNA提取 DNA提取采用方法参考李治华等[4],取10 g蚕豆瓣酱醅到90 mL生理盐水,充分振荡洗脱出样品中的微生物,用无菌金属网过滤,取滤液在4 ℃条件下10 000 r/min离心10 min,收集菌体沉淀,按照上海生工试剂盒(Ezup Column Soil DNA Purification Kit)使用说明提取菌体DNA。
1.2.2 宏基因组DNA检测 提取DNA在1 %琼脂糖凝胶电泳检测,2 μl上样,观察DNA质量。用Nanodrop2000超微量分光光度计检测DNA纯度和浓度 (NanoDrop Technologies, Del., U.S.A.)。
1.2.3 文库构建和测序 使用 NEBNext®UltraTMDNA Library Prep Kit for Illumina®试剂盒进行文库构建。
使用Illumina HiSeq平台进行测序,测序长度2×150 bp。测序得到的原始二进制basecalling数据经Illumina bcl2fastq软件转化为序列数据Raw data,结果以 fastq 文件格式存储。测序数据产量统计使用Illumina RTA软件进行图像识别和Basecalling,Illumina bcl2fastq 2.17根据每个样本的index信息进行demultiplexing并统计产出reads数和碱基测序质量(Q30),质控统计结果见表1。
M:1kb DNA ladder, 1为样本(2 μl),2位空白对照M:1kb DNA ladder, 1: Test sample(2 μl),2:Control sample图1 宏基因组DNA提取结果Fig.1 Metagenomic DNA extraction
1.2.4 数据分析 数据分析方法采用 Sunagawa 等2013年发表在《Nature methods》的mOTUs方法[3]和Senavirathne 等2015年发表在《Nature methods 》的Metaphlan2方法[5]
2 结果与分析
2.1 宏基因组DNA提取
电泳检测如图1,样品条带明亮。Nanodrop2000分析核酸浓度为73.5 ng/μl, 核酸质量为3.3 μg,A260/280=1.74,A260/230=0.47。电泳和Nanodrop2000分析显示样品满足建库要求,且总量可以满足2次或者2次以上建库需要的样品。
2.2 测序结果与数据分析
mOTUs方法发现共有12个种的微生物相对丰度大于1.00 %,Metaphlan2方法发现共有11个种的微生物相对丰度大于1.00 %,其中嗜盐四联球菌(Tetragenococcushalophilus)被2种方法同时确定为优势菌种, mOTUs方法显示其相对丰度为64.26 %,Metaphlan2分析其丰度为50.80 %, Metaphlan2进一步分析得出其菌株为NBRC 12172,是酱油和鱼酱的一个重要耐盐菌株[6]。破布子乳酸菌(Lactobacilluspobuzihii)仅仅被Metaphlan2分析到,而且其相对丰度为17.60 %,但没有被mOTUs方法分析出,具体菌株为E100301,首次是在台湾的传统发酵食品破布子中分离得到[7]。约氏不动杆菌(Acinetobacterjohnsonii)2种方法显示其丰度分别为4.43 %和4.17 %,但是具体菌株无法在NCBI数据库比配,暗示有可能是一株新的菌株。
在mOTUs相对丰度大于1.00 %的菌群里,Metaphlan2揭示到菌株水平的还有3株,分别为溶酪大球菌(Macrococcuscaseolyticus),菌株为JCSC5402,有报道该菌株对甲氧西林(methicillin)具有抗性,在进化上与金黄色葡萄球菌高度相关[8];肠杆菌属种暂定编号638(Enterobactersp. 638),因与目前该种下的菌株有明显区别,菌株暂定为638;类肠膜魏斯氏菌(Weissellaparamesenteroides),菌株为ATCC 33313,是一株典型的乳酸菌。除此以外,都不能被确定到菌株水平(unclassified),暗示蚕豆瓣酱醅里存在较多未被认识的微生物资源,对这些菌株的进一步分离有利于揭示蚕豆瓣酱醅的微生态基础。另外,融合魏斯氏乳酸菌(Weissellaconfusa),菌株为LBAE C39-2,仅仅被Metaphlan2检测到。
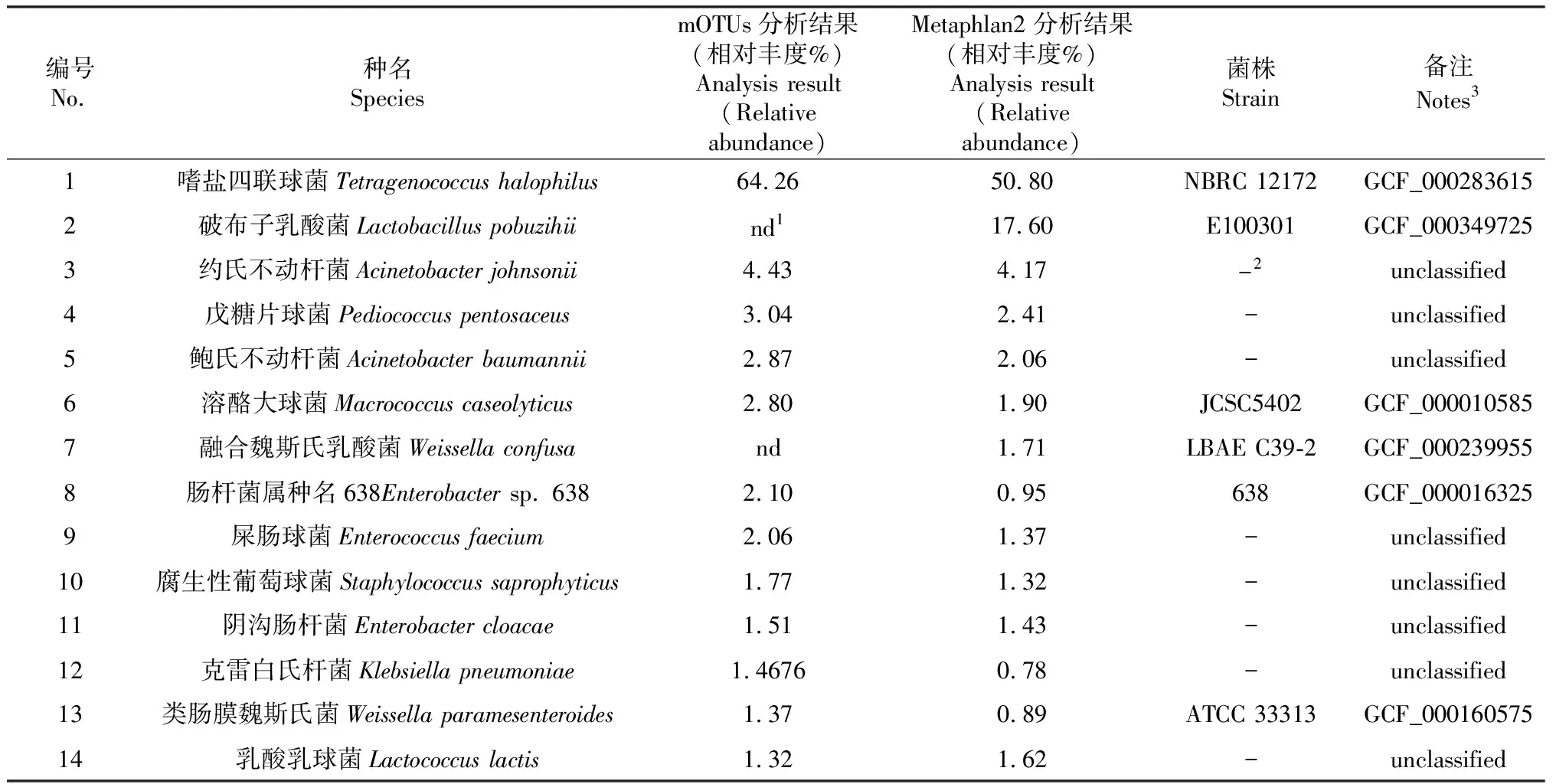
表2 mOTUs和 Metaphlan2分析蚕豆瓣酱醅微生物多样性Table 2 Analysis of microbial diversity in doubanjiang-meju using mOTUs and Metaphlan2 methods, respectively
注:“nd”表示没有被检测到;“-”表示 Metaphlan2方法在NCBI数据库里没有对应的菌株;对应菌株NCBI查询编号;“ unclassified”表示没有对应编号。
3 讨 论
3.1 de novo测序与扩增子测序
相比扩增子测序,denovo测序精确到了种水平,而且一次测序同时可以分析真菌和细菌,避免扩增子的2对引物,多次PCR过程产生的偏差[9]。
3.2 mOTU方法和Metaphlan2方法比较
2种方法结果较高程度的吻合,可以相互印证,Metaphlan2更进一步分析到了菌株级别。
3.3 蚕豆瓣酱醅的优势微生物
嗜盐四联球菌(T.halophilus)是蚕豆瓣酱醅的优势微生物,相对丰度占一半以上(mOTUs方法为64.26 %,Metaphlan2方法为50.80 %),Metaphlan2方法发现破布子乳酸菌(L.pobuzihii)也是优势菌株,其相对丰度为17.60 %,这与作者先前报道的一致[9]。另外,大量未分离到的微生物有待进一步研究。
4 结 论
denovo全基因组测序能分析到蚕豆瓣醅微生物中种或菌株的水平,研究结果不仅为后续深入研究提供了技术支撑,而且对郫县豆瓣的微生物安全性评价提供了有效的方法。
参考文献:
[1]李幼筠. “郫县豆瓣”剖析[J].中国酿造,2008,11:83-86.
[2]李 昂,崔 迪,王继华,等. 适用于第2代测序技术的宏基因组DNA 提取方法[J]. 哈尔滨工业大学学报,2012(6):20-23.
[3]Sunagawa S, Mende D R, Zeller G, et al. Metagenomic species profiling using universal phylogenetic marker genes[J]. Nature Methods, 2013, 10(12): 1196-1199.
[4]李治华,黄 驰,王自鹏,等. 发酵豆瓣酱微生物宏基因组DNA提取研究[J]. 西南农业学报,2013,26(6):2663-2665.
[5]Truong D T, Franzosa E A, Tickle T L, et al. MetaPhlAn2 for enhanced metagenomic taxonomic profiling[J]. Nature Methods, 2015, 12(10): 902-903.
[6]Satomi M, Furushita M, Oikawa H, et al. Diversity of plasmids encoding histidine decarboxylase gene inTetragenococcusspp. isolated from Japanese fish sauce[J]. International Journal of Food Microbiology, 2011, 148(1): 60-65.
[7]Chen Y S, Miyashita M, Suzuki K, et al.Lactobacilluspobuzihiisp. nov., isolated from pobuzihi (fermented cummingcordia)[J]. International Journal of Systematic and Evolutionary Microbiology, 2010, 60(8): 1914-1917.
[8]Baba T, Kuwahara-Arai K, Uchiyama I, et al. Complete genome sequence of Macrococcus caseolyticus strain JSCS5402, reflecting the ancestral genome of the human-pathogenic staphylococci[J]. Journal of Bacteriology, 2009, 191(4): 1180-1190.
[9]Li Z, Rui J, Li X, et al. Bacterial community succession and metabolite changes during doubanjiang-meju fermentation, a Chinese traditional fermented broad bean (ViciafabaL.) paste[J]. Food Chemistry, 2017, 218: 534-542.

