脑卒中lncRNA与mRNA表达谱分析
王玺,王凡,宁浩宇,白雪峰
(哈尔滨医科大学大庆校区医学信息学院,黑龙江 大庆 163319)
0 引言
脑卒中(Stroke)又称“中风”、“脑血管意外”(cerebralvascular accident,CVA)。是一种急性脑血管疾病,脑卒中是发达国家严重的长期神经功能障碍的主要原因,发病率约为0.25-4%,死亡率约为30%[1]。大量的证据表明,缺血性脑卒中伴随着一系列的神经事件,如缺氧、氧化应激和炎症反应[2],最终导致了神经细胞急性坏死、细胞凋亡和自我死亡[3]。随着分子生物技术的快速发展,人们发现lncRNA和mRNA的表达异常在脑卒中中起到了重要的作用,因此,我们通过研究来确定引起脑卒中新的发病机制,探索有效的预防和治疗脑卒中手段。本文就通过表达谱分析,对lncRNA和mRNA的交互作用与脑卒中关系进行研究,初步探讨了脑卒中发病机制中lncRNA和mRNA的作用,对lncRNA的功能进行注释分析。
(1) mRNA与脑卒中
随着近年来研究发现,mRNA在脑卒中疾病发生起着关键的角色。最新研究发现分化群(CD46)是心源性脑卒中的潜在生物标志物。并以CD46 mRNA为目标,这一结果通过荧光酶的报告分析进一步证实[4]。随着更多的研究发现大量的mRNA与脑卒中的发生有着十分密切的联系,如:Toll-Like受体3和干扰素βmRNA表达在缺血性卒中患者的外周血中增加,且预后良好[5]。通过以上的证明mRNA在脑卒中的各个阶段都扮演着重要的角色。
(2)LncRNA与脑卒中
LncRNA(long noncoding RNA,lncRNA)指 的 是长度超过200nt。目前研究,超过一半的lncRNAs在大脑中表达[6],它们调节了许多中枢神经系统[7]。研究证实,lncRNA H19通过驱动组蛋白脱乙酰酶,促进缺血性中风的神经炎症。这表明找到一种基于H19的诊断和治疗缺血性中风的可能方法[8]。目前,研究表明,通过调节特定的lncRNA,可以改变后缺血性神经元的死亡和/或再生/再生。这些研究对治疗疾病很有意义,通过调节这些目标,能够进一步防止中风后脑损伤[9]。随着科研的进步,发现越来越多的lncRNA与脑卒中的并发有着千丝万缕的关联,MEG3通过激活p53介导缺血性神经元死亡脑卒中,MEG3作为缺血细胞死亡启动子在生理和功能上与p53进行交互调解缺血性损伤[10]。对于以上的研究,我们不难发现lncRNA在脑卒中中有着非常重要的功能及作用,对lncRNA调节脑缺血性损伤的分子机制的研究提供潜在的新治疗靶点。
1 脑卒中表达谱分析
1.1 数据准备
从 NCBI上下载mRNA数据(GSE16561)和,并利用GPL570平台blast重注释得到lncRNA,数据包括39个病人样本和24个正常样本,差异基因的选取通过SAM算法获得,通过R代码进行数据处理,选取差异表达的lncRNA和mRNA(p-value<0.05),得到1038个lncRNAs和6085mRNAs,采取皮尔森系数方法(cor>0.8),构建lncRNAs-mRNAs互作网,并从HMDD数据库下载Protein-Protein互作对进行网络优化,最终形成lncRNAs-mRNAs互作网络。用Cytoscape插件对网络进行功能模块挖掘,获得5个功能显著性模块,最后通过David进行基因的功能注释,数据分析见图1。

图1 数据分析流程图
1.2 表达谱网络分析
对于形成lncRNA-mRNA互作网络,结点数2223个,4431条边。在网络中,度越大的基因连接的边越多,另外,还有一些结点虽然度小,但在结点间传输起到重要作用的桥梁作用,这些结点的关系,可能作为脑卒中的潜在的致病基因。结果见图2。
1.3 网络模块挖掘
利用Cytoscape中的JActivemodule插件进行模块挖掘。获得5个功能性显著模块(module1-module5),结果见图3。
1.4 Gene ontology(GO)分析
通过David在线平台完成对模块中重要结点进行功能分析,筛选出具有诊断意义的显示lncRNA-mRNA关系对。通过以上步骤进一步对脑卒中lncRNAs的功能进行分析。
2 结果

图2 网络度分布
2.1 差异表达lncRNAs、mRNAs
对表达谱数据进行处理,得到差异表达的 mRNAs、miRNAs, 取 表 达 谱 差 异 的 mRNAs、lncRNAs前5%做热图,结果见图4。
在mRNAs表达谱的热图中,module1中显著表达的有 VIPR1,MEOX2;module2中含有 DDHD2;在lnc表达谱的热图中,显著表达的有LINC00314。LINC00315脑卒中的发生都起到重大作用。
2.2 lncRNAs-mRNAs网络模块
通过模块挖掘来研究lncRNAs、mRNAs在癌症当中的共调控方式、以及基于局部策略来分析mRNAs的在脑卒中中的功能,见表1。

图3 lncRNAs-mRNAs模块图

图4 lncRNAs和mRNAs表达谱热图
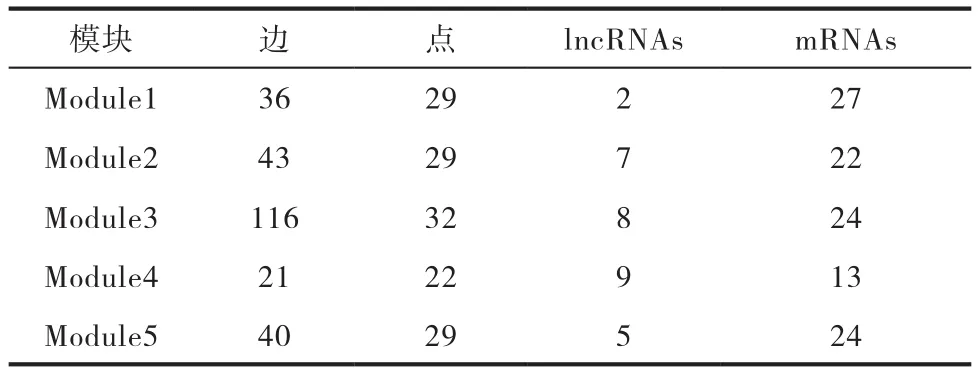
表1 各模块中边、点数量和所含lncRNAs,mRNAs数量
在五个模块中,PLAG1别名(PSA)同时存在于模块一和模块二中,可见它是一个十分重要的因子,已有研究证实PLAG1来自人类胚胎干细胞的神经前体细胞促进神经组织的完整性和脑卒中的行为表现。[11]在模块二中,SNHG12作为lncRNA,重复出现在模块五中。另外,一些研究通过RNA排序(RNA-seq)技术,体外模拟缺血性卒中条件,由此证明了SNHG12在大脑血管系统中起着重要的调控作用。模块三和模块四中,VIP是一种血管活性增强肽,广泛分布在中枢神经系统和各种脊椎动物的外围器官,可以保护受感染脑细胞不受损害,其机制是内源性神经保护物质的表达,证实了这些肽的极强的神经保护潜力,表明他可能为中风治疗中提供新的靶点。在模块五中,SNHG12,LINC01215,TMEM161B-AS1都是十分重要的lncRNA,它们相互连接多种基因,形成靶向关系,TERF2IP 通过调控血小板蛋白表达来间接影响脑卒中的发生[12]。
2.3 模块功能分析
对五个模块分别进行GO功能注释,结果见图5,我们可以看出有很多相同功能同时出现在多个模块中,例如:regulation of nucleobase-containing compound metabolic process,表示脑卒中疾病与代谢密切相关。 organic cyclic compound biosynthetic process注释到四个模块中,这一功能与有机环状化合物生物合成有关,可能与血管炎症相关,血管炎症是动脉粥样硬化性疾病的重要发病机制,研究表明促炎基因在缺血性脑卒中起到重要作用。
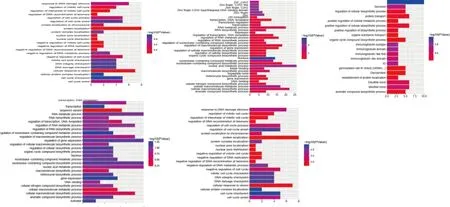
图5 模块功能注释
3 结语
为了明确脑卒中lncRNAs与mRNAs如何行使调控作用,通过对脑卒中芯片数据进行分析,得到lncRNA-mRNAs网络,对网络进行模块挖掘获得5个显著性模块,对模块进行分析,最后,通过本文的研究,发现lncRNAs与mRNAs协同作用的一些结果,为今后更深入的研究脑卒中提供指导和借鉴。众多研究已表明,lncRNA及mRNA通过多种途径参与脑卒中的发生及发展过程,并提供新的治疗靶点,对脑卒中的发生,治疗,预后都有必要的作用。例如,我们发现SNHG12这个重要的lncRNA,通过去除脑内皮组织抑制缺血性中风脑血管功能障碍,可与多个mRNA相互作用,对脑卒中产生影响,VIP这种血管肽也为脑卒中的治疗提供线索,与药物靶点,而且通过功能注释,生物体代谢存在密切的关系。由此看来,lncRNA与mRNA的相互作用对疾病发生和发展产生了不可忽视的影响,我们要对此进行进一步的研究与探索,为诊断和治疗脑卒中等提供新的方向。
[1] Siniscalchi A, Gallelli L, Malferrari G, et al. Cerebral stroke injury:the role of cytokines and brain inflammation[J]. Journal of basic and clinical physiology and pharmacology, 2014, 25(2): 131-137.
[2] Mehta S L, Manhas N, Raghubir R. Molecular targets in cerebral ischemia for developing novel the rapeutics[J]. Brain research reviews, 2007, 54(1): 34-66.
[3] Fayaz S M, Suvanish Kumar V S, Rajanikant G K. Necroptosis:who knew there were so many interesting ways to die?[J]. CNS& Neurological Disorders-Drug Targets (Formerly Current Drug Targets-CNS & Neurological Disorders), 2014, 13(1): 42-51.
[4] Tan J R, Tan K S, Yong F L, et al. MicroRNAs regulating cluster of differentiation 46 (CD46) in cardioembolic and non-cardioembolic stroke[J]. Plos One, 2017, 12(2):1-16.
[5] Işık M, Beydemir Ş, Yılmaz A, et al. Oxidative stress and mRNA expression of acetylcholinesterase in the leukocytes of ischemic patients[J]. Biomedicine & Pharmacotherapy, 2017, 87: 561-567..
[6] Mercer T R, Dinger M E, Sunkin S M, et al. Specific expression of non-coding RNAs in mouse brain[J]. 2008, 105(2):716-721.
[7] Qureshi IA, Mehler MF. Emerging roles of non-coding RNAs in brain evolution, development, plasticity and disease.[J]. Nature reviews. Neuroscience, 2012, 13(8):528-541.
[8] Wang J, Zhao H, Fan Z, et al. Long Noncoding RNA H19 Promotes Neuroinflammation in Ischemic Stroke by Driving Histone Deacetylase 1–Dependent M1 Microglial Polarization[J]. Stroke,2017, 48(8): 2211-2221.
[9] Vemuganti R. All’s well that transcribes well: non-coding RNAs and post-stroke brain damage[J]. Neurochemistry international,2013, 63(5): 438-449.
[10] Yan H, Yuan J, Gao L, et al. Long noncoding RNA MEG3 activation of p53 mediates ischemic neuronal death in stroke.[J].Neuroscience, 2016, 337:191-199.
[11] Zee R Y L, Ridker P M, Chasman D I. Genetic variants in eleven telomere-associated genes and the risk of incident cardio/cerebrovascular disease: The Women’s Genome Health Study[J].Clinica Chimica Acta, 2011, 412(1–2):199-202.
[12] Tourdot B E, Conaway S, Niisuke K, et al. Mechanism of Race-Dependent Platelet Activation Through the Protease-Activated Receptor-4 and Gq Signaling AxisSignificance[J]. Arteriosclerosis,thrombosis, and vascular biology, 2014, 34(12): 2644-2650.

