牛源粪肠球菌esp基因的表达及生物信息学分析
高 昇 ,杨宇泽 ,马 倩 ,樊 杰
(1.甘肃农业大学动物医学院,兰州 730070;2.北京市畜牧总站,北京 100101)
奶牛乳房炎是奶牛养殖业中最常见而且危害最为严重的三大疾病之一,也是对乳品加工业危害最大的疾病。奶牛乳房炎多发生于产后期,其中最主要的因素是病原微生物感染[1]。临床实验研究已发现,粪肠球菌(Enterococcus faecalis)、屎肠球菌(Enterococcus faecium)等环境致病菌在奶牛乳房炎发生中作用明显[2]。
esp(Enterococcus surface protein,esp)是编码粪肠球菌表面蛋白的基因,GenBank序列号为AF034779,全长5 791 bp。esp结构独特,核心区域由多个碱基序列重复组合而成,其所编码的表面蛋白是肠球菌众多致病因素之一,其在肠球菌对宿主细胞的黏附定植和逃避宿主免疫清除方面起到一定作用[3]。肠球菌表面蛋白是肠球菌表面分子量最大的一种蛋白质,属于黏附素的一种,与肠球菌在内置导管表面形成生物膜有关,在肠球菌感染时有利于延长致病菌在感染动物体内的停留时间,但本身并不损伤宿主细胞[4],肠球菌表面蛋白因核心氨基酸重复次数不同而大小不一,在感染初期大分子肠球菌表面蛋白有利于肠球菌对宿主细胞的黏附,定植完成后,小分子肠球菌表面蛋白有利于肠球菌逃避宿主免疫系统的清除作用[5]。
传统的抗生素治疗常常因为耐药性而疗效不佳,同时造成严重的乳中抗生素残留而危害人类健康。因此,近些年来人们开始研究和开发有效的免疫制剂来防制乳房炎。研究表明,牛源粪肠球菌(Bovine Enterococcus faecalis)溶血素基因(cylA)、表面蛋白基因(esp)、胶原蛋白黏附素基因(ace)、心内膜炎有关抗原基因(efaA)等在奶牛乳房炎的发展过程中具有重要作用,被认为是疫苗发展的良好靶位点[6-7]。由于esp较大,很难在体外完全表达,笔者等通过基因克隆得到esp的部分CDS,并将esp与载体相连接构建原核表达载体,并分析esp及其编码蛋白的信息,为奶牛乳房炎的防治提供参考。
1 材料与方法
1.1 材料
1.1.1 菌株与质粒 牛源粪肠球菌、质粒pBluescriptⅡKS(+)、克隆菌株E.coli DH5α和表达菌株E.coli BL21为甘肃农业大学动物医学院保藏。
1.1.2 培养基 培养大肠埃希氏菌DH5α、BL21的培养基为LB培养基,2×YT培养基用于重组菌的诱导。
1.1.3 主要试剂 限制性内切酶BamHⅠ、XhoⅠ购自TaKaRa公司;pfu酶和T4 DNA Ligase购于Thermo fisher公司;镍柱购自GE公司;异丙基-β-D-硫代半乳糖苷(Isopropyl-β-D-1-thiogalactopyranoside,IPTG)、卡那霉素购自北京全式金生物技术有限公司;质粒DNA小提试剂盒、胶回收试剂盒购自于天根生化科技有限公司。
1.2 方法
1.2.1 引物设计 参照GenBank中粪肠球菌esp序列(序列号AF034779)设计引物,并送金唯智科技有限公司合成。引物序列如下:
F:5'-CCGCTCGAG AAATCGTTTCTCCAGGTTTTGA-3'(下划线为XhoⅠ酶切位点);R:5'-CGGGATCCTTGAGGTTTATTCGGTGCTTTT-3'(下划线为BamHⅠ酶切位点)。
1.2.2 细菌培养 粪肠球菌在LB培养基中过夜培养,收集菌体后采用CTAB法提取基因组DNA。
1.2.3 esp基因扩增 以粪肠球菌基因组为模板应用pfu酶进行扩增。反应条件:94℃预变性8 min,94℃30 s,48℃ 30 s,72 ℃ 3 min,共 35 个循环;72℃再延伸 10 min;PCR产物进行1%琼脂糖凝胶电泳检测并用琼脂糖凝胶纯化回收试剂盒纯化,将回收后PCR产物和用BamHⅠ、XhoⅠ双酶切过的pBluescriptⅡKS(+)质粒通过T4 DNA Ligase进行连接,转化至DH5α感受态细胞,提取质粒,经PCR和质粒双酶切鉴定,筛选阳性克隆命名为pBluescriptⅡKS::esp。
1.2.4 原核表达载体的构建 将测序正确的pBluescriptⅡKS::esp和pET-28a同时经BamHⅠ和XhoⅠ酶切,酶切后的目的片段与pET-28a通过T4 DNA Ligase进行连接,并将连接产物转化至DH5α接感受态细胞,阳性重组质粒命名为pET-28a::esp,并送金唯智生物科技有限公司测序。
1.2.5 重组蛋白的表达及纯化 将测序正确的重组质粒pET-28::esp转化至大肠杆菌BL21(DE3)感受态细胞中,挑取单菌落过夜培养后按1∶100比例转接入100 mL 2×YT(Kan+)培养基中,37℃培养2.5h至菌液OD值(600nm)达到0.4~0.6,加入终浓度为1 mmol/L的IPTG,在26℃诱导表达12 h,7 000 r/min离心10 min,收集菌体,PBS洗涤2次后超声波破碎30 min(40%,工作5 s,间歇5 s),7 000 r/min离心10 min。收集上清,用GE公司的Ni-NTA柱进行蛋白纯化,以诱导前的重组菌株为对照进行SDS-PAGE电泳,分析重组蛋白表达情况。
1.2.6 esp序列及其编码蛋白序列的生物信息学分析选取esp序列,利用在线软件Prot-Param(http://www.expasy.org/tools)分析该基因序列编码氨基酸序列组成与理化性质;应用软件ProtScale(http://www.espasy.org/cgi-bin/protscale.pl)分析其氨基酸残基疏水性;应用软件 TMHMM Server v.2.0(http://www.cbs.dtu.dk/services/TMHMM)预测跨膜区;应用软件NetPhos 2.0 Serve(http://www.cbs,dtu.dk/services/NetPhos/)预测磷酸化位点;利用软件 NetNGlyc 1.0(http://www.cbs.dtu.dk/Services/NetNGlyc/)和 NetOGlyc 3.1 Serve(http://www.cbs.dtu.dk/services/NetOGlyc/)预测糖基化位点;利用软件SignalP3.0(http://www.Cbs.dtu.dk/services/SignalP-3.0/)预测蛋白信号肽;利用软件PORTER(http://distill.ucd.ie/porter)预测蛋白质二级结构;利用EMBOSS中的antigenic(http://emboss.bioinfo-rmatics.nl/cgi-bin/emboss/anigenic)及在线软件SYFPEITHI和ProPred预测抗原表位。
2 结果与分析
2.1 esp基因的克隆和表达载体的构建
通过PCR方法克隆得到了粪肠球菌的部分esp,将得到的部分esp连接到pET-28a,转化大肠杆菌DH5α,构建的重组质粒pET-28a::esp经BamHⅠ和XhoⅠ酶切验证无误后转化到大肠杆菌BL21,PCR结果和质粒双酶切验证图谱表明成功构建了表达载体(图1)。最后对重组质粒pET-28a::esp进行测序,测序结果表明,选取的部分esp大小为3 795 bp,所得esp序列与GenBank登录号(AF034779)序列比对同源性为68%。
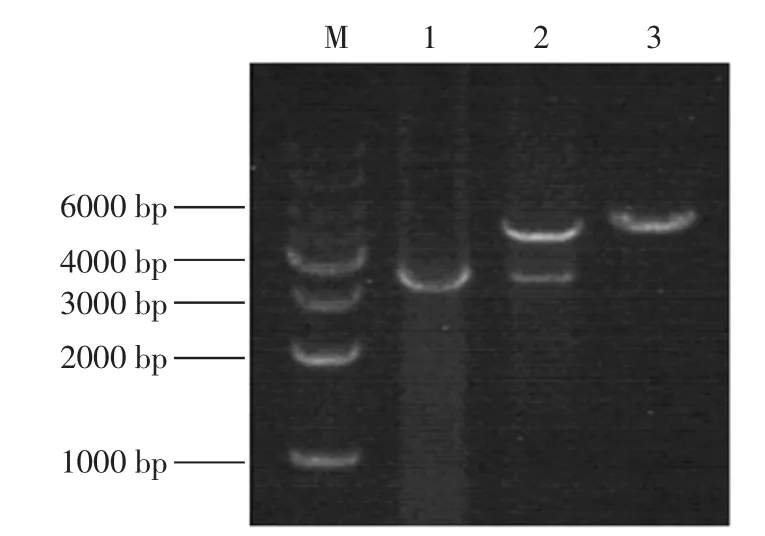
图1 目的基因的PCR扩增产物及重组质粒双酶切
2.2 esp诱导表达及SDS-PAGE电泳
pET-28a::esp重组菌株经IPTG诱导,超声破碎后进行纯化,得到esp,进行SDS-PAGE电泳检测。SDSPAGE分析表明,诱导前无特异条带,诱导后出现特异条带,纯化后在110 ku附近出现单一条带。详见图2。
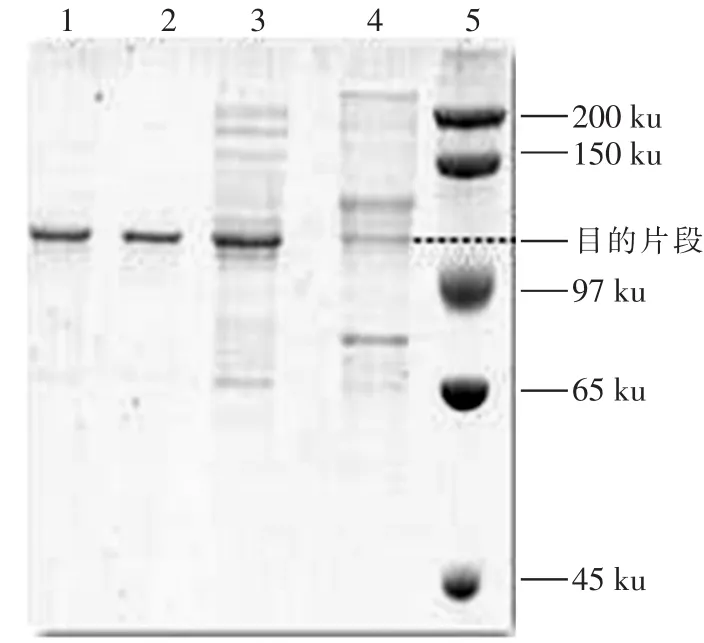
图2 表达产物的SDS-PAGE分析
2.3 esp序列及其编码蛋白序列的生物信息学分析
2.3.1 esp序列及编码蛋白序列的分子特征 通过DNAMAN和在线软件EXPASY分析得到选取的部分esp,长度为 3 795 bp,其中 A 碱基 1 475个(38.87%)、T碱基 816个(21.50%)、G 碱基 844个(22.24%)、C碱基660个(17.39%),G+C占39.63%,A+T占60.37%。esp编码1 207个氨基酸,蛋白分子质量约110 ku,等电点为9.67,总共包括19 799个原子,分子式为C6132H10027N1741O1848S51。在组成esp蛋白的20种氨基酸中,谷氨酸(Glu)所占比例最高,达到11.4%,而胱氨酸(Cys)所占的比例最低,为0.7%。
2.3.2 esp编码蛋白质的疏水性分析 蛋白质的疏水性在其三级结构的形成和稳定中起重要作用。利用软件ProtScale对esp编码氨基酸残基进行疏水性分析,结果表明,其疏水性最大值为3.500,最小值为-3.267,脂肪系数为82.34,总平均亲水性为-0.713,不稳定指数为43.99,表明为亲水蛋白。潜在的亲水区为8~54、71~79、92~114、186~350、361~502、515~536、581~611、623~813、819~836、843~977、983~1 113、1 119~1 192、1 200~1 203位氨基酸,其中623~813位氨基酸亲水性强。疏水区分别位于 1~7、55~70、80~91、115~185、351~360、503~514、537~580、612~622、814~818、837~842、978~982、1 114~1 118、1 193~1 199 位氨基酸,其中 55~70位氨基酸疏水性最强。
2.3.3 esp基因编码蛋白跨膜区、信号肽、糖基化位点、磷酸化位点的预测 经预测,esp蛋白的跨膜结构可能存在区域为1~200位氨基酸,不存在信号肽,有6个肽糖基化位点分别位于 504、749、759、823、905、987。细胞内参与磷酸化的氨基酸残基主要有Thr、Tyr、Ser。预测结果发现,当潜在磷酸位点阈值取0.5时,esp蛋白潜在的磷酸位点有108个(52个Thr位点,分别位于45、117、347、646、849、950、999、1 109 等位氨基酸;44 个Ser位 点 ,分 别 位 于 57、158、200、440、540、654、756、1 196等位氨基酸;12个Tyr位点,分别位于803、861、942、1 049等位氨基酸)。
2.3.4 esp基因编码蛋白二级结构预测 应用Predict Protein工具分析及PORTER软件预测esp的二级结构,发现esp二级结构以α-螺旋、无规则卷曲和β-折叠为主,属于混合型蛋白(图3)。同时对esp在表皮葡萄球菌、屎肠球菌中编码蛋白二级结构氨基酸残基进行比较发现,esp在粪肠球菌中的无规则卷曲和β-折叠比在表皮葡萄球菌略多一些(表1)。应用SWISS-M ODEL/SWISS-PdbView等工具,按照同源建模法构建出esp基因在表皮葡萄球菌、屎肠球菌中所编码蛋白的三级结构具有较大的不同(图4)。

图3 esp基因编码二级结构的预测
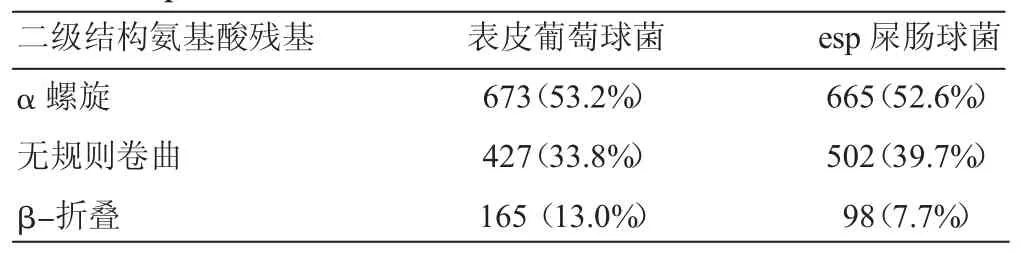
表1 esp在屎肠球菌、表皮葡萄球菌所编码蛋白的二级结构比较
2.3.5 esp的抗原表位分析 经过EMBOSS中的antigenic网站在线预测esp的B淋巴细胞抗原表位。预测结果如图5所示,esp的B淋巴细胞抗原表位分别位于1~32、62~72、78~114、144~154、162~196、226~236、242~316、324~332、336~398、402~410、418~424 位氨基酸。
分别用SYFPEITHI和ProPred对esp编码的T淋巴细胞抗原表位进行预测分析。其中应用SYFPEITHI分析时,一般认为,得分在前2%的多肽成为抗原肽的可能性超过80%,esp有1 207个氨基酸,所以得分取靠前的24个抗原多肽作为优势抗原(表2)。ProPred预测结果见表3。结果显示,VLMQTLVLL、LILLTLLIV和LTLLIVRKV在两种软件中预测结果均在前24位氨基酸,所以确定 VLMQTLVLL、LILLTLLIV和 LTLLIVRKV为esp的最佳MCHⅠ类分子抗原表位。
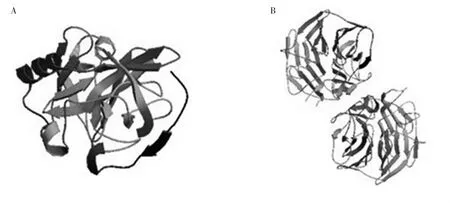
图4 esp编码蛋白三级结构预测
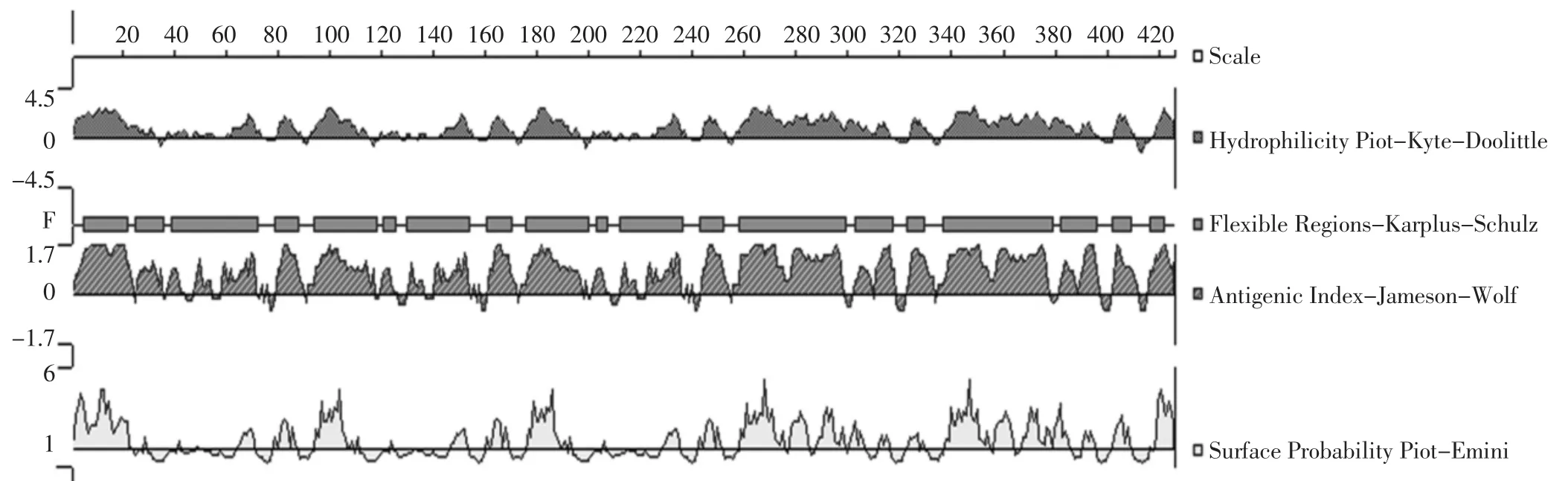
图5 esp编码蛋白B淋巴细胞的抗原表位预测
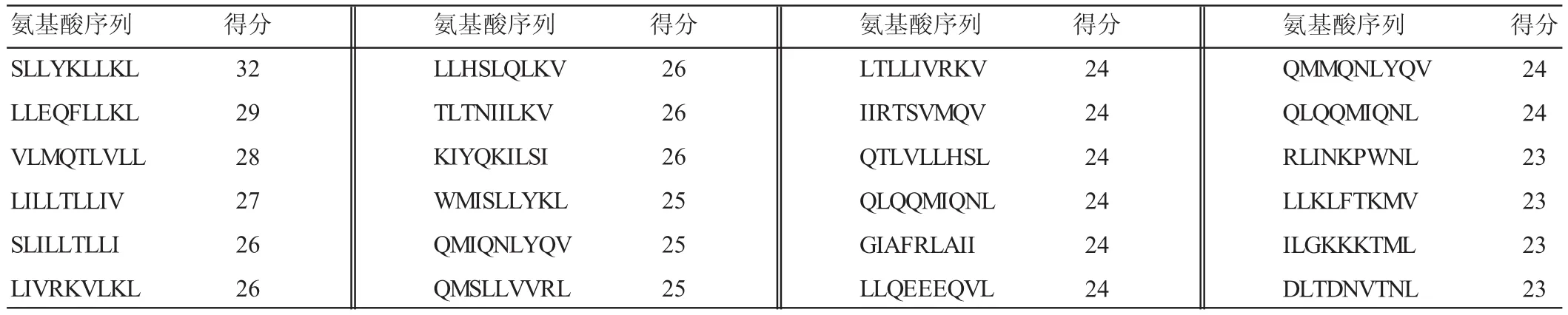
表2 esp编码蛋白T淋巴细胞的抗原表位预测(SYFPEITHI软件)
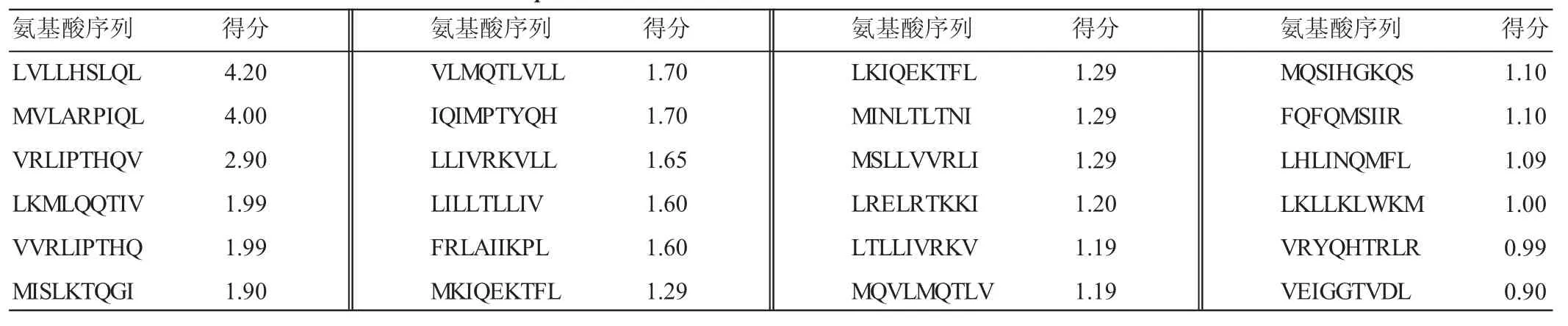
表3 esp编码蛋白T淋巴细胞的抗原表位预测(ProPred软件)
3 讨论
肠球菌表面蛋白是分子量最大的一种肠球菌表面蛋白质,属于黏附素。研究表明,esp位于肠球菌致病岛的基因簇里,是肠球菌主要的毒力因子之一,主要与肠球菌对宿主细胞的定值、黏附、产生生物被膜及宿主免疫清除逃逸有关。esp基因结构独特,核心区域由多个碱基序列重复组合而成,也被称为A重复,A重复的3'端是相邻的2个B重复序列,B重复序列紧接着是8个C重复序列,第8个C重复序列位与其他的C重复序列的前30个核苷酸相同。C末端的156-残基编码序列可编码一个跨膜形疏水区及一个有较小变异的LPXTGX基因序列[8]。研究表明,成熟的esp存在特殊的重复结构,串联重复片段构成核心区,并且拥有大量高度保守的重复片段[9]。本研究通过生物信息学对部分esp编码蛋白的基本理化性质进行了分析,结果显示选取的部分esp长度为3 795 bp,编码1 207个氨基酸,其中谷氨酸(Glu)所占比例最高,达到11.4%,而胱氨酸(Cys)所占的比例最低,为0.7%。esp为酸性亲水性蛋白,有利于后续的异源性表达,其跨膜结构可能存在区域为1~200位氨基酸,说明该蛋白可能是表面结构蛋白,不存在信号肽,说明esp为非分泌蛋白。
蛋白质二级结构不仅是一级结构和三级结构之间的枢纽,也是预测三级结构的关键点,经过预测esp蛋白二级结构以α-螺旋、无规则卷曲和β-折叠为主,属于混合型蛋白[10],通常,转角和无规则卷曲区域决定蛋白的功能,可能与抗原表位有关[11]。较多的α-螺旋的氨基酸残基的存在有利于蛋白质结构的稳定,β-折叠中存在脯氨酸,增加了蛋白质的刚性,无规则卷曲氨基酸残基的存在位点则与形成抗原表位有关[12]。转角及无规则卷曲等二级结构比较松散,易于扭曲、盘旋并展示在蛋白表面,因此,该区域常含B细胞优势抗原表位[13]。本研究结果表明,esp抗原指数较高,具有亲水性,esp抗原表位应该是优势抗原表位所在。
[1]李方华,冯士彬,李孝文.奶牛乳房炎的发病机理及防治研究进展[J].中国动物保健,2008(8):40-42.
[2]Patrick K D,Usuf K,Hannington B,et al.Prevalence and antimicrobial susceptibility patterns of bacteria from milkmen and cows with clinical mastitis in and around Kampala,Uganda [J].PLoS One,2013,8(5):e63413.
[3]Coque,Teresa M,Willems,et al.High occurrence of esp among ampicillin-resistant and vancomycin-susceptible Enterococcus faecium clones from hospitalized patients[J].The Journal of Antimicrobial Chemotherapy,2002,50(6):1035.
[4]马立艳,许淑珍,马纪平.肠球菌致病机制的研究进展[J].中华医院感染学杂志,2005,15(3):356-360.
[5]强华,林建银,蒋明森,等.肠球菌溶血素与其致病力的关系[J].中国人兽共患病学报,2001,17(6):67-80.
[6]狄婷婷,高原.粪肠球菌主要毒力因子研究进展[J].中国病原生物学杂志,2012(3):231-234.
[7]De V S,Barkema H W,Stryhn H,et al.Impact of early lactation somatic cell count in heifers on milk yield over the first lactation[J].Journal of Dairy Science,2005,88(3):938.
[8]Shankar V,Baghdayan A S,Huycke MM,et al.Infection-derived Enterococcus faecalis strains are enriched in esp,a gene encoding a novel surface protein[J].Infection&Immunity,1999,67(1):193.
[9]Toledo-Arana A,Valle J,Solano C,et al.The enterococcal surface protein,Esp,is involved in Enterococcus faecalis biofilm formation[J].Applied&Environmental Microbiology,2001,67(10):4538.
[10]Skolnick J,Fetrow J S.From genes to protein structure and function:novel applications of computational approaches in the genomic era[J].Trends in Biotechnology,2000,18(1):34.
[11]宋建勋,朱锡华,陈克敏.人Fas抗原表位预测[J].免疫学杂志,1999,15(1):14-16.
[12]Scandurra R,Consalvi V,Chiaraluce R,et al.Protein thermostability in extremophiles[J].Biochimie,1998,80(11):933-941.
[13]Blythe M J,Flower D R.Benchmarking B cell epitope prediction:Underperformance of existing methods[J].Protein Science A Publication of the Protein Society,2005,14(1):246.

