CHB患者PegIFN治疗前差异表达microRNAs的检测及意义
杨艳琳 刘明 邓樱 郭艳 张绪清 向德栋 蒋黎 游忠岚 伍艺 李茂仕 毛青
400038 重庆,陆军军医大学(第三军医大学)西南医院全军感染病研究所感染病研究重庆市重点实验室
乙型病毒性肝炎(hepatitis B virus, HBV)呈世界性流行,据估计全球HBsAg携带者有2.4亿人,每年有近1百万人死于HBV相关性疾病[1-2]。早期获得HBsAg清除可降低患者发生肝硬化、肝癌的风险[3]。但是,临床上HBsAg清除率很低,干扰素和核苷(酸)类似物(nucleos(t)ide analogue, NAs)治疗的HBsAg清除率分别为3%~7%/年、0%~3%/年[1]。研究发现干扰素治疗时通过优化CHB患者的基线特征可提高HBsAg清除率,如OSST研究和NEW SWITCH研究发现NAs经治的CHB患者基线HBsAg <1500 IU/ml时序贯接受PegIFN治疗后HBsAg清除率分别为22.2%、28.5%[4-5]。因此研究HBsAg清除的影响因素对于改进CHB治疗具有重要意义。MicroRNA(miRNA)作为生物体内源性基因表达调控分子(19~23nt),等研究报道,其可参与HBV治疗的免疫应答、影响病毒复制如miR-199 a-3p和miR-210分别靶向HBsAg和pre-S1编码区从而降低HBsAg的表达水平[6]。由于细胞中miRNA表达水平相对于血浆、血清较稳定和肝组织标本难以获取等原因,不容易受其他因素的影响,故本研究选择外周血单个核细胞(peripheral blood mononuclear cell, PBMC)中的差异miRNAs作为研究对象。
1 对象与方法
1.1研究对象从2015年4月30日开始从重庆西南医院门诊招募符合入组要求的病例,随访观察PegIFN应答结局。研究对象入组标准为:①年龄18~65岁;②HBsAg阳性至少6个月,基线HBsAg水平 < 1500 IU/ml,HBeAg(-),HBV DNA < 1000IU/ml;③ALT ≤ 2ULN(正常值上限,我们医院为42IU/ml);④干扰素初治,核苷(酸)经治或者未治疗。排除标准:①合并感染HAV、HCV、HDV、HEV、HIV;②有酒精性肝病、非酒精性脂肪肝、药物性肝炎、自身免疫性肝病等其他慢性肝病的证据;③近6个月内接受免疫调节药物治疗;④有生育计划。所有患者入组前均签署知情同意书,该研究已通过西南医院伦理委员会批准,并在中国临床试验注册中心注册(注册号ChiCTR-ROC-16008735)。
1.2治疗方案招募的患者接受PegIFNα-2 a治疗(180 μg/w),疗程至少48周,单药或者联合NAs治疗,最终疗程及药物剂量由门诊医师根据患者具体病情决定,但不超过96周。若干扰素治疗后HBsAg无明显下降甚至上升,可提前终止治疗,停药后每3个月进行复查随访。
1.3实验室检测方法
1.3.1 临床指标检测:乙肝标志物用Abbott公司配套试剂盒进行检测,HBsAg检测范围为0.05 IU/ml~> 250 IU/ml,HBsAg < 0.05 IU/ml认为是HBsAg清除。血清HBV DNA运用上海复星长征医学科学有限公司的试剂盒进行检测,HBV DNA检测下限为<500 IU/ml;全自动生化仪(7600-020,HITACHI公司)测定血清ALT、TBIL、ALB水平;全自动五分类血液分析仪(XT-2000i,Sysmex公司)测定WBC、PLT。
1.3.2 总RNA提取:外周静脉血PBMC用淋巴细胞分离液(LymphoprepTM)分离,胎牛血清重悬(HyCloneTM, Cat.N:SV30087.03)储存在液氮中备用。PBMC的总RNA根据Qiagen试剂盒说明书提取(Cat No/ID: 217004),RNA的浓度及质量用Nano Drop 2 000 C分光光度计进行检测并进行凝胶电泳。
1.3.3 miRNAs芯片检测:芯片检测委托上海康成生物公司完成,芯片根据miRCURYTMHy3TM/Hy5TMPower labeling(Exiqon, Vedbaek, Denmark, Cat#208032-A)和miRCURYTM LNA Array(v.19.0)(Exiqon)试剂盒说明书进行标记和杂交。图像扫描导入GenePix Pro 6.0(Axon)软件进行数据提取,根据以下标准筛选两组间差异表达的miRNAs:差异倍数>1.5,P< 0.05。
1.3.4 miRNAs的实时荧光定量:根据miRNA在两组间的表达差异程度,选择差异倍数 > 3,P< 0.05的前7条miRNAs进行实时荧光PCR定量验证,其中3条上调,4条下调。实验过程按照microRNA通用定量试剂盒说明书进行(Cat#GMRS-001,吉玛基因,上海)。荧光定量上游引物序列见表1,差异miRNAs的表达水平用ΔCT方法表示(ΔCT=CT目标miRNA - CT内参U6)。
1.3.5 差异miRNAs的靶向基因预测:使用TargetScan 7.1和miRDB V5.0在线数据库预测差异表达的miRNAs靶基因,对预测到的所有靶基因根据以下原则进行排序筛选:TargetScan 7.1数据库靶基因cumulativeWeightedContext++Score ≤-0.3,miRDB V5.0数据库靶基因targetScore ≥ 70。并对预测到的靶向基因进行生物信息学分析,即Pathway分析,预测其与HBsAg清除潜在的联系。
表 1 miRNAs引物序列
Tab.1Primers sequence of miRNAs

表2 研究队列的基线特征
Tab.2Baseline characteristic of study cohort
基线特征筛选队列(n=20)验证队列(n=47)清除组未清除组P清除组未清除组P例数1010—1730—男,例数(%)9(90)7(70)058215(88)24(80)0692a年龄,岁29±89464±6800001∗∗∗39±77387±930921既往用NAs史——————是67110220305否43—78—HBVDNA,IU/ml——————低于检测于下限(<500)79058216230228500~100031—17—b基线HBsAg,IU/ml18609(2931⁃72435)18428(6275⁃53669)111654(3336⁃19526)50436(15635⁃87549)001<500,No.(%)770549$1415009$500~1000,No.(%)23—210—1000~1500,No.(%)10—15—a基线ALT,IU/ml364±17833069±9903883151±1403721±30650474
$a`=(2*a)/[k*(k-1)+1]=0.014,k=3,a=0.05; i*P< 0.05,**P< 0.01,***P< 0.001
a数据用均值±标准差表示;b数据用中位数表示(四分位间距);ALT谷丙转氨酶;既往用药史,表示干扰素治疗前是否用核苷(酸)类似物治疗,NAs包括所有常规抗病毒药物:替诺福韦酯,恩替卡韦,拉米夫定,替比夫定,阿德福韦酯等,可以单药或者联合用药,平均用药时间是64.60周 (33.3-82.97)(中位数-四分位间距)
$a`=(2*a)/[k*(k-1)+1]=0.014,k=3,a=0.05;*P< 0.05,**P< 0.01,***P< 0.001;
Notes.adata are presented as mean±SD;bdata are presented as median (IQR range); ALT denotes alanine aminotransferase; WBC, white blood count; History of NAs means whether they have used nucleos(t)ide analogs (NAs) before PegIFN treatment, the NAs include all kinds of conventional medicines: TDF, ETV, LAM, LdT, ADV. They were used by means of monotherapy or combined treatment. The median duration time of NAs was 64.60 w (33.3-82.97) (median±IQR)
1.4统计学方法所有统计分析运用SPSS 23.0软件进行分析。两组之间的差异比较用卡方检验或者Fisher精确概率检验方法,独立样本t检验,曼-惠特尼U检验。双侧检验P< 0.05表示两组间差异有统计学意义。
2 结果
2.1基线特征本研究病例数共67例,分成两个队列:筛选队列(n=20例)和验证队列(n=47例)。停药时HBsAg水平低于0.05 IU/ml认为是HBsAg清除,在表面抗原清除组中,有21例获得HBsAg血清学转换,各基线参数在两队列中差异均无统计学意义,如性别、基线HBV DNA、ALT,但在筛选队列中年龄差异具有统计学意义,基线特征详见表2。
2.2芯片结果筛选队列中10例HBsAg清除与10例未清除患者PBMC抽提的总RNA与miRNA芯片杂交后,获得差异miRNAs芯片图像。结果显示两组间存在明显miRNAs差异表达(差异倍数 > 1.5,P< 0.05):共417条miRNAs差异表达,其中在清除组上调的有342条,如miR-196b-3p, miR-548ah-5p, miR-5583, miR-122;下调的有75条,如miR-3960, miR-126-3p, miR-146 a, miR-199 a。
2.3差异miRNAs的验证对选择的7条差异显著的miRNAs在验证队列中进行实时荧光定量验证,验证队列由17例HBsAg清除和30例HBsAg未清除病例组成。结果显示有4条miRNAs(miR-3960, miR-126-3p, miR-23 a-3p, miR-335-5p)在验证队列中有统计学差异(P< 0.05),四条均在HBsAg清除组下调,其余3条miR-196b-3p, miR-548ah-5p, miR-5583差异无统计学意义(P> 0.05),各条差异miRNAs相对表达水平见图1。
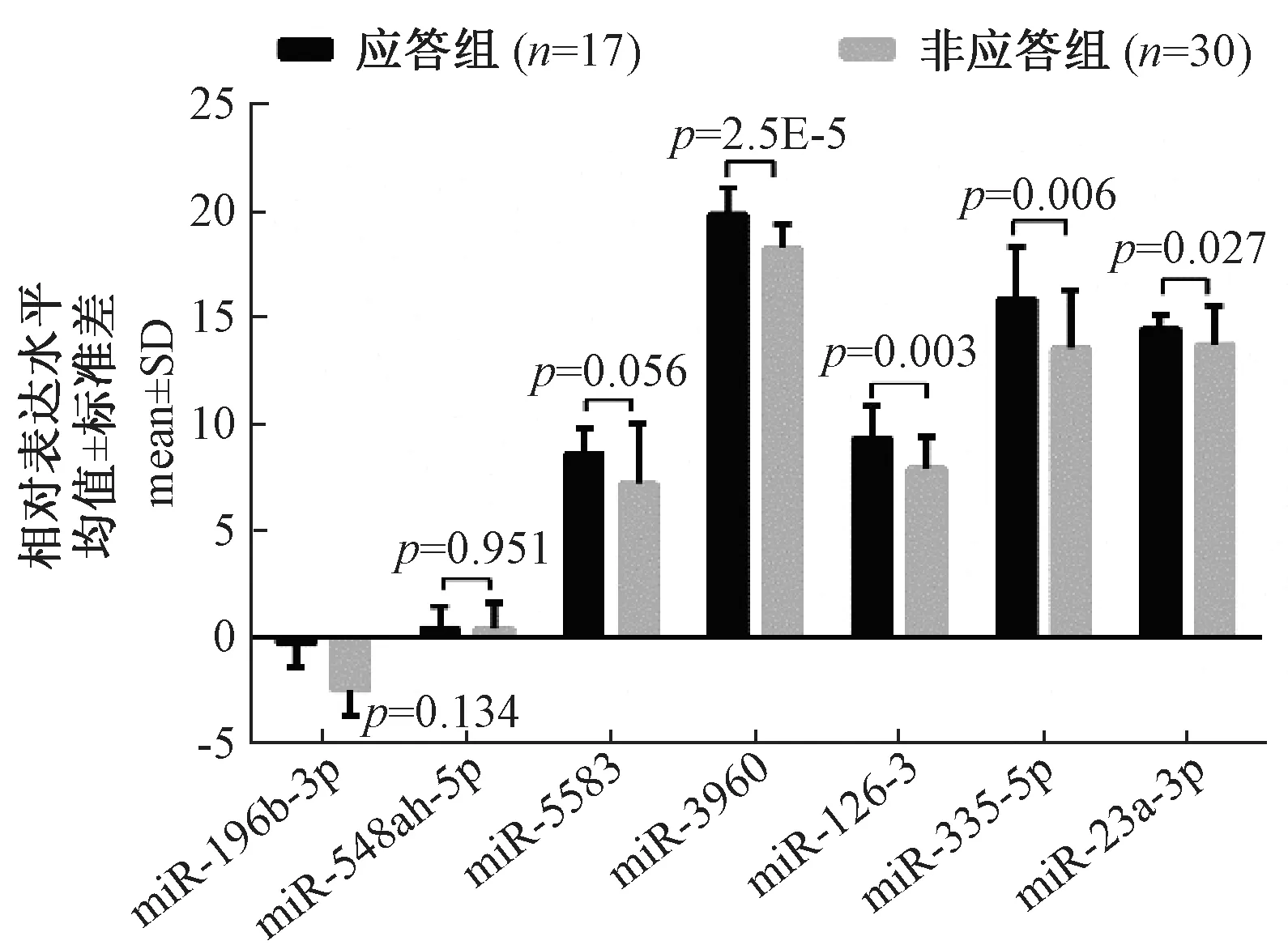
图1 差异miRNAs的相对表达水平,相对表达水平用ΔCT法表示:ΔCT=CT目标miRNA-CT 内参U6Fig.1 The relative expression levels of the differential miRNAs between the two groups. The relative level is shown as ΔCT (ΔCT=CT of interest miRNA-CT of internal control U6)
2.4差异miRNAs靶基因预测对miR-3960, miR-126-3p, miR-23 a-3p和miR-335-5p通过在线数据库TargetScan 7.1和miRDB V5.0预测靶基因,并取两数据库重复出现的部分,筛选出可能与HBsAg清除免疫应答相关的靶基因。结果显示miRNAs的可能靶基因与免疫调节等生物过程相关,如CCL-7、IGSF8、CASP7等。
2.5差异miRNAs靶基因功能预测对差异miRNAs的靶基因进行通路分析,探究其可能参与的生物过程,其中4条差异microRNAs的靶向基因相关的信号通路包括AMPK信号通路、NOD样受体信号通路、NF-kappa B信号通路、mTOR通路等。
3 讨论
本研究通过前瞻性建立PegIFN治疗队列,获得不同应答组之间差异表达的miRNAs,为了排除病毒、宿主免疫因素对HBsAg清除的影响,本研究选择肝功能正常、低基线HBsAg和低病毒载量的CHB患者。研究发现CHB的不同感染阶段具有不同的miRNA表达模式[7],这可能与他们不同的免疫状态相关。我们推测经PegIFN治疗后获得HBsAg清除的患者,可能在治疗前已经处于一种有利于免疫清除的状态,经PegIFN的免疫调节推动作用后进一步发生HBsAg清除,而用药前差异表达的miRNAs可能与这种免疫状态相关。在验证出的4条miRNAs中,miR-3960被报道可与长非编码RNA HOTAIRMI竞争性结合调节单核细胞/树突状细胞(dendritic cell,DC)的分化[8],DC作为专性抗原提呈细胞在抗原加工、处理、呈递过程中具有重要作用,是连接固有免疫和适应性免疫的纽带。4条差异miRNAs靶基因的通路分析结果显示与多个重要信号通路相关,如AMPK信号通路、NOD样信号通路、NF-kappa B信号通路、mTOR通路等。AMPK是生物能量代谢调节的关键分子,通过调节组织器官的能量代谢进而影响机体功能,研究发现AMPK信号可影响T细胞的激活和细胞因子的产生[9],而NF-kappa B被报道在肿瘤早期发展的免疫监视过程中具有重要作用[10]。另外miR-126-3p[11], miR-23 a-3p[12]和miR-335-5p[13]均被报道与肿瘤的侵袭和转移相关,而免疫系统对于肿瘤的监视发挥重要作用,免疫系统的失调可能导致肿瘤的侵袭和转移。综合相关文献报道及差异miRNAs靶基因、信号通路的分析,我们推测miRNAs可能通过调控免疫相关基因的表达,增强HBV清除相关的免疫应答,促进HBsAg清除。
miRNAs除了调控免疫因素外,也可能通过直接抑制HBV DNA的复制或者靶向HBsAg转录本降低HBsAg水平。例如miR-199 a-3p,miR-122,miR-146 a在芯片结果中差异表达,研究报道miR-199 a-3p与HBsAg编码区特定位点结合降低HBsAg的表达水平[7];miR-122作为肝细胞特异表达的miRNA,可抑制HBV DNA的复制[14],而miR-146 a通过下调肝细胞内STAT1损伤IFN诱导的免疫应答能力[15]。本研究选择的7条miRNAs中,miR-548ah-5p, miR-196b-3p和miR-5583在验证队列中未验证出差异,可能是该基因本身表达量低以致芯片检测结果容易出现假阳性,或者是验证样本量较少的结果。
综上所述,miRNA对HBsAg清除具有一定的研究价值,miRNA可能通过调节免疫相关基因的表达、直接抑制HBV DNA复制或者HBsAg转录本的翻译获得HBsAg清除。本研究尚存在不足之处:整个研究队列的样本数较少,需要进一步扩大样本进行验证;另外缺乏对差异miRNAs的靶基因的具体分子机制的研究,这将是我们今后的研究方向。
利益冲突:无
参考文献
[1] Lampertico P, Agarwal K, Berg T, et al. EASL 2017 Clinical Practice Guidelines on the management of hepatitis B virus infection[J]. J Hepatol, 2017,67(2):370-398. DOI: 10.1016/j.jhep.2017.03.021.
[2] Sarin SK, Kumar M, Lau GK, et al. Asian-Pacific clinical practice guidelines on the management of hepatitis B: a 2015 update[J]. Hepatol Int, 2016,10(1):1-98. DOI: 10.1007/s12072-015-9675-4.
[3] Tseng TC, Liu CJ, Yang HC, et al. Serum hepatitis B surface antigen levels help predict disease progression in patients with low hepatitis B virus loads[J]. Hepatology, 2013,57(2):441-450. DOI: 10.1002/hep.26041.
[4] Ning Q, Han M, Sun Y, et al. Switching from entecavir to PegIFN alfa-2a in patients with HBeAg-positive chronic hepatitis B: A randomised open-label trial (OSST trial)[J]. J Hepatol, 2014,61(4):777-784.DOI: 10.1016/j.jhep.2014.05.044.
[5] Increased and sustained HBsAg loss in HBeAg positive CHB patients switched from NUC to Peg-IFN alfa-2a: A randomised open label trial (NEW SWITCH study).2014, AASLD, LB-10.
[6] Zhang G, Li Y, Zheng S, et al. Suppression of hepatitis B virus replication by microRNA-199a-3p and microRNA-210[J]. Antiviral Res, 2010,88(2):169-175.DOI: 10.1016/j.antiviral.2010.08.008.
[7] Ji F, Yang B, Peng X, et al. Circulating microRNAs in hepatitis B virus-infected patients[J]. J Viral Hepat, 2011,18(7):e242-e251.DOI: 10.1111/j.1365-2893.2011.01443.x.
[8] Xin J, Li J, Feng Y, et al. Downregulation of long noncoding RNA HOTAIRM1 promotes monocyte/dendritic cell differentiation through competitively binding to endogenous miR-3960[J]. Onco Targets Ther, 2017,Volume 10:1307-1315. DOI: 10.2147/OTT.S124201.
[9] Rao E, Zhang Y, Li Q, et al. AMPK-dependent and independent effects of AICAR and compound C on T-cell responses[J]. Oncotarget, 2016,7(23):33783-33795.DOI: 10.18632/oncotarget.9277.
[10] Ratnam NM, Peterson JM, Talbert EE, et al. NF-κB regulates GDF-15 to suppress macrophage surveillance during early tumor development[J]. J Clin Invest, 2017, 127(10):3796-3809. DOI: 10.1172/JCI91561.
[11] Du C, Lv Z, Cao L, et al. MiR-126-3p suppresses tumor metastasis and angiogenesis of hepatocellular carcinoma by targeting LRP6 and PIK3R2[J]. J Transl Med, 2014,12(1):259. DOI: 10.1186/s12967-014-0259-1.
[12] Wen Y, Lee W, Tan P, et al. By inhibiting snail signaling and miR-23a-3p, osthole suppresses the EMT-mediated metastatic ability in prostate cancer[J]. Oncotarget, 2015,6(25):21120-21136. DOI10.18632/oncotarget.4229.
[13] Wang Y, Yang T, Zhang Z, et al. Long non-coding RNA TUG1 promotes migration and invasion by acting as a ceRNA of miR-335-5p in osteosarcoma cells[J]. Cancer Sci, 2017,108(5):859-867. DOI:10.1111/cas.13201.
[14] Qiu L, Fan H, Jin W, et al. miR-122-induced down-regulation of HO-1 negatively affects miR-122-mediated suppression of HBV[J]. Biochem Biophys Res Commun, 2010,398(4):771-777. DOI: 10.1016/j.bbrc.2010.07.021.
[15] Hou Z H, Han Q J, Zhang C, et al. miR146a impairs the IFN-induced anti-HBV immune response by downregulating STAT1 in hepatocytes[J]. Liver Int, 2014,34(1):58-68. DOI: 10.1111/liv.12244.

