多聚ADP-核糖结合蛋白的鉴定
沈莉芳,张 弛,吴家雪
(复旦大学 生命科学学院,上海200438)
ADP-核糖基化是一种在高等真核生物中普遍存在的蛋白翻译后修饰.该修饰由ADP-核糖基转移酶家族的成员催化,从底物β-NAD+转移ADP-核糖部分到受体蛋白上,从而调节这些蛋白的功能.ADP-核糖基团可以以单个的ADP-核糖(MAR)或以聚合链多聚ADP-核糖(PAR))的形式存在.目前已经发现PAR在染色质重塑、DNA损伤修复、基因转录、蛋白质降解和细胞死亡中发挥重要作用[1-3].
PAR的功能除了调节底物外,还可以作为介导两个蛋白之间相互作用的平台起作用.例如,许多DNA损伤应答因子,如ALC1[4-5]和CHFR[6]在DNA损伤时能够被招募到DNA损伤位点,这种募集作用取决于它们与DNA损伤位点上被PAR修饰蛋白的结合,并且能被ADP-核糖基转移酶(poly(ADP-ribose)polymerases, PARPs)抑制剂抑制.这种相互作用主要由PAR和PAR结合结构域的结合来介导.目前已经报道了一些PAR结合结构域.这些结构域包括Marco结构域[4],WWE结构域[7],PAR结合锌指结构域(PBZ)[6,8-10],BRCT结构域[11],FHA结构域[11],OB-折叠结构域[12],PIN结构域[13]和RNA识别基序(RRM)[14].
鉴定PAR修饰蛋白或PAR结合蛋白对于探索PAR的功能非常重要.但是由于目前大规模检测PAR修饰位点技术的缺乏,在文献和数据库中报道的PAR修饰蛋白或PAR结合蛋白还比较少.基于质谱的蛋白质组学已经成为鉴定复杂的PAR结合蛋白的关键技术[15-17].最近有研究表明利用抗体,PAR结合结构域或NAD+类似物沉淀相关的PAR修饰蛋白,并进行质谱分析可以得到大量的PAR修饰或者PAR结合蛋白[18-22].然而,通过这些方法鉴定的蛋白绝大部分是被PAR修饰的蛋白,缺少直接与PAR结合蛋白的信息.
本论文中,我们通过利用PAR修饰的PARP1或PARP1分别进行pull-down试验和质谱分析来比较分别与PARP1或PAR-PARP1结合的蛋白,得到PAR特异结合蛋白.结果表明,我们鉴定到大量PAR结合蛋白,并进行进一步的验证.
1 材料和方法
1.1 材料
1.1.1 细胞系和质粒
人肾上皮细胞来源的HEK 293T细胞系和人肝癌细胞来源的QGY-7703细胞系获自中国科学院上海细胞库.载体pGEX-4T-1或修饰的SBP载体由本实验室保存.
1.1.2 主要试剂
DNA聚合酶,限制性内切酶,T4DNA连接酶体系(New England Biolabs),质粒小量抽提试剂盒,PCR产物纯化试剂盒和柱离心式凝胶回收纯化试剂盒(Axygen),谷胱甘肽琼脂糖珠4B(Amersham),胎牛血清(Gibco),100U/mL青霉素和100μg/mL链霉素(Euroclone),DMEM培养基(Hyclone),辣根过氧化物酶偶联羊抗兔抗体(Calbiochem),抗体抗-PAR多抗(Thermo),ECL显色试剂盒(Santa Cruze).
1.1.3 siRNA
HRP2干扰siRNA寡核苷酸购自上海吉玛生物公司.HRP-2的siRNA寡核苷酸正向序列是: si-HRP-2#1: 5’-GCCCAACAAGAGGAAAGGC-3’;si-HRP-2#2: 5’-GCUGCACAGUGAGAUCAAG-3’.阴性对照序列为: 对照: 5’-ACAGACUUCGGAGUACCUG-3’.根据说明书,使用Oligofectamine(Invitrogen)将siRNA转染入细胞.
1.2 方法
1.2.1 体外ADP-核糖基化
GST-PARP1融合蛋白与PAR核糖基化缓冲液(100mmol/L Tris-HCl(pH7.8),12.5mmol/L NAD+,10mmol/L MgCl2,50μg寡脱氧核糖核苷酸(5’-GGAATTCC-3’)和10mmol/L DTT)或者不含NAD+的PAR核糖基化缓冲液混合,并于30℃条件下孵育30min.通过GST抗体或者PAR抗体进行免疫印迹分析PARP1的自我修饰.
1.2.2 PAR的合成和纯化
PAR合成缓冲液1mL(100mmol/L Tris-HCl pH 7.8, 10mmol/L MgCl2,12.5mmol/L NAD+,10mmol/L DTT,50μg寡脱氧核糖核苷酸(5’-GGAATTCC-3’),100μg GST-PARP1),37℃孵育1h,加入20mL预冷的20% TCA终止.加入DNA酶Ⅰ去除寡核苷酸DNA,并经蛋白酶K消化蛋白后,用乙醇沉淀并纯化[6].
1.2.3 GST pull-down实验
收集HEK 293T细胞,用含有0.5% NP-40,100mmol/L NaCl,20mmol/L Tris-HCl(pH 8.0)和1mmol/L EDTA的细胞裂解缓冲液裂解细胞.将细胞裂解液与GST融合蛋白(GST-PARP1或被PAR修饰的GST-PARP1)在4℃孵育2h,然后加入谷胱甘肽琼脂糖珠4B再结合2h.通过离心的方法,收集谷胱甘肽琼脂糖珠,并用细胞裂解液将非特异结合的蛋白去除.加入SDS上样液,98℃加热10min后,进行SDS-PAGE电泳,并用考马斯亮蓝(CBB)染色.最后将相应的泳道进行切割,并送到质谱分析平台进行质谱鉴定.
1.2.4 蛋白的表达和纯化
按照之前报道的方法[6]在昆虫细胞中表达并纯化得到GST-PARP1蛋白.而GST-HRP2,CDK9,EWSR,FXR1,PWWP和PWWP突变蛋白在大肠杆菌E.coliBL21中表达并纯化.简而言之,将转入PGEX-4T-HRP2或其他基因的E.coliBL21培养到OD到0.6左右,加入异丙基-β-D-硫代半乳糖苷(IPTG),并在20℃诱导表达诱导表达6h.离心收集细菌后,通过超声破碎细胞,高速离心后收集上清液体,再加入谷胱甘肽琼脂糖珠4B在4℃结合2h,然后用含有还原性谷胱甘肽的洗脱液将GST融合蛋白从谷胱甘肽琼脂糖珠4B洗脱下来,并通过SDS-PAGE凝胶电泳和考马斯亮蓝染色来分析融合蛋白.
1.2.5 斑点印迹和体外结合实验
将GST融合蛋白各2.5μL点样在0.22μm硝酸纤维素膜上,自然风干后,用1×TBST(20mmol/L Tris-HCl,0.3mmol/L NaCl,0.05% Tween-20,pH 8.0)配置的5%的脱脂牛奶在室温下封闭40min;室温下将PAR聚合物与结合GST融合蛋白的膜孵育1h,然后用含高浓度氯化钠(50mmol/L)的1×TBST洗膜4次,自然风干;将膜与用TBST稀释的抗PAR多克隆抗体(HRP标记)孵育1h,然后用TBST溶液洗膜3次,最后进行显色爆光.
1.2.6 细胞存活率测定
将细胞接种到60mm的培养皿上,然后分别用对照siRNA或HRP-2 siRNA转染.转染24h后,加入DNA损伤药物CPT或MMS处理细胞.每3天更换1次培养基,将细胞培养10d.通过比较培养皿中形成的细胞克隆数来计算存活的细胞数量.
1.2.7 统计分析
数据取自3个独立实验的均值±标准差.有意义的差异通过Studentt检验进行评估.P<0.05被认为具有统计学意义.
2 结 果
2.1 多聚ADP-核糖结合的蛋白的鉴定
从昆虫细胞中表达并纯化GST-PARP1,考马斯亮蓝染色表明获得的GST-PARP1比较纯,大小为140kDa左右,与预期一致(图1(a)).通过体外PAR反应,并分别利用PAR抗体和GST抗体进行Western blot分析,表明获得的GST-PARP1在体外具有很强的酶活性,几乎所有的GST-PARP1能够在体外进行自我PAR修饰(图1(b)).
为了鉴定PAR结合蛋白,我们将GST-PARP1或PAR修饰的GST-PARP1蛋白分别与HEK 293T细胞的裂解液孵育,然后用谷胱甘肽琼脂糖珠进行沉淀.通过质谱分析GST-PARP1和PAR修饰的GST-PARP1结合的蛋白.表1列出了部分质谱分析得到的特异与被PAR修饰的GST-PARP1结合的蛋白.在这些仅特异性结合PAR修饰的GST-PARP1的蛋白中,有部分已被报道与PAR结合,如SFPQ,NONO,FUS,TAF15,RBMX,PARP2,CHFR和ALC1[5-6,21,23-24],部分是第一次发现与PAR结合.为了进一步验证这些蛋白与PAR的结合,我们表达并纯化了GST融合的CDK9,FXR2,EWSR1和HRP2蛋白(图1(c)).然后通过Dot blot方法体外检测GST-CDK9,FXR2,EWSR1和HRP2与PAR的相互作用,并用抗GST和抗PAR的抗体进行Western blot分析,结果表明这4个蛋白都能在体外与PAR结合(图1(d)).

图1 PAR结合蛋白的鉴定Fig.1 Identification of PAR-binding proteins(a) GST-PARP1的SDS-PAGE凝胶电泳及考马斯亮蓝染色;(b) Western blot验证GST-PARP1的体外自我修饰;(c) GST-CDK9,FXR1,HRP2和EWSR1的SDS-PAGE电泳及考马斯亮蓝染色;(d) Dot blot验证GST-CDK9, FXR1, HRP2和EWSR1与PAR体外结合.
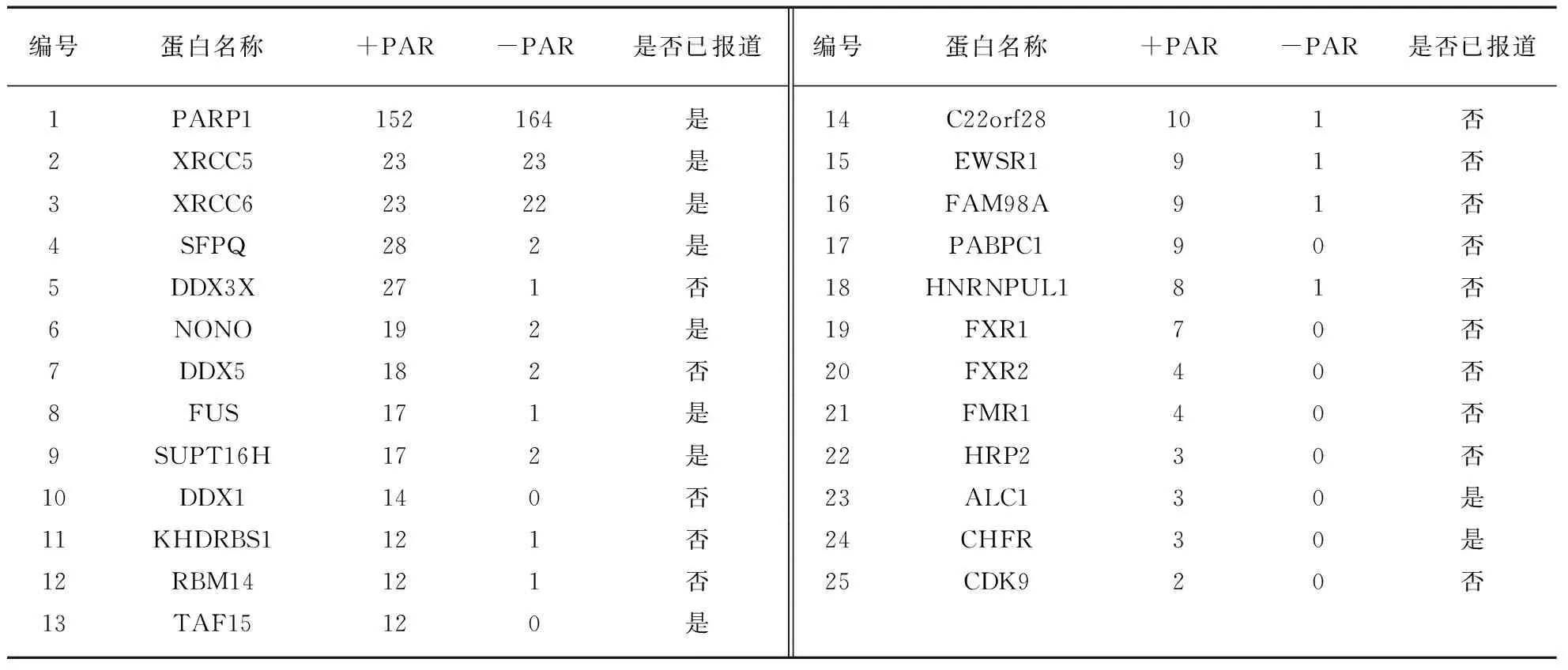
编号蛋白名称+PAR-PAR是否已报道编号蛋白名称+PAR-PAR是否已报道1PARP1152164是14C22orf28101否2XRCC52323是15EWSR191否3XRCC62322是16FAM98A91否4SFPQ282是17PABPC190否5DDX3X271否18HNRNPUL181否6NONO192是19FXR170否7DDX5182否20FXR240否8FUS171是21FMR140否9SUPT16H172是22HRP230否10DDX1140否23ALC130是11KHDRBS1121否24CHFR30是12RBM14121否25CDK920否13TAF15120是
2.2 PWWP域是一个PAR结合结构域
在这些新的PAR结合蛋白中,HRP-2属于肝癌衍生生长因子相关蛋白家族(HDGF家族).该家族包括HDGF,HDGFL1,LEDGF,HRP-2和HRP-3.该家族的蛋白在N末端都有保守的PWWP/HATH结构域[25-27].之前,我们发现HRP-2和HRP-3在肝癌(HCC)细胞中上调,并促进HCC细胞的生长和存活,表明它们在肿瘤发生和发展中起重要作用[28-29].有趣的是,有报道发现HDGF与PARP1相关[30].我们还发现HDGF存在于PAR修饰的GST-PARP1结合的蛋白复合物中,而不存在于GST-PARP1结合的蛋白复合物中.HDGF和HRP-2在N末端都含有非常保守的PWWP结构域,暗示HRP-2与PAR的相互作用很大可能依赖于其PWWP结构域.
为了验证这一假设,我们构建了SBP标签标记的全长HRP-2(HRP-2 WT)和PWWP结构域缺失突变体HRP-2(HRP-2 MT)的质粒,并将其转染到HEK 293T细胞中.将PAR修饰的GST-PARP1分别与表达HRP-2 WT或HRP-2 MT的HEK 293T细胞的细胞裂解液共孵育,然后用谷胱甘肽-琼脂糖珠进行沉淀并用Flag抗体进行Western blot分析.如图2(a)所示,PAR修饰的GST-PARP1能与野生型的HRP-2相互作用,而不与PWWP结构域缺失突变体HRP-2相互作用,表明HRP-2的PWWP结构域是HRP2与PAR相互作用必需的.
为了进一步验证这一假设,我们克隆、表达并纯化了所有HDGF家族成员的PWWP结构域,并进行PAR体外结合实验.如图2(b)和图2(c)(看40页)所示,HDGF家族蛋白的PWWP结构域都与PAR具有强烈的相互作用.为进一步证实这种相互作用,我们表达并纯化了HRP-2的PWWP结构域的截断突变体蛋白,并检测了这些截断体蛋白与PAR的相互作用.如图2(d)和图2(e)(看40页)所示,缺失PWWP结构域的N末端或C末端完全抑制了PWWP结构域和PAR之间的相互作用.这些结果表明,HDGF蛋白家族的PWWP结构域是PAR结合结构域.
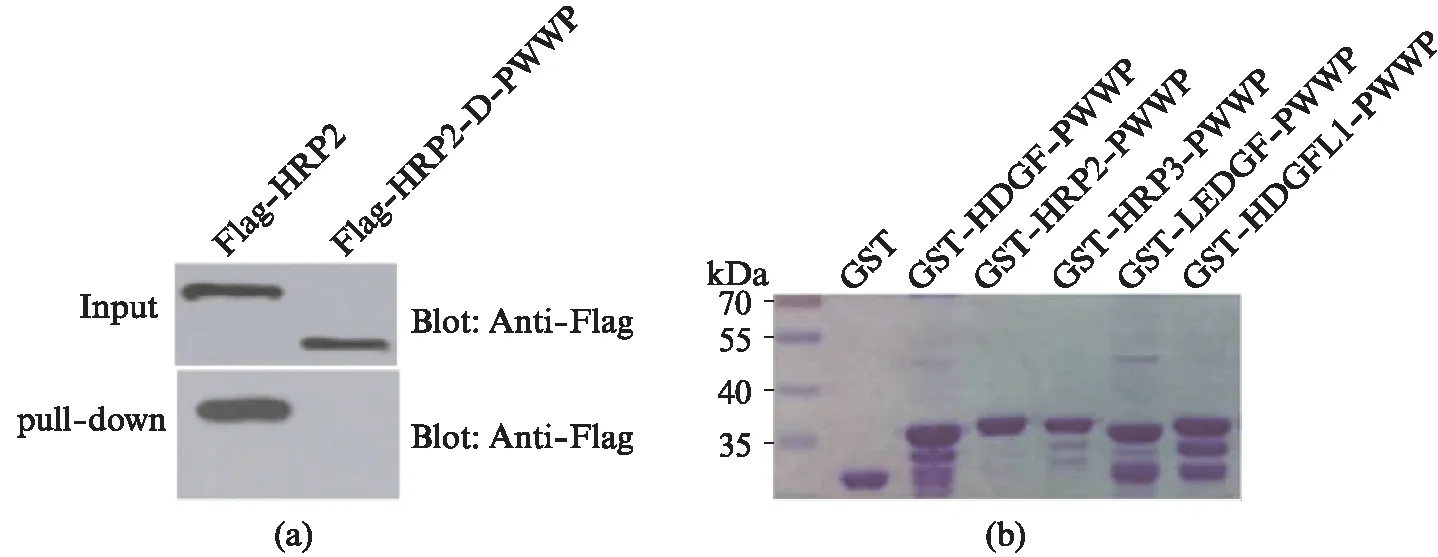

图2 PWWP结构域是个PAR结合结构域Fig.2 PWWP domains is a PAR binding module(a) HRP2的PWWP结构域与PAR修饰的PARP1相互作用;(b) HDGF家族的PWWP结构域的SDS-PAGE电泳及考马斯亮蓝染色;(c) Dot blot鉴定PWWP结构域与PAR的体外结合;(d) PWWP结构域及其突变体的SDS-PAGE电泳及考马斯亮蓝染色;(e) Dot blot鉴定PWWP结构域及其突变体与PAR体外结合.
2.3 HRP-2缺失突变细胞对DNA损伤剂敏感
之前的研究表明很多PAR结合蛋白能在DNA损伤时被招募到DNA损伤位点,从而参与DNA损伤应答[4-5,8,11].我们检测了HRP-2是否能在激光诱导DNA损伤的细胞中被招募到DNA损伤部位.虽然阳性对照CHFR能快速地定位于激光诱导的DNA损伤处,但是在激光处理细胞引起DNA损伤后,GFP-HRP2的定位没有明显变化,表明HRP2在DNA损伤时不能被招募到DNA损伤位点(数据未显示).
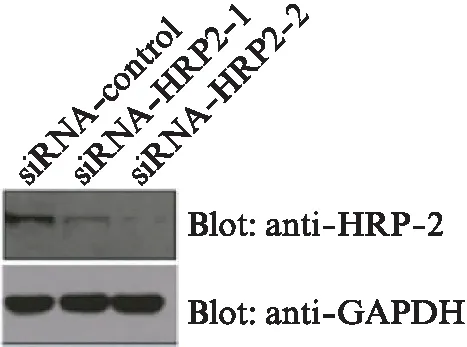
图3 HRP-2表达水平被其 siRNA显著抑制Fig.3 Expression of HRP-2 was inhibited by its siRNA
接着我们检测HRP-2缺失细胞对DNA损伤药物的敏感性.前期的研究中,我们发现HRP-2特异siRNA能显著降低其在肝癌细胞中的表达[29].因此,我们将HRP-2特异siRNA或对照siRNA分别转入QGY-7703,并用Western blot的方法来检测其表达情况.如图3所示,相对于对照细胞,HRP-2特异siRNA转染的细胞,其HRP-2的表达水平显著降低.
然后我们用DNA损伤药物CPT和MMS处理转染HRP-2特异siRNA或对照siRNA的细胞,并通过CKK8试剂盒来检测细胞的存活率.如图4所示,DNA损伤剂CPT或MMS处理后,相对于对照组细胞,HRP-2缺失细胞的存活率明显降低,表明HRP-2缺失细胞对DNA损伤剂非常敏感,HRP-2可能在DNA损伤修复中具有重要的功能.这并不奇怪,许多蛋白参与DNA损伤修复,但并不定位到DNA损伤位点例如,最近报道的53BP1的相互作用蛋白TIRR尽管在DNA损伤后不能被招募到DNA损伤部位,但是TIRR缺失细胞对DNA损伤剂呈现敏感性[31].

图4 HRP-2消减细胞对DNA损伤药物敏感Fig.4 HRP-2 depletion cells are sensitive to DNA damage agent(a) HRP-2消减细胞对DNA损伤药物MMS敏感;(b) HRP-2消减细胞对DNA损伤药物CPT敏感.
3 结 论
本研究中,我们通过亲和纯化和质谱分析的方法鉴定了大量与PAR特异结合的蛋白,并在体外进一步验证了所鉴定的CDK9、EWSR1、FXR1和HRP2与PAR的结合.在此基础上,我们还发现HDGF家族成员的PWWP结构域是一个PAR结合结构域;而且HRP-2消减能导致细胞对DNA损伤药物敏感,表明HRP2可能参与了DNA损伤修复过程.这些结果为进一步研究PAR及其相互作用蛋白的功能打下了坚实的基础.
参考文献:
[1] SCHREIBER V, DANTZER F, AME J C, et al. Poly(ADP-ribose): Novel functions for an old molecule [J].NatureReviewsMolecularCellBiology, 2006,7(7): 517-528.
[2] DIEFENBACH J, BÜSRKLE A. Introduction to poly(ADP-ribose) metabolism [J].CellularandMolecularLifeSciences, 2005,62(7-8): 721-730.
[3] BARKAUSKAITE E, JANKEVICIUS G, AHEL I. Structures and mechanisms of enzymes employed in the synthesis and degradation of PARP-dependent protein ADP-ribosylation [J].MolecularCell, 2015,58(6): 935-946.
[5] PINES A, VROUWE M G, MARTEIJN J A, et al. PARP1 promotes nucleotide excision repair through DDB2 stabilization and recruitment of ALC1 [J].JournalofCellBiology, 2012,199(2): 235-249.
[6] LIU C, WU J, PAUDYAL S C, et al. CHFR is important for the first wave of ubiquitination at DNA damage sites [J].NucleicAcidsResearch, 2013,41(3): 1698.
[7] ZHANG Y, LIU S, MICKANIN C, et al. RNF146 is a poly(ADP-ribose)-directed E3 ligase that regulates axin degradation and Wnt signalling [J].NatureCellBiology, 2011,13(5): 623-629.
[8] AHEL I, AHEL D, MATSUSAKA T, et al. Poly(ADP-ribose)-binding zinc finger motifs in DNA repair/checkpoint proteins [J].Nature, 2008,451(7174): 81-85.
[9] OBEROI J, RICHARDS M W, CRUMPLER S, et al. Structural basis of poly(ADP-ribose) recognition by the multizinc binding domain of checkpoint with forkhead-associated and RING domains(CHFR) [J].JournalofBiologicalChemistry, 2010,285(50): 39348-39358.
[10] LI G Y, MCCULLOCH R D, FENTON A L, et al. Structure and identification of ADP-ribose recognition motifs of APLF and role in the DNA damage response [J].ProcNatAcadSciUSA, 2010,107(20): 9129-9134.
[11] LI M, LU L Y, YANG C Y, et al. The FHA and BRCT domains recognize ADP-ribosylation during DNA damage response [J].Genes&Development, 2013,27(16): 1752-1768.
[12] ZHANG F, CHEN Y, LI M, et al. The oligonucleotide/oligosaccharide-binding fold motif is a poly(ADP-ribose)-binding domain that mediates DNA damage response [J].ProcNatlAcadSciUSA, 2014,111(20): 7278-7283.
[13] ZHANG F, SHI J, CHEN S H, et al. The PIN domain of EXO1 recognizes poly(ADP-ribose) in DNA damage response [J].NucleicAcidsResearch, 2011,43(22): 10782-10794.
[14] KRIETSCH J, CARON M C, GAGNÉ J P, et al. PARP activation regulates the RNA-binding protein NONO in the DNA damage response to DNA double-strand breaks [J].NucleicAcidsResearch, 2012,40(20): 10287-301.
[15] ZHANG Y, WANG J, DING M, et al. Site-specific characterization of the Asp-and Glu-ADP-ribosylated proteome [J].NatureMethods, 2013,10(10): 981-984.
[16] DANIELS C M, ONG S E, LEUNG A K L. The promise of proteomics for the study of ADP-ribosylation [J].MolecularCell, 2015,58(6): 911-924.
[17] DANIELS C M, ONG S E, LEUNG A K L. Phosphoproteomic approach to characterize protein mono-and poly(ADP-ribosyl) ation sites from cells [J].JournalofProteomeResearch, 2014,13(8): 3510-3522.
[18] JUNGMICHEL S, ROSENTHAL F, ALTMEYER M, et al. Proteome-wide identification of poly(ADP-ribosyl) ation targets in different genotoxic stress responses [J].Molecularcell, 2013,52(2): 272-285.
[19] GAGNÉ J P, ISABELLE M, LO K S, et al. Proteome-wide identification of poly(ADP-ribose) binding proteins and poly(ADP-ribose)-associated protein complexes [J].NucleicAcidsResearch, 2008,36(22): 6959-6976.
[20] GAGNÉ J P, PIC E, ISABELLE M, et al. Quantitative proteomics profiling of the poly(ADP-ribose)-related response to genotoxic stress. [J].NucleicAcidsResearch, 2012,40(16): 7788-7805.
[21] ISABELLE M, GAGNÉ J P, GALLOUZI I E, et al. Quantitative proteomics and dynamic imaging reveal that G3BP-mediated stress granule assembly is poly(ADP-ribose)-dependent following exposure to MNNG-induced DNA alkylation [J].JournalofCellScience, 2012,125(19): 4555-4566.
[22] DANI N, STILLA A, MARCHEGIANI A, et al. Combining affinity purification by ADP-ribose-binding macro domains with mass spectrometry to define the mammalian ADP-ribosyl proteome [J].ProcNatlAcadSciUSA, 2009,106(11): 4243-4248.
[23] ADAMSON B, SMOGORZEWSKA A, SIGOILLOT F D, et al. A genome-wide homologous recombination screen identifies the RNA-binding protein RBMX as a component of the DNA-damage response [J].NatureCellBiology, 2012,14(3): 318-328.
[24] ALTMEYER M, NEELSEN K J, TELONI F, et al. Liquid demixing of intrinsically disordered proteins is seeded by poly(ADP-ribose). [J].NatureCommunications, 2014,6(8088): 8088.
[25] IZUMOTO Y, KURODA T, HARADA H, et al. Hepatoma-derived growth factor belongs to a gene family in mice showing significant homology in the amino terminus. [J].Biochemical&BiophysicalResearchCommunications, 1997,238(1): 26-32.
[26] DIETZ F, FRANKEN S, YOSHIDA K, et al. The family of hepatoma-derived growth factor proteins: Characterization of a new member HRP-4 and classification of its subfamilies. [J].BiochemicalJournal, 2002,366(2): 491-500.
[27] BAUDE A, AAES T L, ZHAI B, et al. Hepatoma-derived growth factor-related protein 2 promotes DNA repair by homologous recombination [J].NucleicAcidsResearch, 2016,44(5): 2214-2226.
[28] XIAO Q, QU K, WANG C, et al. HDGF-related protein-3 is required for anchorage-independent survival and chemoresistance in hepatocellular carcinomas [J].Gut, 2013,62(3): 440-451.
[29] GAO K, XU C, JIN X, et al. HDGF-related protein-2(HRP-2) acts as an oncogene to promote cell growth in hepatocellular carcinoma [J].BiochemicalandBiophysicalResearchCommunications, 2015,458(4): 849-855.
[30] ZHAO J, YU H, LIN L, et al. Interactome study suggests multiple cellular functions of hepatoma-derived growth factor(HDGF) [J].JournalofProteomics, 2011,75(2): 588-602.
[31] DRANÉ P, BRAULT M E, CUI G, et al. TIRR regulates 53BP1 by masking its histone methyl-lysine binding function [J].Nature, 2017,543(7644): 211-216.

