沙门菌LAMP可视化检测方法的建立与应用
刘志科,张 洁,杨宁宁,徐锦凤,徐明国,荆明龙,吴文星,曹旭东,任 艳,石 峰,陈创夫*
(1.石河子大学动物科技学院,新疆石河子 832003;2.石河子大学生命科学学院,新疆石河子 832003;3.石河子大学医学院,新疆石河子 832002)
在最新的研究报告中,由沙门菌引起人食物中毒的病例居于首位,其中肠炎沙门菌占85%以上,据不完全统计,全球每年报道的沙门菌食物中毒的病人大约为170万人,死亡人数大约在1 000人以上,且呈上升趋势[1],对公共卫生安全构成威胁。由此造成的医疗费用每年约114亿美元,给各国政府带来了巨大的经济负担[2]。因此,迫切需要建立一种快速、准确诊断沙门菌食物中毒的方法。
目前,对沙门菌的检测方法主要有传统的分离鉴定方法、免疫学方法及分子生物学方法。传统的沙门菌检测方法是根据国家标准(GB4789.4-2016)进行的,其步骤比较繁琐,耗时较长,至少需要4 d~7 d才能得出明确的诊断结果,虽然此方法为“金标准”,但已不能满足实际工作的需要。免疫学方法主要利用抗原-抗体之间的特异性反应,是一种定性或定量的诊断技术,其灵敏性和特异性比较高,但其操作复杂、试验条件要求高等因素,在临床应用上带来了较大的不便 。分子生物学方法需要昂贵的仪器设备,且受人为操作技术影响较大,不能区别核酸来源于死菌还是活菌,在基层单位也很难普及和推广应用[3-7]。Notomi T等[8]研究者开发了一种环介导等温扩增技术(loop-mediated isothermal amplification,LAMP),具有灵敏度高、特异性好、操作简单和方便快捷等优点,其扩增原理是利用一种具有链置换特性的Bst DNA聚合酶,该酶具有5′→3′聚合酶活性,可以针对特异性靶基因序列上的6个不同区域的4条引物和环引物,在简单的恒温装置上即可快速实现扩增核酸的方法。LAMP扩增产物可以通过借助浊度仪、电泳及在反应管中加入荧光染料进行结果判断[9-10],检测时间在60 min左右,与常规PCR方法[11-12]相比,克服了重复的热变性并缩短了反应时间,且灵敏度和特异性在一定程度上也得到了很大的提高,具有良好的应用前景和发展潜力[13]。研究发现,invA基因是沙门菌重要的毒力因子,参与大多数沙门菌的入侵,并与致病性密切相关。在所有沙门菌侵袭机体的过程中,invA基因在毒力岛SPI-1的侵袭功能上发挥不可替代的作用,同时也是沙门菌的主要标记基因之一,该基因在沙门菌属中的同源性高达99%以上,且与其他物种同源性较小[14]。
本研究根据沙门菌invA靶基因的保守区域设计LAMP引物,优化检测条件,建立一种切合实际的沙门菌LAMP检测方法。通过对感染鸡白痢沙门菌的病鸡进行剖检,采集的脏器组织和人工污染沙门菌的鱼粉作为样品,用建立的沙门菌LAMP可视化检测方法与常规PCR和real-time PCR方法进行检测,评价其检出率和灵敏度,旨在建立一种特异性强、敏感性高和操作简便的沙门菌LAMP检测方法,为沙门菌病原的快速检测提供一种更便捷、可靠和适合基层的临床应用技术。
1 材料与方法
1.1 材料
1.1.1 试验菌种及来源 肠炎沙门菌(CVCC 3949)购自中国兽医微生物菌种保藏管理中心;多杀性巴氏杆菌、大肠埃希菌、金黄色葡萄球菌、枯草芽胞杆菌、链球菌、无乳链球菌、变形杆菌、痢疾杆菌、单核细胞增生李斯特菌和放线杆菌均由石河子大学微生物教研室保存。
1.1.2 主要仪器与试剂 紫外凝胶成像分析系统,Molecular Imager公司产品;超级恒温水浴锅,常州金坛精达仪器制造有限公司产品;Nanodrop-2000微量核酸蛋白检测仪,美国Thermo Scientific公司产品;LightCycler®480II 实时荧光定量PCR系统,罗氏公司产品;细菌基因组DNA提取试剂盒、2×ESTaqMasterMix 、dNTP混合液,上海生工生物工程技术服务有限公司产品;Bst 2.0 DNA聚合酶大片段(M0538),New England Biolads(Beijing) 有限公司产品;钙黄绿素、甜菜碱、MgSO4,Sigma公司产品。
1.2 方法
1.2.1 引物及探针的设计与合成 根据NCBI中GenBank公布的沙门菌invA基因(登录号:NC003197.1),并用生物学在线软件分析该基因的保守核苷酸序列。针对invA基因的保守区域,结合环介导等温扩增方法的引物设计原则,利用PrimerExplorer V3(http://primer exp lorer.jp/e/)在线引物设计软件筛选出一套特异性较好的引物(图1);根据real-time PCR引物和探针的设计原则,利用Beacon Designer 软件设计1对引物和探针,预期扩增片段大小为278 bp。试验所用得引物和探针见表1,均有Sangon Biotech (Shanghai) Co.Ltd合成。
1.2.2 细菌培养和DNA模板制备 将试验涉及的沙门菌、大肠埃希菌、金黄色葡萄球菌、枯草芽胞杆菌、变形杆菌、痢疾杆菌、链球菌、多杀性巴氏杆菌、无乳链球菌、单核细胞增生李斯特菌和放线杆菌分别划线接种于LB或血液固体培养基,放到37℃培养箱里,培养16 h~24 h,挑取单个菌落于LB或血液培养液里,在37℃、180 r/min条件下,振荡培养16 h~48 h后,平板涂布培养法对各培养物进行细菌计数,调整菌液浓度至1.0×108CFU/mL,然后按10倍梯度稀释,其浓度分别为1.0×108CFU/mL~1.0×101CFU/mL等8个不同浓度的菌液。分别取不同浓度的菌液1 mL按照细菌基因组DNA提取试剂盒的操作步骤,提取细菌基因组DNA,作为LAMP、PCR及real-time PCR反应模板,于-20℃保存备用。

图1 沙门菌invA基因的LAMP引物设计示意图Fig.1 Schematic diagram of LAMP primer design for Salmonella invA gene表1 本试验检测沙门菌所使用的特异性引物和探针Table 1 The specific primers and probes designed for Salmonella detection
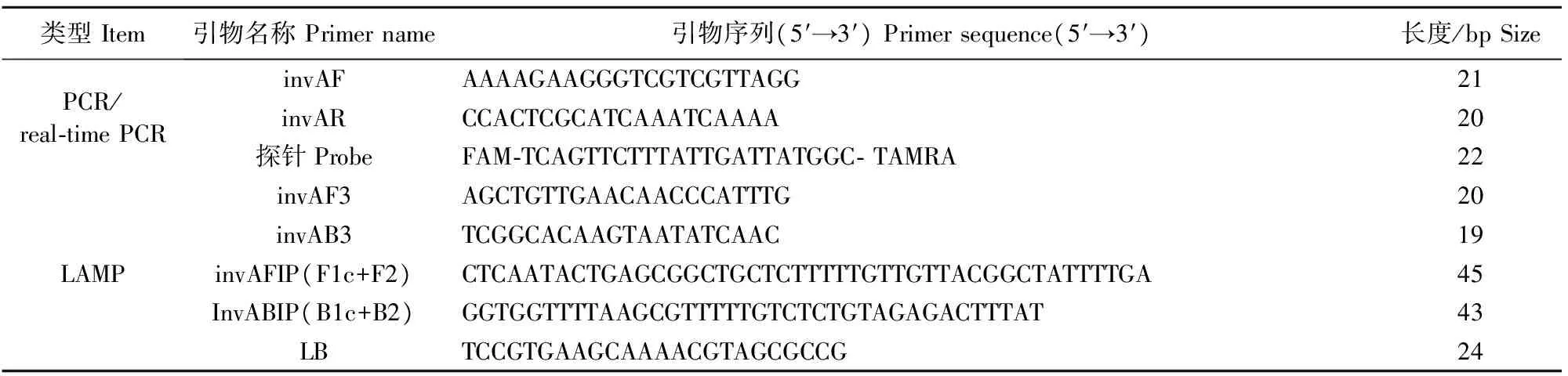
类型Item引物名称Primername引物序列(5′→3′)Primersequence(5′→3′)长度/bpSizePCR/real⁃timePCRinvAFAAAAGAAGGGTCGTCGTTAGG21invARCCACTCGCATCAAATCAAAA20探针ProbeFAM⁃TCAGTTCTTTATTGATTATGGC⁃TAMRA22LAMPinvAF3AGCTGTTGAACAACCCATTTG20invAB3TCGGCACAAGTAATATCAAC19invAFIP(F1c+F2)CTCAATACTGAGCGGCTGCTCTTTTTGTTGTTACGGCTATTTTGA45InvABIP(B1c+B2)GGTGGTTTTAAGCGTTTTTGTCTCTGTAGAGACTTTAT43LBTCCGTGAAGCAAAACGTAGCGCCG24
1.2.3 LAMP检测方法的建立及反应条件优化 根据文献[15],初步确定LAMP反应体系和反应条件,建立沙门菌LAMP检测方法。反应体系为:2.5 μL 10×ThermoPol缓冲液,1.5 μL MgSO4, 3 μL引物(包括内引物(20 μmol/L)、外引物(10 μmol/L)和环引物(15 μmol/L)各1 μL),3.5 μL dNTPs,2.5 μL甜菜碱(10 mol/L),1 μL Bst 2.0 DNA聚合酶,2 μL模板,1 μL钙黄绿素,最后加ddH2O水补足25 μL;同时设置1个空白对照,用水替代模板。放入60℃恒温水浴锅扩增1 h,80℃ 5 min使酶失去活性。取5 μL的扩增产物进行30 g/L琼脂糖凝胶电泳判断结果。在此反应体系和条件的基础上对内外引物浓度比例、dNTPs体积、Mg2+浓度、反应温度和扩增时间进行优化,其中内外引物的比例分别为5∶1、4∶1、3∶1、2∶1;dNTPs的体积为1.0、1.5、2.0、2.5、3.0、3.5、4.0、4.5 μL;MgSO4分别为1.0、1.5、2、2.5、3.0、3.5 μL;反应温度为54、56、58、60、62、64℃;扩增时间为30、40、50、60 min。LAMP反应条件和体系都在前一步优化的基础上进行反应,取5 μL扩增产物进行30 g/L琼脂糖凝胶电泳检测,确定最佳的反应体系和条件。最后,通过电泳方法和荧光染料方法进行判定,确定最佳条件和体系。
1.2.4 沙门菌LAMP的特异性和重复性试验 按照上述1.2.3优化的LAMP反应条件和体系,分别以提取的肠炎沙门菌、多杀性巴氏杆菌、大肠埃希菌、金黄色葡萄球菌、枯草芽胞杆菌、链球菌、无乳链球菌、变形杆菌 、痢疾杆菌、单核细胞增生李斯特杆菌和放线杆菌等11种食源性致病菌的DNA为模板进行扩增,产物用30 g/L琼脂糖凝胶电泳检测。同时,用建立的LAMP方法,以提取的肠炎沙门菌为模板进行扩增,至少重复3次,已确定建立的检测方法的重复性。
1.2.5 LAMP、常规PCR和real-time PCR方法的灵敏度比较 以肠炎沙门菌不同培养物(1.0×108CFU/mL~1.0×101CFU/mL)所对应的DNA为模板,空白对照用水代替。利用上述1.2.3优化的反应体系和反应条件进行LAMP扩增,反应产物通过电泳或钙黄绿素可视化检测,进行方法的灵敏度试验。以invAF和invAR为上、下游引物进行PCR反应, PCR反应体系20 μL,包括2×ESTaqMasterMix 10 μL,上、下游引物invAF和invAR各0.3 μL,模板2 μL, ddH2O 7.4 μL;PCR反应程序为:95℃ 5 min;95℃ 40 s,59℃ 30 s,30个循环;72℃ 1 min 50 s。扩增产物分别进行电泳,根据目的条带的亮度判断其灵敏度。在常规PCR引物基础上增加1个水解探针,进行real-time PCR反应,反应体系10 μL:包括2×PCR Master Mix 5 μL,上、下游引物invAF和invAR各0.2 μL,探针0.2 μL,模板1 μL,ddH2O 3.4 μL;反应条件为95℃ 5 min,94℃ 15 s,58℃ 30 s,72℃ 10 s,同时捕捉FAM信号,进行45个循环。根据实时荧光定量PCR扩增的标准“S”形曲线而变化的荧光信号强度,求出Ct值,然后把Ct值带入标准曲线就可以计算出该样品检测的起始浓度。
1.2.6 沙门菌病原人工污染的鱼粉及临床样品检测 取经检测未被沙门菌污染的鱼粉(10 g)9份,取1.2.2稀释好的不同浓度的肠炎沙门菌菌液(1.0×108CFU/mL~1.0×101CFU/mL)1 mL,与鱼粉匀浆液等体积混匀,其中阴性对照用PBS代替菌液。按照细菌基因组提取试剂盒说明提取细菌核酸,利用Nanodrop-2000微量核酸蛋白检测仪测定其浓度,各取2 μL作为LAMP、PCR和real-time PCR方法检测时的模板。
为了评价所建立的LAMP检测方法的优势,对新疆某种鸡场有一定沙门菌病临床症状的25只病鸡,用平板凝集试验方法进行初筛,结合细菌分离鉴定方法进行确诊。剖检后取肝脏、脾脏和肠内容等样品,并对这些样品进行病原分离鉴定,同时用细菌基因组DNA提取试剂盒分别提取这些组织的核酸。分别进行LAMP、PCR和real-time PCR检测,比较3种方法的检出率,并与病原分离的结果进行比较。
2 结果
2.1 沙门菌LAMP检测方法的建立
以提取的肠炎沙门菌(浓度为1.0×105CFU/mL)DNA为模板,进行LAMP反应,其产物进行荧光染料显色观察和30 g/L琼脂糖凝胶电泳分析。阳性结果荧光染料显色呈绿色,而空白对照为橙黄色(图2A)。琼脂糖凝胶电泳可见明显的特异性大小不一的梯形条带(图2B),而空白对照无梯形条带。结果表明,所建立的LAMP方法扩增的梯形条带为肠炎沙门菌invA的基因片段。
2.2 LAMP扩增最佳反应条件的确定
当内外引物浓度比例为3∶1(图3A)时,目的条带最亮;通过对反应试剂中加入的不同dNTPs和MgSO4的体积进行摸索,最终确定dNTPs为3.5 μL(图3B),MgSO4为1.5 μL(图3C),扩增温度为62℃(图3D),其LAMP扩增效率最佳,呈现典型的梯形条带;同时对反应的时间进行优化结果表明,当扩增反应时间在40 min时,即可以出现明显的梯形条带,50 min时条带几乎没有任何的变化趋于饱和(图3E),而在试验检测时可能会遇到样品模板浓度较低的情况,为了确保在较低浓度的沙门菌病原作为模板进行检测时,依然可以扩增出条带,因此确定最佳的反应时间为50 min。通过对反应体系和条件优化,最终确定25 μL的最佳反应体系为:2.5 μL 10×ThermoPol缓冲液,1.5 μL MgSO4, 4 μL引物(包括内外引物比例为2∶1和环引物为1 μL),3.5 μL dNTPs,2.5 μL甜菜碱(10 mol/L),1 μL Bst 2.0 DNA聚合酶,2 μL模板质粒,1 μL钙黄绿素,最后加ddH2O水补足25 μL;混匀并简短瞬离后,在60℃恒温水浴条件下扩增50 min后,80℃ 5 min使酶失去活性。

M.DNA标准DL 1 000;1. LAMP产物;2.阴性对照;A.钙黄绿素-可视化LAMP扩增产物;B.AMP扩增产物电泳分析
M.DNA Marker DL 1 000;1. LAMP products;2.Negative control;A.Calcein-visualization LAMP products;B.LAMP product electrophoresis
图2沙门菌LAMP检测方法的建立
Fig.2 The establishment of LAMP forSalmonelladetection
2.3 LAMP检测方法的特异性和重复性试验
采用已经优化好的沙门菌LAMP反应体系和条件,对 11株试验菌株进行LAMP扩增,来确认方法的特异性。图4电泳结果显示,只有沙门菌有预期的特征性梯形条带,而其他非沙门菌均没有条带,此方法的特异性较好。以沙门菌基因组DNA为模板进行多次重复试验,LAMP产物电泳结果均为阳性,空白对照结果为阴性 ,表明该方法重复性较好(图5)。
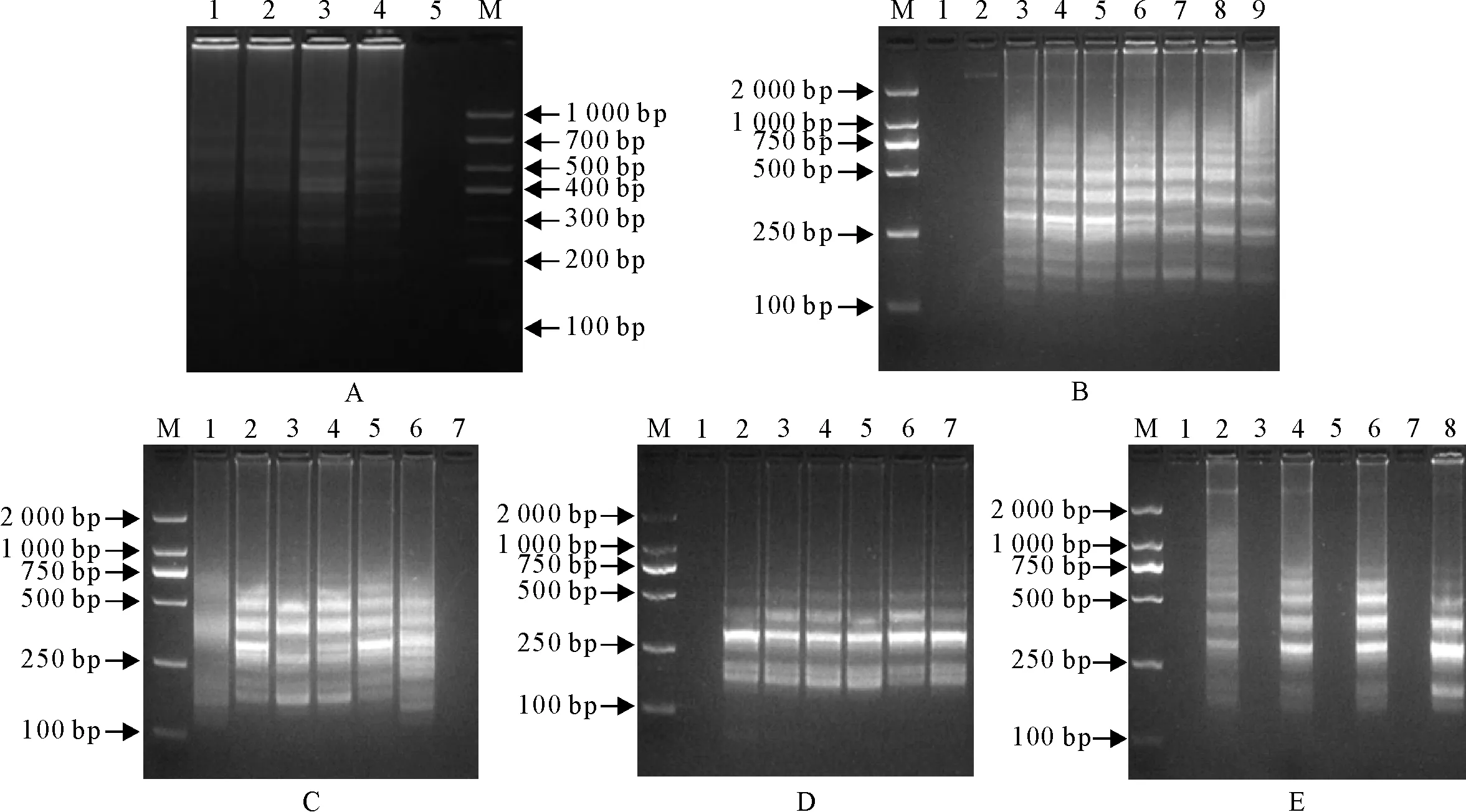
A.内外引物浓度比例的优化;M.DNA标准DL 1 000;1~4.引物比例分别为5∶1、4∶1、3∶1、2∶1;5.阴性对照;B.反应体系中dNTPs的优化;M.DNA标准DL 2 000;1.阴性对照;2~9. 1.0 μL、1.5 μL、2.0 μL、2.5 μL、3.0 μL、3.5 μL、4.0 μL、4.5 μL;C.反应体系中MgSO4的优化;M.DNA标准DL 2 000;1~6.1.0 μL、1.5 μL、2 μL、2.5 μL、3.0 μL、3.5 μL;7.阴性对照;D.反应温度的优化;M.DNA标准DL 2 000;1.阴性对照;2~7.54℃、56℃、58℃、60℃、62℃、64℃的扩增产物;E.反应时间的优化;M.DNA标准DL 2 000;1.阴性对照;2、4、6、8. 30 min、40 min、50 min、60 min的扩增产物;3,5,7.空白对照
A.Optimization of the primer ratios;M.DNA Marker DL 1 000; 1-4.Primer ratios of 5∶1,4∶1,3∶1,2∶1;5.Negative control; B.Optimization of the dNTPs concentration; M. DNA Marker DL 2 000; 1:Negative control; 2-9.dNTPs of 1.0 μL,1.5 μL,2.0 μL,2.5 μL,3.0 μL,3.5 μL,4.0 μL; C:Optimization of MgSO4;M.DNA Marker DL 2 000;1-6. MgSO41.0 μL,1.5 μL,2 μL,2.5 μL,3.0 μL,3.5 μL; 7.Negative control;D.Optimization of reaction temperature;M.DNA Marker DL 2 000; 1.Negative control; 2-7.Temperature 54℃,56℃,58℃,60℃,62℃,64℃ amplification products; E.Optimization of reaction time;M.DNA Marker DL 2 000; 1.Negative control; 2,4,6,8. 30 min,40 min,50 min,60 min amplification products; 3,5,7. The blank control
图3 LAMP方法检测沙门菌最适反应条件的优化
Fig.3 Optimization of reaction conditions of the LAMP for detectionSalmonella

M.DNA标准DL 1 000;1.以无菌水作为模板;2.以肠炎沙门菌的核酸作为模板;3~12.分别以多杀性巴氏杆菌、大肠埃希菌、金黄色葡萄球菌、枯草芽胞杆菌、链球菌、无乳链球菌、变形杆菌 、痢疾杆菌、单增李斯特菌和放线杆菌的基因组DNA作为模板
M.DNA Marker DL1 000; 1.Sterile water as template; 2.Salmonellaenteritidisas template; 3-12.Templates fromPasteurellamultocida,Escherichiacoli,Staphylococcusaureus,Bacillussubtilis,Streptococcus,Streptococcusagalactiae,Proteus,Shigella,ListeriamonocytogenesandActinobacillussp,respectively
图4 LAMP检测方法特异性分析结果
Fig.4 Analysis of specificity results of LAMP detection method

M.DNA标准DL 1 000;1.以无菌水为模板作为阴性对照;2~11.分别以肠炎沙门菌CVCC 3949的基因组DNA为模板进行扩增
M.DNA Marker DL 1 000; 1.Sterile water as a template as a negative control; 2-11.Template fromSalmonellaenteritidisCVCC 3949 genomic DNA,respectively as positive control
图5 LAMP检测方法的重复性分析结果
Fig.5 Analysis of reproducibility of results of LAMP detection method
2.4 LAMP、PCR和real-time PCR灵敏度比较
以提取的不同浓度的肠炎沙门菌基因组为模板,进行LAMP反应,肉眼可视化和电泳检测发现(图6A和6B),其最低检测浓度为1.0×102CFU/mL;而PCR最低可检测到1.0×105CFU/mL(图6C)。real-time PCR最低可检测模板的浓度为1.0×101CFU/mL,比LAMP方法灵敏度高1个数量级(图6D)。结果表明,建立的沙门菌LAMP方法的灵敏度与real-time PCR方法的灵敏度基本相当,比常规PCR方法高1 000倍。
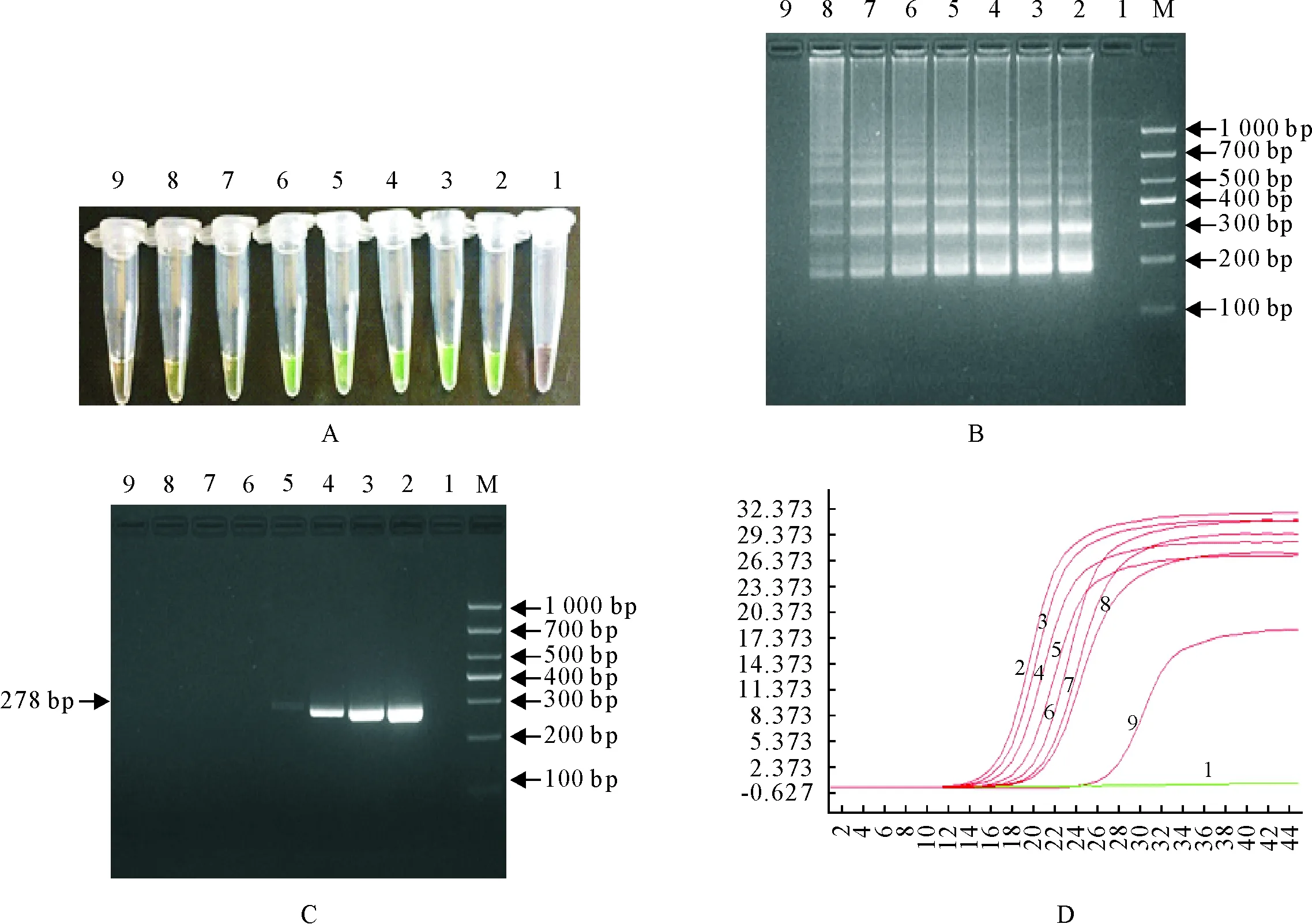
M.DNA标准 DL 1 000;1.阴性对照;2~9.肠炎沙门菌CVCC 3949浓度分别为1×108、1×107、1×106、1×105、1×104、1×103、1×102、1×101CFU/mL; A.钙黄绿素-LAMP;B.LAMP产物电泳;C.普通PCR产物电泳;D.real-time PCR
M. DNA Marker DL 1 000; 1.Negative control; 2-9.SalmonellaCVCC 3949 diluted of 1×108,1×107,1×106,1×105,1×104,1×103,1×102,1×101CFU/mL; A.Calcium chlorophyll-LAMP products; B.Electrophoresis of LAMP products; C.Electrophoresis of conventionl PCR products;D.real-time PCR
图6 LAMP(A,B)、普通PCR(C)和real-time PCR(D)方法检测沙门菌的灵敏度分析
Fig.6 Sensitivity comparison analyses of LAMP (A,B),common PCR(C) and real-time PCR (D) methods for detectingSalmonella
2.5 LAMP、PCR和real-time PCR方法检测沙门菌人工污染鱼粉及临床样品结果
不同浓度的肠炎沙门菌人工污染鱼粉后,用所建立的LAMP方法和real-time PCR方法进行检测其结果(表3),两种方法均可以从5.0×102CFU/mL的人工污染的沙门菌鱼粉中检测到该菌,而阴性对照鱼粉中未能检测到该菌;且与传统的细菌分离方法检测结果一致。而常规PCR方法检测的灵敏度为5.0×105CFU/mL ,阴性对照也未检测出该病原体。

表3 肠炎沙门菌人工污染鱼粉检测结果
注:+.阳性;-.阴性。
Note:+.Postive;-.Negative.
对临床疑似沙门菌病的病鸡,解剖后取肝脏、脾脏和肠内容物等共获的75份可疑病料。传统的细菌分离方法结果显示(表4)49份为阳性,26份为阴性,阳性率为65.4%;应用PCR检测方法得到阳性46份,阴性为29份,阳性率为61.3%;而LAMP技术和real-time PCR方法的检测结果一致,其阳性率为65.4%。说明LAMP检测方法对临床样品的检出率比常规PCR方法的高,与传统的细菌分离结果相符。3种方法检测结果与国家标准方法(GB4789.4-2016)检测结果的符合率分别为100%、93.7%、100%,LAMP检测方法的临床检出率比常规PCR方法的要高。

表4 细菌分离鉴定、LAMP、PCR和real-time PCR方法对临床样本的检测结果
3 讨论
沙门菌可以引起不同动物的多种多样的急性和慢性疾病,给人类经济和公共卫生造成了巨大的威胁。据美国疾病控制和预防中心(CDC)报告,美国公民每年由于感染沙门菌病而住院的病人达到1.5万多人,其中400多人死亡 ,而大多数感染者为老人和幼龄儿童;而这些食物中毒病例中大多数源于食用了污染沙门菌的禽产品和牛奶[15]。因此,在食品安全或者沙门菌食物中毒的临床诊断中,非常有必要建立一种快速、可靠和高灵敏的沙门菌检测方法。
本研究以沙门菌的invA为靶基因,利用软件设计了两对引物和一个环引物,建立了一种快速、高效检测沙门菌的LAMP方法,该方法操作简单,可目视化,通过电泳和荧光染料等途径都可以判定结果。其优点是其他分子生物学方法不可替代的,用该方法对临床样本的检测结果与行业内标准方法的检出率一致;对人工污染的鱼粉样品检测的灵敏度为5.0×102CFU/mL,比常规PCR方法高1 000倍,比real-time PCR方法检测灵敏度结果低一个数量级,这可能是real-time PCR的引物设计增加了一个特异性的水解探针的缘故[11];但荧光定量PCR对试验技术和条件要求相对较高。LAMP技术的核心环节是引物设计、稳定的反应体系和反应条件以及操作加样无菌区域的控制。引物必须同时满足特异性好和高度保守的核苷酸特异性区域,是保证LAMP检测方法成功与否的一个关键因素。而本研究选择的靶基因invA高度保守,国内外学者利用该基因,设计PCR和LAMP特异性引物的相关研究报道比较多。Kokkios P A等[16]学者建立了LAMP方法用于检测食品中沙门菌的含量;Abdelaziz N M等[17]建立PCR方法检测鸡肉中沙门菌,以上两种方法都是以高度保守的invA基因为检测靶标。本试验对LAMP反应组分中的Mg2+浓度、dNTP浓度、扩增温度和反应时间等都进行了优化,确定了最佳的反应体系和反应条件。LAMP扩增反应效率较高,当反应液中加入钙黄绿素后,有扩增产物时为绿色,反之,为橙黄色。另外,在对临床采集的75个样本(阴性26个)进行检测时,发现所建立的LAMP方法的检出率与常规的细菌分离鉴定方法一致。
目前,判定某种检测方法的优劣主要从灵敏度、操作方便和实用性这3个指标去衡量,能否在基层推广应用也是重要因素。传统的检测方法是基于微生物学知识,通常需要增菌培养,得出确切的诊断结果至少需要1周左右。就目前对沙门菌病诊断技术的研究趋势来看,其检测方法的研究方向主要围绕经济和高效这两个方面。而当前的分子生物学检测方法主要包括PCR、real-time PCR、套式PCR及PCR-基因芯片等方法,都具有较高的灵敏度和精确度,但需要借助昂贵的仪器设备和专业的技术人员操作等,一般的基层单位很难满足[18-20]。Chen Z等[21]基于沙门菌的目标invE基因和3个血清型特异性的基因(flic、lygD和STM4495)建立了LAMP技术,最低可检测出2.0×101CFU/mL沙门菌,而常规PCR方法的最低检测限为2.33×103CFU/mL。本研究建立的沙门菌LAMP方法其灵敏度为1.0×102CFU/mL。Less D等[22]利用免疫胶体金技术原理,建立了一种环介导等温扩增联合横向流动试纸条检测ZiKa病毒的方法,该方法无需电泳,35 min内肉眼直接可以读出结果,其灵敏度高,对于2 μL的LAMP反应产物作为样品,其最低可检测到单个拷贝数。但目前该方法仅局限于血液样本的检测。Wang D等[23]应用氧化还原反应建立了快速检测沙门菌的电化学方法,其最低检测限为7.6×102CFU/mL~6.0×102CFU/mL。Wu G P等[24]将LAMP技术与荧光定量PCR方法结合,结合各自方法的优缺点并进行改善,建立了一种Rti-LAMP方法应用于食物表面沙门菌检测,最低可以检测出6 CFU/2.5 μL(模板),整个反应在3.5 h内就可以完成。Yang Q等[25]建立的LAMP-BART技术,需要在反应体系中加入特定的荧光素酶,其数据处理复杂,反应时间较长,容易出现假阳性结果,应用于食品和饲料样品的检测,其灵敏度达到了104CFU/25 g~106CFU/25 g样品。本研究建立的可目视化LAMP方法,增加了一条环引物,提高了扩增效率,缩短了反应时间,扩增反应在1 h内就可以完成,其灵敏性和特异性比较好;并且整个反应结束后,无需开盖通过颜色变化即可判断结果,避免了二次污染产生的假阳性问题。
本研究以沙门菌高度保守且特异性较强的invA基因为检测靶标,建立的可目视化环介导等温扩增方法具有特异性强、敏感度高和结果可目视等优点,可以在62℃恒温条件下,60 min内完成对沙门菌的检测,对仪器和试验条件要求比较低,其灵敏度可以达到1.0×102CFU/mL,该方法对于沙门菌的临床检测、食品安全和公共卫生机构监控提供了一个有效的现场检测技术保障。
参考文献:
[1] Chandrajit L,Shrikant P,Radhakrishnan S,et al.Interactome analyses ofSalmonellapathogenicity islands reveal SicA indispensable for virulence[J].J Theor Biol,2014,363:188-197.
[2] Ying J,Li W,Ying Z,et al.QseB mediates biofilm formation and invasion inSalmonellaentericaserovartyphi.[J].Microb Pathogenesis,2017,104:6-11.
[3] 陆承平.兽医微生物学.[M].4版.北京:中国农业出版社,2007.
[4] Sabatino R,Perrone A,Cuomo M,et al.Analytical criticalities associated to different immunological methods for serum free light chain detection in plasma cell dyscrasias:a description of particular clinical cases.[J].Int J Mol Sci,2017,18(4):1-9.
[5] Wang L F,Xu L,Luo S Q,et al.Diagnosis ofStrongyloidesstercoralisby morphological characteristics combine with molecular biological methods.[J].Parasitol Res,2017,116(4):1159-1163.
[6] Malorny B,Hoorfar J,Bunge C,et al.Multicenter validation of the analytical accuracy ofSalmonellaPCR:towards an International Standard[J].Appl Environ Microb,2003,69(1):290-296.
[7] 陈小玲,李永清,孙慧玲,等.沙门氏菌fimY基因的原核表达及禽沙门氏菌抗体检测ELISA研究[J].中国家禽,2012(13):18-22.
[8] Notomi T,Okayama H,Masubuchi H,et al.Loop-mediated isothermal amplification of DNA.[J].Nucleic Acids Res,2000,28(12):E63.
[9] Haridas D V,Pillai D,Manojkumar B,et al.Optimisation of reverse transcriptase loop-mediated isothermal amplification assay for rapid detection ofMacrobrachiumrosenbergii,noda virus and extra small virus inMacrobrachiumrosenbergii[J].J Virol Methods,2010,167(1):61-67.
[10] Techathuvanan C,Draughon F A,D'Souza D H.Loop-mediated isothermal amplification (LAMP) for the rapid and sensitive detection ofSalmonellatyphimuriumfrom pork.[J].J Sci Food Agr,2010,75(3):165-172.
[11] Cossu A,Levin R E.Rapid conventional PCR and real-time-qPCR detection of low numbers of from ground beef without enrichment[J].Food Sci Biotechnol,2014,28(2):96-105.
[12] Nde C W,Fakhr M K,Doetkott C,et al.An evaluation of conventional culture,invA PCR,and the real-time PCR iQ-Check kit as detection tools forSalmonellain naturally contaminated premarket and retail turkey[J].J Food Prot,2008,71(2):386-391.
[13] Parker W D,Lungu B,Berghaus R D,et al.Comparison of real-time PCR with conventional PCR and culture to assess the efficacy of a live attenuatedSalmonellaentericaserovartyphimuriumvaccine againstSalmonellaentericaserovarenteritidisin commercial leghorn chicks vaccinated under field an[J].Avian Dis,2011,55(2):248-254.
[14] Lefebre M D,Galán J E.The inner rod protein controls substrate switching and needle length in aSalmonellatype III secretion system.[J].P Natl Acad Sci USA,2013,111(2):817-822.
[15] Kokkinos P A,Ziros P G,Bellou M,et al.Loop-mediated isothermal amplification (LAMP) for the detection ofSalmonellain food[J].Food Anal Method,2014,7(2):512-526.
[16] Abdelaziz N M.Detection ofSalmonellaspecies in chicken carcasses using genus specific primer belong to invA gene in Sohag city,Egypt[J].Vet World,2016,9(10):1125-1128.
[17] Hoenigl M,Prattes J,Spiess B,et al.Performance of galactomannan,beta-d-glucan,Aspergilluslateral-flow device,conventional culture,and PCR tests with bronchoalveolar lavage fluid for diagnosis of invasive pulmonary aspergillosis[J].J Clin Microbiol,2014,52(6):2039-2045.
[18] Akbarmehr J,Salehi T Z,Brujeni G H N.Identification ofSalmonellaisolated from poultry by MPCR technique and evaluation of their hsp groEL gene diversity based on the PCR-RFLP analysis[J].Afr J Microbiol Res,2010,4(15):112-118.
[19] Islam M A,Großebrinkhaus C,Pröll M J,et al.PBMC transcriptome profiles identifies potential candidate genes and functional networks controlling the innate and the adaptive immune response to PRRSV vaccine in Pietrain pig[J].PLoS One,2017,12(3):e0171828-e0171851.
[20] Haddad M H F,Ghasemi E,Maraghi S,et al.Identification ofLeishmaniaspecies isolated from human cutaneous leishmaniasis in Mehran,Western Iran using nested PCR[J].IRAN J Parasitol,2016,11(1):65-72.
[21] Chen Z,Zhang K,Yin H,et al.Detection ofSalmonellaand several commonSalmonellaserotypes in food by loop-mediated isothermal amplification method[J].Food Sci Human Wellness,2015,4(2):75-79.
[22] Lee D,Yong S,Chung S,et al.Simple and highly sensitive molecular diagnosis of zika virus by lateral flow assays[J].Anal Chem,2016,88(24):12272-1229.
[23] Wang D,Wang Z,Chen J,et al.Rapid detection ofSalmonella,using a redox cycling-based electrochemical method[J].Food Control,2016,62:81-88.
[24] Wu G P,Chen S H,Levin R E.Rapid real-time loop-mediated isothermal amplification combined with coated activated carbon for detection of low numbers ofSalmonellaenterica,from lettuce without enrichment[J].Food Control,2015,56:47-52.
[25] Yang Q,Domesle K J,Wang F,et al.Rapid detection ofSalmonellain food and feed by coupling loop-mediated isothermal amplification with bioluminescent assay in real-time[J].BMC Microbiol,2016,16(1):1-10.

