一株源自深海沉积物产中温α-淀粉酶菌株的鉴定及其酶学性质的研究
,,,,, ,,*,
(1.扬州大学环境科学与工程学院,海洋科学技术研究所,江苏扬州 225100; 2.河北农业大学植物保护学院,河北保定 071001)
α-淀粉酶是能够随机切割α-1,4-糖苷键降解淀粉和其他碳水化合物的内切酶类。α-淀粉酶占据了工业酶制剂市场的25%~30%,在淀粉加工、酿造、酒精生产、纺织和其他工业领域具有广泛的应用[1]。传统的淀粉糖化生物工艺通常涉及液化和糖化两个过程。研究发现,这两个过程均在高温下进行:液化所需的温度为80~90 ℃,而糖化所需的温度为55~60 ℃[2]。嗜热α-淀粉酶在节约成本、溶解底物、降低粘度和减少微生物污染风险方面具有明显的优势。极端嗜热菌的最适生长温度高于80 ℃,是产生嗜热α-淀粉酶的重要来源[3],从而引起了国内外研究者的关注。因此,嗜热α-淀粉酶在淀粉糖化生物工艺方面具有潜在的应用价值。发掘具有新型功能的α-淀粉酶,特别是嗜热性强的α-淀粉酶,对于科学研究以及工业应用都具有重大的研究意义。而由于极端嗜热α-淀粉酶的最佳活性温度超出了工业需求的温度,所以中度嗜热α-淀粉酶比极端嗜热α-淀粉酶在工业方面具有更大的应用价值。例如,最适温度在50~65 ℃的α-淀粉酶能够有效地减少副产品的形成和降低能源需求,在淀粉液化方面比极端嗜热α-淀粉酶更具有潜在优势。
大多数α-淀粉酶来源于陆地微生物的芽孢杆菌属,但是从海洋环境中获得的α-淀粉酶具有独特的性质,受到越来越多的关注。与陆地环境相比,海洋环境具有高压、低温或高温(热液口)、高盐、寡营养等典型的环境特征,蕴藏着丰富、性质独特的海洋微生物。深海微生物由于具有独特的代谢途径,从而成为提供新型极端酶的重要资源库。目前,已有一些源自深海细菌α-淀粉酶的报道:Bacillussp. SCSIO 15121[4-5],Nocardiopsissp. 7326[6],Geobacillussp. 4j[7],Flammeovirgapacifica[8],LuteimonasabyssiXH031[9],Thermotogamaritima[10]和Rhodothermusmarinus[11]。此外,几种源自深海极端嗜热古菌火球菌属和热球菌属的嗜热α-淀粉酶也已经被研究[2,12]。这些源自海洋环境微生物的α-淀粉酶在热稳定性、耐盐性和pH稳定性等方面具有特殊的性质,不同于源自陆地微生物的α-淀粉酶。最近,Homaei等人综述了源自海洋微生物的淀粉酶[13];Hiteshi和Gupta探讨了α-淀粉酶的热适应性机制[14]。
本文从西南印度洋洋中脊非活动热液区的深海沉积物中分离出一株能够有效地降解淀粉的菌株,研究其生长的最适温度和最适pH,并对其所产生的α-淀粉酶的酶学性质进行了初步研究,为淀粉糖化过程中进行淀粉的降解提供了深海微生物资源。
1 材料与方法
1.1 材料与仪器
深海沉积物样本编号为DY11-15-20 TVG11,是由大洋一号于2009年1月巡航期间,在西南印度洋洋中脊通过电视捕获取样器在1985 m水深处收集得到;蛋白胨及酵母提取物分析纯,英国OXOID公司;基因组提取试剂盒美国Omega Bio-tek公司;2×Taq PCR MasterMix美国New England Biolab公司;淀粉分析纯,美国Sigma公司;Tris、甘氨酸分析纯,美国AMRESCO公司;氯化钠、氯化钾、琼脂粉、碘、碘化钾、醋酸、磷酸钠、氢氧化钠、盐酸、戊二醛、磷酸二氢钾、磷酸氢二钠、乙醇、3,5-二硝基水杨酸、苯酚分析纯,中国国药集团;稀释LB 培养基蛋白胨1.0 g,酵母提取物0.5 g,氯化钠1.0 g,并加入人工海盐将盐度调到35‰;完全溶解后,1×105Pa湿热灭菌20 min。
THZ-92 C恒温振荡器上海博迅实业有限公司;SHP-160型生化培养箱上海三发科学仪器有限公司;722可见紫外分光光度计上海菁华科技仪器有限公司;S-4800电子显微镜日本日立公司;C1000 Touch PCR 仪美国Bio-Rad公司;Heraeus Pico17微型台式离心机美国Thermo Fisher 公司;FE20pH计梅特勒-托利多仪器(上海)公司;CT15RE台式微量高速离心机日本HITACHI公司。
1.2 实验方法
1.2.1淀粉降解菌的筛选称取0.5 g深海沉积物样本,接种到20 mL稀释LB液体培养基中,28 ℃、180 r/min培养24 h。取新鲜培养物,划线接种于稀释LB 固体培养基,倒置于恒温培养箱中培养。挑取生长较好的单菌落,进一步划线分离纯化3~4次。将多次划线分离得到的单菌落,接种于稀释LB培养基,28 ℃、180 r/min培养24 h。分别取2 μL培养物接种在含有1%可溶性淀粉的稀释LB培养基平板上,过夜培养。碘液染色5 min,轻轻倒去碘液,菌落周围形成透明圈的菌株即为能够降解淀粉的菌株。选取形成透明圈最大的菌株,命名为TVG11-1,进行后续的研究。
1.2.2菌株TVG11-1的形态鉴定将菌株TVG11-1接种到35‰盐度的稀释LB液体培养基中,在最适温度45 ℃振荡培养至OD600为0.5~0.6。取1 mL菌液至无菌1.5 mL离心管,8000 r/min离心5 min,去上清。取无菌去离子水重悬洗涤细胞,重复两次,使用2.5%戊二醛在4 ℃温度下固定样品2 h。取1 mL 0.1 mol/L PBS缓冲液重悬洗涤细胞,重复三次,分别使用30%、50%、70%、80%、90%及100%乙醇对样品进行梯度脱水,每个梯度加入乙醇后静置10 min。经过临界点干燥和喷金处理后,通过扫描电镜对菌株TVG11-1的形态学特征包括细胞形态,大小和表面纹理进行分析。
1.2.3菌株TVG11-1最适生长温度和pH的确定将过夜活化的菌株TVG11-1按1%接种量接种到35‰盐度的稀释LB液体培养基中。选择30~80 ℃温度范围,振荡培养6 h。通过测定不同温度条件下菌株TVG11-1培养物在600 nm处吸光度值,以确定其最适生长温度。
选择不同缓冲体系(浓度20 mmol/L):pH5.0和pH5.5(醋酸-乙酸钠体系),pH6.0、pH6.5、pH7.0和pH7.5(磷酸钠-氢氧化钠体系),pH8.0和pH8.5(Tris-盐酸体系),pH9.0和pH9.5(甘氨酸-氢氧化钠体系),对生长培养基的pH进行调整。45 ℃振荡培养6 h,通过测定不同pH条件下菌株TVG11-1培养物在600 nm处吸光度值,以确定其最适生长pH。
1.2.4菌株TVG11-1的分子生物学鉴定在最适温度45 ℃和pH7.0条件下,培养菌株TVG11-1 18 h。取2 mL过夜培养物,利用OMEGA基因组提取试剂盒提取菌株TVG11-1的基因组DNA。以菌株TVG11-1的基因组DNA为模板,以gyrB-F(5′-TTG RCG GHR GYG GHT ATA AAG T-3′)和gyrB-R(5′-TCC DCC STC AGA RTC WCC CTC-3′)为引物,进行gyrB基因的PCR扩增[15]。PCR反应体系为25 μL:12.5 μL 2×Taq PCR MasterMix,0.2 μmol/L的正向引物和反向引物,10 ng基因组DNA。PCR程序为:95 ℃ 3 min,94 ℃ 30 s,55 ℃ 45 s,72 ℃ 60 s 共35个循环,72 ℃ 5 min。将PCR产物进行1%琼脂糖凝胶电泳分析,并且由生工生物技术有限公司(中国上海)进行测序。
通过BLAST,检索GenBank数据库中与菌株TVG11-1的gyrB基因序列相似的序列。选取具有95%相似性的序列,构建系统发育树。利用CLUSTAL X 2.0对相似性最近的芽孢杆菌属序列进行多重比对[16]。以相似性低、部分注释的芽孢杆菌gyrB基因序列作为外群。采用邻接法和最大似然法构建系统发育树[17],bootstrap设为1000次。
1.2.5粗酶液制备和酶活分析取不同条件下菌株TVG11-1的培养物,进行冷冻离心(4 ℃,10000 r/min,20 min),取上清液作为粗酶液。
采用3,5-二硝基水杨酸(DNS)法[18],测定淀粉水解过程中还原糖的产量,从而进行α-淀粉酶活力的分析。反应体系为2 mL:1.5 mL 1.0%的可溶性淀粉和0.5 mL的粗酶液。在不同条件下进行反应,加入2 mL DNS,终止反应,混匀后100 ℃下加热10 min。反应物冷却至室温后测定540 nm处的OD值。α-淀粉酶的活力单位定义为每分钟产生1 μmol还原糖的酶量。实验重复三次,取平均值,并计算标准差。
1.2.6菌株TVG11-1的生长温度对α-淀粉酶产量的影响将过夜活化后的菌株TVG11-1按1%的比例接种到pH7.0,盐度为35‰的稀释LB液体培养基,在不同温度下(25、30、40、45、50、55、60 ℃)过夜培养18 h。按照上述方法制备粗酶液,测定α-淀粉酶活性,从而分析菌株TVG11-1的生长温度对α-淀粉酶产量的影响。
1.2.7淀粉含量对于菌株TVG11-1α-淀粉酶产量的影响将过夜活化菌株TVG11-1,分别接种到含有不同可溶性淀粉含量(0.2%~1.0%)并且盐度为35‰的稀释LB液体培养基中,在最适温度和pH的条件下过夜培养18 h。按照上述方法制备粗酶液,测定α-淀粉酶活性,计算相对酶活,从而确定淀粉含量对菌株TVG11-1α-淀粉酶产量的影响。相对酶活计算公式,如式(1):
相对酶活力(%)=不同淀粉浓度实验组测得OD540值/不含淀粉实验组测得OD540值×100
式(1)
1.2.8菌株TVG11-1的生长周期与α-淀粉酶产量关系将过夜活化后的菌株TVG11-1在最适温度和pH的条件下,按1%的比例接种到pH7.0,盐度为35‰的LB稀释液体培养基振荡培养。通过测定菌株TVG11-1培养物在不同时间(2.0、3.0、3.5、4.0、4.5、5.0、5.5、6.0、6.5、7.0、7.5、8.0、8.5、9.0、9.5、10.0、11.0、12.0、13.0、14.5 h)的OD600值,绘制其生长曲线。每次取样测定菌液OD600时,按照上述方法制备粗酶液,测定α-淀粉酶活性,从而确定菌株TVG11-1的生长周期与α-淀粉酶产量的关系。
1.2.9反应温度对菌株TVG11-1α-淀粉酶活性的影响将菌株TVG11-1在最适温度和pH的条件下,过夜培养18 h。按照上述方法制备粗酶液,测定在pH7.0时,菌株TVG11-1α-淀粉酶在35~85 ℃的酶活,从而确定该酶的最适反应温度。使用以下缓冲液(终浓度为20 mmol/L)调整反应的pH:pH5.0和pH5.5(醋酸-乙酸钠体系),pH6.0、pH6.5、pH7.0和pH7.5(磷酸钠-氢氧化钠体系),pH8.0和pH8.5(Tris-盐酸体系),pH9.0和pH9.5(甘氨酸-氢氧化钠体系),然后进行菌株TVG11-1α-淀粉酶酶活力分析,计算相对酶活,从而确定该酶酶活的最适pH。相对酶活计算公式,如式(2):

式(2)
1.2.10反应pH对菌株TVG11-1α-淀粉酶活性的影响将菌株TVG11-1在最适温度和pH的条件下,过夜培养18 h。按照上述方法制备粗酶液,测定在最适温度下,菌株TVG11-1α-淀粉酶在pH在5.0~9.5条件下酶活,从而确定该酶的最适反应pH。使用以下缓冲液(终浓度为20 mmol/L)调整反应的pH:pH5.0和pH5.5(醋酸-乙酸钠体系),pH6.0、pH6.5、pH7.0和pH7.5(磷酸钠-氢氧化钠体系),pH8.0和pH8.5(Tris-盐酸体系),pH9.0和pH9.5(甘氨酸-氢氧化钠体系)。相对酶活计算同式(2)。
1.3 数据处理
采用Excel处理数据,计算平均值和标准差;利用CLUSTAL X 2.0软件进行序列多重比对,并用MEGA 5.0软件构建系统发育树。
2 结果与分析
2.1 菌株TVG11-1的特征
通过划线分离获得多株可降解淀粉的菌株,其中TVG11-1菌落生长最快,菌落最大。菌株TVG11-1革兰氏染色呈阳性,在电子显微镜观察下呈杆状(图1A)。对含有淀粉的琼脂培养基上过夜培养的TVG11-1菌落,使用革兰氏碘溶液染色后,能够观察到明显的透明圈(图1B),表明菌株TVG11-1能够产生水解淀粉的α-淀粉酶。进一步的研究发现,菌株TVG11-1的最适生长温度为45 ℃(图2A),最适生长pH为7.0(图2B)。菌株TVG11-1已保存在中国农业菌种保藏中心(编号ACCC19897)。

图1 菌株TVG11-1的电镜照片(A)及其在含有淀粉的平板上形成的透明圈(B)Fig.1 Scanning electron micrograph of the strain TVG11-1(A)and its clear zone in starch agar-iodine plate(B)

图3 菌株TVG11-1与相关菌株的系统发育树Fig.3 Phylogenetic tree of the strain TVG11-1 and its relatives注:括号内为GenBank登录号;线段0.05表示序列差异的分支长度;发育树节点的数值表示Bootstrap值。
根据上海生工分析测序结果,菌株TVG11-1包含了894个核苷酸的gyrB基因序列,已被递交至GenBank数据库,登录号为KX265578。用BLAST程序,对菌株TVG11-1的gyrB基因序列与GenBank 数据库中已收录的其他相似序列进行核酸序列同源性比较。选取了17个具有代表性的gyrB基因序列用于构建系统发育树,其中10条序列与KX265578的相似度在98%以上,其余7条序列与KX265578同源性在90%。所构建的系统发育树(图3)表明,在这18个gyrB基因序列组成的系统发育树中,菌株TVG11-1(KX265578)与解淀粉酶芽孢杆菌聚在一个分支,该分支的支持率为100%,因此本文将菌株TVG11-1确定为解淀粉酶芽孢杆菌。关于解淀粉酶芽孢杆菌的产α-淀粉酶条件已有相关研究[19-22];也有利用分子技术对来自该菌α-淀粉酶进行分离,进一步对其结构及性质的相关报道[23-26]。

图2 菌株TVG11-1生长的最适温度(A)及最适pH(B)Fig.2 The optimal temperature(A) and pH(B)of strain TVG11-1
2.2 不同温度对TVG11-1 α-淀粉酶产量的影响
如图4所示,随着温度的增加,菌株TVG11-1的OD600值和其α-淀粉酶的OD540值均呈现先增加后减少的趋势,表明菌株的生长量与其α-淀粉酶的产量相吻合。在培养温度为40 ℃时,菌株TVG11-1α-淀粉酶产量最高,而在该菌株最适生长温度45 ℃时,其α-淀粉酶产量减少。由此可见,40 ℃是菌株TVG11-1产生α-淀粉酶的最适温度,不同于该菌株的最适生长温度(45 ℃)。

图4 菌株TVG11-1的生长温度对其α-淀粉酶产量的影响Fig.4 Effect of growth temperature of the strain TVG11-1 on α-amylase production
2.3 淀粉含量对菌株TVG11-1 α-淀粉酶产量的影响
如图5所示,与不加可溶性淀粉的对照组相比,培养基中加入0.2%的可溶性淀粉时,菌株TVG11-1α-淀粉酶的产量最高,并随着可溶性淀粉含量的增加,该酶的产量逐渐减少。当培养基中含有0.8%和1%可溶性淀粉时,菌株TVG11-1α-淀粉酶的产量要低于不加淀粉的对照组的产量。由此可见,菌株TVG11-1α-淀粉酶是一种不依赖于淀粉底物诱导的胞外酶。Konsula和Liakopoulou-Kyriakides研究了枯草芽孢杆菌产中温α-淀粉酶,结果表明该酶也是不依赖淀粉底物诱导[21],与本研究的结果相一致。

图5 淀粉含量对菌株TVG11-1 α-淀粉酶产量的影响Fig.5 Effect of soluble starch contents on the TVG11-1 α-amylase activity
2.4 菌株TVG11-1的生长周期与其α-淀粉酶活性的关系
为了分析菌株TVG11-1的生长周期与其α-淀粉酶活性的关系,测定不同培养时间内的培养物的OD600值和α-淀粉酶活性。如图6所示,菌株TVG11-1α-淀粉酶的活性在培养时间为9 h时达到最大值(122.7 U/mL),而该菌株处于指数生长期。当菌株TVG11-1处于指数生长期后期,随着生长时间的延长,菌株TVG11-1α-淀粉酶活性逐渐下降。由此,菌株TVG11-1α-淀粉酶的活性时间进程曲线与该菌株的生长曲线趋势总体相符。

图6 菌株TVG11-1的生长曲线与其α-淀粉酶活性的关系Fig.6 Relationship between growth curve of the strain TVG11-1 and its α-amylase activity
2.5 反应温度对菌株TVG11-1 α-淀粉酶活性的影响
如图7所示,菌株TVG11-1α-淀粉酶在35~85 ℃内均有活性。随着温度从35 ℃升高到60 ℃,菌株TVG11-1α-淀粉酶活性也随之增加。然而,当温度从60 ℃继续升高至80 ℃时,该α-淀粉酶的活性呈下降趋势。因此,60 ℃是菌株TVG11-1α-淀粉酶反应的最适温度。

图7 反应温度对菌株TVG11-1 α-淀粉酶活性的影响Fig.7 Effect of reaction temperature on the TVG11-1 α-amylase
菌株TVG11-1α-淀粉酶活性的最适温度(60 ℃)不同于该菌株的最适生长温度(45 ℃)。与本研究结果相一致的相关研究已有很多报道,例如,Igarashi等人所分离出一株芽孢杆菌的最适生长温度为30 ℃,但该菌株所产生的α-淀粉酶在55 ℃时具有最大的酶活性[27]。同样,Sodhi等人发现芽孢杆菌PS-7的最适生长温度(37 ℃)也不同于其α-淀粉酶的最适反应温度(60 ℃)[28]。类似的结果还存在于枯草芽孢杆菌TN106(pAT 5)[29]和链霉菌IMD 267[30]。但是,有些芽孢杆菌的最适生长温度与其α-淀粉酶的最适反应温度相同[31-32]。因此,菌株的最适生长温度与其产生的α-淀粉酶最适反应温度是否相同,可能与菌株的生存环境相关。
2.6 反应pH对菌株TVG11-1 α-淀粉酶活性的影响
不同反应pH对TVG11-1菌株α-淀粉酶活性的影响,如图8所示,菌株TVG11-1α-淀粉酶在pH5.5~9.0范围内有活性,而当pH小于5.0时,该菌株α-淀粉酶无法降解淀粉。随着pH的增加,菌株TVG11-1α-淀粉酶的活性呈现先增加后减少的趋势。当反应pH为6.5时,菌株TVG11-1α-淀粉酶的活性最高,因此该酶的最适反应pH为6.5。这些结果表明,菌株TVG11-1α-淀粉酶在中性pH附近活性较高,在酸性条件下活性几乎消失,但在偏碱性环境下仍具有40%以上相对酶活性。在最适条件下培养9 h,该酶平均酶活力为122.7 U/mL。
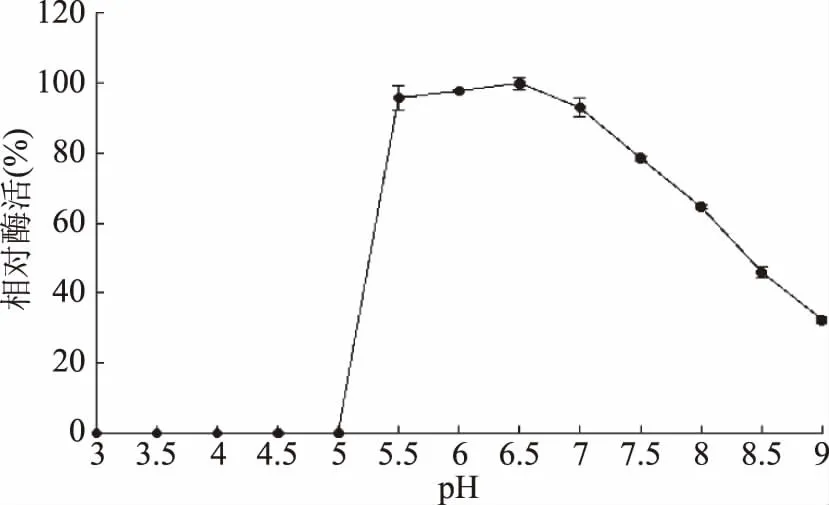
图8 反应pH对菌株TVG11-1 α-淀粉酶活性的影响Fig.8 Effect of the reaction pH on the TVG11-1 α-amylase
与其他芽孢杆菌所产生的α-淀粉酶最适pH相比,菌株TVG11-1α-淀粉酶与已报道的枯草芽孢杆菌、深海芽孢杆菌和地衣芽孢杆菌NH1所产生的α-淀粉酶的最适反应pH相同[21,33-34]。然而,芽孢杆菌GM8901和芽孢杆菌TS-23所产生的α-淀粉酶具有相同的最适反应温度,但是它们的最适反应pH不同于菌株TVG11-1α-淀粉酶。芽孢杆菌种GM8901α-淀粉酶在pH11.0~12.0时表现出了最强活性[35],而芽孢杆菌种TS-23α-淀粉酶的最适pH为9[36]。另外,菌株TVG11-1α-淀粉酶与源自其它海洋微生物的α-淀粉酶具有相似的最适pH以及相同的最适温度[37-39]。与上述研究结果不同的是,芽孢杆菌Bacillussp.Ferdowsicous的α-淀粉酶在pH低至4.5时,该酶仍具有最强的活性[40]。因此,源自不同环境的微生物所产生的α-淀粉酶在pH的适应性方面存在着差异性。
3 结论
本文首次从西南印度洋洋中脊非活动热液区的深海沉积物中分离出一株能够有效降解淀粉的菌株TVG11-1。通过电镜和分子生物学鉴定,该菌株为解淀粉芽孢杆菌。菌株TVG11-1最适的生长温度和pH分别为45 ℃和pH7.0,其所产生的α-淀粉酶是一种不依赖淀粉底物的胞外中温淀粉酶,其最适反应温度为60 ℃,最适反应pH为6.5。在最适条件下培养9 h,该酶平均酶活力为122.7 U/mL,高于王磊[41]分离于芽孢杆菌的α-淀粉酶(6.4~10.4 U/mL);同时高于来源于ThermococcussiciuliHJ21[42]的高温酸性α-淀粉酶(19.6 U/mL)。作为分离自热液区菌株的α-淀粉酶,不仅具有对高温的耐受性,还具有中温的最适反应温度,在轻工业节能生产方面有较大的应用价值。本研究分离获得的菌株TVG11-1为食品工业降解淀粉提供了菌种资源,其对淀粉降解能力的基因资源有待进一步开发和利用。
[1]Pandey A,Nigam P,Soccol CR,et al. Advances in microbial amylases[J]. Biotechnology and Applied Biochemistry,2000,31(Pt 2)(2):135-152.
[2]Vieille C,Zeikus GJ. Hyperthermophilic enzymes:sources,uses,and molecular mechanisms for thermostability[J]. Microbiology and Molecular Biology Reviews,2001,65(1):1-43.
[3]Atomi H. Recent progress towards the application of hyperthermophiles and their enzymes[J]. Current Opinion in Chemical Biology,2005,9(2):166-173.
[4]Li L,Yang J,Li J,Long L,et al. Role of two amino acid residues’ insertion on thermal stability of thermophilicα-amylase AMY121 from a deep sea bacteriumBacillussp. SCSIO 15121[J].Bioprocess and Biosystem Engineering,2015,38(5):871-879.
[5]Yang Jian,Li L,Xiao Y,et al. Identificationand thermoadaptation engineering of thermostability conferring residue of deep sea bacterialα-amylase AMY121[J]. Journal of Molecular Catalysis B:Enzymatic,2016,126:56-63.
[6]Zhang JW,Zeng RY. Purification and characterization of a cold-adaptedα-amylase produced byNocardiopsissp. 7326 isolated from Prydz Bay,Antarctic[J]. Marine Biotechnology,2007 10(1):75-82.
[7]Jiang T,Cai M,Huang M,et al. Characterization of a thermostable raw-starch hydrolyzingα-amylase from deep-sea thermophileGeobacillussp.[J]. Protein Expression and Purification,2015,114:15-22.
[8]Zhou G,Jin M,Cai Y,et al. Characterization of a thermostable and alkali-stableα-amylase from deep-sea bacteriumFlammeovirgapacifica[J]. International Journal of Biological Macromolecules,2015,80:676-682.
[9]Song Q,Wang Y,Yin C,et al. LaaA,a novel high-active alkalophilicα-amylase from deep-sea bacteriumLuteimonasabyssiXH031T[J]. Enzyme and Microbial Technology,2016,90:83-92.
[10]Lim WJ,Park SR,An CL,et al. Cloning and characterization of a thermostable intracellularα-amylase gene from the hyperthermophilic bacteriumThermotogamaritimaMSB8[J]. Research in Microbiology,2003,154(10):681-687.
[11]Seong-Ae Y,Ryu SI,Lee SB,et al. Purification and characterization of branching specificity of a novel extracellular amylolytic enzyme from marine hyperthermophilicRhodothermusmarinus[J]. Journal of Microbiology and Biotechnology,2008,18(3):457-464.
[12]Wang S,Lu Z,Lu M,et al. Identification of archaeon-producing hyperthermophilicα-amylase and characterization of theα-amylase[J]. Applied Microbiology and Biotechnology,2008,80(4):605-614.
[13]Homaei A,Ghanbarzadeh M,Monsef F. Biochemical features and kinetic properties ofα-amylases from marine organisms[J]. International Journal of Biological Macromolecules,2016,83:306-314.
[14]Hiteshi K,Gupta R. Thermal adaptation ofα-amylases:A review[J]. Extremophiles,2014,18(6):937-944.
[15]Sharga BM,Lyon GD.BacillussubtilisBS 107 as an antagonist of potato blackleg and soft rot bacteria[J]. Canadian Journal of Microbiology,1998,44(8):777-783.
[16]Larkin MA,Blackshields G,Brown NP,et al. Clustal W and Clustal X version 2.0[J]. Bioinformatics,2007,23(21):
2947-2948.
[17]Myung IJ. Tutorial on maximum likelihood estimation[J]. Journal of Mathematical Psychology,2003,47(1):90-100.
[18]Miller GL. Use of dinitrosalicylic acid reagent for determination of reducing sugar[J]. Analytical Chemistry,1959,31(3):426-428.
[19]Hewitt DCJ,Solomons GL.The production ofα-amylase(E.C.3.2.1.1.)byBacillusamyloliquefaciens,in a complex and a totally defined synthetic culture medium[J]. Journal of Industrial Microbiology & Biotechnology,1996,17(2):96-99.
[20]Hillier P,Wase DAJ,Emery AN,et al. Instability ofα-amylase production and morphological variation in continuous culture ofBacillusamyloliquefaciens,is associated with plasmid loss[J]. Process Biochemistry,1997,32(1):51-59.
[21]Konsula Z,Liakopoulou-Kyriakides M. Hydrolysis of starches by the action of anα-amylase fromBacillussubtilis[J]. Process Biochemistry,2004,39(11):1745-1749.
[22]Gangadharan D,Sivaramakrishnan S,Nampoothiri KR,et al. Response surface methodology for the optimization of alpha amylase production byBacillusamyloliquefaciens[J]. Bioresource Technology,2008,99(11):4597-4602.
[23]Demirkan ES,Mikami B,Adachi M,et al.α-Amylase fromB.amyloliquefaciens:purification,characterization,raw starch degradation and expression inE.coli[J]. Process Biochemistry,2005,40(8):2629-2636.
[24]Alikhajeh J,Khajeh K,Ranjbar B,et al. Structure ofBacillusamyloliquefaciensα-amylase at high resolution:Implications for thermal stability[J]. Acta Crystallographica,2010,66(Pt 2):121-129.
[25]Liu Y,Shen W,Shi GY,et al. Role of the calcium-binding residues Asp231,Asp233,and Asp438 in alpha-amylase ofBacillusamyloliquefaciensas revealed by mutational analysis[J]. Current Microbiology,2010,60(3):162-166.
[26]Deb P,Talukdar SA,Mohsina K,et al. Production and partial characterization of extracellular amylase enzyme fromBacillusamyloliquefaciens,P-001[J]. Springer Plus,2013,2(1):154.
[27]Zonouzi R,Khajeh K,Monajjemi M,et al. Role of the salt bridge between Arg176 and Glu126 in the thermal stability of theBacillusamyloliquefaciensα-amylase(BAA)[J]. Journal of Microbiology and Biotechnology,2013,23(1):7-14.
[28]Igarashi K,Hatada Y,Hagihara H,et al. Enzymatic Properties of a novel liquefyingα-amylase from an alkaliphilicBacillusisolate and entire nucleotide and amino acid sequences[J]. Applied and Environmental Microbiology,1998,64(9):3282-3289.
[29]Sodhi HK,Sharma K,Gupta JK,et al. Production of a thermostableα-amylase fromBacillus,sp. PS-7 by solid state fermentation and its synergistic use in the hydrolysis of malt starch for alcohol production[J]. Process Biochemistry,2005,40(2):525-534.
[30]Lee J,Parulekar SJ. Enhanced production ofα-amylase in fed-batch cultures ofBacillussubtilis TN106[pAT5][J]. Biotechnology and Bioengineering,1993,42(10):1142-1150.
[31]Mc Mahon HE,Kelly CT,Fogarty WM. Effect of growth rate on alpha-amylase production by Streptomyces sp. IMD 2679[J]. Applied Microbiology and Biotechnology,1997,48(4):504-509.
[32]Lin Long-Liu,Chyau Charng-Cherng,Hsu Wen-Hwei. Production and properties of a raw-starch-degrading amylase from the thermophilic and alkaliphilicBacillussp. TS-23[J]. Biotechnology and Applied Biochemistry,1998,28(Pt 1)(1):61-68.
[33]Teodoro,Souzamartins CED,Leal ML. Culture conditions for the production of thermostable amylase byBacillussp[J]. Brazilian Journal of Microbiology,2000,31(4):298-302.
[34]HatadaY,Masuda YN,AkitaM,et al. Oxidatively stable maltopentaose-producingα-amylase from a deep-seaBacillus,isolate,and mechanism of its oxidative stability validated by site-directed mutagenesis[J]. Enzyme & Microbial Technology,2006,39(6):1333-1340.
[35]Kim TU,Gu BG,Jeong JY,et al. Purification and characterization of a maltotetraose-forming alkalineα-amylase from an alkalophilicBacillusstrain,GM8901[J]. Applied & Environmental Microbiology,1995,61(8):3105-3112.
[36]Lo HF,Lin LL,Chen HL,et al. Enzymatic properties of a SDS-resistantBacillussp. TS-23α-amylase produced by recombinantEscherichiacoli[J]. Process Biochemistry,2001,36(9):743-750.
[37]Arellanocarbajal F,Olmossoto J. Thermostable alpha-1,4-and alpha-1,6-glucosidase enzymes fromBacillussp. isolated from a marine environment[J]. World Journal of Microbiology and Biotechnology,2002,18(8):791-795.
[38]Vidilaseris K,Hidayat K,Retnoningrum DS,et al. Biochemical characterization of a raw starch degradingα-amylase from the Indonesian marine bacteriumBacillus,sp. ALSHL3[J]. Biologia,2009,64(6):1047-1052.
[39]Kalpana BJ,Pandian SK. Halotolerant,acid-alkali stable,chelator resistant and raw starch digestingα-amylase from a marine bacteriumBacillussubtilisS8-18[J]. Journal of Basic Microbiol,2014,54(8):802-811.
[40]Asoodeh A,Chamani J,Lagzian M. A novel thermostable,acidophilic alpha-amylase from a new thermophilic “Bacillussp. Ferdowsicous” isolated from Ferdows hot mineral spring in Iran:Purification and biochemical characterization[J]. International Journal of Biological Macromolecules,2010,46(3):289-297.
[41]王磊,陈宇飞,杨柳. 产淀粉酶芽孢杆菌的分离和鉴定[J]. 食品研究与开发,2017,38(6):175-178.
[42]Ying Q,Zhang C,Guo F,et al. Secreted expression of a hyperthermophilicα-amylase gene fromThermococcussp. HJ21 inBacillussubtilis[J]. Journal of Molecular Microbiology & Biotechnology,2012,22(6):392-398.

