犬链球菌M蛋白基因的克隆及生物信息学分析
梁昱,刘燕霏,孙晓莉,杨建德
犬链球菌M蛋白基因的克隆及生物信息学分析
梁昱,刘燕霏,孙晓莉,杨建德通信作者
(天津农学院 动物科学与动物医学学院,天津 300384)
本研究对犬链球菌M蛋白(SCM)基因克隆,并对其进行生物信息学分析,预测其蛋白质结构及功能,为链球菌表位疫苗的研究及链球菌病的诊断提供参考。利用ExPASy ProtParam在线分析软件,预测犬链球菌M蛋白为亲水性蛋白质且属于稳定蛋白。使用SignalP 4.1软件推测该蛋白有信号肽。利用SOPMA中5种相互独立的方法预测其二级结构,其中α螺旋占83.53%,可知α螺旋为二级结构的主要元件。应用SWISS-MODEL在线分析软件,进行三级构造同源建模,发现其结果与二级结构预测结论大致相同。使用DNAStar预测软件中的Protean 部分对SCM亲水性、蛋白质的二级结构、抗原性指数、表面可能性、柔韧性进行归纳比较分析,在第55~59和第377~379氨基酸区段存在B细胞表位。
犬链球菌M蛋白;生物信息学分析;蛋白质结构;B细胞表位
犬链球菌(,SC)属于一类G群链球菌,对动物有较强的致病性。常常引起猫和犬败血症和关节炎,偶尔引起奶牛乳房炎;也可感染人出现脑膜炎等症。近年来,链球菌感染及相关毒力因子研究逐渐得到人们的关注[1]。M蛋白是A群链球菌主要的毒性抗原决定簇,并且能够作为其逃生的一种手段来躲避动物体内的免疫防御机制。M蛋白作为链球菌细胞壁外的一种构造,犹如网球表面的绒毛,可以帮助链球菌逃避吞噬作用并在宿主中存活下来。M蛋白具有很好的抗原性,能够使机体产生特异性抗体,还可以激活细胞使其增殖并释放出淋巴因子,使机体产生免疫保护作用[2]。
对链球菌病的整体防控来看,使用疫苗对动物或人进行预防是降低该病发生、减少经济损失最有前途和最节约的途径[3]。为了更好地筛选动物链球菌疫苗的有效抗原成分,本文通过对SCM基因进行克隆和测序,然后开展其生物信息学研究,如理化性质、蛋白质结构的分析以及B细胞表位预测等,为进一步研制犬链球菌表位疫苗和诊断试剂及阐明其毒力因子致病机理提供参考依据。
1 材料与方法
1.1 材料
1.1.1 菌株
由天津农学院预防兽医学实验室保存的犬链球菌菌株。
1.1.2 主要试剂及软件
大肠杆菌()DH5α感受态细胞和pET vector购于Novagen生物工程公司;胶回收试剂盒、T4 DNA ligase、酶、限制性内切酶H I、I以及质粒的提取试剂盒全部购买于上海生工公司;培养的CNA购自普博欣公司。
分析软件为DNAStar,下载地址为http://www. dnastar.com;Primer Premier 5.0软件。
1.2 方法
1.2.1 引物的设计与合成
使用Primer Premier 5.0软件设计犬基因的全长基因引物,上游引物: 5'-TCGCTCGAGGTT TGTAATAGGGCTGATAG-3',下游引物: 5'-TTA GGATCCTGATTCATTCGTTGATGGTAATTG-3'。
1.2.2 SCM基因克隆
分离菌模板DNA的提取,按文献提供的方法进行[4]。PCR反应程序:94 ℃预变性3 min,94 ℃变性30 s,54 ℃退火30 s,72 ℃延伸80 s,30个循环;72 ℃延伸10 min。使用限制性内切酶I和H I将PCR产物酶切后回收。将回收得到的片段进行1%琼脂糖凝胶电泳检测,37 ℃ 4 h,纯化之后再用T4连接酶将目的片段与pET载体进行连接,使其能够进入到大肠杆菌中,转化为DH5α感受态。涂于培养皿上,次日观察菌落形态。根据培养结果选出呈现阳性的菌落,对该菌落的质粒实行PCR扩增和酶切鉴定,然后对鉴定阳性的菌株测序,得到序列用于下面分析。
1.2.3 SCM基因生物信息学分析
利用生物信息学软件及在线工具,应用ExPASy ProtParam(http://web.expasy.org/protparam/)计算犬链球菌M蛋白的氨基酸构成、理论分子质量、等电点等。通过在线分析软件SignalP 4.1(http://www.cbs.dtu.dk/services/SignalP/)预测SCM的信号肽序列。利用SOPMA中5种相互独立的方法预测其二级结构。三级结构分析利用SWISS MODEL(http://swissmodel.expasy.org/)程序进行建模,分析SCM的三级结构[5-6]。通过使用DNA Star软件Protean程序这一在线工具,对SCM的B细胞表位进行预测。
2 结果与分析
2.1 SCM基因克隆
PCR结果与预期片段大小相符,所获得重组质粒用限制性内切酶I和H I进行酶切鉴定,最后获得大小约为1.1 kb的基因片段,见图1。

图1 PCR扩增结果
注:1和3为DNA Marker;2为PCR扩增结果;4为酶切鉴定
SCM基因的测序结果:(ATGACTAGAAAAAATACAATTAAGAAGCTTAGCGTTGGAACAGCATCACTGCTTGCCGCGACAACTGTTCTAGGAGCAGCTACCACAACAACTGTAAAGGCAGAAAGTAATAGGGCTGATAGGGCAAGAATGGAAGTCTTATTAAAGTATGTGCCGTATGTTTCTTCTAATTACTGGTACCAGGAAGTACTTAAAAATGATGCACTGCGAGAAGAAGTGCAACGTTTAGAAGATTTAGTTGAATCAGAAGTAAAAGATTACAATGCTCTACTCGACAAGAAAGAATCCGTTGAAAAGCAGCTTAAGAGCACAGAGGACAGCTTAGAAGTAACTGAAAAAGCTAACAAAGGTCTTACTAAAGAAAGAGATCTTTTAACAGATTTTTTAGAAACCACCAAAAAAGCTCTTGAAGATAGCCAAAAAGAAGTTCAAGCTAACCTTGACGCTTTGAACCATAAGAACGAGCAAATTGCCAGCTTAGTCGGTGAAAGAGATAGCTTGTCAGCACAACTATCTGCTAGCCAAGAGCGTAACGCTGAGTTGGAGCGTAACCTTGAAAGCTACGACCGCTTGATTGAGTCTGCAAAATTTGAAATGCAACAAAAACTAGCAGAAATTGAGCGCTTGACAGCTGAAAATGCAGACGCAAAAGATCAAATCGAGAAACTTCAAGCAGAAGTGTCAACACTTCGTGATCAAGTAGCAAGCCTAGACCGCTTGGTTGAATCAGCTAAGTATGACCTAGCTCAAAAACAAGCTGAAATCGATACCCTTACAAAACAAAAAGCAGAGGCAGAGCAAGCTCTTGCCGCAGAGCAAGCTAAAGTAGCTGAGCTTGAAAAACAACTTGAAGCATCAAACGCTAAAGTAGCTGAGCTTGAAAAACAAAAAGCAGAAGCAGAAGCTAAGATTGCTGTTCTTGAAAAAGACCTTGAAACAGCTCAAGCTGAAAACGCTAAGTACAAAGAACAATTAGCTAAACAAGCTGAAGAGCTTGAAGCAGCTAAGAAAGCAAAAGCAGAGGCAGAAGCTAAGATTGAAGAACTCAAAGGCATGCTTGACAAGAAAGCTCAAGACAACGCAAGTCTTCAAGCAGAGATTAACCGCTTGAAACAAGGTATTTCTGACAAGATGAAGTCAATGCCAGGTCAAGGCACTGCTAAAGCAAACATGTCATCTACTGACGCTAAGAAAGATAGCAAATCAATTACCATCAACAGGTGA)。
2.2 SCM理化性质分析
利用ExPASy ProtParam在线分析软件推测SCM的理化性质及氨基酸构成。按照推测出的数值显示,这段SCM基因编码氨基酸的数量为407个。亲水性氨基酸(D、E、H、K、Q、R、S、T)共有223个、疏水性氨基酸(A、F、I、L、M、P、V、W、Y)共有158个,犬链球菌M蛋白的平均亲水性为-0.774,其脂肪系数为85.28,表明犬链球菌M蛋白可能为亲水性蛋白质。该蛋白质分子质量约为45 281.91 kDa,分子结构式为C1944H3249N553O668S7,理论等电点值pI=5.13,负电荷残基数(Asp+Glu)为78,占19.2%。该M蛋白在大肠杆菌中的半衰期达到10 h以上,其在溶液中的不稳定指数为31.22,属于稳定蛋白。
2.3 SCM信号肽特性
使用SignalP 4.1软件对SCM推测,分析SCM蛋白是否存在信号肽。从图2可以看出,值最大切割点在第35位氨基酸,其分值为0.668,综合剪切位点值(值)最高数值处也在第35位氨基酸上,数值为0.642,信号肽最大分值(Max)出现在第16位氨基酸上,分值为0.960。SCM从第1到第34 个氨基酸,所得出的平均值为0.806,平均值为0.706,截断值是0.450,由图2可知,SCM有明显的信号肽切除位点,推测M蛋白存在35个氨基酸的信号肽[7]。
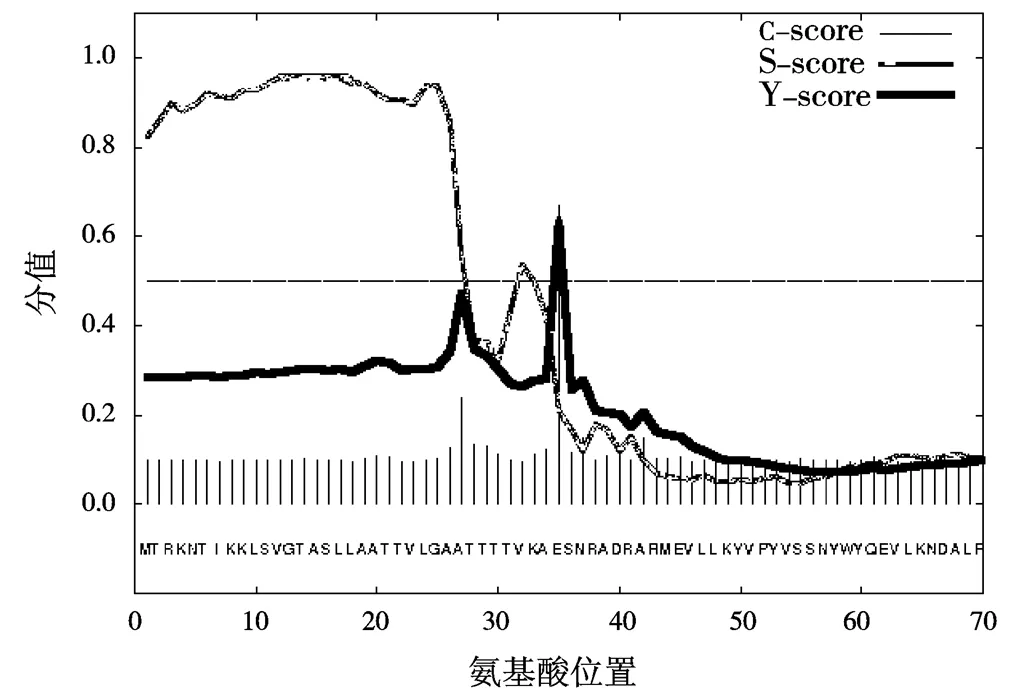
图2 SCM蛋白信号肽预测结果
2.4 SCM二级结构预测
利用SOPMA中5种不同、且彼此之间互相独立的方法预测SCM二级结构,汇总数据如表1。显示SCM氨基酸序列中约包括340个α螺旋,0个β折叠,20个延展片段,其余47个为无规则卷曲。由此可知α螺旋为二级结构的主要元件。Hh、Cc、Ee贯穿于整个氨基酸链中,而Tt则主要分布于氨基酸链的第50~60个之间,以及第380个氨基酸以后。
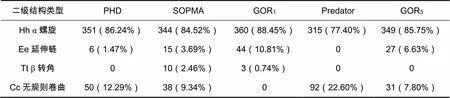
表1 SOPMA中5种方法预测犬链球菌M蛋白二级结构结果
2.5 SCM三级结构预测
应用SWISS-MODEL软件,在SCM氨基酸与其家族蛋白质之间进行三级结构模型的建构,发现其三级结构与Gp7-MYH7(1 361~1 425)-Eb1 嵌合体蛋白(4xa3.1.A)的一致性为23.91%,三维结构如图3,由图可见其三维空间结构主要以螺旋为主,存在少量无规则区域,与二级结构预测结果大致相同。

图3 SCM蛋白质三级结构预测模式图
2.6 SCM B细胞表位预测
使用DNAStar软件Protean工具中的Kyte-Doolittle法、Jameson-Wolf法、Emini法预测SCM亲水性、抗原性指数、表面可能性。在所得结果中依据亲水性指数≥0、抗原性指数≥0、表面可能性指数≥1的要求,筛选出链球菌M蛋白在第1~10、33~44、90~124、130~148、175~218、292~303、306~404区段符合条件[8-9]。
选用3种程序分别测定其柔性构造区域,按Chou.Fas-man法、Gamier-Robson法、Karplus-Schulz法预测的结果表明,链球菌M蛋白的二级结构存在丰富α-螺旋和β-折叠,结果如图4,位于N-端的第55~59和第377~379区段是3种方法共同判定的氨基酸区域中存在柔性结构的位置,并结合所筛选出的区段,推测两个区域很有可能存在B细胞表位。

图4 SCM B细胞表位预测
注:A为亲水性;B为指数抗原性指数;C为表面可能性指数;D为二级结构转角和无规则区域预测;E为二级结构柔性区域预测
3 讨论
应用软件分析SCM基因序列,推测其编码蛋白为稳定的亲水性蛋白质,且存在35个氨基酸的信号肽。对其编码蛋白的亲水性、二级结构、抗原性指数、表面可能性及柔韧性一一进行数据分析预测,得到部分区域可能存在B细胞抗原表位,据此可以设计一些表位疫苗[10-11]。SCM氨基酸组成中大部分是亲水性氨基酸且存在于细胞膜外侧,可作为研发疫苗的候选蛋白。预测结果显示其半衰期较长,能够在一段时间内存在于体内,进而可以持续刺激机体产生大量的抗体,这可能成为疫苗制备的重要候选蛋白[12-13]。SCM的三级构造推测出的模式图与其二级结构的预测结果大致相同,可以通过利用该模式图进行进一步分析,对可以结合佐剂的位点进行实验室验证,为日后利用该蛋白制作亚单位疫苗打下基础[14-15]。也可以利用该基因所表达的蛋白抗原制作酶联反应试剂盒及亚单位疫苗,对犬链球菌病的预防、诊断以及后续的治疗具有重要意义。
[1] 刘燕霏,杨建德. 犬链球菌重组M蛋白的免疫学分析[J]. 黑龙江畜牧兽医,2013(7):87-89.
[2] Fulde M,Rohde M,Polok A,et al. Cooperative plasminogen recruitment to the surface ofvia M protein and enolase enhances bacterial survival[J]. M Bio,2013,4(2):629-641.
[3] Steer A C,Carapetis J R,Dale J B,et al. Status of research and development of vaccines for[J].Vaccine,2016,34(26):2953-2958.
[4] 杨建德,刘燕霏,徐军,等. 犬链球菌保护性抗原的鉴定[J]. 中国预防兽医学报,2009,31(9):680-683.
[5] Kuo C F,Tsao N,Hsieh I C,et al. Immunization with a streptococcal multiple-epitope recombinant protein protects mice against invasive group A streptococcal infection[J]. Plos One,2017,12(3):e0174464.
[7] 颜其贵,雷燕,张志和,等. 大熊猫轮状病毒CH-l株外衣壳蛋白(VP7)基因的克隆和生物信息学分析[J].畜牧兽医学报,2010,41(6):705-710.
[8] 冯旭芳,张双翔,周碧君,等. 猪轮状病毒贵州株VP7基因克隆及生物信息学分析[J]. 畜牧与兽医,2016,48(4):87-93.
[9] 胡晓维,靳聪飞,刘新峰,等. 牛ACOT11基因克隆及其编码蛋白生物信息学分析[J]. 天津农学院学报,2015,22(3):1-6.
[10] Thigpen M C,Whitney C G,Messonnier N E,et a1. Bacterial meningitis in the United States[J].N Engl J med,2011,364(21):2016-2025.
[11] 朱晓琳,王劭,陈少莺,等. 猪传染性胃肠炎病毒福建株M基因的克隆与生物信息学分析[J]. 福建农业学报,2011,26(5):925-929.
[12] Converso T R,Goulart C,Rodriguez D,et al. Systemic immunization with rPotD reducesnasopharyngeal colonization in mice[J]. Vaccine,2017, 35(1):149-155.
[13] Williamson D A,Smeesters P R,Steer A C,et al. Comparative M-protein analysis offrompharyngitis and skin infections in New Zealand:Implications for vaccine development[J]. BMC Infect Dis,2016,16(1):561.
[14] 许宇彪,杨建荣,李碧锦. 人类miR-425靶基因预测及生物信息学特征分析[J]. 河北医药,2017,39(7):977-980.
[15] Cornick J E,Özlem Tastan Bishop,Yalcin F,et al. The global distribution and diversity of protein vaccine candidate antigens in the highly virulentserotype 1[J]. Vaccine,2017,35(6):972-980.
责任编辑:张爱婷
Cloning and bioinformatics analysis of M gene in
LIANG Yu, LIU Yan-fei, SUN Xiao-li, YANG Jian-deCorresponding Author
(College of Animal Science and Veterinary Medicine, Tianjin Agricultural University, Tianjin 300384, China)
In order to provide the reference for developing epitope vaccine and diagnosis of, M protein of(SCM)gene was cloned and analyzed by bioinformatics software to predict its protein structure and function in this study. SCM was a stable hydrophilic protein and transmembrane protein proved by analysis of ExPASy ProtParam on-line software. This protein was predicted to contain signal peptide by SignalP 4.1 software. Five separate methods of SOPMA showed that SCM had α helix as the main component of secondary structure, accounted for 83.53%. The three-dimensional structure of SCM determined by using SWISS-MODEL work-space was predicted to have the same result with the secondary structure. By using Protean modules of DNAStar software, SCM hydrophilicity plot, secondary structure, antigenicity index, surface probability plot and flexible regions are analyzed and compared to find that B cell epitopes were predicted at the region No. 55~59 and No.377~379.
SCM; bioinformation analysis; protein structure; B cell epitope
S852.61
A
1008-5394(2018)01-0044-05
10.19640/j.cnki.jtau.2018.01.010
2017-04-10
天津自然科学基金项目(07JCYBJC16000);天津农学院附属动物医院关键技术集成与学生综合能力提升项目(ZH004901)
梁昱(1996-),女,本科在读,主要从事生物信息学方面研究。E-mail:582605713@qq.com。
杨建德(1969-),男,教授,博士,主要从事预防兽医学方面的教学与研究。E-mail:jiandeyang@126.com。

