水稻WRKY转录调控因子研究进展
郑超,郑二松,王栩鸣,李冬月,杨勇,余初浪,周洁,严成其,陈剑平
1.浙江师范大学 化学与生命科学学院,浙江 金华 321004;2.浙江省农业科学院 病毒学与生物技术研究所,浙江省有害生物防控国家重点实验室培育基地,农业部植保生物技术重点实验室,浙江省植物病毒重点实验室,浙江 杭州 310021
水稻(Oryza sativaL.)是世界上最重要的粮食作物之一,是全世界半数以上(50%~80%)人口的主粮[1]。在人类几千年的育种过程中,高产一直是水稻品种选育的主要目标,也是农业生产追求的主要目标。在粮食生产过程中,生物或非生物胁迫往往会严重影响水稻的产量。由于水稻种植方式单一,在栽培过程中很容易受到外界多种病原菌的侵害,干旱和盐渍等非生物胁迫也是影响水稻产量的主要因素。近年来,大量研究表明WRKY转录因子家族在水稻抗病耐逆中发挥重要作用。因此,深入了解这些基因在水稻抗病耐逆中的作用,探索相关的信号调控途径及代谢过程,理解相应的分子调控机理,对我们进一步发展抗病耐逆材料,创制水稻新品种具有重要的现实意义。
1 WRKY转录调控因子
转录调控因子是一类在生物中调节基因表达的蛋白。真核生物中的转录调控因子又称反式作用因子,是一类与靶基因启动子区域顺式作用元件特异性结合的DNA结合蛋白,结合后使得靶基因在转录水平发生显著变化,激活或抑制靶基因的表达[2]。这种结合的特异性反映在转录调控因子特殊的空间结构和顺式作用元件基本对应的核酸序列上。转录调控因子在调控基因表达上具有重要作用,据报道在拟南芥和水稻中分别鉴定到2100和2300个转录调控因子[3]。在水稻基因组中有许多参与编码转录调控因子的基因,而且有的转录调控因子家族拥有上百个基因,比如bZIP家族、MYC家族、MYB家族、WRKY家族、AP2/EREBP家族和NAM家族[4],这些转录调控因子参与水稻生长、发育和抗病耐逆,具有极其重要的生物学功能。
1.1 WRKY转录调控因子的发现
WRKY转录调控因子家族是植物中研究最广泛的转录因子之一,属于高等植物10大转录因子家族[5]。20多年前,Ishiguro等从甘薯(Ipomoea batatas)中鉴定得到一个新的DNA结合蛋白,命名为SWEET POTATO FACTOR 1(SPF1),这是发现的第一个WRKY转录调控因子[6],其后许多WRKY家族的基因在不同植物种类中也被确认。虽然在一些非植物种类的基因组(单细胞真核生物Giardia lamblia[7]和黏菌Dictyostelium dis⁃coideum[8])中也鉴定得到WRKY转录调控因子,但目前并没有在动物基因组中鉴定到WRKY转录因子,有研究报道WRKY转录调控因子起源于单细胞真核生物,在植物中得到发展且普遍存在于植物中[8]。随着越来越多的植物基因组测序完成,WRKY转录调控因子的全基因组鉴定及其功能分析也已完成[9-11]。在拟南芥(Arabidopsis thali⁃ana)中有74个WRKY基因[12],大豆(Glycine max)中有197个[13],番茄(Solanum lycopersicum)中有81个[14-15],黄瓜(Cucumis sativus)中有55个[16],玉米(Zea mays)中有119个[17],水稻中有109个[18]。
1.2 WRKY转录调控因子的结构特征及分类
WRKY转录调控因子的命名是因其N端氨基酸序列中有保守的WRKY序列[19]。所有WRKY转录调控因子的一个共同特点是至少包含1个约由60个氨基酸残基组成的高度保守的WRKY结构域,每个WRKY结构域包含1个位于C端的锌指结构模型(C2H2或C2-HC)和位于N端的严格保守的七肽序列WRKYGQK[20-21]。虽然WRKYGQK序列是高度保守的,但也有1或2个残基被替换的9个突变体在19个结构域中被发现,几个WRKY突变体模式存在于水稻WRKY蛋白中,大多属于第Ⅱc亚组和第Ⅲ组[8],例如WRKYGKK[20]和WRKYGEK[22]。WRKY转录调控因子通过与靶基因启动子区域的DNA序列(T)TGAC(C/T)(W盒)特异性结合来调节目的基因的表达[23]。一些含有TGAC核心结构的类似W盒也被提议作为WRKY转录调控因子的顺式作用元件,并且邻近TGAC核心结构的序列也决定了WRKY转录调控因子结合位点的优先权[24]。根据WRKY转录调控因子所含的WRKY结构域的个数和锌指型结构的特征,一般将WRKY转录调控因子分为3组[25]:第Ⅰ组含有2个WRKY结构域,且其锌指型结构的类型为C2H2(CX4-5-C-X22-23-H-X1-H)型,根据WRKY结构域位于转录因子的N端和C端分别被命名为N端WRKY结构域(NTWD)和C端WRKY结构域(CTWD)[25],如OsWRKY24和Os⁃WRKY53属于第Ⅰ组[26];第Ⅱ组WRKY转录调控因子含有1个WRKY结构域,其锌指型结构为C2H2(CX4-5-C-X22-23-H-X1-H)型,分为Ⅱa、Ⅱb、Ⅱc和Ⅱd等4个亚组,如OsWRKY62和OsWRKY76属于WRKY第Ⅱa亚组[27];第Ⅲ组WRKY转录调控因子也只含有1个WRKY结构域,但其锌指型结构为C2-HC(C-X7-C-X23-HX)型[5],水稻中有28个OsWRKY基因属于第Ⅲ组,其中OsWRKY90编码最大的蛋白(633个氨基酸残基),OsWRKY55编码最小的蛋白(210个氨基酸残基)[28]。
2 水稻WRKY转录调控因子的生物学功能
转录调控是通过细胞或生物有机体来控制其基因表达的重要机制[8]。胁迫诱导引起相关目的基因的转录调控,在植物生长发育和抗病耐逆性上具有重要作用,这些调节主要依赖于转录调控因子在时间和空间上的功能,胁迫分为生物胁迫和非生物胁迫[29]。WRKY转录调控因子家族作为植物中最大的转录家族之一,在调控植物应对生物胁迫和非生物胁迫的响应中起重要作用[30]。迄今,在经过全面基因组序列分析之后,已在栽培稻日本晴中预测到至少103个WRKY基因[31],已有研究显示其大量成员参与了植物对病毒[32]、细菌和真菌[33]等多种病原的防卫反应及水杨酸(SA)和茉莉酸(JA)相关的抗病信号转导途径。如过表达OsWRKY12(OsWRKY03)[34]、OsWRKY13[35]、OsWRKY53[36]和OsWRKY71[37]等基因后,水稻内源的抗病相关基因OsNPR1、OsNH1、病程相关(patho⁃genesis-related,PR)基因OsPR1等的表达量显著增强,进而表现出对白叶枯病或(和)稻瘟病的抗性。此外,除参与生物逆境的应答反应外,一些WRKY基因还参与非生物逆境的调控,如内源性增强表达OsWRKY45除了能提高水稻对稻瘟病和白叶枯病的抗性外,在冷、干旱和盐胁迫中也起重要作用[38],有研究报道外源性增强OsWRKY45基因在拟南芥中的表达,可提高转基因植株对干旱胁迫的耐性[39],表明OsWRKY45基因在植物对非生物胁迫的应答反应中也有重要作用。大量研究表明WRKY转录调控因子不仅参与植物对生物和非生物胁迫的响应,同时也参与调控植物的生长发育、各种生理过程和形态学建成[19,40],其功能具有多样性和复杂性。我们对已报道的水稻WRKY转录因子基因的生物学功能进行了归类与整合(表1),期望为水稻WRKY基因家族的研究工作提供参考和帮助。
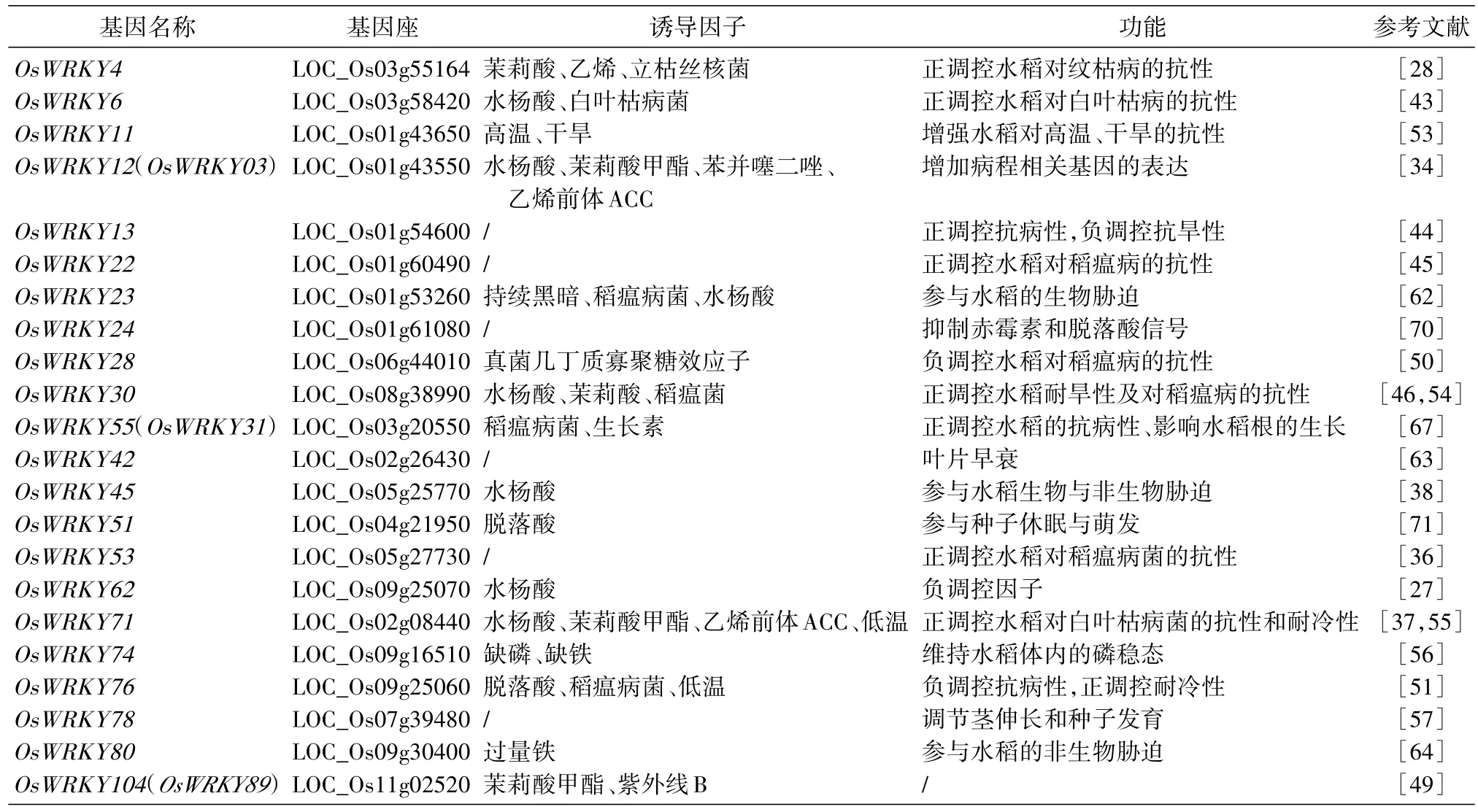
表1 本文涉及的水稻WRKY转录因子的功能
2.1 WRKY在水稻生物胁迫中的调控作用
许多研究表明WRKY转录调控因子家族中的大多数成员在水稻防御病原菌的侵害中起重要作用,是植物免疫反应的重要因子[41-42]。WRKY转录调控因子通过与靶基因的W盒、类似W盒的顺式作用元件和其他顺式作用元件特异性结合,特异调节靶基因的表达,在植物与病原菌的互作中起重要作用。有研究报道OsWRKY6基因的表达量在受白叶枯病菌诱导后迅速提高,表明其可能参与了水稻对病原菌的防御反应,在过表达OsWRKY6的转基因水稻中发现叶片上出现病斑,引起了细胞的程序性死亡,以阻止病原菌在植株中的继续扩散,从而提高水稻对白叶枯病菌的抗性,而在OsWRKY6RNA干扰的转基因水稻中发现病程相关基因的表达量降低,表现出对白叶枯病菌的易感性,实验证明OsWRKY6是水稻病程相关基因10a(PR10a)的正向调控因子,可直接与OsPR10a启动子的WLE1结合并调控OsPR10a的表达,从而提高水稻对白叶枯病菌的抗性[43]。由稻瘟病菌造成的稻瘟病是水稻最具毁灭性的疾病之一,对感染稻瘟病的水稻的OsWRKY基因的表达进行了系统性分析,通过RT-PCR的初筛发现测试的45个OsWRKY基因中有15个被稻瘟病菌诱导[33],之后研究证实至少有11个水稻WRKY转录因子参与调控水稻对稻瘟病菌的应答反应,如OsWRKY13[44]、OsWRKY22[45]、OsWRKY30[46]、Os⁃WRKY45[47-48]、OsWRKY104(OsWRKY89)[49]等正调控水稻对稻瘟病菌的应答反应,OsWRKY28[50]和Os⁃WRKY76[51]负调控水稻对稻瘟病菌的应答反应。研究报道等位基因OsWRKY45-1和OsWRKY45-2编码的蛋白有10个氨基酸残基的差异,它们在水稻抵抗细菌性病原菌中起相反的作用,Os⁃WRKY45-1超表达植株对水稻细菌性条斑病菌(Xoc)和水稻白叶枯病菌(Xoo)表现易感症状,而OsWRKY45-2超表达植株对Xoc和Xoo却表现极强的抗性,但它们在水稻抵抗稻瘟病菌中起相同的作用,OsWRKY45-1和OsWRKY45-2超表达植株均对稻瘟病菌表现出极强的抗性[48]。OsWRKY53也是一个稻瘟菌的正调控因子,它的过量表达和磷酸化突变体都能增强对稻瘟菌的抗性,它受到丝裂原活化蛋白激酶基因OsMPK3/OsMPK6的正向调控,同时又反过来抑制了它们的活性,从而减少食草动物侵害植物诱导的防卫反应,增加了二化螟的侵害[52]。也有一些WRKY基因在防御病原菌中起负调控作用,例如同属于Ⅱa亚组的Os⁃WRKY62和OsWRKY76基因,最新研究发现它们都有组成型和诱导性可变剪切OsWRKY62.1、Os⁃WRKY62.2和OsWRKY76.1、OsWRKY76.2,过表达OsWRKY62.1和OsWRKY76.1抑制相关防御基因的激活,增强水稻对稻瘟病菌和白叶枯病菌的易感性;而其RNA干扰和敲除材料中,由于防御相关基因的表达和植物抗毒素的积累增加,则表现出水稻对稻瘟病菌和白叶枯病菌的抗性[27]。
2.2 WRKY在水稻非生物胁迫中的调控作用
WRKY转录调控因子不仅在植物抵抗病原菌、虫害等生物胁迫中起重要作用,而且在植物应对干旱、冷害、盐害等非生物胁迫中发挥重要的调控功能。在筛选水稻受非生物逆境诱导的WRKY转录因子的研究中发现,OsWRKY11[53]、Os⁃WRKY30[54]和OsWRKY45[39]等在高温、干旱、高盐、低温等逆境胁迫中不同程度地被诱导。Os⁃WRKY11在水稻幼苗中受高温和干旱胁迫的诱导,用热激蛋白HSP101基因启动子驱动Os⁃WRKY11表达时,转基因植株对高温和干旱的抗性显著增加[53],因此OsWRKY11在水稻应对高温和干旱非生物胁迫的响应中起重要作用。Os⁃WRKY30可被OsMAPK3、OsMAPK7和OsMAPK14磷酸化,从而激活自身的转录活性以行使功能,在其过表达的转基因株系中OsWRKY30可被MAPK激活,提高了水稻对高温逆境的抗性,在水稻应对非生物胁迫中起正向调控作用[54]。OsWRKY71在水稻受到冷害胁迫后诱导表达,而且在其转基因株系中发现有2个冷害应答基因OsTGFR和WSI76的表达是上调的,因此OsWRKY71可能是通过调节下游目的基因的表达,在水稻应对冷害胁迫中起正调控作用[55]。研究表明水稻OsWRKY45的表达在脱落酸(ABA)、NaCl、PEG和甘露醇等胁迫处理时显著增加,外源性高表达OsWRKY45能增强转基因拟南芥对干旱胁迫的耐受性[39]。进一步研究发现,OsWRKY45的 2个等位基因Os⁃WRKY45-1和OsWRKY45-2在脱落酸信号途径及非生物胁迫过程中所起的作用并不完全相同,Os⁃WRKY45-1负调控而OsWRKY45-2正调控脱落酸信号途径,OsWRKY45-2而非OsWRKY45-1负调控对盐胁迫的应答,但在低温和干旱胁迫应答中两者的表现相似[38]。也有WRKY转录调控因子在水稻应对非生物胁迫应答反应中起负调控作用,如OsWRKY13可以与SNAC1和WRKY45-1启动子区域的WBOX1(TTGACT)和WBOXa(TTGAC)特异结合,抑制SNAC1和WRKY45-1的表达,负调控水稻的抗干旱过程[44]。OsWRKY74主要在水稻根部和叶片中表达,并受磷酸盐饥饿的诱导,参与调控水稻对缺磷胁迫的耐受性,其超表达植株对缺磷环境的耐受性显著增强,在缺磷条件下种植时,超表达植株根和地上部的生物量以及磷含量均比野生型增加。并且在缺磷条件下,OsWRKY74超表达水稻还表现出铁元素的积累和低温应答基因的上调,这些发现揭示了OsWRKY74在水稻调控植物体内磷素平衡、缺磷与缺铁之间潜在的关系以及寒冷胁迫中的重要作用[56]。OsWRKY104(OsWRKY89)受紫外线B辐射强烈诱导,过表达植株增强了水稻对紫外线B辐射的抗性[49]。
2.3 WRKY在水稻生长发育中的调节作用
WRKY转录调控因子不仅参与生物胁迫和非生物胁迫,而且还在植物生长发育、种子发育及衰老等一系列生命活动起重要的调节作用。有研究发现OsWRKY78的表达量在伸长茎中最为丰富,其过表达植株的株型与野生型一样,而其RNAi植株可能由于细胞长度的缩减而表现为半矮秆,且谷粒变小,在对其进行谷粒品质分析时发现胚乳淀粉晶体结构发生轻微变化但米质并没有明显的变化,说明OsWRKY78在水稻茎的伸长和种子发育中起重要作用[57]。也有研究发现在水稻种子萌发阶段中,赤霉素(GA)可从胚中分泌到糊粉层细胞中,促进水解酶(α-淀粉酶)的表达,利于种子的萌发和种子萌发后的生长,而ABA则会抑制相关酶的表达,从而影响种子的正常萌发,OsWRKY51和OsWRKY71是2个在胚和糊粉层细胞中受ABA诱导GA抑制的水稻WRKY基因,过表达这2个基因会特异并协同抑制α-淀粉酶的表达,说明它们可能参与了种子的休眠与萌发[58]。研究发现在模式植物拟南芥中存在与衰老相关的WRKY转录调控因子基因AtWRKY6[59]、At⁃WRKY53[60]和AtWRKY70[61]。AtWRKY6在幼叶和成熟叶片中几乎不表达,但在衰老叶片中表达很高,而且还发现AtWRKY6不仅能调节相关防御基因(ELI3、PR1和PR5)的表达,而且还能调节衰老相关基因(SAG12和SAG13)的表达[59]。在水稻中也发现了与植物衰老相关的WRKY转录调控因子,例如水稻OsWRKY23在拟南芥中过量表达会加速黑暗诱导的叶片衰老,同时改变了衰老相关基因SAG12和SEN1的表达[62]。OsWRKY42通过抑制活性氧清除基因OsMT1d的表达,从而诱导水稻叶片的衰老[63]。OsWRKY80转录调控因子在铁过量处理的水稻叶片、茎干和根中表达上调,而且这种上调表达在暗诱导的衰老和干旱胁迫过程中也能被检测到,说明OsWRKY80可能调控水稻营养胁迫诱导的衰老过程[64]。
2.4 WRKY参与水稻的形态建成
有研究表明WRKY转录调控因子还参与植物的形态建成。拟南芥中AtWRKY44转录调控因子基因可以通过与其他基因相互作用,调控毛状体的发生、毛状体的数目及其分叉情况,表明At⁃WRKY44参与拟南芥叶毛的形态建成[65],进一步研究发现AtWRKY44还参与调控拟南芥根毛细胞的分化[66]。目前在水稻中也发现了形态建成相关的WRKY转录调控因子,OsWRKY31受稻瘟菌和生长素诱导表达,提高了水稻的抗病性,但会影响水稻根的生长,研究发现OsWRKY31的过表达转基因植株的苗期侧根减少并且变短,抽穗期株高较野生型矮且根数目减少,说明OsWRKY31会抑制水稻不定根的形成,进而影响水稻根的形态建成,同时还发现这种影响伴随着对生长素响应反应的干预[67]。最新研究表明,OsWRKY53正调控油菜素内酯的信号途径,从而调控了水稻叶片角度及种子的大小,其过表达植株的叶角增加,种子变大。说明OsWRKY53转录调控因子通过与植物激素的互作参与调控水稻株型的形态建成[68]。
2.5 WRKY与激素信号途径的相互作用
植物在应对不同病原菌的长期侵害过程中进化出一套自己独特的防御反应体系,这个防御反应体系有很多信号分子参与,如SA、JA、GA和乙烯(ET)等。OsWRKY6是受SA诱导并在SA介导的防御信号途径中起重要作用的正调控因子,水稻在受病原菌侵害后可迅速激活SA信号途径,并引起OsWRKY6基因的表达,OsWRKY6不仅可与异分支酸合成酶1(OsICS1)启动子区域的W盒直接结合,调控OsICS1基因的表达以增加体内SA的浓度,最终激活SA介导的信号途径,还可以直接调控防御相关基因如OsPR10a和OsNPR1的表达,从而增强ROS和NPR1介导的防御反应,抵御病原菌的侵害[43]。OsWRKY4可被立枯丝核菌、外源JA和ET诱导表达,是水稻依赖JA/ET防御信号途径中的正向调控因子,在水稻对抗纹枯病的防御反应中起重要作用[69]。也有WRKY转录因子在激素信号途径中起负调控作用,OsWRKY24是水稻GA和ABA途径中的负调控因子,这2种激素调控了种子成熟、休眠及萌发的过程,在糊粉层细胞中瞬时过表达OsWRKY24基因能够抑制受GA诱导的Amy32b基因和受ABA诱导的HVA22基因的表达,因此OsWRKY24基因可能参与了水稻调控种子的萌发和萌发后生长过程[70]。大量研究显示激素信号途径在植物体内并不是单独起作用的,它们之间存在互作。OsWRKY13基因在SA和JA的互作中起重要作用,过表达OsWRKY13基因可激活SA合成相关基因和SA响应相关基因的表达,但会抑制JA合成相关基因和JA响应相关基因的表达,说明OsWRKY13基因是SA信号途径的激活子、JA信号途径的抑制子,通过直接或间接调控SA和JA上下游基因的表达,进而调控病程相关基因的表达,在水稻对白叶枯病菌和稻瘟病菌的防御反应中起正调控作用[35]。
3 结语
WRKY转录因子在植物对生物和非生物胁迫的反应中起关键作用。以上综述了WRKY在生物和非生物胁迫中的功能以及它们的下游基因和信号通路方面的主要研究。这些研究,不仅有助于我们发现WRKY基因在抗病抗逆方面的重要作用,还能帮助我们发展新的抗病抗逆材料,以用于水稻的育种工作。WRKY转录因子自我调控模式和它们之间的信号传导途径及相互作用机制目前尚不清晰。相信随着基因组学和转录组学研究的进展,相关信息越来越多时,WRKY转录因子在植物胁迫反应中的作用模式将会越来越明确。鉴于WRKY转录因子在水稻抗病信号调控途径中起非常重要的作用,新的研究必然会帮助我们进一步了解水稻抗病抗逆的机理,同时也会为将来水稻遗传改良提供重要帮助。
[1] Sasaki T,Burr B.International rice genome sequenc⁃ing project:the effort to completely sequence the rice genome[J].Curr Opin Plant Biol,2000,3(2):138-142.
[2] Riechmann J L.Transcription factors of Arabidopsis and rice:a genomic perspective[M]//Grasser K D.An⁃nual plant reviews volume 29:Regulation of transcrip⁃tion in plants.Blackwell Publishing Ltd,2007.
[3] Perez-Rodriguez P,Riano-Pachon D M,Correa L G, et al.PlnTFDB:updated content and new features of the plant transcription factor database[J].Nucleic Ac⁃ids Res,2010,38:D822-D827.
[4] Alves M,Dadalto S,Gonçalves A,et al.Transcription factor functional protein-protein interactions in plant defense responses[J].Proteomes,2014,2(1):85-106.
[5] Llorca C M,Potschin M,ZentgrafU.bZIPsand WRKYs:two large transcription factor families execut⁃ing two different functional strategies[J].Front Plant Sci,2014,5:169.
[6] Ishiguro S,Nakamura K.Characterization of a cDNA encoding anovelDNA-bindingprotein,SPF1,that recognizes SP8 sequences in the 5'upstream regions ofgenescoding forsporamin and β-amylasefrom sweet potato[J].Mol Gen Genet,1994,244(6):563-571.
[7] Ulker B,Somssich I E.WRKY transcription factors: from DNA binding towards biological function[J].Curr Opin Plant Biol,2004,7(5):491-498.
[8] Zhang Y,Wang L.The WRKY transcription factor su⁃perfamily:its origin in eukaryotes and expansion in plants[J].BMC Evol Biol,2005,5:1.
[9] Jiang Y,Duan Y,Yin J,et al.Genome-wide identifi⁃cation and characterization of the Populus WRKY tran⁃scription factor family and analysis of their expression in response to biotic and abiotic stresses[J].J Exp Bot,2014,65(22):6629-6644.
[10]Ding M,Chen J,Jiang Y,et al.Genome-wide investi⁃gation and transcriptome analysis of the WRKY gene family in Gossypium[J].Mol Genet Genomics,2015, 290(1):151-171.
[11]Ma J,Lu J,Xu J,et al.Genome-wide identification of WRKY genes in the desert poplar populus euphrati⁃ca and adaptive evolution of the genes in response to salt stress[J].Evol Bioinform Online,2015,11(Suppl 1): 47-55.
[12]Eulgem T,Somssich I E.Networks of WRKY tran⁃scription factors in defense signaling[J].CurrOpin Plant Biol,2007,10(4):366-371.
[13]Zhou Q Y,Tian A G,Zou H F,et al.Soybean WRKY-type transcription factor genes,GmWRKY13, GmWRKY21,and GmWRKY54,confer differential tol⁃erance to abiotic stressesin transgenic Arabidopsis plants[J].Plant Biotechnol J,2008,6(5):486-503.
[14]Hofmann M G,Sinha A K,Proels R K,et al.Clon⁃ing and characterization of a novel LpWRKY1 tran⁃scription factorin tomato[J].PlantPhysiolBiochem, 2008,46(5-6):533-540.
[15]Huang S,Gao Y,Liu J,et al.Genome-wide analysis of WRKY transcription factors in Solanum lycopersi⁃cum[J].Mol Genet Genomics,2012,287(6):495-513.
[16]Ling J,Jiang W,Zhang Y,et al.Genome-wide analy⁃sis ofWRKY gene family in Cucumis sativus[J]. BMC Genomics,2011,12:471.
[17]Wei K,Chen J,Chen Y,et al.Multiple-strategy anal⁃yses of ZmWRKY subgroups and functional explora⁃tion of ZmWRKY genes in pathogen responses[J].Mol BioSystems,2012,8(7):1940-1949.
[18]Ross C A,Liu Y,Shen Q J.The WRKY gene family in rice(Oryza sativa)[J].J Integr Plant Biol,2007,49(6): 827-842.
[19]Jiang W,Wu J,Zhang Y,etal.Isolation ofa WRKY30 gene from Muscadinia rotundifolia(Michx) and validation of its function under biotic and abiotic stresses[J].Protoplasma,2015,252(5):1361-1374.
[20]Eulgem T,Rushton P J,Robatzek S,etal.The WRKY superfamily ofplanttranscription factors[J]. Trends Plant Sci,2000,5:199-206.
[21]Rushton D L,Tripathi P,Rabara R C,et al.WRKY transcription factors:key components in abscisic acid signalling[J].Plant Biotechnol J,2012,10(1):2-11.
[22]Qiu Y,Jing S,Jian F U,et al.Cloning and analysis of expression profile of 13 WRKY genes in rice[J]. Chin Sci Bull,2004,49(20):2159-2168.
[23]Maleck K,Levine A,Eulgem T,et al.The transcrip⁃tome of Arabidopsis thaliana during systemic acquired resistance[J].Nat Genet,2000,26(4):403-410.
[24]Ciolkowski I,Wanke D,Birkenbihl R P,et al.Stud⁃ies on DNA-binding selectivity of WRKY transcrip⁃ tion factors lend structural clues into WRKY-domain function[J].Plant Mol Biol,2008,68(1-2):81-92.
[25]Wu K L,Guo Z J,Wang H H,et al.The WRKY family of transcription factors in rice and Arabidopsis and their origins[J].DNA Res,2005,12(1):9-26.
[26]Zhang L,Gu L,Ringler P,et al.Three WRKY tran⁃scription factors additively repress abscisic acid and gibberellin signaling in aleurone cells[J].PlantSci, 2015,236:214-222.
[27]Liu J,Chen X,Liang X,et al.Alternative splicing of rice WRKY62 and WRKY76 transcription factor genes in pathogen defense[J].Plant Physiol,2016,171 (2):1427-1442.
[28]Wang Y,Feng L,Zhu Y,et al.Comparative genomic analysis of the WRKY III gene family in populus, grape,arabidopsisand rice[J].BiolDirect,2015,10: 48.
[29]Roychoudhury A,Gupta B,Sengupta D N.Trans-act⁃ing factor designated OSBZ8 interacts with both typi⁃cal abscisic acid responsive elements as well as ab⁃scisic acid responsive element-like sequences in the vegetative tissues of indica rice cultivars[J].Plant Cell Rep,2008,27(4):779-794.
[30]Tripathi P,Rabara R C,Choudhary M K,et al.The interactome of soybean GmWRKY53 using yeast 2-hy⁃brid library screening to saturation[J].Plant Signal Be⁃hav,2015,10(7):e1028705.
[31]Ramamoorthy R,Jiang S Y,Kumar N,et al.A com⁃prehensive transcriptional profiling of the WRKY gene family in rice under various abiotic and phytohormone treatments[J].Plant Cell Physiol,2008,49(6):865-879.
[32]Shimizu T,Satoh K,Kikuchi S,et al.The repression of cell wall-and plastid-related genes and the induc⁃tion of defense-related genes in rice plants infected with Rice dwarf virus[J].Mol Plant Microbe Interact, 2007,20(3):247-254.
[33]Ryu H S,Han M,Lee S K,et al.A comprehensive expression analysis of the WRKY gene superfamily in rice plants during defense response[J].PlantCell Rep,2006,25(8):836-847.
[34]Liu X Q,Bai X Q,Qian Q,et al.OsWRKY03,a rice transcriptional activator that functions in defense signaling pathway upstream of OsNPR1[J].Cell Res, 2005,15(8):593-603.
[35]Qiu D,Xiao J,Ding X,et al.OsWRKY13 mediates rice disease resistance by regulating defense-related genes in salicylate-and jasmonate-dependent signaling [J].Mol Plant Microbe Interact,2007,20(5):492.
[36]Chujo T,Takai R,Akimoto-Tomiyama C,et al.In⁃volvement of the elicitor-induced gene OsWRKY53 in the expression of defense-related genes in rice[J].Bio⁃chim Biophys Acta,2007,1769(7):497-505.
[37]Liu X,Bai X,Wang X,et al.OsWRKY71,a rice transcription factor,isinvolved in rice defense re⁃sponse[J].J Plant Physiol,2007,164(8):969-979.
[38]Tao Z,Kou Y,Liu H,et al.OsWRKY45 alleles play differentroles in abscisic acid signalling and salt stress tolerance but similar roles in drought and cold tolerance in rice[J].J Exp Bot,2011,62(14):4863-4874. [39]Qiu Y,Yu D.Over-expression of the stress-induced OsWRKY45 enhances disease resistance and drought tolerance in Arabidopsis[J].Environ Exp Bot,2009,65 (1):35-47.
[40]Rushton P J,Somssich I E,Ringler P,et al.WRKY transcription factors[J].TrendsPlantSci,2010,15(5): 247-258.
[41]Ren X,Chen Z,Liu Y,et al.ABO3,a WRKY tran⁃scription factor,mediates plant responses to abscisic acid and drought tolerance in Arabidopsis[J].Plant J, 2010,63(3):417-429.
[42]Pandey S P,Somssich I E.The role of WRKY tran⁃scription factors in plant immunity[J].Plant Physiol, 2009,150(4):1648-1655.
[43]Choi C,Hwang S H,Fang I R,et al.Molecular char⁃acterization of Oryza sativa WRKY6,which binds to W-box-like element 1 of the Oryza sativa pathogene⁃sis-related(PR)10a promoter and confers reduced sus⁃ceptibility to pathogens[J].New Phytol,2015,208(3): 846-859.
[44]Xiao J,Cheng H,Li X,et al.Rice WRKY13 regu⁃lates cross talk between abiotic and biotic stress sig⁃naling pathways by selective binding to different ciselements[J].Plant Physiol,2013,163(4):1868-1882.
[45]Abbruscato P,Nepusz T,Mizzi L,et al.OsWRKY22, a monocot WRKY gene,plays a role in the resis⁃tance response to blast[J].Mol Plant Pathol,2012,13 (8):828-841.
[46]Peng X,Hu Y,Tang X,et al.Constitutive expression of rice WRKY30 gene increases the endogenous jas⁃monic acid accumulation,PR gene expression and re⁃ sistance to fungal pathogens in rice[J].Planta,2012, 236(5):1485-1498.
[47]Shimono M,Sugano S,Nakayama A,etal.Rice WRKY45 plays a crucial role in benzothiadiazole-in⁃ducible blast resistance[J].Plant Cell,2007,19(6):2064-2076.
[48]Tao Z,Liu H,Qiu D,et al.A pair of allelic WRKY genes play opposite roles in rice-bacteria interactions [J].Plant Physiol,2009,151(2):936-948.
[49]Wang H,Hao J,Chen X,et al.Overexpression of riceWRKY89 enhancesultravioletB toleranceand diseaseresistancein riceplants[J].PlantMolBiol, 2007,65(6):799-815.
[50]Chujo T,Miyamoto K,Shimogawa T,etal.Os⁃WRKY28,a PAMP-responsive transrepressor,negative⁃ly regulates innate immune responses in rice against rice blast fungus[J].Plant Mol Biol,2013,82(1-2):23-37.
[51]Yokotani N,Sato Y,Tanabe S,et al.WRKY76 is a rice transcriptional repressor playing opposite roles in blast disease resistance and cold stress tolerance[J].J Exp Bot,2013,64(16):5085-5097.
[52]Hu L,Ye M,Li R,et al.The rice transcription fac⁃torWRKY53 suppressesherbivore-induced defenses by acting as a negative feedback modulator of mito⁃gen-activated protein kinase activity[J].Plant Physiol, 2015,169(4):2907-2921.
[53]Wu X,Shiroto Y,Kishitani S,et al.Enhanced heat and drought tolerance in transgenic rice seedlings over⁃expressing OsWRKY11 under the control of HSP101 promoter[J].Plant Cell Rep,2009,28(1):21-30.
[54]Shen H,Liu C,Zhang Y,et al.OsWRKY30 is acti⁃vated by MAP kinases to confer drought tolerance in rice[J].Plant Mol Biol,2012,80(3):241-253.
[55]Kim C Y,Vo K T X,Nguyen C D,et al.Functional analysis of a cold-responsive rice WRKY gene,Os⁃WRKY71[J].Plant Biotechnol Rep,2016,10(1):13-23.
[56]Dai X,Wang Y,Zhang W H.OsWRKY74,a WRKY transcription factor,modulates tolerance to phosphate starvation in rice[J].J Exp Bot,2016,67(3):947-960.
[57]Zhang C Q,Xu Y,Lu Y,et al.The WRKY transcrip⁃tion factor OsWRKY78 regulates stem elongation and seed development in rice[J].Planta,2011,234(3):541-554.
[58]Xie Z,Zhang Z L,Zou X,et al.Interactions of two abscisic-acid induced WRKY genes in repressing gib⁃berellin signaling in aleurone cells[J].Plant J,2006,46 (2):231-242.
[59]Rob atzek S,Somssich I E.A new member of the Ara⁃bidopsis WRKY transcription factor family, At⁃WRKY6,is associated with both senescence-and de⁃fence-related processes[J].Plant J,2001,28(2):123-133.
[60]Miao Y,Laun T,Zimmermann P,et al.Targets of the WRKY53 transcription factor and its role during leaf senescence in Arabidopsis[J].Plant Mol Biol,2004,55 (6):853-867.
[61]Besseau S,Li J,Palva E T.WRKY54 and WRKY70 co-operate as negative regulators of leaf senescence in Arabidopsis thaliana[J].J Exp Bot,2012,63(7):2667-2679.
[62]Jing S,Zhou X,Song Y,et al.Heterologous expres⁃sion of OsWRKY23 gene enhances pathogen defense and dark-induced leaf senescence in Arabidopsis[J]. Plant Growth Regulation,2009,58(2):181-190.
[63]Han M,Kim C Y,Lee J,et al.OsWRKY42 repress⁃es OsMT1d and induces reactive oxygen species and leaf senescence in rice[J].Mol Cells,2014,37(7):532-539.
[64]Ricachenevsky F K,Sperotto R A,Menguer P K,et al.Identification of Fe-excess-induced genes in rice shoots reveals a WRKY transcription factor responsive to Fe,drought and senescence[J].Mol Biol Rep,2010, 37(8):3735-3745.
[65]Johnson C S,Kolevski B,Smyth D R.TRANSPAR⁃ENT TESTA GLABRA2,a trichome and seed coat de⁃ velopment gene of Arabidopsis,encodes a WRKY tran⁃scription factor[J].Plant Cell,2002,14(6):1359-1375.
[66]Ishida T,HattoriS,Sano R,etal.Arabidopsis TRANSPARENT TESTA GLABRA2 is directly regulat⁃ed by R2R3 MYB transcription factorsand isin⁃volved in regulation of GLABRA2 transcription in epi⁃dermal differentiation[J].Plant Cell,2007,19(8):2531.
[67]Zhang J,Peng Y L,Guo Z J,et al.Constitutive ex⁃pression of pathogen-inducible OsWRKY31 enhances disease resistance and affects root growth and auxin response in transgenic rice plants[J].Cell Res,2008,18 (4):508-521.
[68]Tian X,Li X,Zhou W,et al.Transcription factor Os⁃WRKY53 positively regulates brassinosteroid signaling and plantarchitecture[J].PlantPhysiol,2017,175(3): 1337.
[69]Wang H,Meng J,Peng X,et al.Rice WRKY4 acts as a transcriptionalactivatormediating defense re⁃sponses toward Rhizoctonia solani,the causing agent of rice sheath blight[J].Plant Mol Biol,2015,89(1-2): 157-171.
[70]Zhang Z L,Shin M,Zou X,et al.A negative regula⁃tor encoded by a rice WRKY gene represses both ab⁃scisic acid and gibberellins signaling in aleurone cells [J].Plant Mol Biol,2009,70(1-2):139-151.
[71]Xie Z,Zhang Z L,Zou X,et al.Annotations and functional analyses of the rice WRKY gene superfami⁃ly reveal positive and negative regulators of abscisic acid signaling in aleurone cells[J].Plant Physiol,2005, 137(1):176-189.

