Ku70基因稳定敲除HeLa细胞株的建立及其生物学功能研究
李达,沈雪莲,李少华,丁红梅,李慧,黄皑雪,耿介,王超男,白琛俊,张坦,董洁,邵宁生
军事医学研究院 军事认知与脑科学研究所,北京 100850
Ku蛋白是一种含量丰富的核蛋白,进化上高度保守,分布广泛,从细菌到人类均有表达[1-2]。人Ku蛋白是异二聚体,由Ku70和Ku80两个亚基组成。研究发现,Ku蛋白具有不同寻常的DNA结合特性,其以非序列依赖性的方式紧密结合在双链DNA的断端,是经典非同源末端连接(classi⁃cal non-homologous end joining,C-NHEJ)途径中的DNA末端结合因子,招募DNA依赖的蛋白激酶催化亚基(DNA-PKcs)与其结合形成DNA依赖的蛋白激酶全酶,催化双链断裂DNA的修复[2-7]。
此外,Ku蛋白还可能参与其他重要的生物学过程,比如Ku蛋白被证实与染色体端粒结构的维持有密切联系。研究发现在失去任意一个Ku蛋白亚基后,酿酒酵母就会出现端粒结构缺失的现象[8-9]。还有研究显示Ku70与细胞凋亡有直接联系,即Ku70能够与促凋亡因子Bax结合,抑制其促进细胞凋亡的作用[10]。然而,Ku蛋白的生物学功能尚未得到完全阐释,有待深入研究。
我们利用CRISPR/Cas9基因编辑技术破坏HeLa细胞Ku70基因开放读框,同时在其中插入潮霉素B抗性基因,然后通过潮霉素B抗性筛选获得Ku70基因稳定敲除的HeLa细胞株,再进一步利用此细胞株研究Ku70蛋白的生物学功能。
1 材料与方法
1.1 材料
HeLa细胞由本实验室保存;pCas-guide和pSilencer2.1-U6hygro质粒由军事医学研究院郑晓飞教授惠赠;感受态大肠杆菌DH5α、T4DNA连接酶、细胞基因组DNA提取试剂盒、琼脂糖凝胶DNA回收试剂盒、质粒小提试剂盒购自康为世纪生物科技有限公司;PCR平末端产物加A试剂盒、pBackZero-T载体为TaKaRa公司产品;pfuDNA聚合酶购自北京全式金生物技术公司;限制性核酸内切酶BamHⅠ、BsmBⅠ为Thermo Scientific公司产品;PRIME jet转染试剂为PolyPlus公司产品;潮霉素B为Roche公司产品;Ku70抗体购自Pro⁃teintech公司;GAPDH抗体购自中杉金桥公司;CCK-8试剂购自上海碧云天生物技术有限公司;2×SYBR Green Mix购自Toyobo公司;引物由生工生物技术公司合成。
1.2 构建pCas-gRNA质粒
用CRISPR DESIGN程序设计针对人Ku70基因的向导RNA(guide RNA,gRNA)序列(表1),合成编码gRNA的DNA序列,退火形成双链DNA。用限制性内切酶BamHⅠ、BsmBⅠ对pCas-guide载体和退火产物进行双酶切,胶回收酶切产物。将酶切后的pCas-guide载体和退火产物用T4DNA连接酶于20℃连接4 h,连接产物转化大肠杆菌感受态细胞DH5α,涂布于氨苄西林(Amp)抗性的细菌培养板上,37℃培养过夜。挑取细菌单克隆,菌液PCR鉴定后选取阳性克隆测序。

表1 合成的引物序列
1.3 构建供体DNA质粒pBackZero-T-Ku70
以HeLa细胞基因组为模板,Ku70-F1/Ku70-R1为引物扩增Ku70左同源臂片段Ku70-L,以Ku70-F2/Ku70-R2为引物扩增Ku70右同源臂片段Ku70-R;以pSilencer2.1-U6hygro质粒为模板,Hy-F/Hy-R为引物扩增潮霉素B抗性基因片段Hy;琼脂糖凝胶回收试剂盒分别回收片段Ku70-L、Ku70-R和Hy。以片段Ku70-L、Hy为模板,Ku70-F1/Hy-R为引物,重叠PCR扩增出Ku70-LHy;再以Ku70-L-Hy和Ku70-R为模板,Ku70-F1/ Ku70-R2为引物,扩增获得片段Ku70-KO,回收目的片段。用A-Tailing Kit在Ku70-KO片段平末端添加单个碱基A后,将该片段连接到pBackZero-T载体,转化感受态大肠杆菌DH5α并挑取单克隆菌株进行菌液PCR鉴定,选取阳性克隆测序。
1.4 细胞转染、抗性筛选与鉴定
将HeLa细胞以3×105/mL的密度接种于6孔细胞培养板中,细胞生长至30%~40%时转染0.8 μg pCas-gRNA质粒和1.2 μg pBackZero-T-Ku70质粒,转染后72 h收集细胞,以4×104/mL的密度接种至6孔板中,待细胞完全贴壁后加入潮霉素B(200 μg/mL),2~3 d更换一次含同浓度潮霉素B的新鲜培养基,培养至第7 d提取细胞基因组做PCR鉴定。以提取的细胞基因组为模板,Ku70左同源臂基因上的引物K-F和潮霉素B抗性基因上的引物K-R进行PCR扩增。之后将细胞扩大培养,冻存。
1.5 单克隆稳定细胞株筛选与鉴定
用有限稀释法筛选Ku70稳定敲除的HeLa单克隆细胞株,胰酶消化细胞,以新鲜培养基稀释细胞浓度至10/mL,将稀释的细胞悬液接种至96孔板,100 μL/孔,培养1周后显微镜下观察,选取含单克隆细胞的孔进行扩大培养。扩大培养至24孔板时改用含200 μg/mL潮霉素B的培养基继续培养,1周后收集细胞进行Western印迹鉴定。
1.6 Western免疫印迹实验
收集细胞,用RIPA提取细胞总蛋白,BCA蛋白定量,行12%SDS-PAGE,每泳道35 μg蛋白,然后湿转至PVDF膜,用5%脱脂奶粉室温封闭1 h;一抗室温孵育2 h,0.1%TBS-T洗3次;二抗室温孵育1 h,0.1%TBS-T洗3次;ECL曝光显影。
1.7 CCK-8法测定Ku70稳定敲除细胞株的增殖能力
将处于对数生长期的Ku70稳定敲除细胞和野生型HeLa细胞以3×104/mL的密度接种于96孔细胞培养板,每孔100 μL,分别在0、24、48、72、96 h加入CCK8试剂,每孔10 μL,继续培养1 h后检测D450nm值,制作细胞的96 h生长曲线。
1.8 Transwell方法检测Ku70稳定敲除细胞株的迁移能力
将处于对数生长期的Ku70稳定敲除细胞和野生型HeLa细胞以2×105/mL的密度种于Tran⁃swell小室的上室,下室加入500 μL完全培养基,培养12 h后擦去未穿膜的细胞,用4%多聚甲醛固定穿膜细胞,0.1%结晶紫染色后在显微镜下观察并拍照。
1.9 Ku70稳定敲除细胞的miRNA表达水平检测
用TRIzol法提取Ku70稳定敲除细胞和野生型HeLa细胞的总RNA,取1 μg RNA反转录为cDNA。qPCR反应体系包括1 μL cDNA、1 μL引物(表1)、10 μL 2×qPCR SYBR Green mix、8 μL ddH2O。反应条件:预变性95℃ 10 min,变性95℃ 20 s,退火55℃ 20 s,延伸72℃ 20 s,50个循环,采集熔解曲线。用 StrataGene公司的Mx3000P qPCR仪检测,Mx3000P软件分析结果。
1.10 统计分析
所有实验均重复3次以上,数据以x±s表示,统计分析使用SAS 9.2软件的Student'sttest,P<0.05视为具有统计学差异。
2 结果
2.1 设计并构建pCas-gRNA质粒
用 CRISPR DESIGN程 序(http://crispr.mit. edu/)设计gRNA序列。选取Ku70编码区100~300 bp序列输入对话框,从输出的结果中选择合适的靶位点序列。为了防止脱靶,我们共设计了3条gRNA序列。将合成的gRNA互补链退火形成双链,与双酶切的线性化pCas-guide连接,获得重组质粒pCas-gRNA。转化铺板后挑取单克隆菌落进行PCR鉴定,将阳性克隆测序(图1),结果显示3条gRNA序列均正确插入pCas-guide载体,pCasgRNA质粒构建成功。
图1 pCas-gRNA质粒菌液PCR鉴定M:DNA marker;1~10:单克隆
2.2 构建供体DNA质粒pBackZero-T-Ku70
以HeLa细胞基因组为模板,分别以Ku70-F1/ Ku70-R1、Ku70-F2/Ku70-R2为引物,扩增长度分别为500和540 bp的Ku70左右同源臂Ku70-L、Ku70-R;以质粒pSilence2.1-U6 hygro为模板,Hy-F/Hy-R为引物,扩增长度为1026 bp的潮霉素抗性基因片段Hy;进而,以DNA片段Ku70-L、Hy为模板,Ku70-F1和Hy-R为引物,利用搭桥PCR扩增片段Ku70-L-Hy;再以Ku70-L-Hy和Ku70-R为模板,Ku70-F1和Ku70-R2为引物,扩增获得2066 bp的Ku70同源臂供体DNA片段Ku70-KO。琼脂糖凝胶切胶回收PCR产物中的目的片段,在其末端添加单个碱基A,再将该片段连接到pBackZero-T载体上,菌液PCR鉴定重组菌,将阳性克隆测序(图2),序列比对结果表明重组质粒序列正确,同源臂供体DNA载体pBackZero-TKu70构建成功。
图2 pBackZero-T-Ku70质粒菌液PCR鉴定M:DNA marker;1~10:单克隆
2.3 筛选鉴定Ku70敲除的多克隆细胞株
将重组质粒pBackZero-T-Ku70分别与3种pCas-gRNA质粒共转染HeLa细胞,72 h后加入终浓度为200 μg/mL的潮霉素进行抗性筛选,15 d后收集细胞,提取细胞总蛋白,Western印迹检测Ku70蛋白的表达水平。结果显示,与对照细胞相比,转染质粒pCas-gRNA3的细胞Ku70条带明显减弱,提示gRNA3敲除效果最佳(图3)。
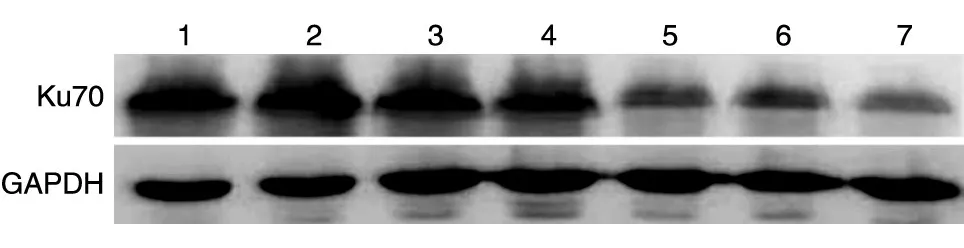
图3 Western印迹检测多克隆细胞株中Ku70的表达1:未转染的对照细胞;2:pBackZero-T-Ku70载体和pCas-gRNA1载体共转染的细胞;3:pBackZero-T-Ku70载体和pCas-gRNA2载体共转染的细胞;4~7:pBackZero-T-Ku70载体和pCas-gRNA3载体共转染的细胞
2.4 Ku70敲除细胞的基因组PCR鉴定
提取细胞基因组DNA,以K-F/K-R为引物进行PCR鉴定,结果见图4,共转染载体pBackZero-T-Ku70和pCas-gRNA3的细胞基因组DNA,经PCR扩增后在800 bp左右出现目的条带,而未转染的对照细胞没有此条带,表明潮霉素B抗性基因正确插入Ku70基因的特定位点。
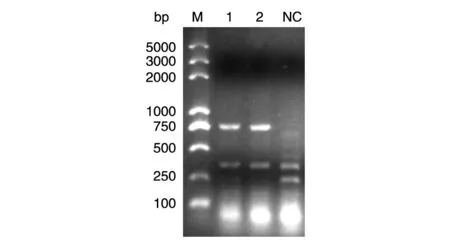
图4 基因组PCR鉴定敲除Ku70基因的单克隆细胞株1,2:共转染pBackZero-T-Ku70和pCas-gRNA3的细胞;NC:未转染的细胞
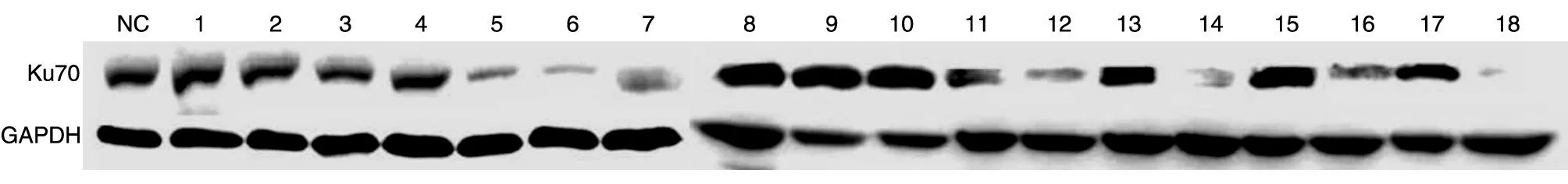
图5 敲除Ku70基因的单克隆细胞株Western印迹鉴定NC:野生型HeLa细胞;1~18:单克隆HeLa细胞株
2.5 分离并鉴定Ku70稳定敲除的单克隆细胞株
用有限稀释法筛选Ku70稳定敲除的HeLa单克隆细胞株。将细胞接种至96孔板,培养1周后选取单克隆细胞进行扩大培养,继而收集细胞,提取细胞提取总蛋白进行Western印迹鉴定(图5)。本次实验共获得18株细胞,其中14号和18号2株为敲除Ku70的单克隆细胞株。
2.6 敲除Ku70基因的稳定细胞株生物学功能检测
2.6.1 细胞增殖实验 用CCK-8法检测敲除Ku70基因的18号细胞株的增殖能力,得到18号Ku70稳定敲除细胞(KO)和野生型HeLa细胞(WT)的生长曲线(图6),与野生型比较,敲除Ku70基因的HeLa细胞增殖能力明显减弱。
2.6.2 细胞迁移实验 用Transwell方法检测18号细胞株的迁移能力,显微镜下观察并拍照(图7A);分别统计实验组和对照组穿膜的HeLa细胞数,并进行统计学分析(图7B)。实验结果表明,与野生型细胞相比,敲除Ku70基因的HeLa细胞迁移能力明显减弱。
2.6.3 实时荧光定量PCR分析敲除Ku70基因后HeLa细胞5种miRNA的表达水平 用TRIzol法提取HeLa细胞总RNA,反转录成cDNA,用qPCR仪检测5种miRNA的表达水平。结果见图8,与WT比较,敲除Ku70基因后hsa-miR-649、hsa-miR-562、hsa-miR-544a的表达水平显著上调,hsamiR-548a、hsa-miR-492水平没有显著变化。
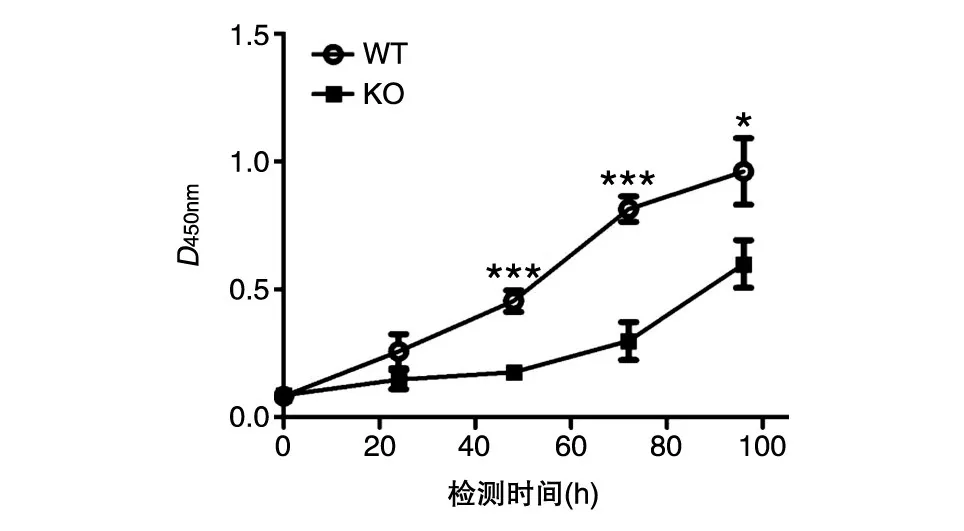
图6 Ku70基因稳定敲除细胞株生长曲线(n=3)WT:野生型HeLa细胞;KO:Ku70稳定敲除细胞;*P<0.05,***P<0.001
3 讨论
作为C-NHEJ途径中的重要因子,Ku蛋白的DNA断裂修复功能已经有了较为详尽的研究报道。与DNA断裂修复功能类似,Ku蛋白还在保持染色体端粒结构的完整性方面发挥重要作用。在酵母中的研究发现,Ku蛋白能够通过结合端粒酶RNA元件TLC1的48 nt颈环结构,协助招募端粒酶,完成端粒的延伸[11-12]。但随后又有研究表明,人Ku蛋白能够结合端粒酶47 nt的RNA元件hTR,而hTR与TLC1并没有相似序列[13]。Ku70还被证实能够通过结合细胞凋亡促进因子Bax,抑制细胞凋亡。在个体水平上,Ku蛋白还被证实与衰老有关。单独敲除Ku70或Ku80,或者二者都敲除的小鼠,均出现了类似的加速衰老现象[14]。猜测这可能与影响了C-NHEJ途径有关,因为衰老的大鼠神经元细胞内和阿尔兹海默患者脑组织内的C-NHEJ途径均明显减弱。缺失了Ku蛋白的作用后,C-NHEJ途径将进一步减弱,这或许最终导致细胞和个体衰老加速[15-16]。这些研?究结果提示,Ku蛋白的生物学功能十分广泛,还需要进一步挖掘。
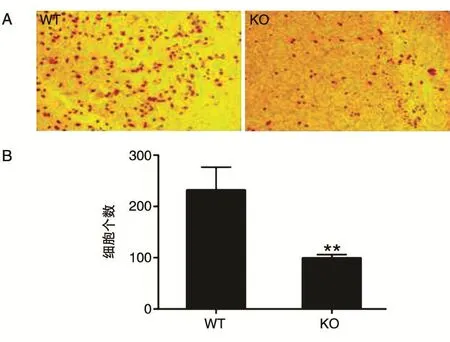
图7 Ku70基因稳定敲除细胞株迁移能力检测(n=3)WT:野生型HeLa细胞;KO:Ku70稳定敲除细胞;**P<0.01
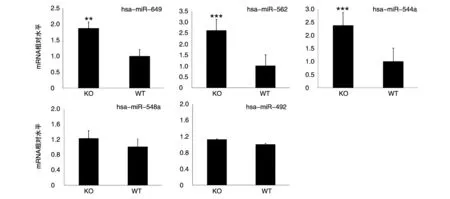
图8 qPCR检测敲除Ku70后HeLa细胞5种miRNA的表达水平(n=3)WT:野生型HeLa细胞;KO:Ku70稳定敲除细胞;**P<0.01,***P<0.001
Ku蛋白结合DNA的特异性早已确定,并且已经有研究证明Ku蛋白与DNA的结合并不是依赖特定序列或碱基的[17]。生物化学分析结果显示,Ku70亚基的3个结构域(ɑ/β结构域、DNA结合结构域、Ku80结合结构域)对于Ku蛋白聚集在双链断裂DNA末端是必需的[18]。细胞实验表明Ku70蛋白能够作为细胞内的模式受体识别外来的病毒DNA,进而介导Ⅲ型干扰素的产生[19]。此外,Ku蛋白的RNA结合特异性也被证实。比如上面提到的Ku蛋白在维持端粒结构的过程中,就发挥了招募端粒酶RNA元件的功能。有报道称Ku70蛋白能够与一类具有颈环结构并且带有一个突出基序的RNA特异结合,这表明Ku70与RNA的结合是空间特异性的[20-21]。
为了便于深入研究Ku70蛋白的生物学功能,我们构建了Ku70基因稳定敲除的HeLa细胞株,为后续实验奠定了基础。我们检测了Ku70稳定敲除细胞株的增殖和迁移能力等生物学功能,结果表明敲除Ku70基因后HeLa细胞增殖和迁移能力均有所减弱,提示Ku70可能参与了HeLa细胞的增殖和迁移过程。此外,我们前期的实验结果提示Ku70蛋白可能调节miRNA表达(结果未显示),而本研究中,我们尝试检测Ku70稳定敲除细胞株中几种可能被Ku70蛋白调节的miRNA表达水平。RT-qPCR结果显示,3种miRNA在敲除Ku70基因的HeLa细胞中明显上调,提示Ku70可能参与了这些miRNA的表达调控,相关的分子机制还有待进一步研究。
[1] Mimori T,Akizuki M,Yamagata H,et al.Characteriza⁃tion of a high molecular weight acidic nuclear protein recognized by autoantibodies in sera from patients with polymyositis-scleroderma overlap[J].Clin Invest, 1981,68(3):611-620.
[2] Mimori T,Steitz H J A.Characterization of the DNA-binding protein antigen Ku recognized by autoantibod⁃iesfrom patients with rheumatic disorders[J].Biol Chem, 1986,261(5):2274-2278.
[3] Vries E,Driel W,Bergsma W G,et al.HeLa nuclear protein recognizing DNA termini and translocating on DNA forming a regular DNA-multimeric protein com⁃plex[J].Mol Biol,1989,208(1):65-78.
[4] Griffith A J,Blier P R,Mimori T,et al.Ku polypep⁃tides synthesized in vitro assemble into complexes which recognize ends of double-stranded DNA[J].Biol Chem,1992,267(1):331-338.
[5] Grundy G J,Moulding H A,Caldecott K W,et al. One ring to bring them all-the role of Ku in mamma⁃lian non-homologous end joining[J].DNA Repair, 2014,17:30-38.
[6] Hammel M,Yu Y,Mahaney B L,et al.Ku and DNA-dependent protein kinase dynamic conformations and assembly regulate DNA binding and the initial non-homologousend joiningcomplex[J].BiolChem, 2010,285(2):1414-1423.
[7] Kragelund B B,Weterings E,Hartmann P R,et al. TheKu70/80 ringin non-homologousend-joining: easy to slip on,hard to remove[J].Front Biosci,2016, 21:514-527.
[8] Boulton S J,Jackson S P.Identification of a Saccharo⁃myces cerevisiae Ku 80 homologue:roles in DNA dou⁃ble strand break rejoining and in telomeric mainte⁃nance[J].Nucleic Acids Res,1996,24(23):4639-4648.
[9] Gravel S,Larrivee M,Labrecque P,et al.Yeast Ku as a regulator of chromo somal DNA end structure[J]. Science,1998,280(5364):741-744.
[10]Sawada M,Sun W,Hayes P,et al.Ku70 suppresses the apoptotic translocation of Bax to mitochondria[J]. Nat Cell Biol,2003,5(4):320-329.
[11]Stellwagen A E,Haimberger Z W,Veatch J R,et al. Ku interactswith telomeraseRNA topromotetelo⁃mere addition at native and broken chromosome ends [J].Genes Dev,2003,17(19):2384-2395.
[12]Fisher T S,Taggart A K,Zakian V A.Cell cycle-de⁃pendent regulation of yeast telomerase by Ku[J].Nat Struct Mol Biol,2004,11(12):1198-1205.
[13]Ting N,Yu Y,Pohorelic B,et al.Human Ku70/80 in⁃teracts directly with hTR,the RNA component of hu⁃man telomerase[J].Nucleic Acids Res, 2005,33(7): 2090-2098.
[14]Li H,Vogel H,Holcomb V B,et al.Deletion of Ku70,Ku80,or both causes early aging without sub⁃stantially increased cancer[J].Mol Cell Biol,2007,27 (23):8205-8214.
[15]Vyjayanti V N,Rao K S.DNA double strand break repair in brain:reduced NHEJ activity in aging rat neurons[J].Neurosci Lett,2006,393(1):18-22.
[16]Shackelford D A.DNA end joining activity is reduced in Alzheimer's disease[J].Neurobiol Aging,2006,27(4): 596-605.
[17]Walker J R,Corpina R A,Goldberg J.Structure of the Ku heterodimer bound to DNA and its implica⁃tions for double-strand break repair[J].Nature,2001, 412(6847):607-614.
[18]Koike M,Yutoku Y,Koike A.Accumulation of Ku70 at DNA double-strand breaks in living epithelial cells [J].Exp Cell Res,2011,317(17):2429-2437.
[19]Zhang X,Brann T W,Zhou M,et al.Ku70 is a nov⁃el cytosolic DNA sensor that induces type III rather than type I IFN[J].J Immunol,2011,186(8):4541-4545.
[20]Andrey N A,Ekaterina S K,Timofey S Z,et al.Hu⁃man Ku70 protein bindshairpin RNA and double stranded DNA through two different sites[J].Biochimie, 2017,132:85-93.
[21]Andrew B D,Karen J G,Jennifer S P,et al.RNA recognition by the DNA end-binding Ku heterodimer [J].RNA,2013,19(6):841-851.

