奶牛乳腺上皮细胞中SCAP和SREBP1蛋白调控SCD基因表达的作用研究
韩立强,孙 宇,付 彤,廉红霞,高腾云
(河南农业大学牧医工程学院/农业部动物生化与营养重点实验室,郑州 450002)
固醇调节元件结合蛋白(SREBPs)是调控细胞内胆固醇和甘油三酯合成,维持脂肪稳态的主要调控因子[1-2]。固醇调节元件结合蛋白裂解激活蛋白(SREBP cleavage activating protein,SCAP)是SREBP的结合蛋白。哺乳动物SCAP蛋白一般由跨膜螺旋(TM)和羧基(C)末端组成,其中SCAP的TM构成了一个固醇敏感结构域,C末端结构域与SREBP前体在内质网膜形成复合物[3-4]。在细胞内胆固醇减少后,SREBP-SCAP复合物从内质网转移到高尔基体,随后SREBP前体蛋白的N末端发生蛋白酶水解,生成的SREBP片段进入细胞核调控靶基因转录,而SREBP前体蛋白的C末端通过未知机制从SCAP中移除,SCAP蛋白再从高尔基体回到内质网,然后与新合成的SREBP前体蛋白循环结合[5]。
已经有多个研究发现,SCAP-SREBP是调控固醇和脂肪代谢的关键信号通路[6],介导了细胞内cAMP等多种分子对机体脂肪代谢的调节[7-8]。对反刍动物的研究发现,奶牛乳腺作为分泌乳脂的主要器官,胰岛素及一些信号分子能够调控乳腺上皮细胞的SREBP基因表达[9-10]。本实验室前期通过构建奶牛SREBP表达载体及硬脂酰辅酶A 去饱和酶基因(SCD)启动子载体发现,在奶牛乳腺上皮细胞中SREBP1可以促进SCD基因的转录[11-12],但在SCAP作用下对SREBP1调控SCD基因转录的影响还不清楚。
本研究通过在奶牛乳腺上皮细胞转染SCD启动子载体,共转染SCAP和SREBP1真核表达载体,分析对SCD启动子活性及其基因表达的影响,为阐明奶牛乳腺细胞中SCAP-SREBP1通路对于脂肪代谢基因的转录调控机制打下基础。
1 材料与方法
1.1 试验材料
奶牛乳腺上皮细胞由实验室保存,奶牛SCAP真核表达载体(pcDNA3.1-SCAP)、SREBP1真核表达载体(pcDNA3.1-SREBP1)、奶牛pGL3-SCD2/SCD3启动子载体为实验室前期构建的SCD基因启动子荧光素酶报告基因表达载体[11](序列长度分别为381和417 bp,其中奶牛pGL3-SCD3含有SRE元件,结构见图1) 由实验室构建,荧光定量PCR仪(Eppendorf,德国),Opti-mem无血清培养基(Gibco,美国),DMEM培养基(Hyclone),Lipofectamine3000(Thermo,美国),双荧光素酶检测试剂盒(Promega,美国),SYBR Green(百泰克),c-MYC Antibody(9E10)(Santa Cruz,美国),Alexa Fluor®488 Donkey Anti-Mouse IgG (H+L)(Invitrogen,美国),二氧化碳培养箱(Sanyo,日本),Fluroskan Ascent FL荧光和化学发光检测仪(Thermo,美国),激光共聚焦显微镜(Carl Zeiss LSM 5 PASCAL,德国)。

图1 奶牛pGL3-SCD2/SCD3启动子的结构Fig.1 Structure of dairy pGL3-SCD2/SCD3 promoters
1.2 方 法
1.2.1双荧光素酶报告基因系统检测SCD启动子活性将奶牛乳腺上皮细胞接种于24孔板培养,放入5% CO2、37 ℃的培养箱中进行培养。将构建好的pGL3-SCD2和pGL3-SCD3启动子用Lipofectamine3000转染至细胞,进行不同的转染处理分组:对照组(转染1.0 μg pcDNA3.1质粒),SCAP组(转染1.0 μg SCAP质粒),SREBP1组(转染1.0 μg SREBP1质粒),SCAP+SREBP1组(转染1.0 μg SCAP+1.0 μg SREBP1质粒),作用24 h后收集细胞加入裂解液,同时转染内参海肾荧光素酶质粒,采用荧光和化学发光检测仪检测荧光素酶活性并进行分析,相对启动子荧光素酶活性=萤火虫荧光素酶活性/海肾荧光素酶活性。
1.2.2荧光素酶活性检测SCAP与SCD启动子的量效关系乳腺上皮细胞接种于24孔板,将pGL3-SCD3质粒瞬时转染细胞,进行不同的转染处理分组:对照组(2.0 μg pcDNA3.1),SREBP1组(1.0 μg pcDNA3.1+1.0 μg SREBP1),SCAP(0.1)组(0.1 μg SCAP+0.9 μg pcDNA3.1+1.0 μg SREBP1),SCAP(0.5)组(0.5 μg SCAP+0.5 μg pcDNA3.1+1.0 μg SREBP1),SCAP(1.0)组(1.0 μg SCAP+1.0 μg SREBP1质粒),作用24 h后,收集细胞进行启动子活性检测,采用回归分析检测SCAP质粒含量与SCD基因启动子活性之间的量效关系。
1.2.3免疫荧光观察SREBP1蛋白在细胞核的表达在24 孔板中加入细胞爬片后接种培养乳腺上皮细胞,进行不同的转染处理分组:对照组(Control,转染1.0 μg pcDNA3.1 +1.0 μg SREBP1),SCAP组(SCAP,转染1.0 μg SCAP+1.0 μg SREBP1质粒),培养12 h后用PBS漂洗3次,多聚甲醛固定后用PBS漂洗,室温脱脂奶粉封闭2 h后,c-myc(9E10)一抗(1∶500)4 ℃孵育过夜,PBS漂洗后用绿色荧光标记的驴抗鼠IgG二抗(1∶1 000)37 ℃孵育1 h, 进行免疫荧光标记,PBS漂洗后,DAPI复染细胞核,封片后在激光共聚焦显微镜下观察细胞核中SREBP1的荧光表达情况。
1.2.4荧光定量PCR检测SCD基因mRNA的表达将乳腺上皮细胞经过胰酶消化后分散到12孔板中培养,进行不同的转染处理分组:对照组(转
染1.0 μg pcDNA3.1),SCAP组(转染1.0 μg SCAP质粒),SREBP1组(转染1.0 μg SREBP1质粒),SCAP+SREBP1组(转染1.0 μg SCAP+1.0 μg SREBP1质粒),培养48 h后Trizol提取RNA,反转录成cDNA。在NCBI上搜索奶牛SCD基因序列(NM_173959),并用Primer3 Plus设计荧光定量引物(表1),以cDNA为模板,利用SYBR Green扩增荧光定量PCR检测SCD基因mRNA的表达。同时以牛UXT基因(NM_001037471)作为内参基因,利用相对定量法计算不同处理对SCD基因mRNA表达的倍数差异。
表1荧光定量PCR引物
Table1Primersusedforquantitativereal-timePCRanalysis

引物Primer序列(5'-3')Sequence产物长度/bpSizeSCD-FCGACGTGGCTTTTTCTTCTC158SCD-RCACAACAACAGGACACCAGGUXT-FCAGCTGGCCAAATACCTTCAA125UXT-RGTGTCTGGGACCACTGTGTCAA
1.2.5数据统计试验数据采用SPSS10.0软件进行统计学分析,Sigmaplot作图,*表示P<0.05,**表示P<0.01,***表示P<0.001。
2 结 果
2.1 奶牛乳腺上皮细胞中SCAP对SCD启动子转录的影响
由图2可知,在乳腺上皮细胞中转染 pGL3-SCD2启动子后,与对照组相比,SREBP1组和SCAP组的SCD2启动子活性值有所下降,而SCAP+SREBP1组SCD2启动子活性值显著下降(P<0.05)。在转染 pGL3-SCD3启动子的乳腺上皮细胞中,与对照组相比,SCAP组对启动子活性无显著影响,SREBP1组启动子活性值极显著升高到114.53 (P<0.01),SCAP+SREBP1组启动子活性值极显著升高到192.81(P<0.001),并且SCAP+SREBP1组与SREBP1组之间SCD3启动子活性值也达到极显著差异(P<0.01)。

图2 SCAP对SCD基因启动子活性的影响Fig.2 Effect of SCAP on promoter activity of SCD gene
2.2 奶牛乳腺上皮细胞中SCAP与SCD启动子转录的量效关系
由图3可知,与对照组转染pcDNA3.1相比,转染SREBP1组启动子活性值为55.66 (P<0.01),随着SCAP质粒含量的增加,SCD基因启动子活性值从64.41(SCAP= 0.1 μg)增加到169.07(SCAP=1.0 μg),回归分析发现,SCAP质粒浓度与SCD基因启动子活性呈极显著的量效关系(P<0.01)。
2.3 SCAP对SREBP1蛋白在奶牛乳腺上皮细胞核表达的影响
由图4可知,对照组乳腺上皮细胞核中只有少量SREBP1表达绿色荧光,与DAPI染的细胞核蓝色融合(Merge)后主要呈现蓝色。转染SCAP组细胞核中SREBP1绿色荧光表达明显增多,与DAPI重叠后呈现出融合的青光。
2.4 SCAP及SREBP1蛋白对奶牛乳腺上皮细胞中SCD基因mRNA表达的影响
由图5可知,与对照组相比,SCAP组细胞中SCD基因的表达量无明显变化。SREBP1组显著上调1.23倍(P<0.05),SCAP+SREBP1组显著上调1.54倍(P<0.05),而SREBP1组与SCAP+SREBP1组SCD基因表达相比差异不显著(P=0.21)。
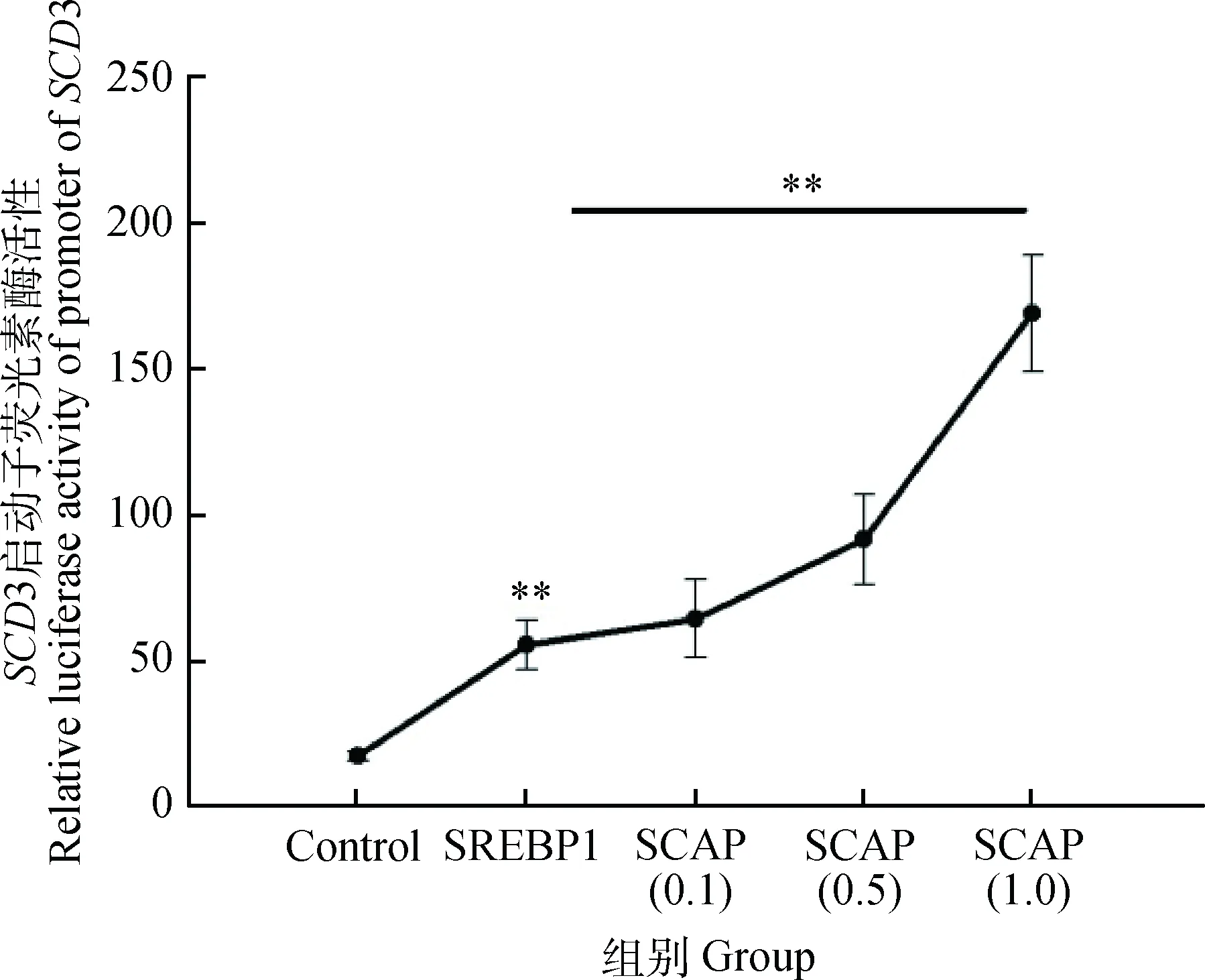
Control. 2.0 μg pcDNA3.1;SREBP1. 1.0 μg pcDNA3.1+1.0 μg SREBP1;SCAP(0.1). 0.1 μg SCAP+0.9 μg pcDNA3.1+1.0 μg SREBP1;SCAP(0.5). 0.5 μg SCAP+0.5 μg pcDNA3.1+1.0 μg SREBP1;SCAP(1.0). 1.0 μg SCAP+1.0 μg SREBP1图3 SCAP调控SCD基因启动子活性的量效关系Fig.3 Dose-response relationship between SCAP and SCD promoter activity
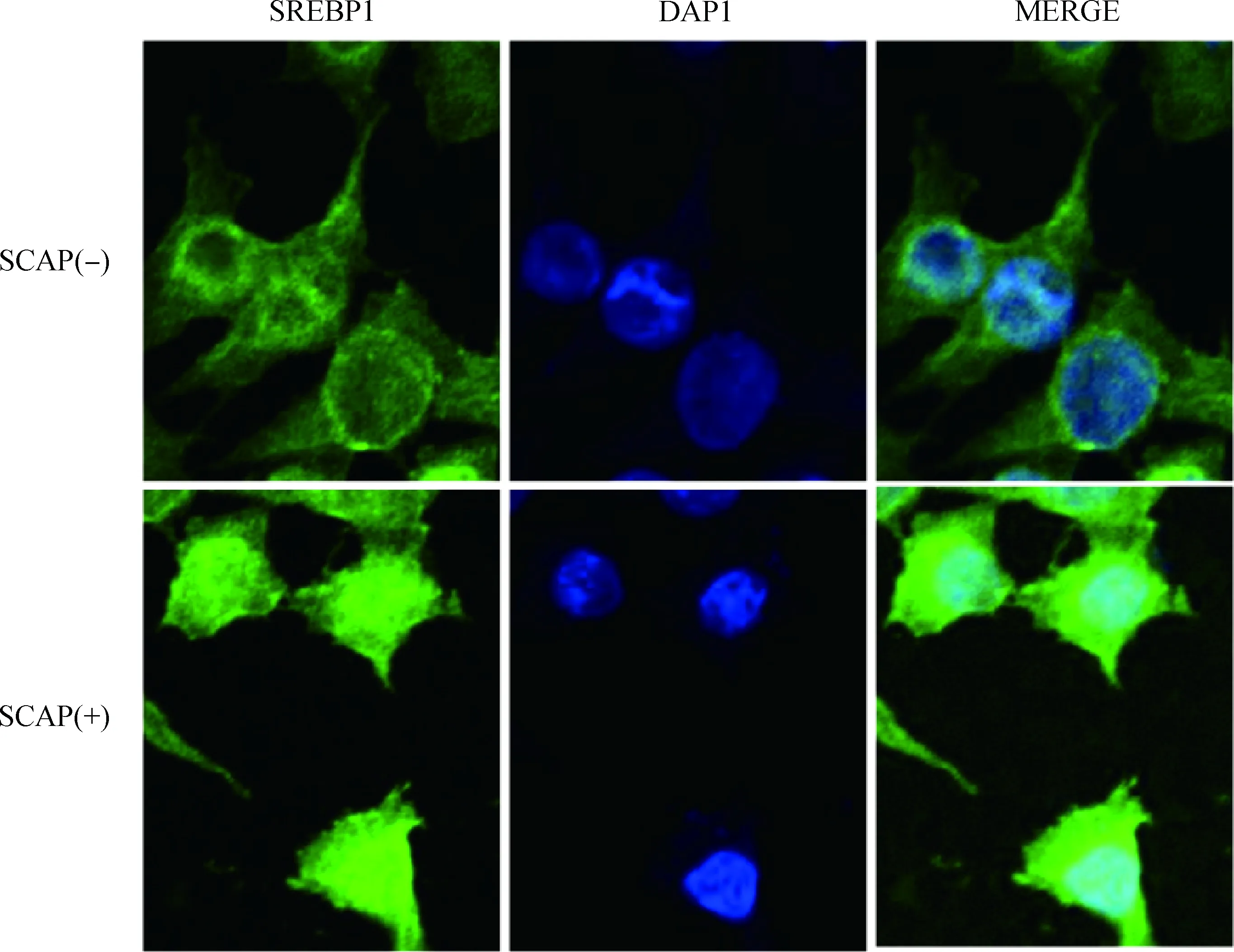
SCAP(-) . 1.0 μg pcDNA3.1 +1.0 μg SREBP1;SCAP(+). 1.0 μg SCAP+1.0 μg SREBP1图4 SCAP对SREBP1核蛋白表达的影响 600×Fig.4 Effect of SCAP on the nuclear SREBP1 expression 600×
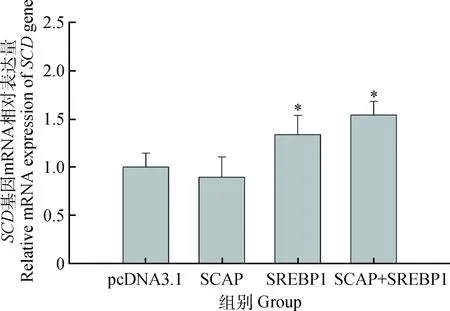
图5 SCAP与SREBP1对乳腺上皮细胞SCD基因mRNA表达的影响Fig.5 Effects of SCAP and SREBP1 on mRNA expression of SCD in mammary epithelial cells
3 讨 论
SREBPs是一种调控脂代谢的转录因子,通过包含两个跨膜序列的中心结构域锚定在内质网膜上,其N端结构域是碱性环-螺旋-亮氨酸拉链结构,能够结合靶基因启动子上的增强子序列以激活转录,这些增强子序列被称为固醇应答元件(SRE)[13-14]。本研究前期构建的SCD基因启动子序列,SCD3启动子比SCD2启动子多的36 bp片段中含有SRE序列(图1),转染SREBP1后发现SCD3启动子的活性显著升高,这符合SREBP1能够结合在基因启动子SRE上促进转录的性质,其他研究也发现过表达SREBP能够促进SCD基因的转录[15]。SREBP的蛋白活性受到多种分子的调控[16],其中包括SCAP蛋白[17]。研究表明,采用siRNA沉默小鼠SCAP基因后发现其肝的SREBP信号通路及下游靶基因表达受到抑制[18]。本试验在转染SCAP+SREBP1质粒时发现,SCD3启动子的活性比单独转染SREBP1的活性显著升高,结合SCAP质粒的浓度与SCD3启动子活性呈现出的量效关系,表明SCAP作为SREBP1的结合蛋白,能够增强SREBP1促进SCD基因转录的作用。
SCAP作为SREBP的相互作用蛋白,其调控作用主要体现在SCAP能够通过与SREBP结合转运其到高尔基体进行水解,进一步释放SREBP进入细胞核[19-20]。在SCAP基因敲除的MA10细胞中重新转染SCAP质粒,能够显著增加核SREBP蛋白的表达[7]。本研究采用免疫荧光观察发现,SCAP组的细胞核内有更多的SREBP1蛋白表达(图4),表明SCAP增强了SREBP1前体蛋白向核的转运,相应的也会增加下游靶基因的转录,与本研究中SCD基因启动子的结果相互印证(图2、图3)。C.M.Cheng等[21]通过免疫荧光发现,在葡萄糖刺激下增加细胞SCAP的表达,也能够提高SREBP在细胞核的表达。SREBP1作为调控奶牛乳腺细胞中脂肪合成的主要转录因子[22],在乳腺细胞中转染SREBP能够增加脂类基因的表达[23-24]。为进一步验证SCAP及SREBP对SCD基因表达的作用,本研究在乳腺细胞中分别转染这两种质粒,结果发现,SREBP1能够显著上调乳腺细胞SCD基因的表达,这与在羊乳腺上皮细胞中的研究结果相一致[25],同时发现,SCAP+SREBP1组也能够显著上调细胞中SCD基因的表达,并且其倍数高于SREBP1组。虽然SCAP+SREBP1与SREBP1组之间统计分析差异不显著,但这一结果也佐证了SCAP能够促进SREBP1对于SCD基因的调控作用。
X.Gong等[26]采用晶体衍射法发现,在裂殖酵母SCAP蛋白C端有7个WD重复序列形成的结构域,在与SREBP的C-端结合以促进SREBP前体蛋白转运过程中发挥重要作用,因此进一步研究奶牛等反刍动物SCAP蛋白结构和功能的关系,对于阐明乳腺细胞脂肪合成调控机制具有重要意义。
4 结 论
本研究发现,SCAP可以通过增加SREBP1蛋白在细胞核中的表达,促进对SCD基因的转录激活作用,为深入阐明奶牛乳腺细胞中SCAP-SREBP1通路对乳脂肪合成的表达调控机制提供了理论基础。
参考文献(References):
[1]SHAO W, ESPENSHADE P J. Expanding roles for SREBP in metabolism[J].CellMetab, 2012, 16(4): 414-419.
[2]MOON Y A, LIANG G S, XIE X F, et al. The Scap/SREBP pathway is essential for developing diabetic fatty liver and carbohydrate-induced hypertriglyceridemia in animals[J].CellMetab, 2012, 15(2): 240-246.
[4]SHIMANO H. SREBPs: physiology and pathophysiology of the SREBP family[J].FEBSJ, 2009, 276(3): 616-621.
[5]SHAO W, ESPENSHADE P J. Sterol regulatory element-binding protein (SREBP) cleavage regulates Golgi-to-endoplasmic reticulum recycling of SREBP cleavage-activating protein (SCAP)[J].JBiolChem, 2014, 289(11): 7547-7557.
[6]MCFARLANE M R, CANTORIA M J, LINDEN A G, et al. Scap is required for sterol synthesis and crypt growth in intestinal mucosa[J].JLipidRes, 2015, 56(8): 1560-1571.
[7]SHIMIZU-ALBERGINE M, VAN YSERLOO B, GOLKOWSKI M G, et al. SCAP/SREBP pathway is required for the full steroidogenic response to cyclic AMP[J].ProcNatlAcadSciUSA, 2016, 113(38): E5685-E5693.
[8]RU P, GUO D L. microRNA-29 mediates a novel negative feedback loop to regulate SCAP/SREBP-1 and lipid metabolism[J].RNADis, 2017, 4(1): e1525.
[9]赵艳坤, 邵伟, 王立文, 等. 与脐带间充质干细胞共培养对乳腺上皮细胞乳脂合成及关键基因表达的影响[J]. 畜牧兽医学报, 2017, 48(5): 846-853.
ZHAO Y K, SHAO W, WANG L W, et al. Effect of Co-culturing with umbilical cord mesenchymal stem cells on milk fat synthesis and expression of key genes in bovine mammary gland epithelial cells[J].ActaVeterinariaetZootechnicaSinica, 2017, 48(5): 846-853. (in Chinese)
2.4 两组患儿瓣膜反流情况 对照组患儿术后新发瓣膜反流13例(32.5%),主动脉瓣反流6例(15.0%);观察组术后新发瓣膜反流6例(14.6%),主动脉瓣反流4例(9.8%)。两组患儿新发瓣膜反流发生率对比差异有统计学意义(χ2=7.933,P<0.05)。
[10]王皓宇, 秦彤, 郝海生, 等. 胰岛素对体外培养奶牛乳腺上皮细胞乳蛋白、乳脂肪合成相关基因mRNA表达的影响[J]. 畜牧兽医学报, 2013, 44(5): 710-718.
WANG H Y, QIN T, HAO H S, et al. Effects of insulin on mRNA expression of genes related to milk protein and fat synthesis in bovine mammary epithelial cells culturedinvitro[J].ActaVeterinariaetZootechnicaSinica, 2013, 44(5): 710-718. (in Chinese)
[11]韩立强, 王月影, 王林枫, 等. 奶牛SREBP1蛋白在乳腺上皮细胞的表达定位及对SCD1基因启动子的转录调控[J]. 中国农业科学, 2016, 49(24): 4797-4805.
HAN L Q, WANG Y Y, WANG L F, et al, Expression and localization of bovine SREBP1 protein and regulation of the transcription of SCD1 promoter in bovine mammary epithelial cell[J].ScientiaAgriculturaSinica, 2016, 49(24): 4797-4805. (in Chinese)
[12]韩立强, 曹菁菁, 付彤, 等. 奶牛硬脂酰辅酶A去饱和酶基因(SCD)启动子的克隆及活性分析[J]. 农业生物技术学报, 2013, 21(4): 435-440.
HAN L Q, CAO J J, FU T, et al. Cloning and activity analysis of promoter of bovine stearoyl-CoA desaturase gene (SCD)[J].JournalofAgriculturalBiotechnology, 2013, 21(4): 435-440. (in Chinese)
[13]NOHTURFFT A, DEBOSE-BOYD R A, SCHEEK S, et al. Sterols regulate cycling of SREBP cleavage-activating protein (SCAP) between endoplasmic reticulum and Golgi[J].ProcNatlAcadSciUSA, 1999, 96(20): 11235-11240.
[14]SAKAI J, NOHTURFFT A, CHENG D, et al. Identification of complexes between the COOH-terminal domains of sterol regulatory element-binding proteins (SREBPs) and SREBP cleavage-activating protein[J].JBiolChem, 1997, 272(32): 20213-20221.
[15]ZULKIFLI R M, PARR T, SALTER A M, et al. Regulation of ovine and porcine stearoyl coenzyme A desaturase gene promoters by fatty acids and sterols[J].JAnimSci, 2010, 88(8): 2565-2575.
[16]王苗, 罗军, 许会芬, 等. 山羊INSIG1基因超表达对乳腺上皮细胞中脂质合成的影响[J]. 畜牧兽医学报, 2016, 47(9): 1806-1816.
WANG M, LUO J, XU H F, et al. Effect ofINSIG1 overexpression on the lipid synthesis in goat mammary gland epithelial cells[J].ActaVeterinariaetZootechnicaSinica, 2016, 47(9): 1806-1816. (in Chinese)
[17]XU D Q, WANG Z, ZHANG Y X, et al. PAQR3 modulates cholesterol homeostasis by anchoring Scap/SREBP complex to the Golgi apparatus[J].NatCommun, 2015, 6: 8100.
[18]JENSEN K K, TADIN-STRAPPS M, WANG S P, et al. Dose-dependent effects of siRNA-mediated inhibition of SCAP on PCSK9, LDLR, and plasma lipids in mouse and rhesus monkey[J].JLipidRes, 2016, 57(12): 2150-2162.
[19]KUWABARA P E, LABOUESSE M. The sterol-sensing domain: multiple families, a unique role?[J].TrendsGenet, 2002, 18(4): 193-201.
[20]MOON Y A. The SCAP/SREBP pathway: a mediator of hepatic steatosis[J].EndocrinolMetab(Seoul), 2017, 32(1): 6-10.
[21]CHENG C M, RU P, GENG F, et al. Glucose-mediated N-glycosylation of SCAP is essential for SREBP-1 activation and tumor growth[J].CancerCell, 2015, 28(5): 569-581.
[22]BIONAZ M, LOOR J J. Gene networks driving bovine milk fat synthesis during the lactation cycle[J].BMCGenomics, 2008, 9: 366.
[23]RUDOLPH M C, MONKS J, BURNS V, et al. Sterol regulatory element binding protein and dietary lipid regulation of fatty acid synthesis in the mammary epithelium[J].AmJPhysiolEndocrinolMetab, 2010, 299(6): E918-E927.
[24]MA L, CORL B A. Transcriptional regulation of lipid synthesis in bovine mammary epithelial cells by sterol regulatory element binding protein-1[J].JDairySci, 2012, 95(7): 3743-3755.
[25]XU H F, LUO J, ZHAO W S, et al. Overexpression of SREBP1 (sterol regulatory element binding protein 1) promotes de novo fatty acid synthesis and triacylglycerol accumulation in goat mammary epithelial cells[J].JDairySci, 2016, 99(1): 783-795.
[26]GONG X, LI J X, SHAO W, et al. Structure of the WD40 domain of SCAP from fission yeast reveals the molecular basis for SREBP recognition[J].CellRes, 2015, 25(4): 401-411.

