肾细胞癌的DNA甲基化研究新进展
陈小龙 肖跃海 孙发
肾细胞癌作为人类最常见的恶性肿瘤之一,其在成人恶性肿瘤中约占2%~3%,平均每年全球超过30万人被确诊,发病率大约以2%的速度递增[1]。它起源于近曲小管上皮细胞,较易发生转移,其发病由环境变化因素和遗传物质的异常共同决定[2]。目前对于早期肾癌的治疗主要是通过外科手术的方式,但早期诊断十分困难,约33%的肾癌患者在首诊时就有转移,40%的局限性肾癌最后也会出现远处转移[3]。转移性肾癌患者的预后较差,其5年生存率多数低于10%[4]。因此揭示肾癌潜在的致瘤机制而进行早期诊断显得尤为关键。最新研究发现,基因的表观遗传调控在肿瘤发生、发展中起主导作用[5]。关于肾癌的发病机制、早期诊断和预后等方面的DNA甲基化研究越来越受到重视,本文对DNA甲基化与肾癌之间的关系以及抑癌基因甲基化在肾癌中的相关研究做一综述。
一、DNA甲基化与肾癌之间的关系
在过去15年中,大多数研究集中在确定CpG岛或启动子区域与基因沉默的相互关系而发生的变化。但是许多其他基因组区域也具有特异性(组织特异性)或异常(肿瘤)甲基化[6-7]。这里我们将介绍肾癌特异性CpG甲基化与基因沉默之间的具体关系。
1.启动子甲基化:通过在CpG岛甲基化中发现减少的基因组,因而建立了第一种肿瘤特异性表观遗传学异常现象的机制,表明所有CpG岛低甲基化可能会诱导肿瘤的基因组的不稳定状态,这将造成肿瘤基因经历进化选择性应激,从而导致启动子的高甲基化和抑癌基因的沉默。RB1即是通过这种机制确定的第一个沉默基因[8]。癌症中许多关键的肿瘤抑制基因(如CDKN2A、RASSF1A、MLH1、CDH1等)会被启动子甲基化失活。研究表明,肾透明细胞癌(clear cell renal cell carcinoma,ccRCC)和乳头状肾细胞癌(papillary renal cell carcinoma,pRCC)中CpG岛甲基化的比例分别高达31%[9]和7%[10]。存在CpG岛表型(CpG island methylator phenotype, CIMP)的组织大多数具有肿瘤侵袭和糖酵解活性,因此CIMP的pRCC和晚期低生存率密切相关。特定区域的甲基化取决于DNA甲基转移酶1(DNMT1)、DNMT3a和DNMT3b,它们通常在癌症中过表达。这些酶与相关的CpG结合蛋白(如MBD2、MBD3和MeCP2)一起将组蛋白脱乙酰酶和其他组蛋白修饰的蛋白质转移至基因的启动子区域,从而诱导组蛋白3,4的脱乙酰化以促进染色质凝聚、基因沉默。
5-甲基胞嘧啶可被氧化成由TET家族成员(TET1、TET2和TET3)催化的5-羟甲基胞嘧啶(5-hydroxylmethylcytosine, 5hmC)。许多特异性组织聚集了大量的5hmC,表明它不仅参与基因表达的调控,而且也可能是一种特异性的表观遗传学标记。与癌旁组织相比,ccRCC组织中5hmC基因组水平显著降低[1]。除了作为ccRCC诊断的重要生物标志物外,5hmC聚集表明它在转录调控中也起重要作用[12]。然而,5hmC的缺失、信号转导及其在肾癌中的确切机制和功能尚不清楚。
2.频繁异常调节通路:WNT-β连环蛋白和MET信号通路。
两种类型的抑制蛋白调节WNT信号通路:一种与WNT产生的分泌型卷曲相关蛋白(secreted frizzled-related protein, SFRP)直接结合,并阻止其与FZ结合;另一种是Dickkopf相关蛋白(Dickkopf-related proteins, DKK),其结合WNT受体复合物LRP-5和LRP-6,是重要的组成部分。此外,胰岛素样生长因子结合蛋白(insulin-like growth factor-binding proteins, IGFBP)1,2,4,6也会通过WNT途径阻断LRP-5、LRP-6和FZ的活性。作为WNT途径的下游靶点,β-连环蛋白促进致瘤性蛋白(原癌基因c-MYC、细胞周期蛋白D1等)的表达,最终导致细胞增殖和组织浸润。研究表明,β-连环蛋白的降解是VHL抑制蛋白的关键缺氧诱导因子之一[13]。由于关键途径的把控基因启动子发生甲基化的缘故,WNT/β-连环蛋白信号通路常常被中断,并且不稳定β-连环蛋白的活化可导致肾细胞的肿瘤发生[14]。据报道,在肾细胞癌中,通过激活WNT通路中一些抑癌基因启动子甲基化可最终破坏β-连环蛋白的正常修饰[5]。研究表明,肾细胞癌中SFRP1、SFRP2、SFRP4、SFRP5、WIF1、DKK1、DKK2、DKK3的甲基化频率分别为47%、53%、53%、57%、73%、44%、58%、50%,并且蛋白质表达的抑制也将导致β-连环蛋白的积累及其相应的靶基因上调[15]。Ibanez等[16]检测20例ccRCC和10例pRCC的甲基化频率分别为35%和20%,而IGFBP1也出现沉默。
MET(也称为HGF受体)是调节某些关键致瘤过程的重要受体,例如通过PI3K/AKT/mTOR和RAS/RAFMET途径调节细胞增殖、转移和代谢。研究表明,MET中的突变与遗传性和散发性pRCC密切相关,但在散发性ccRCC中很少见[17]。SPINT2编码Kunitz型蛋白酶抑制剂2,其通过抑制HGF介导MET活化。有研究学者发现SPINT2在64例ccRCC和38例pRCC中的甲基化频率分别为30%和45%,这些研究表明在肾细胞癌中MET介导的信号往往受到干扰[18]。
EMT与细胞黏附:肾细胞癌中的VHL抑制蛋白有利于保持CDH1的表达,而CDH1编码降钙素E具有维持肾上皮形态的作用。降钙素E水平降低既可增加致瘤性,又能促进上皮-间质转化(EMT)作用[19]。多项研究发现,由于CDH1启动子的甲基化,ccRCC和pRCC中降钙素E发生沉默现象[10,15]。编码纤维蛋白原2的原纤维蛋白2(fibrillin-2,FBN2)基因通常在肾细胞癌中发生甲基化,体内FBN2表达的丧失与肾细胞癌的致瘤性密切相关。来自癌症基因组图谱的数据分析发现,FBN2是肾细胞癌中常见的表观遗传标记,200例患者中有34%被检测到该基因甲基化,3%的患者出现FBN2突变[20]。有研究表明,原钙黏蛋白8(protocadherin 8, PCDH8)是钙粘蛋白家族的第2个成员,在ccRCC中不仅会通过启动子甲基化状态表达沉默,而且其表观遗传学标志物与患者生存相关[20-21]。
虽然PCDH8的功能尚未阐明,但越来越多的研究证据表明,这种黏附分子在细胞信号转导、细胞黏附和表观遗传表型的维持中起重要作用,而4个胶原蛋白家族成员COL1A1、COL1A2、COL14A1、COL15A1还通过肾细胞癌中的启动子甲基化表现出基因下调或沉默[15-16,22-23],作为赖氨酸氧化酶同源物LOXL1基因在48例ccRCC中的作用大约1/3表现出类似现象[23],但这些蛋白质的缺失与肾细胞癌的发展有何种关系尚不清楚。然而可明确的是细胞外基质重塑过程是致瘤性和发生转移的中心环节。
除了上述信号传导通路改变外,还存在着其他因基因甲基化而沉默的信号传导通路,如APAF1、BNC1、CASP8、CDKN2A、FHIT、GREM1、MGMT、TU3A、UCHL1等[9-10,15,20]。但是对于这些基因所编码蛋白质的缺失与肾癌发生、发展是否有关,目前尚不清楚。
3.非启动子DNA甲基化:大多数甲基化分析主要针对直接起始转录起始位点的启动子区CpG甲基化。然而,该项目的大规模全基因组分析表明,远程调控增强子也会产生CpG甲基化。在基因组水平分析肾细胞癌中增强子甲基化,可发现异常增强子甲基化的富集与缺氧细胞的活性相关[24]。该研究还发现增强子甲基化与预后显著相关。目前,增强子元件的改进映射及DNA甲基化新型微阵列(包括这些区域的探针)等技术的研究,进一步拓展了非启动子DNA甲基化方面的研究途径。
二、抑癌基因甲基化与肾细胞癌的研究
1.肾细胞癌的经典抑癌基因甲基化研究:①VHL基因:VHL是肾癌中抑癌基因的典型代表,编码213个氨基酸残基组成的蛋白质pVHL。因此其失活会导致pVHL合成障碍。值得注意的是,作为多功能蛋白质,pVHL与家族性和散发性肾细胞癌的发病机制密切相关,这与其在泛素化过程中的作用有关。此外,由于体细胞突变或启动子甲基化,pVHL在肾细胞癌中的表达异常是非常多见的。据报道,VHR缺陷的ccRCC细胞系显示出加速细胞凋亡和异常细胞周期等特征。当pVHL表达正常时细胞系恢复初始生长,说明其在细胞周期调控中起着关键作用[25]。Yang等[26]研究团队通过荟萃分析,在1 998例肾细胞癌患者和294例对照组中发现在固定效应模型下,596例肾细胞癌和294例非恶性样本显示VHL启动子甲基化明显高于对照组。这项研究表明,VHL启动子甲基化可能在肾细胞癌的发生中起重要作用,并且与肾细胞癌的风险增加相关。然而,VHL启动子高甲基化与特定的临床病理特征无关。最近的研究也发现VHL突变或甲基化状态是病程较差患者的重要指标,而对肾细胞癌预后判断评估并无明显价值[27]。②p16:p16基因由2个内含子和3个外显子组成,由于其产物p16蛋白与CDK4结合发生失活可明显抑制细胞的分裂与增殖,所以p16基因在多种肿瘤发生、发展过程中失活。p16未发生基因突变时,p16 5'CpG岛再次甲基化与其完全失活相关。Maestro等[28]通过对40例散发性肾癌患者的研究发现p16甲基化发生率为20%,并且其甲基化主要集中在肾癌发生的早期。与此同时,另一项研究发现在60例肾癌患者中p16 CpG岛甲基化率约占73.3%[29],表明p16基因的突变缺失和甲基化与肾癌病理分级、临床分期呈正相关,该研究还表明其甲基化发生在肾癌发展的早期阶段。因此p16可能作为早期诊断肾癌的潜在表观遗传标志物之一。③APC基因:APC基因包含15个外显子,该基因启动子区因过度甲基化而在部分肿瘤组织中呈现出表达异常的状态。作为WNT信号传导通路中重要的抑癌基因之一,APC基因失活可诱导β连环蛋白降解发生障碍,致使细胞发生恶变。Hauser等[30]发现APC的高甲基化与晚期肿瘤分期相关。然而治疗癌症的关键在于肿瘤的早期诊断,Skrypkina等[31]研究发现APC甲基化可能有助于肾癌的早期诊断,在27例肾癌和15例健康对照样本中APC甲基化频率分别为51.9%和6.7%,表明APC基因CpG岛甲基化可作为癌症无创诊断标志物之一。然而,APC作为单细胞基因分析对肾细胞癌的诊断敏感性仍较低,研究报告显示35例肾细胞癌患者APC甲基化频率和灵敏度均为54.3%,多个基因(包括APC、PTGS2和GSTP1)诊断灵敏度提高到62.9%[30]。因此,APC不仅在肾细胞癌的发展和进展中起着重要的作用,而且在早期诊断中作为非侵入性诊断标记同样扮演着十分重要的角色。
2.肾细胞癌的新近发现抑癌基因甲基化:①GPX3基因:位于5q23的GPX3基因是肿瘤中常见的染色体杂合缺失位点。基因编码的蛋白质是谷胱甘肽过氧化物酶3,GPXs是唯一已知的细胞外糖基化酶,并且被氧化损伤。因此,GPX3基因的失活可能破坏细胞对内、外源性遗传毒性ROS的清除作用。GPX3能够催化分解过氧化物,从而破坏细胞的防御能力诱发基因突变,导致肿瘤的发生。目前GPX3已被确定为许多癌症中的肿瘤抑制因子。据报道,在6株ccRCC细胞系中的5株中检测到GPX3甲基化及下调现象,并且ccRCC中的GPX3 mRNA和蛋白表达水平显著低于相邻的非恶性肾脏组织[32]。结果表明,GPX3甲基化状态和高级别肿瘤具有显著相关性。因此,ccRCC中GPX3启动子频繁发生高甲基化导致其失活,可能作为肾肿瘤发生的关键因素,而且GPX3肿瘤特异性甲基化还可作为ccRCC早期检测和判断预后的生物标志物。②CHD5基因:CHD5位于1号染色体的短臂3区6带3亚带(1 p36.3),是CHD染色质重塑蛋白(CHD1~CHD9)家族的第5个成员。CHD5不但拥有由染色质结合域、锌指结构域、解旋酶样域、ATP结合域等重要的组件,还具有功能未知的其他结构域。CHD5染色质域和解旋酶样域与染色质空间结构的形成密不可分,可依赖于TAP提供能量而使染色质结构发生调整,因此其具有染色质重构功能。研究显示在小鼠中,由于p19 arf和p53的上调,CHD5作为剂量依赖性TSG可抑制MDM2(人同源HDM2)途径进而降解p53,从而上调p53蛋白水平,促使细胞增殖、凋亡和衰老,最终抑制肿瘤发生、发展[33]。癌症基因组数据库中CHD5甲基化在肾细胞癌组的总频率为49%(334/688),其中ccRCC最高(100%,320/320),其次为pRCC(4.1%,12/292)和嫌色肾细胞癌(3%,2/66)[34]。Du等[35]研究发现CHD5在多数正常的泌尿系器官组织(包括肾脏)中广泛表达,但在9株肾细胞癌细胞系和55例原发性肿瘤中的甲基化频率分别是78%和44%,其表达通过启动子CpG甲基化常常出现沉默或下调现象。此外,该研究还证明CHD5作为肾细胞癌的新型TSG,主要通过原发性肿瘤中的启动子甲基化而发生失活。③其他相关抑癌基因:近年来,随着DNA甲基化备受关注,研究了许多抑癌基因的异常甲基化位点并得到相关基因甲基化谱,见表1。包括MSH2、GREM-1、BTG3、DKK1、KILLIN、TCF21、FHIT、RIZ1、ADAMTS18、ECRG4、ASC/TMS1和SEMA3B等[36-43],尽管这些基因甲基化在肾癌中的作用逐渐得到认识,但是针对它们的研究大多数在少量临床样本中进行,缺乏大型临床证据的支持,因此多数作用机制并未得到阐明,有待进一步研究证实。
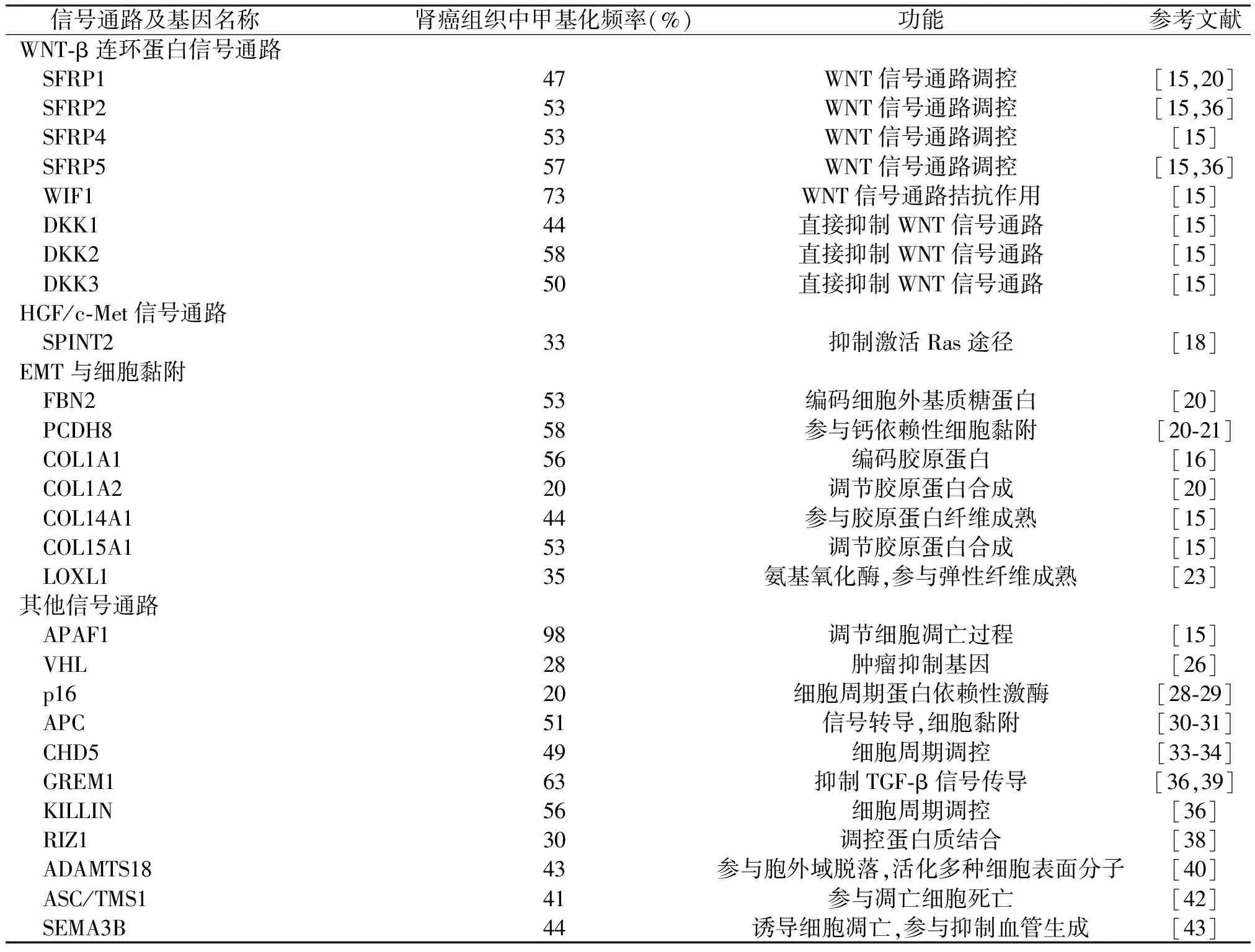
表1 肾细胞癌相关基因甲基化表达谱
三、结论
综上所述,DNA甲基化生物标志物的检测可作为鉴别肾脏肿瘤良恶性的手段之一,其具有较高的灵敏度和特异性。这些生物标志物可用于肾癌患者的早期诊断,同时DNA甲基化在肾癌的发生、发展中起着重要作用,在临床上亦可作为对肾癌患者治疗后复发监测的指标。
[1]Siegel RL, Miller KD, Jemal A. Cancer statistics, 2015[J]. CA Cancer J Clin,2015,65(1):5-29.
[2]Navai N, Wood CG. Environmental and modifiable risk factors in renal cell carcinoma[J]. Urol Oncol,2012,30(2):220-224.
[3]Flanigan RC, Campbell SC, Clark JI, et al. Metastatic renal cell carcinoma[J]. Curr Treat Options Oncol,2003,4(5):385-390.
[4]Alt AL, Boorjian SA, Lohse CM, et al. Survival after complete surgical resection of multiple metastases from renal cell carcinoma[J]. Cancer,2011,117(13):2873-2882.
[5]Morris MR, Latif F. The epigenetic landscape of renal cancer[J]. Nat Rev Nephrol,2017,13(1):47.
[6]Easwaran H, Baylin SB. Epigenetic abnormalities in cancer find a “home on the range”[J]. Cancer Cell,2013,23(1):1-3.
[7]Stirzaker C, Zotenko E, Song JZ, et al. Methylome sequencing in triple-negative breast cancer reveals distinct methylation clusters with prognostic value[J]. Nat Commun,2015,6:5899.
[8]Ohtani-Fujita N, Fujita T, Aoike A, et al. CpG methylation inactivates the promoter activity of the human retinoblastoma tumor-suppressor gene[J]. Oncogene,1993,8(4):1063-1067.
[9]Tian Y, Arai E, Gotoh M, et al. Prognostication of patients with clear cell renal cell carcinomas based on quantification of DNA methylation levels of CpG island methylator phenotype marker genes[J]. BMC Cancer,2014,14(1):772.
[10]Cancer Genome Atlas Research Network, Linehan WM, Spellman PT, et al. Comprehensive Molecular Characterization of Papillary Renal-Cell Carcinoma[J]. N Engl J Med,2016,374(2):135-145.
[11]Munari E, Chaux A, Vaghasia AM, et al. Global 5-Hydroxymethylcytosine Levels Are Profoundly Reduced in Multiple Genitourinary Malignancies[J]. Plos One,2016,11(1):e0146302.
[12]Sérandour AA, Avner S, Oger F, et al. Dynamic hydroxymethylation of deoxyribonucleic acid marks differentiation-associated enhancers[J]. Nucleic Acids Res,2012,40(17):8255-8265.
[13]Chitalia VC, Foy RL, Bachschmid MM, et al. Jade-1 inhibits Wnt signaling by ubiquitinating β-catenin and mediates Wnt pathway inhibition by pVHL[J]. Nat Cell Biol,2008,10(10):1208-1216.
[14]Kruck S, Eyrich C, Scharpf M, et al. Impact of an altered Wnt1/β-catenin expression on clinicopathology and prognosis in clear cell renal cell carcinoma[J]. Int J Mol Sci,2013,14(6):10944-10957.
[15]Morris MR, Maher ER. Epigenetics of renal cell carcinoma: the path towards new diagnostics and therapeutics[J]. Genome Med,2010,2(9):59.
[16]Ibanez de Caceres I, Dulaimi E, Hoffman AM, et al. Identification of novel target genes by an epigenetic reactivation screen of renal cancer[J]. Cancer Res,2006,66(10):5021-5028.
[17]Cancer Genome Atlas Research Network. Comprehensive molecular characterization of clear cell renal cell carcinoma[J]. Nature,2013,499(7456):43-49.
[18]Morris MR, Gentle D, Abdulrahman M, et al. Tumor suppressor activity and epigenetic inactivation of hepatocyte growth factor activator inhibitor type 2/SPINT2 in papillary and clear cell renal cell carcinoma[J]. Cancer Res,2005,65(11):4598-4606.
[19]Lamouille S, Xu J, Derynck R. Molecular mechanisms of epithelial-mesenchymal transition[J]. Nat Rev Mol Cell Biol,2014,15(3):178-196.
[20]Ricketts CJ, Hill VK, Linehan WM. Tumor-specific hypermethylation of epigenetic biomarkers, including SFRP1, predicts for poorer survival in patients from the TCGA Kidney Renal Clear Cell Carcinoma (KIRC) project[J]. Plos One,2014,9(1):e85621.
[21]Morris MR, Ricketts CJ, Gentle D, et al. Genome-wide methylation analysis identifies epigenetically inactivated candidate tumour suppressor genes in renal cell carcinoma[J]. Oncogene,2011,30(12):1390-1401.
[22]McRonald FE, Morris MR, Gentle D, et al. CpG methylation profiling in VHL related and VHL unrelated renal cell carcinoma[J]. Mol Cancer,2009,8:31.
[23]Morris MR, Ricketts C, Gentle D, et al. Identification of candidate tumour suppressor genes frequently methylated in renal cell carcinoma[J]. Oncogene,2010,29(14):2104-2117.
[24]Hu CY, Mohtat D, Yu Y, et al. Kidney cancer is characterized by aberrant methylation of tissue-specific enhancers that are prognostic for overall survival[J]. Clin Cancer Res,2014,20(16):4349-4360.
[25]Arjumand W, Sultana S. Role of VHL gene mutation in human renal cell carcinoma[J]. Tumour Biol,2012,33(1):9-16.
[26]Yang L, Zhao Z, Zhao S, et al. The Clinicopathological Significance of Epigenetic Silencing of VHL Promoter and Renal Cell Carcinoma: A Meta-Analysis[J]. Cell Physiol Biochem,2016,40(6):1465-1472.
[28]Maestro ML, Del Barco V, Sanz-Casla MT, et al. 5'CpG island methylation of the suppressor gene p16/CDKN2/MTS1 in renal carcinoma[J]. Arch Esp Urol,2000,53(5):417-423.
[29]Arai E, Kanai Y, Ushijima S, et al. Regional DNA hypermethylation and DNA methyltransferase (DNMT) 1 protein overexpression in both renal tumors and corresponding nontumorous renal tissues[J]. Int J Cancer,2006,119(2):288-296.
[30]Hauser S, Zahalka T, Fechner G, et al. Serum DNA hypermethylation in patients with kidney cancer: results of a prospective study[J]. Anticancer Research,2013,33(10):4651.
[31]Skrypkina I, Tsyba L, Onyshchenko K, et al. Concentration and Methylation of Cell-Free DNA from Blood Plasma as Diagnostic Markers of Renal Cancer[J]. Dis Markers,2016,2016:3693096.
[32]Liu Q, Jin J, Ying J, et al. Frequent epigenetic suppression of tumor suppressor gene glutathione peroxidase 3 by promoter hypermethylation and its clinical implication in clear cell renal cell carcinoma[J]. Int J Mol Sci,2015,16(5):10636-10649.
[33]Bagchi A, Papazoglu C, Wu Y, et al. CHD5 is a tumor suppressor at human 1p36[J]. Cell,2007,128(3):459-475.
[34]Davis CF, Ricketts CJ, Wang M, et al. The somatic genomic landscape of chromophobe renal cell carcinoma[J]. Cancer Cell,2014,26(3):319-330.
[35]Du Z, Li L, Huang X, et al. The epigenetic modifier CHD5 functions as a novel tumor suppressor for renal cell carcinoma and is predominantly inactivated by promoter CpG methylation[J]. Oncotarget,2016,7(16):21618-21630.
[36]Shenoy N, Vallumsetla N, Zou Y, et al. Role of DNA methylation in renal cell carcinoma[J]. J Hematol Oncol,2015,8:88.
[37]Lakshminarasimhan R, Liang G. The Role of DNA Methylation in Cancer[J]. Adv Exp Med Biol,2016,945:151-172.
[38]Ge P, Yu X, Wang ZC, et al. Aberrant Methylation of the 1p36 Tumor Suppressor Gene RIZ1 in Renal Cell Carcinoma[J]. Asian Pac J Cancer Prev,2015,16(9):4071-4075.
[39]van Vlodrop IJ, Baldewijns MM, Smits KM, et al. Prognostic significance of Gremlin1 (GREM1) promoter CpG island hypermethylation in clear cell renal cell carcinoma[J].Am J Pathol,2010,176(2):575-584.
[40]Xu B, Zhang L, Luo C, et al. Hypermethylation of the 16q23.1 tumor suppressor gene ADAMTS18 in clear cell renal cell carcinoma[J]. Int J Mol Sci,2015,16(1):1051-1065.
[41]Luo L, Wu J, Xie J, et al. Downregulated ECRG4 is associated with poor prognosis in renal cell cancer and is regulated by promoter DNA methylation[J]. Tumor Biol,2016,37(1):1121-1129.
[42]Liu Q, Jin J, Ying J, et al. Epigenetic inactivation of the candidate tumor suppressor gene ASC/TMS1 in human renal cell carcinoma and its role as a potential therapeutic target[J]. Oncotarget,2015,6(26):22706-22723.
[43]Loginov VI, Dmitriev AA, Senchenko VN, et al. Tumor Suppressor Function of the SEMA3B Gene in Human Lung and Renal Cancers[J]. Plos One,2015,10(5):e0123369.

