食管鳞癌中miR-204、miR-205相关ceRNA网络构建及部分lncRNA功能分析
马 军, 李雨濛, 李 南, 任凯凯, 马佳康, 孙 佳, 张 进, 苏彦河,袁 翎, 段芳龄
1.郑州大学第二附属医院检验科,河南 郑州 450014;2.郑州大学消化疾病研究所;3.郑州大学第二附属医院肿瘤科;4.郑州大学第二附属医院胸外科;5.河南省肿瘤医院放疗科
食管癌是世界上最常见的恶性肿瘤之一,发病率位于第8位,占所有癌症死亡率的第6位。中国食管癌病例占全球一半以上,每年约有15万人死于这一疾病,是我国癌症相关死亡的第4大病因。食管癌主要有食管鳞癌(esophageal squamous cell carcinoma, ESCC)和食管腺癌2个亚型,我国ESCC的发病率在90%以上[1]。高死亡率是食管癌的一个典型特征,因此迫切需要新的生物标记物来更精确地诊断和治疗食管癌,尤其是ESCC。
microRNA (miRNA) 是一类由内源基因编码的、长度为20~24个核苷酸的小RNA,在细胞分化、生物发育及疾病发生、发展过程中发挥巨大作用。既往研究表明,miR-204、miR-205可能参与肿瘤的形成与进展[2],但在食管癌中的研究仍较少。
PANDOLFI等[3]于2011年提出的ceRNA(竞争性内源RNA)假说认为,内源性RNA分子具有miRNA作用位点,可以竞争性地与miRNA结合,从而间接调控miRNA靶基因的作用。ceRNA假说的提出赋予mRNA和非编码RNA新的、更为广泛的生物学功能。不同类型的RNA之间,包括长链非编码RNA(lncRNA)、环状RNA(circRNA)、mRNA等,通过RNA-RNA相互作用所构成的调控网络在病理生理过程中发挥重要作用,包括肿瘤的生成和发展[4]。
lncRNA是长度>200个核苷酸的非编码 RNA[5],在细胞分化、个体发育过程中发挥着重要作用,与人类众多疾病的发生、发展都有着密切的联系。有研究[6]表明,lncRNA可能在肿瘤的发生、发展、浸润、转移中发挥关键作用。
本研究用TCGA数据库中ESCC相关转录组数据,筛选出差异表达的mRNA、lncRNA及miRNA,成功构建了miR-204、miR-205相关lncRNA-miRNA-mRNA的ceRNA调控网络,并找出与亚洲ESCC患者生存相关的lncRNA。通过对7个同时调节miR-204、miR-205的lncRNA的靶基因进行GO、KEGG、蛋白互作(PPI)网络等分析,预测这些lncRNA在亚洲ESCC中的临床意义、分子调控机制,为ESCC的诊断和治疗提供新的靶点。
1 资料与方法
1.1TCGA数据下载及差异基因筛选从TCGA(https://cancergenome.nih.gov/)数据库下载ESCC mRNA、miRNA的测序数据及临床信息。截至2018年2月23日,数据库中收录ESCC mRNA测序数据92例,其中81例ESCC组织样本及11例癌旁正常组织样本;收录miRNA ESCC测序数据108例,包括ESCC组织样本95例,癌旁正常组织样本13例。其中亚洲ESCC mRNA测序样本37例,miRNA测序样本44例。使用R语言“edgeR”包筛选差异基因,筛选条件为:log 2-fold change (logFC)>2且P<0.05。
1.2ceRNA网络构建筛选出的差异lncRNA、差异miRNA在miRcode数据库中进行配对。利用starBase在线软件(http://starbase.sysu.edu.cn/)对筛选出的差异miRNA进行靶基因预测,miRDB、miRTarBase、TargetScan 3个数据库预测靶基因,从而得到lncRNA-miRNA-mRNA 的ceRNA调控网络,利用Cytoscape v3.5.1软件制图。
1.3生存分析利用TCGA数据库中亚洲人ESCC的生存时间,采用的是R语言中的“Survival”软件包,对特异的lncRNA进行生存分析,P<0.05被认为差异有统计学意义。
1.4lncRNA靶基因预测、GO及KEGG分析lncRNA靶基因预测利用MEM(https://biit.cs.ut.ee/mem/)在线分析,每个lncRNA选取200个靶基因进行下一步分析。GO、KEGG使用Cytoscape中的“ClueGO”、“CluePedia”APP完成,筛选标准为P<0.05。
1.5蛋白互作网络利用在线软件STRING(https://string-db.org/)寻找蛋白质之间的相互作用关系。相关图表利用Cytoscape中的“stringAPP”完成,并删除未出现在互作网络中的蛋白质。
2 结果
2.1ESCC差异基因筛选及miR-204、miR-205相关ceRNA网络构建TCGA数据库中ESCC的转录组测序数据分析,共筛选出差异lncRNA 1 160个、mRNA 2 064个、miRNA 69个。通过ceRNA网络构建发现,miR-204、miR-205是ESCC中重要的节点基因,提取其相关的lncRNA 38个、mRNA 13个,并成功构建了以miR-204/205为节点的ceRNA调控网络。其中FER1L6-AS1、MIR205HG、TM4SF19-AS1、ADAMTS9-AS2、HOTTIP、LINC00520、SOX2-OT、LINC00524和 RMST这9个lncRNA可同时调控miRNA-204及miRNA-205(见图1)。
2.2miR-204、miR-205相关lncRNA生存分析为了研究差异基因与亚洲ESCC患者总生存期的关系,我们对miR-204、miR-205相关的lncRNA、mRNA进行OS分析,结果显示,FER1L6-AS1和LINC00504与亚洲ESCC患者生存显著相关(见图2),未发现与亚洲ESCC患者生存相关的mRNA(见表1)。
2.3lncRNA靶基因预测及GO、KEGG分析用MEM在线软件对9个lncRNA进行靶基因预测,LINC00524和TM4SF19-AS1未预测出下游靶基因,其余7个lncRNA根据相关性选取前200个作为目标靶基因,去除重复值后共有1 165个靶基因。1 165个预测靶基因在TCGA中2 064个差异表达mRNA中验证,筛选出141个mRNA作为最终靶基因。GO分析共发现9个细胞成分、7个分子功能相关富集,主要参与细胞间连接、突触、通道活性、被动跨膜转运蛋白活性等功能。KEGG分析发现4条相关通路,包括神经活性配体-受体相互作用、逆行内源性大麻素信号、GABA能突触、吗啡成瘾等通路(见图3)。
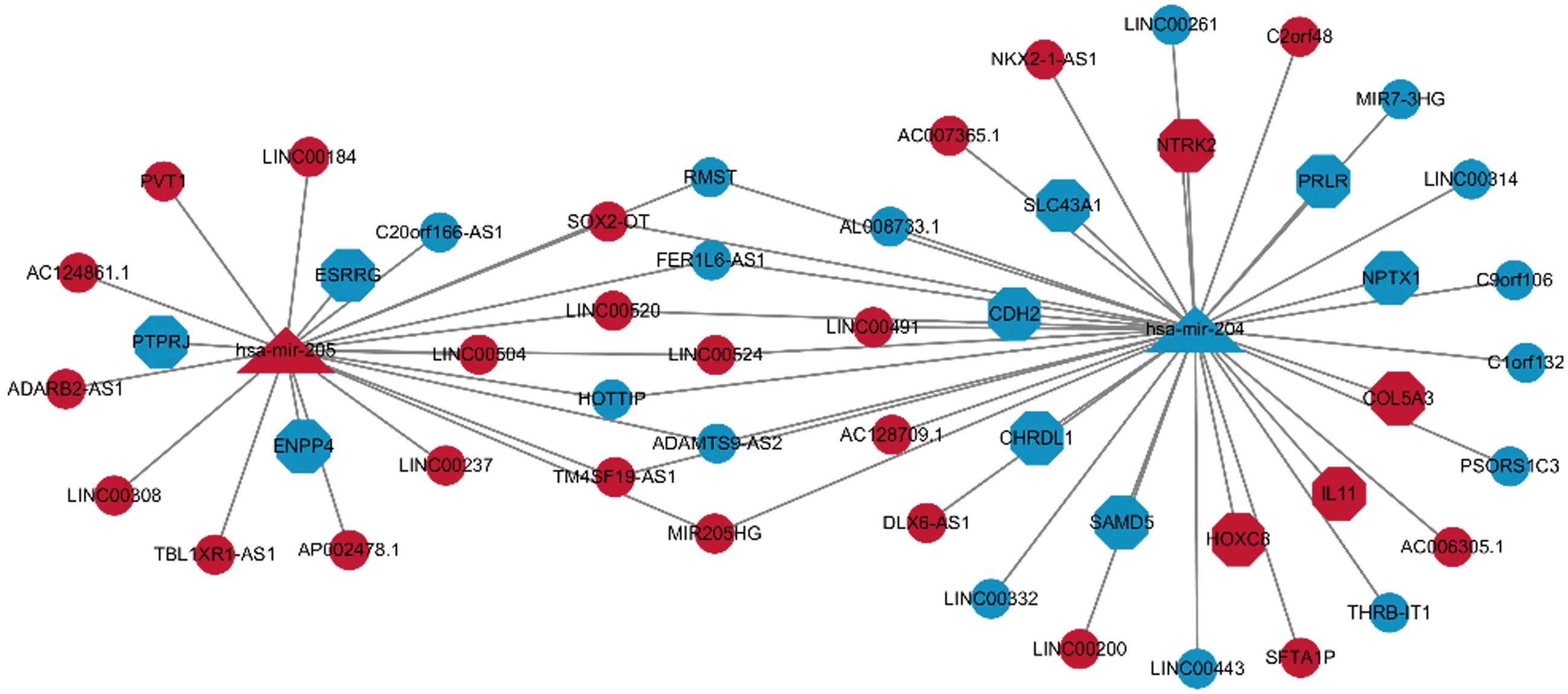
注:红色代表上调基因,蓝色代表下调基因。三角形、圆形及八边形分别代表miRNA、lncRNA和mRNA。

lncRNAGeneID基因坐标(hg19)表达水平P值FDRFER1L6-AS1ENSG00000181171chr8:123,984,138-124,040,782下调2.06E-075.24E-06LINC00504ENSG00000248360chr4:14,470,465-14,888,169上调0.00130.0085
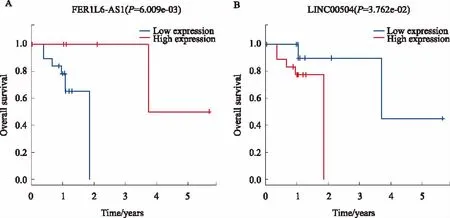
图2 亚洲人ESCC生存相关lncRNA Kaplan-Meier生存曲线
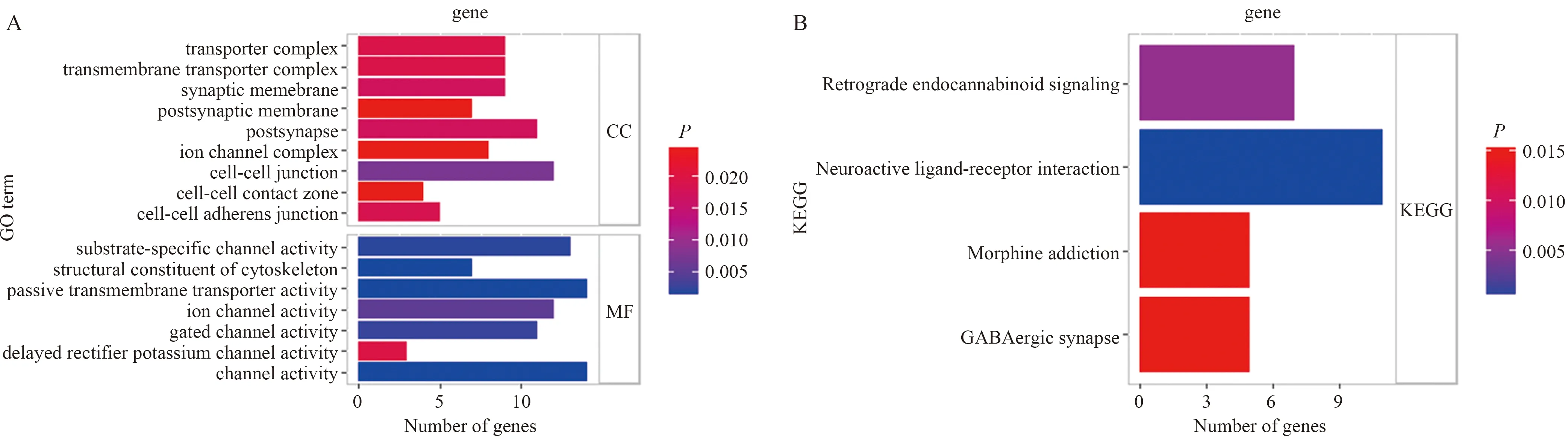
图3 lncRNA靶基因GO、KEGG分析
2.4PPI网络构建141个靶基因通过STRING在线分析其相应编码蛋白质之间的相互作用关系,结果显示,54个编码蛋白质形成包含67个互相作用关系的蛋白互作网络(见图4)。从图中可以看出:GNG7、ADCY5、NMUR2、PTGER3、KPT5位于网络的中心,与多种蛋白质之间存在相互作用。
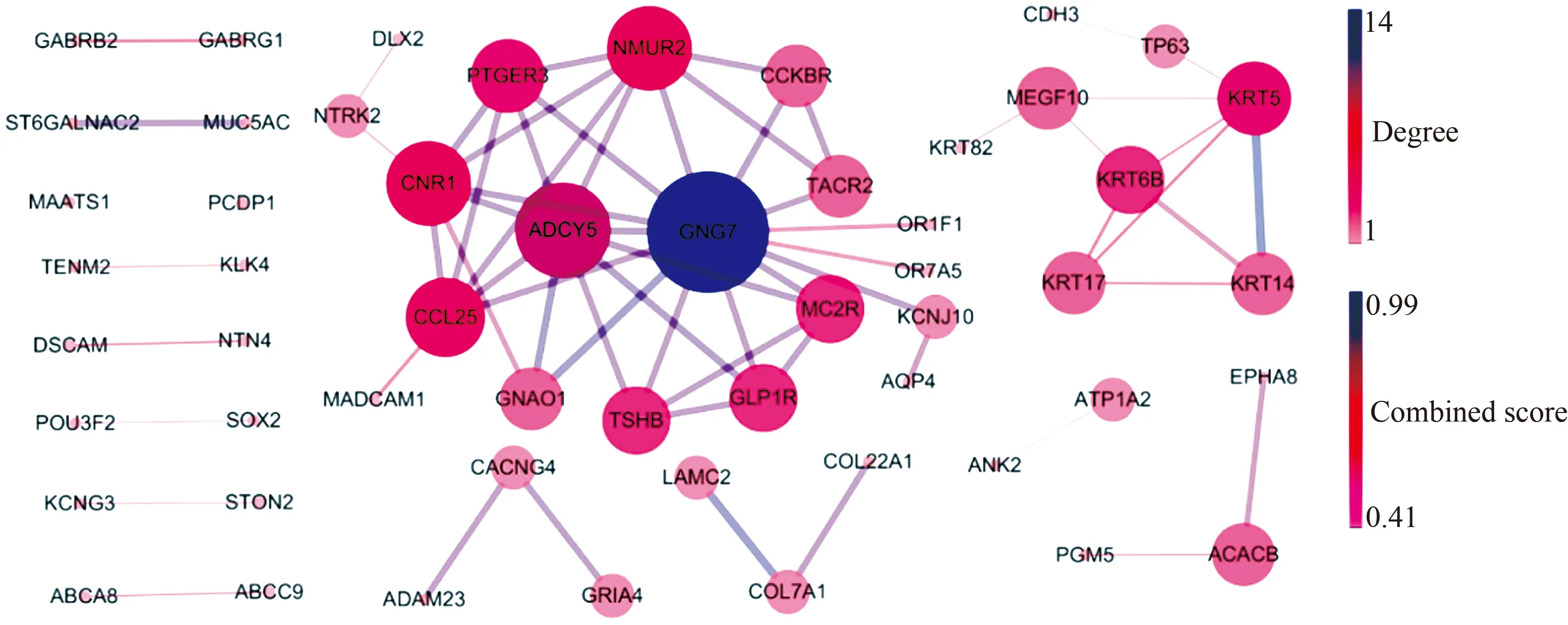
图4 lncRNA靶基因编码蛋白PPI网络
3 讨论
miRNA是目前研究最多的非编码RNA,它可以作为癌基因或抑癌基因[7],在肿瘤的生成、侵袭、转移等病理过程中发挥重要作用。既往研究[2]表明,miR-204、miR-205与多种肿瘤的形成与进展关系密切。
miR-204在ESCC细胞(EC109、TE10)中,可以下调(FOXM1),从而抑制瘤细胞增殖[8];术后复发的ESCC患者miR-204表达降低[9];在ESCC中,miR-204作为ceRNA成员,其作用可被lncRNA UCA1竞争,从而解除miR-204对SOX4基因的抑制,促进ESCC细胞增殖[10]。我们的分析结果发现,miR-204在ESCC中表达下调,这表明miR-204可能作为抑癌基因参与ESCC的形成与进展。
在ESCC中,生活方式可能改变miR-205在肿瘤组织中的表达,吸烟可以使肿瘤组织miR-205表达增高,而酗酒可以使其表达降低[11]。PAN等[12]发现,miR-205能促进ESCC细胞EMT活性、抑制凋亡,并通过DNA修复信号通路促进放疗抵抗,因此,在ESCC中,miR-205起着癌基因的作用。然而,MATSUSHIMA等[13]基因芯片结果显示,尽管在ESCC中miR-205高表达,但表现为抑癌基因作用,可以抑制瘤细胞侵袭和迁移,并通过抑制其靶基因ZEB2,抑制瘤细胞EMT活性;而且,XU等[14]也发现,miR-205可以在ESCC患者血中表达下降。HEZOVA等[15]的实验显示,miR-205在不组织类型的食管癌中,作用不同,在食管腺癌中表现为抑癌基因,而在ESCC表现为癌基因。
越来越多的证据表明,ceRNA在基因表达的调节中具有重要的作用,并参与肿瘤的生成与进展[16]。目前文献ESCC中ceRNA调控网络已经发现很多,但miR-205在ESCC中的ceRNA网络还未见报道,miR-204的ceRNA网络文献报道的仅有UCA1-miR-204-SOX4,因此,ESCC中lncRNA相关的ceRNA调控机制仍需要进一步深入研究。在本研究中,我们发现,miR-204和miR-205是ESCC ceRNA网络中关键的调控节点,以此为节点,这一调控网络的预测可能对研究ESCC生成、进展相关机制提供新的方法。
对ceRNA网络分析发现,9个lncRNA可同时调控miR-204及miR-205,我们推测这些lncRNA与ESCC关系密切。其中LINC00504与患者的生存呈负相关,其表达量越高患者的生存时间越短;FER1L6-AS1与患者生存呈正相关,表达量越高患者生存时间越长。这表明FER1L6-AS1和LINC00504可能是ESCC患者预后的生物标志物。
另外,这些lncRNA的靶基因与细胞间连接、突触、通道活性、被动跨膜转运蛋白活性等功能及神经活性配体—受体相互作用、逆行内源性大麻素信号、GABA能突触、吗啡成瘾等通路显著相关,可能参与ESCC的形成与进展。PPI网络为我们进一步研究lncRNA对基因的调控和在ESCC中的作用机制有积极的提示价值。
[1] DENG H Y, WANG Y C, NI P Z, et al. Long noncoding RNAs are novel potential prognostic biomarkers for esophageal squamous cell carcinoma: an overview [J]. J Thorac Dis, 2016, 8(8): E653-E659. DOI: 10.21037/jtd.2016.07.01.
[2] LAGES E, IPAS H, GUTTIN A, et al.MicroRNAs: molecular features and role in cancer [J]. Front Biosci (Landmark Ed), 2012, 17: 2508-2540.
[3] SALMENA L, POLISENO L, TAY Y, et al. A ceRNA hypothesis: the Rosetta Stone of a hidden RNA language? [J]. Cell, 2011, 146 (3): 353-358. DOI: 10.1016/j.cell.2011.07.014.
[4] TAY Y, RINN J, PANDOLFI P P. The multilayered complexity of ceRNA crosstalk and competition [J]. Nature, 2014, 505(7483): 344-352. DOI: 10.1038/nature12986.
[5] SPIZZO R, ALMEIDA M I, COLOMBATTI A, et al. Long non-coding RNAs and cancer: a new frontier of translational research? [J]. Oncogene, 2012, 31(43): 4577-4587. DOI: 10.1038/onc.2011.621.
[6] SCHMITT A M, CHANG H Y. Long Noncoding RNAs in Cancer Pathways [J]. Cancer Cell, 2016, 29(4): 452-463. DOI: 10.1016/j.ccell.2016.03.010.
[7] LING H, GIRNITA L, BUDA O, et al. Non-coding RNAs: the cancer genome dark matter that matters! [J]. Clin Chem Lab Med, 2017, 55(5): 705-714. DOI: 10.1515/cclm-2016-0740.
[8] SUN Y, YU X, BAI Q. miR-204 inhibits invasion and epithelial-mesenchymal transition by targeting FOXM1 in esophageal cancer [J]. Int J Clin Exp Pathol, 2015, 8(10): 12775-12783.
[9] OKUMURA T, KOJIMA H, MIWA T, et al. The expression of microRNA 574-3p as a predictor of postoperative outcome in patients with esophageal squamous cell carcinoma [J]. World J Surg Oncol, 2016, 14(1): 228. DOI: 10.1186/s12957-016-0985-3.
[10] JIAO C, SONG Z, CHEN J, et al. lncRNA-UCA1 enhances cell proliferation through functioning as a ceRNA of Sox4 in esophageal cancer [J]. Oncol Rep, 2016, 36(5):2960-2966. DOI: 10.3892/or.2016.5121.
[12] PAN F, MAO H, BU F,et al. Sp1-mediated transcriptional activation of miR-205 promotes radioresistance in esophageal squamous cell carcinoma [J]. Oncotarget, 2017, 8 (4):5735-5752. DOI: 10.18632/oncotarget.13902.
[13] MATSUSHIMA K, ISOMOTO H, YAMAGUCHI N, et al. MiRNA-205 modulates cellular invasion and migration via regulating zinc finger E-box binding homeobox 2 expression in esophageal squamous cell carcinoma cells [J]. J Transl Med, 2011, 9:30. DOI: 10.1186/1479-5876-9-30.
[14] XU H, YAO Y, MENG F, et al. Predictive value of serum miR-10b, miR-29c, and miR-205 as promising biomarkers in esophageal squamous cell carcinoma screening [J]. Medicine, 2015, 94(44):e1558. DOI: 10.1097/MD.0000000000001558.
[15] HEZOVA R, KOVARIKOVA A, SROVNAL J, et al. MiR-205 functions as a tumor suppressor in adenocarcinoma and an oncogene in squamous cell carcinoma of esophagus [J]. Tumour Biol, 2016, 37(6): 8007-8018. DOI: 10.1007/s13277-015-4656-8.
[16] HUANG M, ZHONG Z, LV M, et al. Comprehensive analysis of differentially expressed profiles of lncRNAs and circRNAs with associated co-expression and ceRNA networks in bladder carcinoma [J]. Oncotarget, 2016, 7(30): 47186-47200. DOI: 10.18632/oncotarget.9706.

