大鼠骨髓间充质干细胞分化成肝细胞过程中差异基因的功能研究
姜 华,高建鹏,王 辉,张志波
昆明医科大学附属医院延安医院消化内科,云南 昆明 650051
肝移植作为治疗终末期肝病唯一有效的治疗手段,由于其需求与捐献器官的数量严重不匹配,严重限制了其临床应用[1-2]。研究[3]发现,间充质干细胞(mesenchymal stem cells,MSCs)具有自我更新的能力,可分化成脂肪细胞、成骨细胞、软骨细胞、骨骼细胞和细胞的内脏中胚层,也可分化成外胚层的细胞,如肝细胞[4-5]、神经元[6-7]、心肌细胞[8-9]。由于受伤的器官或组织释放细胞因子,MSCs有能力通过趋化性迁移到受伤组织[10-11]。在受伤的组织微环境,比如一些炎症因素,MSCs增殖分化成内生的肝细胞样细胞(hepatocyte like cells,HLCs)[12]。MSCs向HLCs进行诱导分化培养,可获得HLCs,并应用于肝病的治疗,例如肝纤维化、肝损伤、爆发性肝衰竭和肝再生[13-14]。近年来,肝细胞移植被应用到肝脏疾病的治疗,成为一种有潜力的个性化医疗方向[15]。过去,有一些应用肝细胞疗法的研究[16-17]。事实上,许多临床前和临床研究已经进行了使用这种方法治疗代谢性或终末期肝脏疾病的尝试[18]。利用肝或肝外来源的细胞重建一个有功能的肝脏已经有所努力[19],是一种可应用的治疗方法[20]。移植的HLCs不但形态与肝细胞相同,更重要的是具有成熟正常肝细胞(normal liver cells,NLCs)的功能。然而,HLCs不能完全作为“真正的NLCs”行使作用,细胞培养影响HLCs的生存能力和移植后功能发挥[21]。
全基因组表达研究通过微阵列分析了解复杂生物系统,微阵列表达分析以基于杂交的单链核酸片段互补为研究原理[22]。基因芯片能够研究差异表达基因,找到基因调控网络及其机制,揭示不同层次多基因协同作用的生命过程。基因的表达随着分化的过程也呈现出相应的有规律的表达趋势,通过对差异基因的表达趋势聚类和功能分析,为进一步寻找分化关键基因、阐明分化调节机制提供线索,关键基因可能作为预后生物标记[23]和治疗目标[24]。通过特定的分子机制和信号通路,在肝细胞微环境中影响肝细胞表达谱系,从而按照特定方向进行分化[25]。我们基于基因表达谱芯片数据,通过生物信息学分析研究,利用基因的表达丰度构建MSCs、HLCs、NLCs动态发展过程中基因的表达调控网络,通过对调控网络进行网络学分析,从中筛选出核心基因,了解基因间的相互作用关系,探寻它们在动态变化过程的调控机制。
1 材料与方法
1.1材料本实验选取SD大鼠,雌雄各半,6~8周龄(体质量160~200 g),购自昆明医学院实验动物中心,提取大鼠骨髓MSCs,DMEM/F12培养基为Hyclone公司的瓶装、除菌液体培养基,在培养基中添加肝细胞提取液,经肝细胞提取液诱导分化后的HLCs做基因芯片实验,采用Illumina大鼠全基因组基因表达芯片ratref-12,由上海生物芯片有限公司完成,其实验和数据分析方法如下。
1.2总RNA抽提(1)细胞培养皿中细胞样品用1×PBS洗两次后,用1 ml枪将PBS吸干净,加入1 ml Trizol(Invitrogen)溶液,吹打混匀,并吸至1.5 ml RNase free EP管中使细胞充分裂解,室温静置5 min;组织样品用液氮充分研磨,加入1 ml Trizol(Invitrogen)溶液,混匀,室温放置5 min使其充分裂解(管盖与管壁都需标记样品名称);(2)加入200 μl氯仿,剧烈振荡混匀30 s,使水相和有机相充分接触,室温静置3~5 min(离心时离心管按顺序排放,离心完毕,离心管的顺序也按顺序排好,与第一步的顺序一致);(3)4 ℃下,14 000×g离心15 min,可见分为三层,RNA在上层水相,移至另一个新的RNase free EP管(用20~200 μl的枪吸取上清,吸上清时,枪头应沿着液面上层吸取上清,枪头不可碰到、吸到中间层);(4)沉淀RNA:加入等体积异丙醇,轻柔地充分混匀(颠倒6~8次)(不应用振荡器混匀),室温静置10 min;(5)4 ℃下,14 000×g离心10 min,收集RNA沉淀(如离心后仍不见EP管底部有沉淀,应将EP管放置在-80 ℃冰箱过夜,继续在4 ℃下,14 000×g离心10 min,收集RNA沉淀),去上清;(6)用质量浓度为75 g/L的乙醇洗涤两次(12 000×g离心5 min)(加入乙醇后只需轻轻颠倒EP管即可,不用振荡器或枪头吸打沉淀),超净台风干;沉淀不能过干或过湿,过干则不易溶解,过湿则乙醇残留;(7)视沉淀量加入适量DEPC水(至少15 μl)溶解沉淀。
1.3去基因组使用RNase-free的DNase(Promega),按RNA 30 μl,DNase 20 μl,10×buffer 10 μl,H2O(RNase free)39.5 μl,配置反应液,37 ℃消化30 min,65 ℃灭活10 min。然后按以下步骤操作:(1)加入等体积的苯酚/氯仿,上下颠倒混匀,室温放置5 min,后14 000×g,离心15 min,取上清;(2)加入等体积的氯仿,上下颠倒混匀,静置分层后14 000×g,离心15 min,取上清;(3)加入等体积异丙醇,轻柔地充分混匀(颠倒6~8次),-20 ℃静置15 min;(4)4 ℃下,14 000×g离心15 min,收集RNA沉淀,去上清;(5)用质量浓度为750 g/L的乙醇洗涤两次(12 000×g离心5 min),超净台风干;(6)加入适量DEPC水(至少15 μl)溶解沉淀。
1.4总RNA纯度和完整性检测(1)纯度检测:取1 μl RNA样品50倍稀释,在核酸蛋白检测仪上测定OD值,OD260/OD280的比值>1.8,说明制备的RNA较纯,无蛋白质污染;(2)总RNA完整性检测:取RNA样品1 μl,质量浓度为10 g/L的琼脂糖凝胶电泳80 V×20 min,EB染色10 min,用凝胶成像系统观察并拍照,总RNA的5s rRNA、18s rRNA和28s rRNA条带,三条条带完整即可证明总RNA抽提比较完整。
1.5逆转录(1)在RNase free的PCR管中配置Total RNA H2O 1.0 μl溶液;(2)将上述溶液吹打均匀,置85 ℃保温5 min,使RNA变性。随后立即冰上致冷,以防止RNA复性;(3)在该PCR管中加入oligo(dT)0.5 μl,Random primer 0.5 μl,10 mmol/L dNTP 2.0 μl,RNase inhibitor 0.5 μl,5×buffer 4.0 μl,M-MLV 0.5 μl试剂;(4)将上述20 μl反应溶液30 ℃保温10 min;(5)42 ℃保温50 min;(6)85 ℃保温10 min;(7)-20 ℃保存。
1.6芯片杂交、扫描及数据提取(由上海伯豪生物科技有限公司完成) (1)大鼠全基因组基因表达芯片ratref-12芯片,经检测后发现RNA质量完好,进行标记杂交实验。杂交信号以数据形式输出。芯片结果采用illumina SherlockTM1000分析扫描仪进行扫描,得到Cy3和Cy5的图像文件,通过划格确定杂交点的范围,过滤噪声背景,提取基因表达的荧光信号强度值。用BeadStudio软件读取、分析Cy3和Cy5两种荧光信号的强度和比值。(2)通过芯片图像分析软件对芯片灰度扫描图进行分析,可以得到芯片上每个基因点的原始信号值,即所有有效重复点的前景信号值-背景信号值的平均信号值(Avg-Signal)、Detection Pval等。根据这些参数值进行后续的数值分析。(3)筛选差异表达基因:本实验选择的归一化方法为:Cubic Spline。mRNA使用了limmol/La进行显著性水平的计算,设置参数P<0.05,fold change≥2[Foldchange=Sig-nal MIA-PaCa2(TIChigh)/Signal BxPc-3(TIClow)]。(4)相同表达趋势的基因具有相似的功能或参与同一生物学过程,因此通过对差异基因按照表达丰度进行趋势聚类,得到差异基因随着动态发展过程中各自的表达趋势,并将显著性的表达趋势作为下一步集中研究的目标。(5)在MSCs分化成NLCs的过程中,取分化变化过程中的显著性趋势当中的差异表达基因,基于Gene Ontology数据库,对趋势中的基因进行GO注释,并采用Fisher精确检验和χ2检验,对每个GO进行显著性分析,计算每个GO的显著性水平(P值)和误判率(FDR),得到显著性表达趋势在分化变化过程中体现的基因功能。取显著性趋势当中的基因,根据共表达基因的表达丰度计算基因间的Pearson相关系数,利用基因间的相互作用关系构建共表达调控网络,对表达调控网络进行网联系分析,计算网络当中每个基因的中间中介性(betweenness centrality)。筛选出网络当中最关键的基因。

2 结果
2.1总RNA提取的质检结果样本合格,提取RNA纯度满意,且无明显降解。
2.2基因表达谱差异基因表达本实验选取大鼠骨髓MSCs经肝细胞提取液诱导HLCs做基因芯片实验,采用Illumina大鼠全基因组基因表达芯片ratref-12,由于每组样本在进行芯片扫描时分别有3个生物学重复,所以在筛选差异基因时,我们选择适合小样本数据的差异筛选方法随机方差模型进行多组间样本差异的筛选,利用随机方差模型计算每个基因的显著性水平(P值)和误判率(FDR),按照P<0.001进行筛选,获得差异表达基因,通过筛选得到在分化过程当中共有4 938个差异表达基因。
2.3差异表达谱的生物信息学分析显著性基因表达趋势及信号通路分析:在MSCs分化成NLCs的过程中,基因的表达随着分化的过程也呈现出相应有规律的表达趋势,取分化变化过程中的差异表达基因,按照基因的表达丰度进行趋势聚类,得到有共同表达趋势的各组基因,然后对每个趋势进行显著性分析,得到显著性的表达趋势。显著性的表达趋势就是由于MSCs分化的过程变化而导致的,趋势中的基因就是与MSCs分化成HLCs变化相关基因。图1就是所有的趋势情况,其中趋势15、14、9、16、11和4就是显著性的趋势(见图2)。
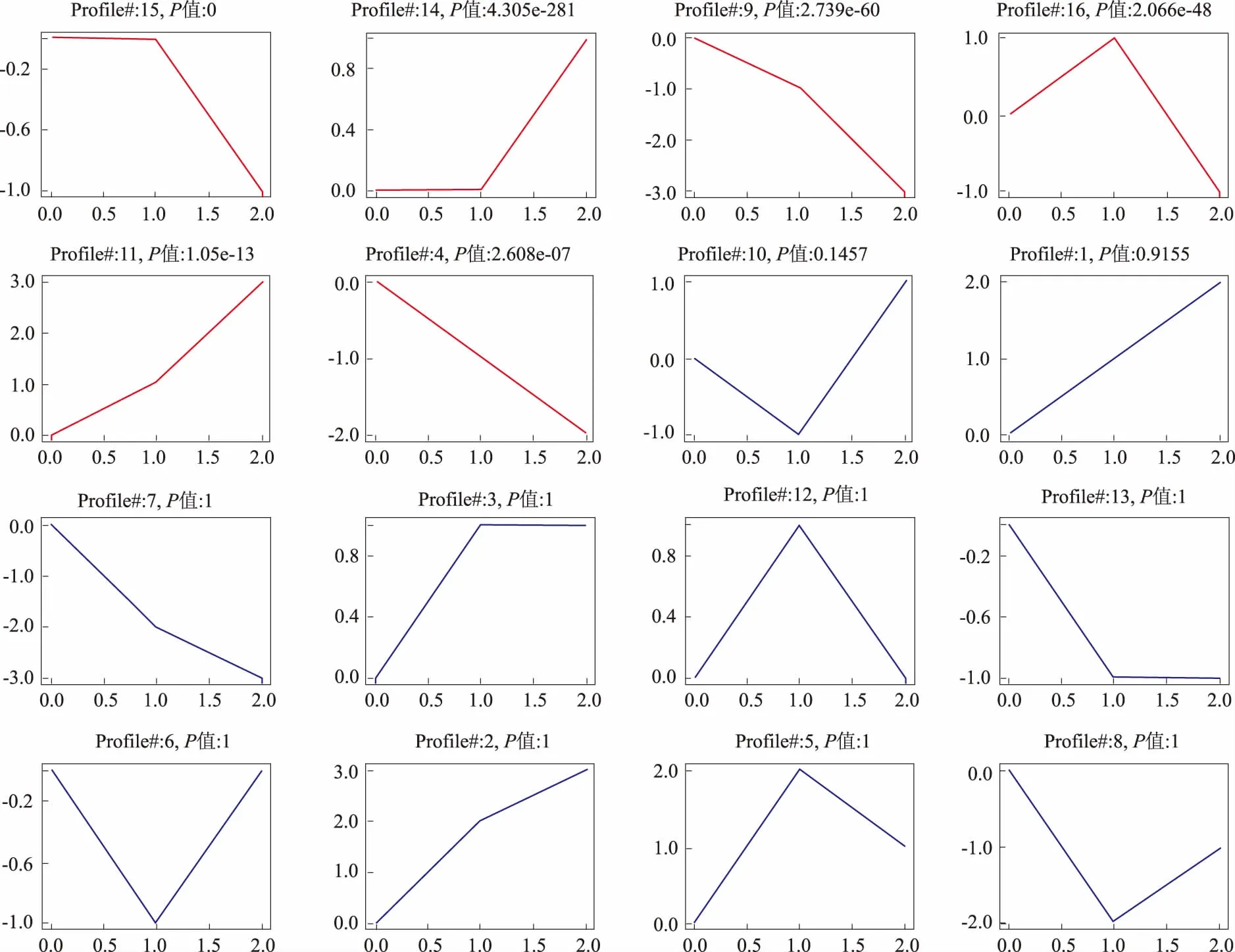
注:图中每个方框代表一个表达趋势,其中框中的线条变化代表一群基因的表达变化趋势。每个方格当中的正上方有趋势的编号和趋势的显著性水平,标红色的趋势是显著性的趋势。
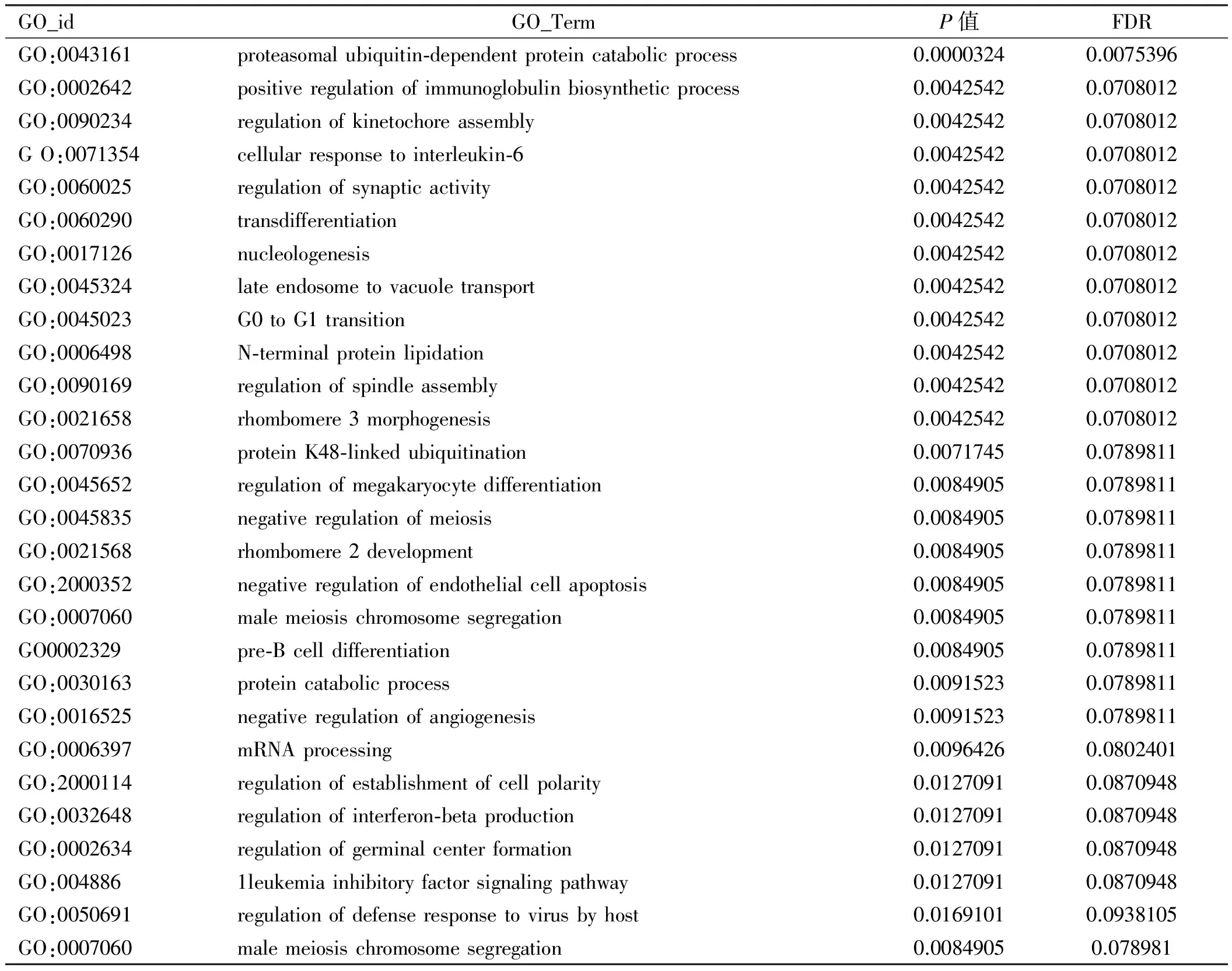
首先以趋势11为例,基于Gene Ontolog数据库,对趋势中的基因进行GO注释,并采用Fisher精确检验和χ2检验,对每个GO进行显著性分析,计算每个GO的显著性水平(P值)和误判率(FDR),得到疾病变化过程中影响的基因所表现的显著性功能(见表1)。
通过对差异基因进行表达趋势的聚类,从中得到了和MSCs诱导分化成HLCs再到成熟的NLCs过程中有关的显著性趋势,取显著性趋势当中的基因,利用基因在每个状态的表达值,计算基因间的共表达相关系数,根据共表达基因间的Pearson相关系数,构建基因间的共表达调控网络,其中点代表基因,线代表基因间的相互作用,实线代表基因间表达呈正相关(激活),虚线代表基因间表达呈负相关(抑制)(见图3)。
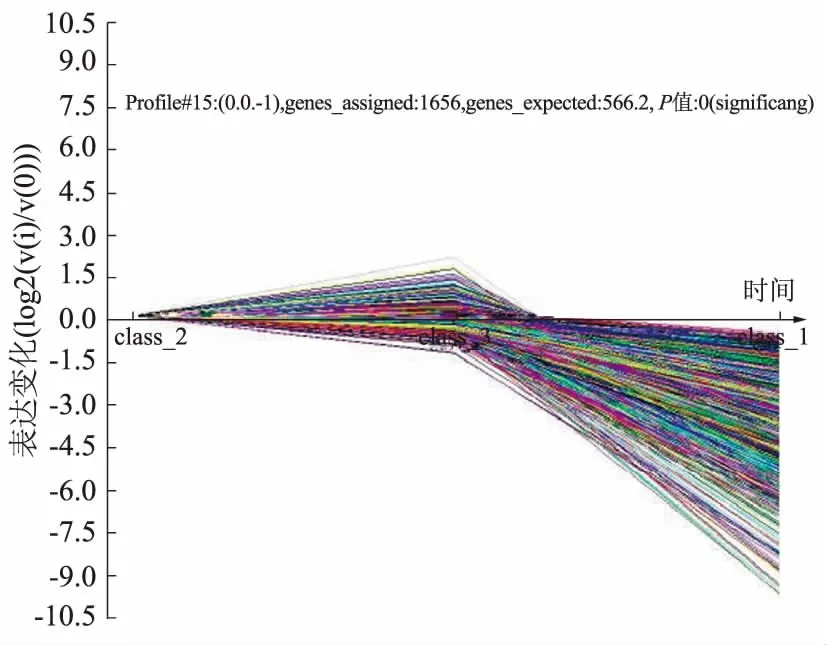
注:横轴代表分化发育时间(也就是诱导分化前后的每个阶段),纵轴代表基因的表达值,其中是以MSCs为初始点,诱导分化的肝细胞和NLCs分别与骨髓MSCs的表达值的比值的对数。一条线代表一个基因。其中class2代表MSCs,class3代表
HLCs,class1代表NLCs。
注:列表当中的GO_id代表GO的ID号,GO_Term表示GO条目,P值代表GO的显著性水平,显著与否的判断标准为P<0.05。FDR表示误判率,FDR越小,就是对每个GO进行显著性判断时错误的概率越小。
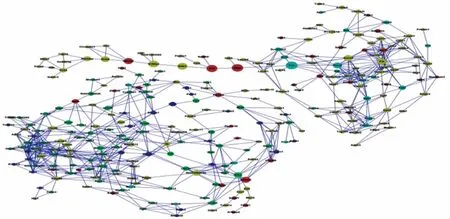
图3 基因调控网络图Fig 3 Gene regulation network diagram
基于差异基因间的相互作用网络,对调控网络进行网络分析,计算网络当中每个基因的中间中介性(betweenness centrality),Betweenness Centrality表示网络中包含节点i的所有最短路的条数占所有最短路条数的百分比。它表示节点i的控制能力,能够描述节点在网络中的重要程度。通过对基因调控网络当中的基因的Betweenness Centrality筛选出网络当中的最关键基因,基因的Betweenness越大,表示基因越处于网络当中的关键位置,对网络的稳定性具有决定性作用。所以可通过网络当中的节点基因的中介性筛选出关键基因(见表2)。
2.4荧光定量PCR实验对结果进行验证分析结合全基因表达的GO分析及Pathwav分析挑选出可信值高的基因做进一步的芯片验证实验,以证实芯片结果的可靠性。挑选基因调控网络当中9个关键基因进行验证。具有代表性的9个差异基因在芯片中的差异倍数和在RT-PCR实验中的差异倍数进行比较,得到RT-PCR结果和基因芯片表达的结果相符(见图4、表3)。
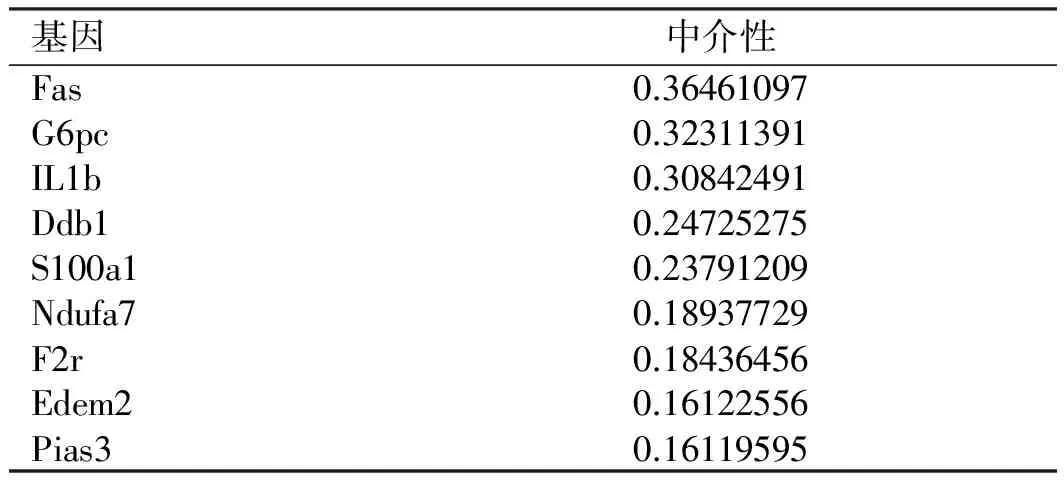
表2 关键基因Tab 2 Key genes
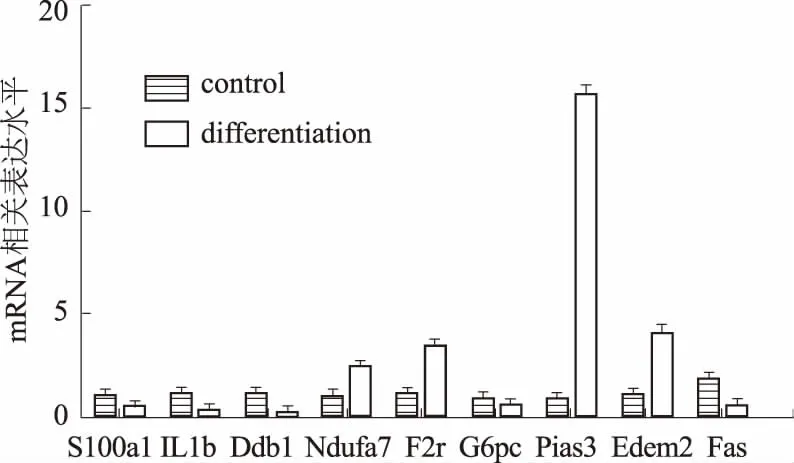
图4 实时定量PCR的结果Fig 4 Results of real-time quantitative PCR
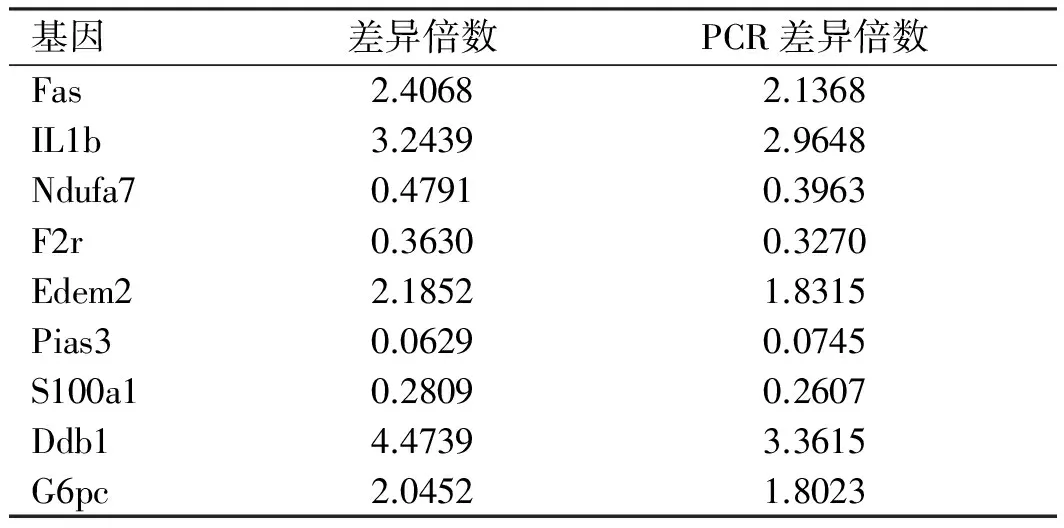
基因差异倍数PCR差异倍数Fas2.40682.1368IL1b3.24392.9648Ndufa70.47910.3963F2r0.36300.3270Edem22.18521.8315Pias30.06290.0745S100a10.28090.2607Ddb14.47393.3615G6pc2.04521.8023
3 讨论
MSCs定向分化为NLCs的动态过程,是一个多基因通路综合变化的复杂网络。本研究归纳出一些重要信号通路上的节点基因在MSCs定向分化过程中出现细胞类群特异性的变化谱式。处于基因网络关键基因与代谢、免疫、增殖、分化等功能密切相关。
脂肪酸合成酶(fatty acid synthase,FAS)是生物体内源性脂肪酸合成过程的关键酶,它通过催化乙酰辅酶A和丙二酰辅酶A而生成长链脂肪酸。抑制脂肪酸合成酶可以减少脂肪酸的合成。亮氨酸可能增加脂肪酸氧化从而减少肥胖,因为亮氨酸减少脂肪酸合酶在组织中的表达[26]。研究[27]发现,菠萝蛋白酶作为在医学上减肥的替代治疗中,发现脂肪酸合酶表达下调。G6pc葡萄糖-6-磷酸酶又称葡萄糖-6-磷酸酶(glucose-6-phosphatase,G-6-pase)是一种水解磷酸化合物的磷酸酶。在肝组织中通过水解葡萄糖-6-磷酸释放葡萄糖入血,因此饥饿时肝糖原既能补充血糖,维持血糖平衡,又可通过其磷酸转移酶活性来合成葡萄糖-6-磷酸,由此可见该酶是糖代谢的关键酶。细胞因子白介素-1,简称白介素-1(IL-1),是一种细胞因子,属于白细胞介素的一种。它是由活化的巨噬细胞所产生,能够刺激参与免疫反应的细胞增殖、分化并提高其功能。S100a1是S100蛋白家族中的一员,参与大量的细胞过程,如细胞周期调控、分化。Ndufa7泛醌氧化还原酶,位于线粒体膜上,它是一个复杂的蛋白质复合体,其功能是从烟酰胺腺嘌呤二核苷酸(NADH)转移电子到呼吸链。参与氧化呼吸链或呼吸代谢,生成ATP。在MSCs分化的肝脏细胞中,通过AMP蛋白激酶调节线粒体呼吸链,如果功能失调、乳酸堆积,引起代谢性疾病[28]。
Fas、Pias3、S100a1为趋势9的基因;G6pc、Edem2为趋势14的基因;Ddb1、F2r为趋势15的基因,而趋势9、14、15均为显著性表达趋势。表明筛选出来的关键基因参与同一个生物学过程,这些基因与MSC发生肝定向分化过程有直接关系,MSCs向HLCs分化提供前提保证。与肝细胞功能有关的基因逐渐表达出来,进一步完善肝细胞的功能。如:肝细胞糖、脂肪酸、胆固醇的代谢及解毒相关基因表达,可能与MSCs发生肝定向分化密切相关。MSCs向肝细胞基因芯片检测出的差异体现在多个方面,反映了MSCs发生肝向分化时复杂广泛的调节模式,通过数据库的生物信息分析,从它们作用的网络关系推导候选的分化关键靶点和通路,以及进一步的全基因组学分析是今后研究的方向。
[1] KIM W R, THERNEAU T M, BENSON J T, et al. Deaths on the liver transplant waiting list: an analysis of competing risks [J]. Hepatology, 2006, 43(2): 345-351. DOI: 10.1002/hep.21025.
[2] LEE S G, MOON D B, HWANG S, et al. Liver transplantation in Korea: past, present, and future [J]. Transplant Proc, 2015, 47(3): 705-708. DOI: 10.1016/j.transproceed.2015.02.015.
[3] KIM E J, KIM N, CHO S G. The potential use of mesenchymal stem cells in hematopoietic stem cell transplantation [J]. Exp Mol Med, 2013, 45: e2. DOI: 10.1038/emm.2013.2.
[4] PETERSEN B E, BOWEN W C, PATRENE K D, et al. Bone marrow as a potential source of hepatic oval cells [J]. Science, 1999, 284(5417): 1168-1170.
[5] SCHWARTZ R E, REYES M, KOODIE L, et al. Multipotent adult progenitor cells from bone marrow differentiate into functional hepatocyte-like cells [J]. J Clin Invest, 2002, 109(10): 1291-1302. DOI: 10.1172/JCI15182.
[6] TROPEL P, PLATET N, PLATEL J C, et al. Functional neuronal differentiation of bone marrow-derived mesenchymal stem cells [J]. Stem Cells, 2006, 24(12): 2868-2876. DOI: 10.1634/stemcells.2005-0636.
[7] COGLE C R, YACHNIS A T, LAYWELL E D, et al. Bone marrow transdifferentiation in brain after transplantation: a retrospective study [J]. Lancet, 2004, 363(9419): 1432-1437. DOI: 10.1016/S0140-6736(04)16102-3.
[8] ROSE R A, KEATING A, BACKX P H. Do mesenchymal stromal cells transdifferentiate into functional cardiomyocytes? [J]. Circ Res, 2008, 103(9): e120. DOI: 10.1161/CIRCRESAHA.108.186908.
[9] PIJNAPPELS D A, SCHALIJ M J, RAMKISOENSING A A, et al. Forced alignment of mesenchymal stem cells undergoing cardiomyogenic differentiation affects functional integration with cardiomyocyte cultures [J]. Circ Res, 2008, 103(2): 167-176. DOI: 10.1161/CIRCRESAHA.108.176131.
[10] GOLZAR F, JAVANMARD S H, BAHRAMBEIGI V, et al. The effect of Kisspeptin-10 on mesenchymal stem cells migration in vitro and in vivo [J]. Adv Biomed Res, 2015, 4: 20. DOI: 10.4103/2277-9175.149851.
[11] LIU W H, SONG F Q, REN L N, et al. The multiple functional roles of mesenchymal stem cells in participating in treating liver diseases [J]. J Cell Mol Med, 2015, 19(3): 511-520. DOI: 10.1111/jcmm.12482.
[12] LIN H, XU R, ZHANG Z, et al. Implications of the immunoregulatory functions of mesenchymal stem cells in the treatment of human liver diseases [J]. Cell Mol Immunol, 2011, 8(1): 19-22. DOI: 10.1038/cmi.2010.57.
[13] ZHANG D, JIANG M, MIAO D. Transplanted human amniotic membrane-derived mesenchymal stem cells ameliorate carbon tetrachloride-induced liver cirrhosis in mouse [J]. PLoS One, 2011, 6(2): e16789. DOI: 10.1371/journal.pone.0016789.
[14] LIN S Z, CHANG Y J, LIU J W, et al. Transplantation of human Wharton’s Jelly-derived stem cells alleviates chemically induced liver fibrosis in rats [J]. Cell Transplant, 2010, 19(11): 1451-1463. DOI: 10.3727/096368910X514198.
[15] IRFAN A, AHMED I. Could stem cell therapy be the cure in liver cirrhosis? [J]. J Clin Exp Hepatol, 2015, 5(2): 142-146. DOI: 10.1016/j.jceh.2014.03.042.
[16] FAUSTO N, CAMPBELL J S, RIEHLE K J. Liver regeneration [J]. J Hepatol, 2012, 57(3): 692-694. DOI: 10.1016/j.jhep.2012.04.016.
[17] MICHALOPOULOS G K, DEFRANCES M C. Liver regeneration [J]. Science, 1997, 276(5309): 60-66.
[18] GRAMIGNOLI R, VOSOUGH M, KANNISTO K, et al. Clinical hepatocyte transplantation: practical limits and possible solutions [J]. Eur Surg Res, 2015, 54(3-4): 162-177. DOI: 10.1159/000369552.
[19] VOSOUGH M, OMIDINIA E, KADIVAR M, et al. Generation of functional hepatocyte-like cells from human pluripotent stem cells in a scalable suspension culture [J]. Stem Cells Dev, 2013, 22(20): 2693-2705. DOI: 10.1089/scd.2013.0088.
[20] CHAMBERLAIN J, YAMAGAMI T, COLLETTI E, et al. Emcient generation of hunmn hepatocytes by the intrahepatic delivery of clonal human mesenchymal stem cells in fetal sheep [J]. Hepatology, 2007, 46(6): 1935-1945. DOI: 10.1002/hep.21899.
[21] GUPTA S, GORLA G R, IRANI A N. Hepatocyte transplantation: emerging insights into mechanisms of liver repopulation and their relevance to potential therapies [J]. J Hepatol, 1999, 30(1): 162-170.
[22] Kimmel A, Oliver B. DNA Microarrays. Academic Press, 2006.
[23] CHEUNG S T, LEUNG K L, IP Y C, et al. Claudin-10 expression level is associated with recurrence of primary hepatocellular carcinoma [J]. Clin Cancer Res, 2005, 11(2 Pt 1): 551-556.
[24] HO J C, IP Y C, CHEUNG S T, et al. Granulin-epithelin precursor as a therapeutic target for hepatocellular carcinoma [J]. Hepatology, 2008, 47(5): 1524-1532. DOI: 10.1002/hep.22191.
[25] LI Y S, HARN H J, HSIEH D K, et al. Cells and materials for liver tissue engineering [J]. Cell Transplant, 2013, 22(4): 685-700. DOI: 10.3727/096368912X655163.
[26] ZEMEL M B, BRUCKBAUER A. Effects of a leucine and pyridoxine-containing nutraceutical on fat oxidation, and oxidative and inflammatory stress in overweight and obese subjects [J]. Nutrients, 2012, 4(6): 529-541. doi:10.3969/j.issn.1006-5709.2018.02.013
[27] DAVE S, KAYR N J, NANDURI R, et al. Inhibition of adipogenesis and induction of apoptosis and lipolysis by stem bromelain in 3T3-L1 adipocytes [J]. PLoS One, 2012, 7(1): e30831. DOI: 10.1371/journal.pone.0030831.
[28] IM I, JANG M J, PARK S J, et al. Mitochondrial respiratory defect causes dysfunctional lactate turnover via AMP-activated protein kinase activation in human-induced pluripotent stem cell-derived hepatocytes [J]. J Biol Chem, 2015, 290(49): 29493-29505. DOI: 10.1074/jbc.M115.670364.

