激酶组学在原核生物中的研究进展
薛 闯, 张 萌, 陈丽杰
(大连理工大学 生命科学与技术学院,辽宁 大连 116023)
激酶组学(kinomics)主要用于研究激酶的丰度、活性、底物特异性、磷酸化状态等特性。蛋白激酶(protein kinase)是指能够使蛋白质发生磷酸化的一类酶,蛋白激酶出现在大多数真核细胞的信号转导过程,同时也可以控制代谢、转录、细胞周期调控、细胞凋亡、运动、分化等其他细胞进程[1]。蛋白质磷酸化是一种常见并且非常重要的蛋白质翻译后修饰(PTM)形式,20世纪50年代首先发现可逆的磷酸化能够调节糖原磷酸化酶的活性,此后对蛋白质磷酸化调控细胞进程的研究逐渐增多。目前对真核生物的激酶组学研究比较透彻,在人类激酶组的鉴定中发现有518个蛋白激酶,通过染色体定位等手段发现有244个激酶与疾病的发生有关[2]。有研究显示,在真核生物中,超过30%的蛋白质在任何时刻都在发生磷酸化进程[3]。其中激酶磷酸化出现最多的有两种形式:一种为丝氨酸/苏氨酸激酶使蛋白质上丝氨酸/苏氨酸残基发生磷酸化,由Hanks等人首先发现,也被称为Hanks家族激酶[4];另一种为酪氨酸激酶使自身酪氨酸残基发生磷酸化,在20世纪80年代早期被发现。在真核生物中丝氨酸磷酸化、苏氨酸磷酸化、酪氨酸磷酸化发生的比例分别约为86.4%、11.8%、1.8%[6]。 最初,人们认为蛋白质磷酸化只在真核生物中出现,原核生物蛋白质磷酸化的研究比真核生物晚。20世纪90年代,科学家在原核生物中发现蛋白激酶也能够起到蛋白质磷酸化的作用。在原核生物中最常见的一类激酶为组氨酸激酶,它出现在双组分信号转导系统(TCS)中,能够使自身的组氨酸残基和其下游响应蛋白的天冬氨酸残基发生磷酸化。而真核生物中经常出现的三类蛋白激酶磷酸化在原核生物的研究中相对较少。
1 丝氨酸/苏氨酸激酶在原核生物中的研究进展
1.1 研究历史
在原核生物中首先发现的被丝氨酸/苏氨酸激酶磷酸化的酶是大肠埃希菌(Escherichiacoli)中的异柠檬酸脱氢酶,它的丝氨酸位点被在真核生物中缺少丝氨酸/苏氨酸激酶同源性的一个双功能激酶/磷酸酶磷酸化。第一个在细菌中被确定与真核生物中丝氨酸/苏氨酸激酶有同源性关系的激酶是黄色粘球菌(Myxococcusxanthus)中的Pkn1激酶[6],编码该激酶的基因pkn1在大肠埃希菌中过表达后会在丝氨酸/苏氨酸位点上发生自磷酸化,而在黄色粘球菌中敲除pkn1基因则会使细胞分化进程提前,说明该激酶在黄色粘球菌的发育中起到重要作用。原核生物基因组测序的完成,基因组序列的获得能够发现更多的丝氨酸/苏氨酸激酶,越来越多激酶的确定为研究丝氨酸/苏氨酸激酶在原核生物的细胞发育和代谢调控等方面提供了支撑[7]。
1.2 丝氨酸/苏氨酸激酶的功能
1.2.1 调控细菌孢子化进程 孢子化是机体响应外界压力或营养源限制启动的进程,孢子化进程受到一系列蛋白质磷酸化事件调控。在枯草芽胞杆菌中,转录因子Spo0A的活化能够启动孢子化进程,它通过组氨酸激酶磷酸化作用使自身发生磷酸化并活化,这类磷酸化易失活且不稳定[8]。最近发现另一类比组氨酸激酶磷酸化更稳定的磷酸化形式:Hanks家族的丝氨酸/苏氨酸激酶使蛋白质上的丝氨酸/苏氨酸残基发生磷酸化,这类磷酸化形式在孢子化进程中扮演重要角色。在枯草芽胞杆菌中,一共有四种丝氨酸/苏氨酸激酶:PrkA、PrkC、PrkD和YabT,除了PrkD外,其他三个激酶均在孢子化的不同阶段发挥重要作用[9],图1为三个激酶在调节孢子化过程中的作用方式。在孢子化起始过程中PrkA和YabT的基因表达量明显增多。PrkA拥有一个催化域,定位在前孢子膜上,在枯草芽胞杆菌中检测到丝氨酸/苏氨酸激酶PrkA能够磷酸化一个60 kDa未知蛋白上的丝氨酸位点,但未检测到自磷酸化现象[10]。YabT包含三个结构域:跨膜区、激酶区和DNA结合区,通过将YabT结合到DNA结合区,能够活化其激酶活性[11]。成熟的孢子从母体释放成为休眠体,休眠体代谢能力明显下调,这受到伸长因子Tu(EF-Tu)磷酸化的调控,而在yabT突变体中不能检测到EF-Tu磷酸化,这也能够证明丝氨酸/苏氨酸激酶YabT调控EF-Tu的磷酸化,进而调控休眠过程。PrkC是一个跨膜蛋白,包括一个内部催化域和外部调控C末端区域。当外界条件适宜生长时,丝氨酸/苏氨酸激酶PrkC通过结合生长细胞释放的胞壁肽进而诱导休眠细胞的再生[12]。因此,枯草芽胞杆菌中的三种丝氨酸/苏氨酸激酶均在孢子化进程中起到重要作用。目前在梭菌中还未见关于丝氨酸/苏氨酸激酶的研究报道。但是梭菌和芽胞杆菌中的很多基因和蛋白有高度同源性,它们在菌株中有着相同或相似的功能。例如,丝氨酸/苏氨酸激酶如PrkA、组氨酸激酶HKs、转录调控因子Spo0A和σ、延伸因子EF-Tu/G和重组酶RecA等在丙丁梭菌和枯草芽胞杆菌中同时存在。并且,已有的研究报道证实两种菌株中的组氨酸激酶HKs、转录调控因子Spo0A和σ在群体效应、磷酸转移和下游蛋白/基因调控均发挥相同或相似的作用[13-14]。因此,丝氨酸/苏氨酸激酶在梭菌中可能发挥着芽胞杆菌中类似的功能。

图1 调节孢子化进程三种丝氨酸/苏氨酸激酶作用方式Fig.1 The regulation of the three serine/threonine kinases in the process of sporulationA:孢子化进程中PrkA和YabT激酶磷酸化作用;B:休眠孢子活化过程,PrkC激酶起到关键作用A:The phosphorylation of PrkA and YabT kinase in the process of sporulation;B:The key role of PrkC kinase in activation of dormancy spores
1.2.2 调控次级代谢进程 丝氨酸/苏氨酸激酶在调控原核细菌次级代谢产物方面起到重要作用。链霉菌为革兰阳性丝状菌,它有复杂的形态学分化并且能合成临床相关抗生素等小分子。放线菌紫素由天蓝色链霉菌在丝氨酸/苏氨酸激酶AfsK的调控下合成[15]。AfsK可以在体外对蛋白AfsR上的丝氨酸/苏氨酸残基进行磷酸化,afsK基因突变的菌株中无法产生放线菌紫素。同时,在afsR下游编码一个与次级代谢相关的蛋白转录子afsS在afsK突变体中的表达也出现了延迟。AfsK磷酸化增强了afsS启动子的启动和AfsR蛋白的ATP水解酶的活性,进而促进afsS的表达,增加放线菌紫素的产量[16]。KbpA蛋白与非磷酸化形式的AfsK激酶相互结合,抑制激酶的自磷酸化,进而促进活化[17]。KbpA编码区在afsK基因上游,afsK的转录能够增加放线菌紫素的产量,进而证明KbpA通过调控AfsK的活性控制其次级代谢的产量,也能够说明激酶在调控次级代谢进程中起到重要作用。
1.2.3 调控细胞分裂和细胞形态 很多实验已经证明丝氨酸/苏氨酸激酶能够调控细胞分裂或细胞形态。细菌的操纵子通常包含功能性相关基因,在细胞分裂中细胞形态和基因的顺序建立了良好的关系。例如,在分枝杆菌(M.tuberculosis)中,丝氨酸/苏氨酸激酶PknA和PknB的操纵子中包含编码青霉素结合蛋白A(PBPA)和RodA的基因, 因此这两个激酶可控制细胞形态。pknA和pknB基因均在细胞生长指数期表达,它们过表达后会使细胞变长[18]。PknA能够使一个真核细胞同源物、分裂隔的主要组成元件的微管蛋白FtsZ上的苏氨酸残基发生磷酸化,PknB在大肠埃希菌中使PBPA的第362位和437位苏氨酸发生磷酸化。因此,丝氨酸/苏氨酸激酶在调控细胞分裂和细胞形态中起到重要作用。
2 酪氨酸激酶在原核生物中研究进展
2.1 研究历史
在发现原核生物酪氨酸激酶前,细菌中的酪氨酸磷酸化已经在大肠埃希菌中被成功发现[19],随后在其他细菌中也发现了酪氨酸磷酸化,但无法确定这些酪氨酸磷酸化是通过核苷酸化产生还是蛋白质的磷酸化作用产生。后来的研究发现通过单克隆抗体等实验技术在青枯假单胞菌(Pseudomonassolanacearum)中能够检测到酪氨酸磷酸化[20]。第一个确定的酪氨酸激酶是在约氏不动杆菌(Acinetobacterjohnsonii)中发现,该蛋白激酶Ptk位于细胞内膜,长为82 kDa,它能够使该蛋白上多个酪氨酸位点发生自磷酸化[21]。Ptk蛋白包含两段沃克A和B序列,这两段序列在真核生物的酪氨酸激酶中常见,在原核生物中蛋白质的ATP结合也起到重要作用,通过研究发现使酪氨酸发生自磷酸化的蛋白激酶一般都具有沃克A和B序列,基于酪氨酸激酶在细菌中的特性,也被称为BY(bacterial tyrosine)激酶。
2.2 细菌酪氨酸激酶结构和催化位点
细菌中的酪氨酸激酶包含一个跨膜区和细胞内催化域,这两个区域通过一个多肽或者通过它们相互作用形成的螺旋连接[22]。革兰阳性菌中的放线菌和厚壁菌门的酪氨酸激酶膜外部环与其他细菌相比序列具有高度的相似性,但是与革兰阴性菌的变形菌门的酪氨酸激酶外部胞质环相比有一些不同。此外,在放线菌和厚壁菌门的酪氨酸激酶外部环更短一些[23]。
细菌中酪氨酸激酶的催化活性位点为沃克A(GXXGXGK[T/S])和沃克B(hhhhD)序列,有的蛋白也有沃克A′序列。沃克A′序列通常不会在核苷酸结合的蛋白中出现,但会出现在某些激酶磷酸化小分子底物中,如莽草酸或葡萄糖酸,这些小分子底物拥有一段退化的沃克B序列[24]。所有的酪氨酸激酶都能够在C末端的位点上检测到酪氨酸的自磷酸化,C末端的区域也为酪氨酸的聚类区域,该区域的七个酪氨酸都有可能被磷酸化。
2.3 细菌酪氨酸激酶功能
细菌酪氨酸激酶具有细胞代谢和基因水平上的调控功能。最初的研究发现BY激酶可能与细胞外多糖的合成有关,BY激酶通常编码控制多糖聚合物合成的操纵子的基因[25],在大肠埃希菌中鉴定到的BY激酶Etk,是荚膜多糖操纵子的组成部分之一[26]。其他蛋白也可以被BY激酶磷酸化,其中包括热休克σ因子RpoH和RpoE[27],这两个σ因子在大肠埃希菌中控制两个热休克调控子的表达。通过突变rpoH基因使其不能发生磷酸化,进而得到一个对温度敏感的表型;类似地,通过磷酸化RpoE的抑制子RseA,也能够改变它对RpoE结合的亲和力。总之,这些研究显示BY激酶能够调节热休克蛋白的响应。
3 组氨酸激酶在原核生物中研究进展
3.1 双组分信号转导系统
组氨酸激酶主要存在于双组分信号转导系统(TCS)中,包括组氨酸激酶(Histidine kinase, HK)和调控响应蛋白(Response regulator, RR)[28]。双组分信号转导系统主要存在于细菌中,在部分真核生物中也被发现[29]。过去30年的研究中发现,TCS不仅仅作为简单的刺激-响应调控体系,很多TCS都包括多个元件,内在或交叉联系很多其他的调控循环。当检测到外界环境变化(例如:盐、温度、营养物等),表观组氨酸激酶的结构域将会发生构象变化,触发ATP介导一个保守的组氨酸残基通过与ATP结合域直接相互作用的磷酸化,接下来磷酸基团会转移到响应蛋白的天冬氨酸上,进一步调控下游基因的表达[30]。大多数双组分系统有一个模块化的架构组织,使它们能够执行复杂和灵活的监管策略。双组分信号转导系统对于调控细菌重要的生存过程,例如代谢、群体效应、孢子化等起到重要作用[31]。双组分信号转导系统与许多细菌的致病性密切相关,使双组分转导系统蛋白成为治疗靶点研究,例如TCS转导被应用在肺炎、中毒性休克综合征、肺结核以及其他急性和慢性感染疾病的研究中[32]。此外,这些修饰在多元化外界环境情况下信号系统会允许这些机体做出快速的改变。相反,哺乳动物细胞体系依赖于跨膜酪氨酸激酶作为外界刺激的回应,在这些细胞中对组氨酸激酶磷酸化的研究非常罕见[29]。
3.2 组氨酸激酶的功能
3.2.1 调控孢子化进程和溶剂产量 在丙丁梭菌(Clostridiumacetobutylicum)中发现组氨酸激酶能够调控孢子化进程和溶剂产量。并且,由组氨酸激酶参与的细胞发育和代谢调控还可能与群体效应有关,与高密度细胞培养条件下的信号分子感应和转导有关[33-34]。生物信息分析中发现在丙酮丁醇梭菌中有五种组氨酸激酶CA_C0323、CA_C0903、CA_C2730、CA_C0437、CA_C3319。Steiner和同事发现了基于上述五种组氨酸激酶的Spo0A磷酸化途径[35]。第一条途径仅依赖于组氨酸激酶CA_C0323,另一条途径依赖于组氨酸激酶CA_C0903和CA_C3319,单独突变编码五种组氨酸激酶的基因发现孢子化频率较亲本菌株减少95%~99%,说明这些基因在孢子化中起到重要作用。此外,双突变cac0323/0903和cac0323/3319基因导致细胞完全失去孢子化能力,cac0903/3319基因双突变与基因单突变相比,只有部分孢子化形成的减少。体外也能够检测到激酶CA_C0323、CA_C0903、CA_C3319自磷酸化,Spo0A的磷酸基团在体外也可以发生转移,激酶CA_C0903、CA_C3319都能够成功自磷酸化并转移Spo0A的磷酸基团,激酶CA_C0323并未检测到自磷酸化的能力。Xu等[36]通过基因敲除技术敲除编码组氨酸激酶的基因cac3319,将敲除菌和野生型分别进行批次发酵培养,敲除菌的丁醇产量达到18.2 g/L,与野生型相比丁醇产量提高了44.4%,实验证明组氨酸激酶能够调节细菌的产溶剂能力。研究表明,梭菌菌株的环境胁迫耐受能力是由多基因调控的复杂性状,组氨酸激酶参与的细胞调控还有助于提高梭菌的环境胁迫耐受能力[37-38]。组氨酸激酶在梭菌细胞中对碳源代谢和胁迫耐性调节存在差异性,组氨酸激酶CA_C3319对目标产物丁醇的合成有正调控作用,而组氨酸激酶CA_C0437对丁醇合成起负调控作用。如果这些组氨酸激酶调控与在线发酵分离耦合解毒策略结合,将有利于提高梭菌或其它微生物生产代谢产物的效率[39-40]。
3.2.2 调节细菌趋化性 细菌趋化性(chemotaxis)是指有运动能力的细菌对有利环境的趋附和对有害环境的避离运动,而二元信号系统则是细菌实现这种功能的主要途径。趋化途径最初在大肠埃希菌和鼠伤寒沙门氏菌中被发现[41]。如图2所示,当甲基化转移酶CheR和甲基酯酶CheB发生甲基化或去甲基化时,细菌能感知到外界环境的信号。当接收到外界信号后,信息将被传递到趋化性组氨酸激酶CheA,通过其保守区域上一个位点的自磷酸化,将磷酸基团传递至下游响应蛋白CheY和CheB的天冬氨酸上。被磷酸化的CheY结合到鞭毛马达上后,鞭毛的旋转运动能够发生改变,进而完成趋化过程。
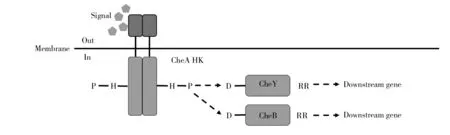
图2 趋化性组氨酸激酶磷酸转移过程Fig.2 The phosphate transfer process of chemotaxis histidine kinaseCheA为趋化性组氨酸激酶,CheY和CheB为响应蛋白CheA is the chemotactic histidine kinase,CheY and CheB are the response proteins
4 激酶磷酸化的研究方法
前对激酶磷酸化的主要研究方法为富集磷酸化肽段,有很多方法能够选择性分离磷酸化肽段。包括:①使用丝氨酸/苏氨酸特异性抗体进行免疫共沉淀富集磷酸化肽段,但该方法具有受抗体特异性限制和价格昂贵的缺点;②通过化学限制迈克尔加成和使用标签来代替磷酸肽上的磷酸基团,但该方法需要较大的工作量,而且还具有由多步反应造成的样品损失和不可避免的副反应增加样品的复杂性等缺点;③与吸附剂发生相互作用,例如金属氧化物TiO2、ZrO2、Al2O3,固定化金属离子Fe3+、Ga3+、Ni2+、Zr4+,亲和吸附IMAC和强离子交换色谱吸附[42]。在以上这些方法中,固定金属离子亲和色谱法(IMAC)是吸附磷酸化肽段的最好方法。质谱分析方法,特别是结合低能量碰撞诱导解离串联质谱已经成为分析共价蛋白修饰的表征功能的工具,用于蛋白质磷酸化的分析和定量[43]。利用质谱分析的方法,磷酸化蛋白将被酶解为肽段,相关富集技术可以将磷酸化肽段选择性分离出来,以避免非磷酸化肽段干扰分析。在质谱分析中,这些磷酸化肽段的序列和磷酸化位点会被鉴定出来。利用Maxcount和Mascot软件,通过搜索对应蛋白组的数据库,可以得到样品中蛋白质组和磷酸化肽段的信息[44]。利用质谱能够对丝氨酸/苏氨酸/酪氨酸激酶的磷酸化肽段进行成功检测,平均每个细胞能够检测到100个磷酸化蛋白[6]。但组氨酸激酶的磷酸化不易检测,因为组氨酸激酶催化的磷酸基团与氨基酸的结合键为P-N键,它在酸性条件下非常不稳定而且丰度很低,在酸性条件下进行质谱分析会丢失组氨酸磷酸化肽段。有研究显示,在49 ℃ 1 mol/L HCl存在的条件下,1-磷酸化组氨酸的半衰期为18 s,3-磷酸化组氨酸半衰期为25 s[45]。这也就意味着很多丝氨酸/苏氨酸磷酸化检测的技术不能应用到组氨酸磷酸化的研究上。因此,激酶的磷酸化研究需要借助质谱技术的发展不断完善和进步。
5 总结与展望
在原核生物中,激酶类型主要以参与双组分信号系统的组氨酸激酶和丝氨酸/苏氨酸激酶为主,它们在细胞发育和代谢调控等方面起重要作用。目前关于原核生物尤其是工业微生物的代谢调控多局限于对代谢网络基因的研究,而对由激酶主导的蛋白翻译后修饰研究很少。由于这些激酶在细胞发育和代谢调控中起重要的作用,它们不仅可以作为基因工程菌株改造的新靶点,还是合成生物学中不可忽视的基因模块。虽然质谱技术和组学分析技术在不断发展,对丝氨酸/苏氨酸激酶的检测已形成较为成熟的技术,目前对组氨酸激酶的检测仍存在很大难题,需要更有效的检测技术。由于激酶在原核生物中的重要作用,相信越来越多的激酶在原核生物中被发现,它们的功能和调控机制将随着激酶磷酸化检测技术的革新不断被深入挖掘和阐明。
[1] Pereira SFF, Goss L, Dworkin J. Eukaryote-Like Serine/Threonine Kinases and Phosphatases in Bacteria[J]. Microbiology and Molecular Biology Reviews, 2011, 75(1): 192-212.
[2] Manning G, Whyte DB, Martinez R, et al. The protein kinase complement of the human genome[J]. Science, 2002,298(5600):1912-1934.
[3] Cohen P. The regulation of protein function by multisite phosphorylation-a 25 year update[J]. Trends in Biochemical Sciences, 2000,25(12):596-601.
[4] Hanks SK, Quinn AM, Hunter T. The Protein-Kinase Family-Conserved Features and Deduced Phylogeny of the Catalytic Domains[J]. Science, 1988,241(4861):42-52.
[5] Olsen JV, Blagoev B, Gnad F, et al. Global,invivo, and site-specific phosphorylation dynamics in signaling networks[J]. Cell, 2006,127(3):635-648.
[6] Cousin C, Derouiche A, Shi L, et al. Protein-serine/threonine/tyrosine kinases in bacterial signaling and regulation [J]. FEMS Microbiology Letters, 2013, 246:11-19.
[7] Kennelly PJ. Protein kinases and protein phosphatases in prokaryotes: a genomic perspective[J]. FEMS Microbiology Letters, 2002,206(1):1-8.
[8] Kornev AP, Taylor SS. Defining the conserved internal architecture of a protein kinase[J]. Biochimica et Biophysica Acta-Proteins and Proteomic, 2010,1804:440-444.
[9] Pompeo F, Foulquier E, Galinier A. Impact of Serine/Threonine Protein Kinases on the Regulation of Sporulation inBacillussubtilis[J]. Frontiers in Microbiology, 2016,7:e1000566.
[10] Fischer C, Geourjon C, Bourson C, et al. Cloning and characterization of theBacillussubtilisprkA gene encoding a novel serine protein kinase[J]. Gene, 1996,168(1):55-60.
[11] Bidnenko V, Shi L, Kobir A, et al.Bacillussubtilisserine/threonine protein kinase YabT is involved in spore development via phosphorylation of a bacterial recombinase[J]. Molecular Microbiology, 2013,88(5):921-935.
[12] Shah IM, Laaberki MH, Popham DL, et al. A Eukaryotic-like Ser/Thr Kinase Signals Bacteria to Exit Dormancy in Response to Peptidoglycan Fragments[J]. Cell, 2008,135(3):486-496.
[13] AI-Hinai MA, Jones SW, Papoutsakis ET. The Clostridium sporulation programs: diversity and preservation of endospore differentiation[J]. Microbiology and Molecular Biology Reviews, 2015, 79:19-37.
[14] Xue C, Zhao JB, Chen LJ, et al. Recent advances and state-of-the-art strategies in strain and process engineering for biobutanol production byClostridiumacetobutylicum[J]. Biotechnology Advances, 2017, 35:310-322.
[15] Matsumoto A, Hong SK, Ishizuka H, et al. Phosphorylation of the Afsr protein involved in secondary metabolism in Streptomyces species by a eukaryotic-type protein kinase[J]. Gene, 1994,146(1):47-56.
[16] Lee PC, Umeyama T, Horinouchi S.afsS is a target ofAfsR, a transcriptional factor with ATPase activity that globally controls secondary metabolism inStreptomycescoelicolorA3(2)[J]. Molecular Microbiology, 2002,43(6):1413-1430.
[17] Umeyama T, Horinouchi S. Autophosphorylation of a bacterial serine/threonine kinase, AfsK, is inhibited by KbpA, an AfsK-binding protein[J]. Journal of Bacteriology, 2001,183(19):5506-5512.
[18] Kang CM, Abbott DW, Park ST, et al. The Mycobacterium tuberculosis serine/threonine kinases PknA and PknB: substrate identification and regulation of cell shape[J]. Gene and Development, 2005,19(14):1692-1704.
[19] Doublet P, Vincent C, Grangeasse C, et al. On the binding of ATP to the autophosphorylating protein, Ptk, of the bacteriumAcinetobacterjohnsonii[J]. FEBS Letters, 1999,445(1):137-143.
[20] Atkinson M, Allen C, Sequeira L. Tyrosine phosphorylation of a membrane-protein fromPseudomonas-Solanacearum[J]. Journal of Bacteriology, 1992,174(13):4356-4360.
[21] Grangeasse C, Cozzone AJ, Deutscher J,et al. Tyrosine phosphorylation: an emerging regulatory device of bacterial physiology[J]. Trends in Biochemical Sciences, 2007,32(2):86-94.
[22] Soulat D, Jault JM, Duclos B, et al.Staphylococcusaureusoperates protein-tyrosine phosphorylation through a specific mechanism[J]. Journal of Biological Chemistry, 2006,281(20):14048-14056.
[23] Kato T, Shirakawa T, Takegawa K, et al. Functional analysis of conserved motifs in a bacterial tyrosine kinase, BtkB, fromMyxococcusxanthus[J]. Journal of Biochemistry, 2015,158(5):385-392.
[24] Grangeasse C, Terreux R, Nessler S. Bacterial tyrosine-kinases: Structure-function analysis and therapeutic potential [J]. Biochimica et Biophysica Acta-Proteins and Proteomic, 2010, 1804:628-634.
[25] Whitfield C. Biosynthesis and assembly of capsular polysaccharides inEscherichiacoli[J]. Annual Review of Biochemistry, 2006,75:39-68.
[26] Peleg A, Shifrin Y, Ilan O, et al. Identification of anEscherichiacolioperon required for formation of the O-antigen capsule[J]. Journal of Bacteriology, 2005,187(15):5259-5266.
[27] Klein G, Dartigalongue C, Raina S. Phosphorylation-mediated regulation of heat shock response inEscherichiacoli[J]. Molecular Microbiology, 2003,48(1):269-285.
[28] Wilke KE, Francis S, Carlson EE. Activity-based probe for histidine kinase signaling[J]. Journal of the American Chemical Society, 2012,134(22):9150-9153.
[29] Stock AM, Robinson VL, Goudreau PN. Two-component signal transduction[J]. Annual Review of Biochemistry, 2000,69:183-215.
[30] Cain JA, Nestor S, Cordwell SJ. Beyond gene expression: The impact of protein post-translational modil cations in bacteria[J]. Journal of Proteomics, 2014, (97):265-286.
[31] Gooderham WJ, Hancock REW. Regulation of virulence and antibiotic resistance by two-component regulatory systems inPseudomonasaeruginosa[J]. FEMS Microbiology Reviews, 2009,33(2):279-294.
[32] Gotoh Y, Eguchi Y, Watanabe T, et al. Two-component signal transduction as potential drug targets in pathogenic bacteria[J]. Current Opinion in Microbiology, 2010,13(2):232-239.
[33] Steiner E, Scott J,Minton NP, et al. An agr quorum sensing system that regulates granulose formation and sporulation inClostridiumacetobutylicum[J]. Applied and Environmental Microbiology, 2012, 78:1113-1122.
[34] Xue C, Zhao JB, Lu CC, High-titer n-butanol production byClostridiumacetobutylicumJB200 in fed-batch fermentation with intermittent gas stripping[J]. Biotechnology and Bioengineering, 2012, 109(11):2746-2756.
[35] Steiner E, Dago AE, Young DI, et al, et al. Multiple orphan histidine kinases interact directly with Spo0A to control the initiation of endospore formation inClostridiumacetobutylicum[J]. Molecular Microbiology, 2011,80(3):641-654.
[36] Xu MM, Zhao JB, Yu L, et al. EngineeringClostridiumacetobutylicumwith a histidine kinase knockout for enhanced n-butanol tolerance and production[J]. Applied Microbiology and Biotechnology, 2015,99(2):1011-1022.
[37] Xue C, Zhao JB, Chen LJ, et al. Recent advances and state-of-the-art strategies in strain and process engineering for biobutanol production byClostridiumacetobutylicum[J]. Biotechnology Advance, 2017, 35:310-322.
[38] Xue C, Zhao XQ, Liu CG, et al. Prospective and development of butanol as an advanced biofuel[J]. Biotechnology Advances, 2013, 31:1575-1584.
[39] Xue C, Zhao JB, Chen LJ, et al. Integrated butanol recovery for an advanced biofuel: current state and prospects[J]. Applied Microbiology and Biotechnology, 2014, 98:3463-3474.
[40] Xue C, Liu FF, Xu MM, et al. A novel in situ gas stripping-pervaporation process integrated with acetone-butanol-ethanol fermentation for hyper n-butanol production[J]. Biotechnology and Bioengineering, 2016, 113(1): 120-129.
[41] Borland S, Prigent-Combaret C, Wisniewski-Dye F. Bacterial hybrid histidine kinases in plant-bacteria interactions[J]. Microbiology, 2016,162(10):1715-1734.
[42] Zhou H, Ye M, Dong J, et al. Specific phosphopeptide enrichment with immobilized titanium ion affinity chromatography adsorbent for phosphoproteome analysis[J]. Journal of Proteome Research, 2008,7(9):3957-3967.
[43] Aebersold R, Mann M. Mass-spectrometric exploration of proteome structure and function[J]. Nature, 2016,537(7620):347-355.
[44] Oslund RC, Kee JM, Couvillon AD, et al. A phosphohistidine proteomics strategy based on elucidation of a unique gas-phase phosphopeptide fragmentation mechanism[J]. Journal of the American Chemical Society, 2014,136(37):12899-12911.
[45] Gonzalez-Sanchez MB, Lanucara F, Helm M, et al. Attempting to rewrite History: challenges with the analysis of histidine-phosphorylated peptides[J]. Biochemical Society, 2013,41:1089-1095.