U87细胞分泌外囊泡中HOTAIR刺激胶质瘤血管新生的机制研究
马鑫 濮剑辰 李婷 许颖 范春雷 田男
浙江中医药大学生命科学学院 杭州 310053
人脑胶质瘤(glioblastoma)是中枢神经系统最常见的原发性恶性肿瘤。因其与正常脑组织没有明确界限,使用传统手术方法难以达到根除目的,同时由于血脑屏障的存在,化疗等治疗手段难以奏效,患者5年生存率不足5%[1-4]。目前脑胶质瘤的发病机制尚不清楚,但越来越多的研究表明,血管化是实体肿瘤发展进程中的重要组成部分,肿瘤新生血管为实体肿瘤提供生长所需的营养和氧气,同时清除肿瘤细胞的代谢废物,促进肿瘤细胞远端转移[5-9]。因此探索肿瘤血管生成的调控分子可为胶质瘤治疗提供新方向。
近年来,关于细胞外囊泡(extracellular vesicles,EVs)与肿瘤关系的研究已成为当前研究的一大热门。肿瘤细胞分泌的EVs能够传递信息到其它肿瘤细胞以及正常基质细胞,营造适合肿瘤生长的微环境,促进血管生成、肿瘤生长发展以及远端转移[10-12]。Giusti等[13]发现人脑胶质瘤细胞分泌的EVs在血管内皮细胞增殖、迁移以及成管的过程中有重要作用。研究还发现长链非编码RNAs(long non-coding RNAs,lncRNAs)在胶质瘤血管新生中扮演重要角色[14-15]。Rinn课题组发现了 HOX转录反义 RNA(HOX transcript antisense intergenic RNA,HOTAIR),其表达与胶质瘤的级别和预后生存均密切相关,是独立于病理诊断指标之外的胶质瘤患者预后指标[16-17]。众多研究证实,HOTAIR在胶质瘤细胞中的高表达,与胶质瘤细胞增殖、细胞周期、细胞凋亡、糖酵解和线粒体功能密切相关[16,18-20],但HOTAIR在胶质瘤血管新生中的作用,以及HOTAIR与EVs的关系至今不明。因此,本研究通过沉默胶质瘤细胞中的HOTAIR基因,观察其对人脑微血管内皮细胞(human brain microvascular endothelial cells,HBMVEC)增殖、迁移和血管形成能力的影响,同时进一步研究胶质瘤细胞中HOTAIR与EVs的关系,探讨HOTAIR参与胶质瘤血管新生的可能机制。
1 材料和方法
1.1 实验材料
1.1.1 细胞与主要试剂 人脑胶质瘤细胞U87和HBMVEC均购自中科院上海细胞所。DMEM高糖培养液、无支原体胎牛血清和含0.25%EDTA胰酶均购于杭州昊天生物技术有限公司(批号:2016122902、20160712、20161019);噻唑蓝(methylthiazolyldiphenyltetrazolium bromide,MTT)购于杭州昊天生物技术有限公司(批号:3068B512);二甲基亚砜(dimethyl sulfoxide,DMSO)购于美国 Sigma公司(批号:67-68-5);姬姆萨染液(Giemsa stain)购自上海源叶生物科技有限公司(批号:L16F8G29355)。Trizol购于美国Invitrogen公司(批号:127706);游离RNA提取试剂盒RNApure Circulating Reagent Kit购于北京康为世纪生物科技有限公司(批号:01731/60138);HiFiScript cDNA第一链合成试剂盒购于北京康为世纪生物科技有限公司(批号:40129);Ultra SYBR荧光定量试剂盒购于北京康为世纪生物科技有限公司(批号:40140)。Lipofectamine2000 Reagent购于美国 Invitrogen公司(批号:1704478),Triton X-100购于美国Sigma公司(批号:92046-34-9),引物由上海生工生物工程有限公司设计合成,过表达质粒LZRSHOTAIR购自美国addgene公司。
1.1.2 仪器 恒温恒湿培养箱为美国Thermo公司产品;荧光定量PCR仪为美国Applied Biosystems公司产品;多功能酶标仪购于美国Molecular Devices公司;倒置光学显微镜及成像系统为日本Nikon公司产品。
1.2 方法
1.2.1 细胞培养 U87细胞和HBMVEC均采用含10%胎牛血清的DMEM高糖培养液,置于37°C、5%细胞CO2饱和湿度培养箱内培养,取对数生长期细胞进行实验。
1.2.2 U87细胞培养上清液的收集 将U87细胞传代后接种于6孔板,培养于37°C、5%CO2饱和湿度培养箱内,24h后观察细胞生长状态,将原细胞培养液吸出至离心管中,4°C、5 000r/min离心20min,离心后取上清液,分装后-80°C保存。
1.2.3 脂质体法转染U87细胞 U87细胞传代培养至生长密度为30%~50%时用于转染。将细胞分为4组:无意义RNA对照组(siGFP组)、siRNA阴性对照组(si-NC组)、siHOTAIR组、回复组(Rescue组)。转染按照说明书方法进行,回复组采用HOTAIR siRNA与过表达质粒LZRS-HOTAIR共转染(按3:5比例混合)。U87细胞转染后,继续置于 37°C、5%CO2饱和湿度培养箱内培养24h。转染24h后收集细胞,于4℃、5 000r/min离心20min,离心后取上清液,分装后-80℃保存。
1.2.4 U87细胞培养上清液处理和RNA提取 取转染前U87细胞培养上清液4mL,分为空白组、RNase组、Triton X-100组、RNase和Triton X-100联合组。空白组不作处理,RNase组加入2mg·mL-1RNase 1mL,Triton X-100组加入 0.1%Triton X-100 1mL,RNase和Triton X-100联合组则分别加入2mg·mL-1RNase和0.1%Triton X-100各1mL,孵育20min。根据试剂盒说明书提取上清液中的RNA,5 000r/min离心10min,除去死细胞,将上清液转移至新的离心管中,加入3倍体积的游离RNA提取试剂。震荡30s使之充分混匀。将处理后的样品在室温放置5min,使蛋白核酸复合物完全分离。加入800μL氯仿,盖好管盖,剧烈震荡 15s,室温放置 2~3min。4°C、12 000r/min离心20min,此时样品分成红色有机相、中间层和上层无色水相3层。因RNA主要在水相中,故把水相转移到新的无RNase的离心管中,在得到的水相溶液中加入等体积异丙醇,颠倒混匀,室温放置30min。4°C、12 000r/min离心20min,弃去上清液。加入75%乙醇(用无RNase的水配制)洗涤沉淀,1mL游离RNA提取试剂加入1mL 75%乙醇。4°C、12 000r/min离心3min,小心弃去上清液,注意不要弃去RNA沉淀。室温放置 2~3min,晾干。加入 30~100μL 无 RNase水充分溶解RNA,-80°C保存,防止降解。
1.2.5 荧光定量PCR检测各组U87细胞培养上清液中HOTAIR基因含量 取转染24h后的U87细胞,用Trizol试剂提取总RNA后,根据HiFiScript cDNA第一链合成试剂盒说明书,逆转录合成cDNA。荧光定量PCR扩增,内参基因及目的基因引物序列见表1。扩增条件:95°C 预变性 10min,95°C 变性 15s、60°C退火1min,72°C延伸30s,扩增40个循环。采用比较CT值的方法计算样本中mRNA的表达水平。Ratio=2-△△Ct,△Ct=目的基因 CT 值-内参基因 CT 值,△△Ct=△Ct实验组-△Ct对照组。

表1 引物序列Tab.1 Primer sequence
1.2.6 MTT法和克隆形成实验检测细胞HBMVEC增殖情况
1.2.6.1 MTT法检测转染后U87细胞上清液对HBMVEC增殖的影响 用含10%胎牛血清的DMEM高糖培养液悬浮HBMVEC,然后将HBMVEC以3×103个/孔的密度接种于96孔板,每孔总体积为100μL。设置siGFP组、si-NC组、siHOTAIR组和回复组,每组6个复孔。培养4~6h后将原培养液分别替换为siGFP组、si-NC组、siHOTAIR组和回复组转染后的U87细胞上清液,再次孵育。孵育6h、12h和24h后,每孔加入20μL浓度为5mg·mL-1的MTT,避光孵育4 h后弃培养液,每孔加入150μL DMSO,置于水平摇床震荡混匀15min。将酶标仪的吸收波长设定为490 nm,测定每孔的OD值。细胞增殖率=实验组OD值/对照组OD值×100%。
1.2.6.2 平板克隆形成实验 取对数生长期的HBMVEC制成单细胞悬液,以5×102个/孔的密度接种于6孔板,设置si-NC组和siHOTAIR组,置于37°C、5%CO2饱和湿度培养箱内培养,12h后将培养液分别替换为转染后si-NC组和siHOTAIR组的U87细胞上清液,2~3d换液1次。14d后弃去培养液,PBS冲洗3次,4%多聚甲醛固定30min,再以Giemsa染液染色15min。缓慢冲洗掉染液后镜下观察拍照并计数细胞克隆数。
1.2.7 Transwell细胞迁移实验 将转染24h后的U87细胞接种于含600μL无血清培养液的Transwell小室的下层,含100μL无血清培养液的HBMVEC悬液置于小室的上层,于37°C、5%CO2饱和湿度培养箱内孵育12h。取出小室,用4%多聚甲醛室温固定小室底部聚碳酸酯微孔膜上的细胞30min,Giemsa染液染色15min。缓慢冲洗掉染液,弃掉上室未固定的细胞。光学显微镜下观察、拍照,计数每个小室特定视野下穿过聚碳酸酯微孔膜底部的细胞数,进行组间比较。
1.2.8 HBMVEC体外成管实验 将基质胶置于4℃条件下冻融过夜。设置阴性对照组和实验组,在预冷的 96孔板中每孔加入 75μL基质胶,37°C放置60min。将HBMVEC悬液以2.5×104个/孔的密度加入96孔板对应孔中,待细胞贴壁后,将培养液替换为转染后的U87细胞上清液继续孵育。4~6h后,镜下选取合适视野进行观察拍照。
1.3 统计学方法 应用SPSS 20.0统计软件进行统计学处理。计量资料以±s表示,常规进行方差齐性检验,组间比较采用单因素方差分析,以P<0.05为差异具有统计学意义。
2 结果
2.1 沉默HOTAIR基因对HBMVEC增殖的影响荧光定量PCR结果显示,siHOTAIR组U87细胞的HOTAIR表达水平明显低于si-NC组,差异有统计学意义(P<0.05),回复组HOTAIR表达水平明显高于siHOTAIR组,差异有统计学意义(P<0.01),见图1A。MTT染色结果显示,用转染后U87细胞培养上清液孵育HBMVEC后,与si-NC组比较,siHOTAIR组细胞上清液孵育的HBMVEC增殖活力降低,差异有统计学意义(P<0.01),而与siHOTAIR组相比,回复组孵育的HBMVEC增殖活力显著增加,差异有统计学意义(P<0.01),见图1B。提示HOTAIR能够特异性调控HBMVEC增殖。进一步的克隆形成实验结果显示,siHOTAIR组HBMVEC形成克隆数量少于si-NC组,差异有统计学意义(P<0.01),见图1C和1D。
2.2 沉默HOTAIR基因对HBMVEC迁移的影响细胞迁移实验结果提示,siHOTAIR组U87细胞诱导的HBMVEC迁移数量少于si-NC组,差异有统计学意义(P<0.01)。见图2A和2B。
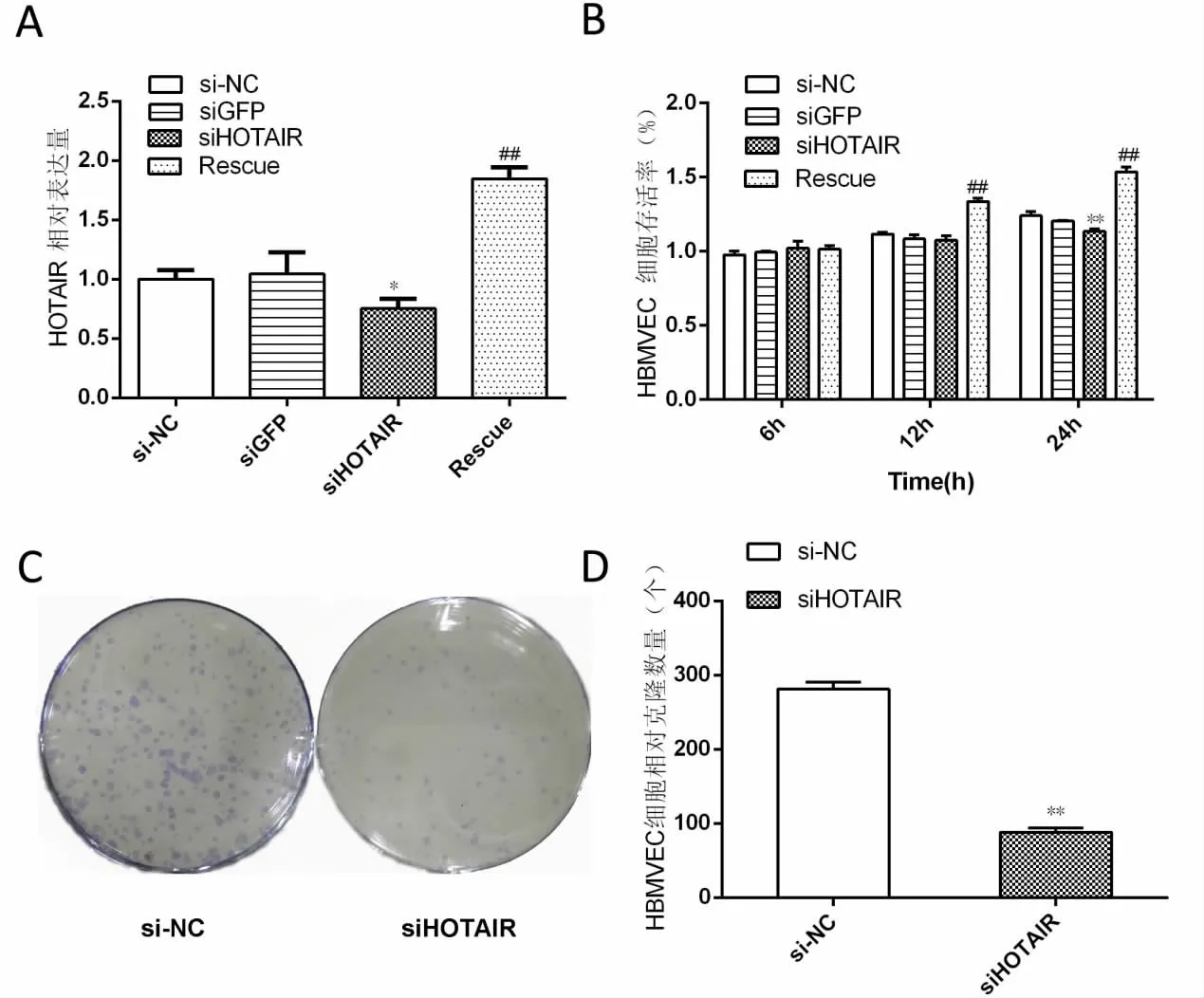
图1 沉默HOTAIR基因对HBMVEC增殖的影响Fig.1 The influence of knockdown HOTAIR gene on HBMVEC proliferation
2.3 沉默HOTAIR基因对HBMVEC细胞成管的影响 实验结果显示,siHOTAIR组U87细胞诱导的HBMVEC细胞成管数量明显少于si-NC组,差异有统计学意义(P<0.01)。见图3A和3B。
2.4 HOTAIR基因与U87细胞分泌EVs的关系 荧光定量PCR结果显示,空白组、RNase组、Triton X-100组3组间HOTAIR基因表达量无明显差异(P>0.05),而RNase和Triton X-100联合组 HOTAIR基因表达量则显著低于其余3组,差异有统计学意义(P<0.01)。见图4。
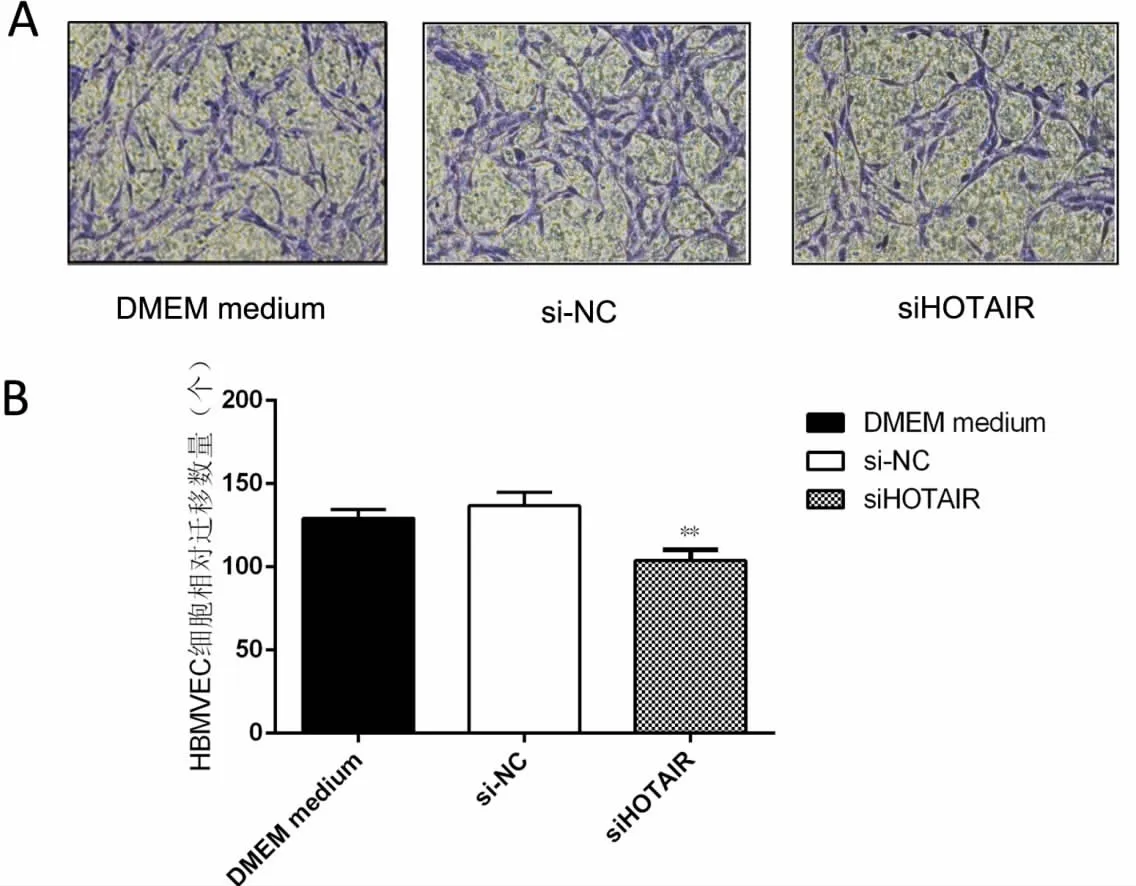
图2 siHOTAIR转染上清液对HBMVEC迁移的影响Fig.2 The influence of transfect supernatant on migration of HBMVEC
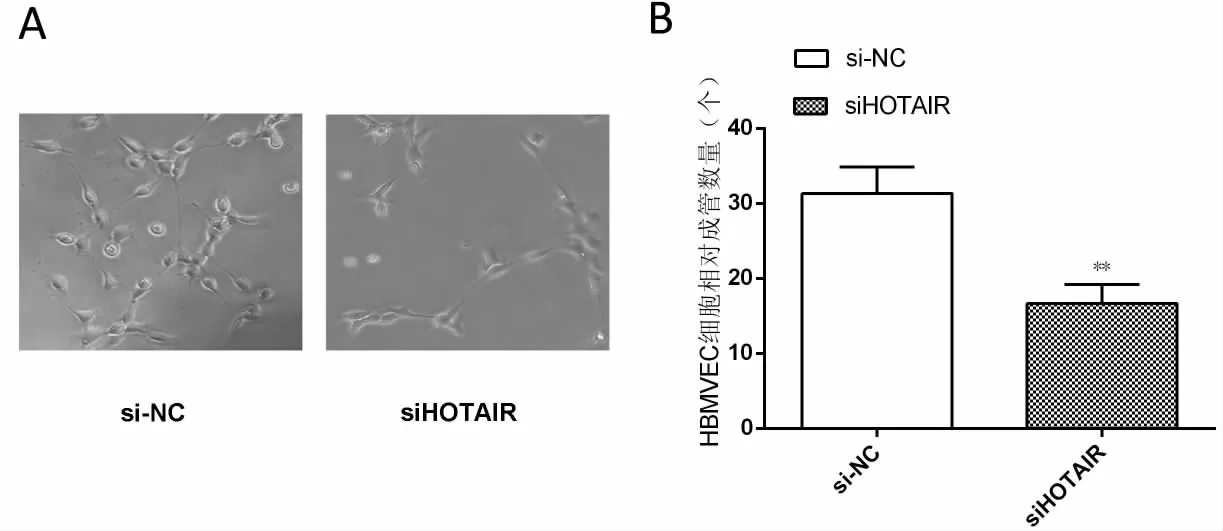
图3 HBMVEC成管图片(200×)Fig.3 The tubes formation of HBMVEC maps(200×)
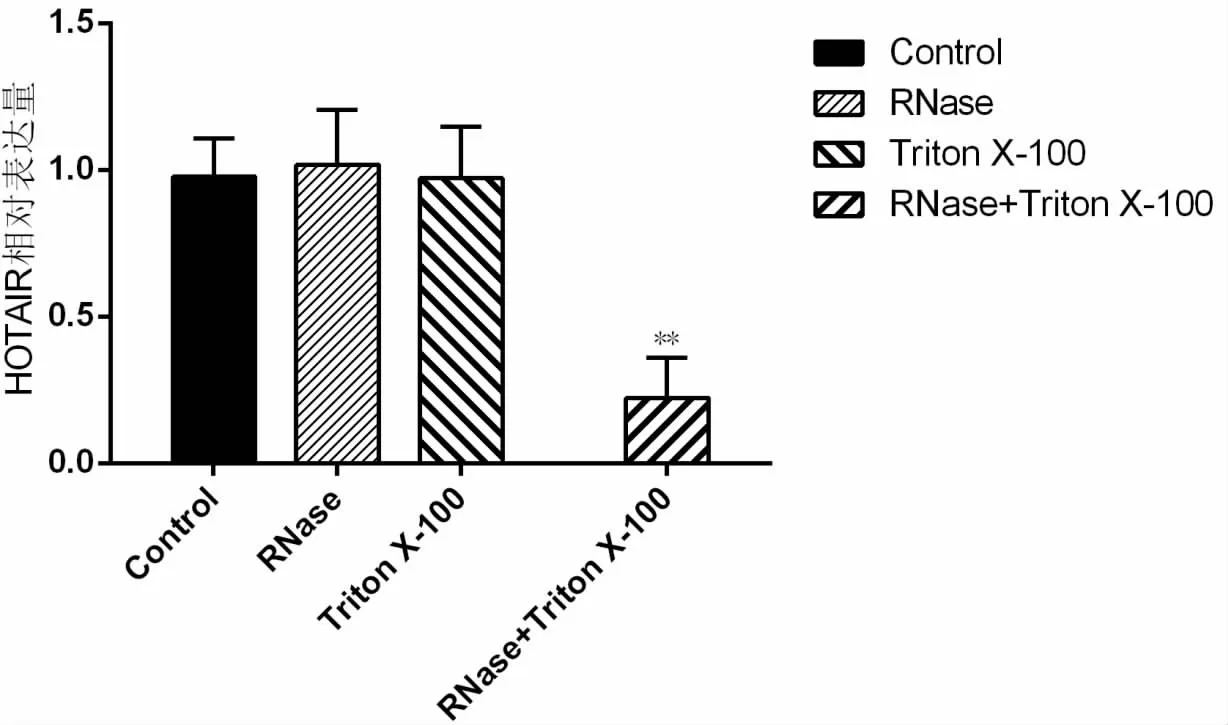
图4 U87细胞分泌的EVs中HOTAIR表达水平Fig.4 The expression levels of HOTAIR in extracellular vesicles derived by U87 cells
3 讨论
lncRNAs是一类转录长度大于200nts、不编码蛋白质的RNA分子,可在转录以及转录后水平调控调节相关基因表达,参与多种生物学活动[21-24]。越来越多的研究表明,lncRNAs与肿瘤之间存在密切关系,在肿瘤发生、肿瘤血管生成以及肿瘤远端转移等发展进程中扮演重要角色[25-29]。肿瘤血管生成与肿瘤生长密切相关,实体肿瘤的生长和转移过程高度依赖肿瘤新生血管[30]。Jia等[31]研究证实,H19可通过抑制miRNA-29a从而促进胶质瘤血管生成和胶质瘤相关内皮细胞的生物学行为。Zhu等[32]研究发现肝癌高表达转录(highly up-regulated in liver cancer,HULC)基因通过磷脂酰肌醇3激酶(phosphatidylinositol 3 kinase,PI3K)/蛋白激酶 B(protein kinase B,PKB)/雷帕霉素靶向基因(mammalian target of rapamycin,mTOR)信号通路调节内皮细胞特异性分子-1(endothelial cell specific molecule-1,ESM-1)表达,从而参与胶质瘤血管生成。但HOTAIR在胶质瘤血管生成中的作用及其可能的分子机制尚未见于报道。本研究通过RNA干扰试验发现沉默U87细胞中HOTAIR基因表达对HBMVEC增殖存在抑制作用,同时设立回复组以排除RNAi中的脱靶效应,反向验证HOTAIR基因表达对HBMVEC增殖的影响。克隆形成、细胞迁移和体外成管实验也证实HOTAIR在U87细胞诱导HBMVEC增殖、迁移以及成管过程中具有调控作用,进一步提示HOTAIR参与了胶质瘤血管新生过程。
大量研究表明,EVs在肿瘤血管生成过程中发挥重要作用,肿瘤细胞通过向细胞外分泌大量EVs进行细胞间信息交流,并创建适合肿瘤生长和转移的微环境[33]。Gezer等[34]研究发现,在人宫颈癌细胞和乳腺癌细胞分泌的EVs中富含HOTAIR、长链非编码RNA-p21(long non-coding RNA-p21,lncRNA-p21)、长链非编码RNA-细胞周期蛋白D1(long non-coding RNA-cyclind1,lncRNA-CCND1)等 RNA 分子。越来越多的研究表明,脑胶质瘤细胞可以分泌大量EVs促进肿瘤生长及发展[35-37]。为了探索HOTAIR是否存在于U87细胞培养上清液中的EVs中,本研究首先通过荧光定量PCR证实在U87细胞上清液中存在HOTAIR分子,其次用去污剂Triton X-100和RNase证实细胞上清液中的HOTAIR分子受膜性囊泡保护。结合以上实验结果,笔者推测沉默HOTAIR基因可以抑制U87细胞促血管生成活性,而HOTAIR分子可能存在于U87细胞分泌的EVs中,参与调控胶质瘤血管生成。
综上所述,肿瘤新生血管是肿瘤细胞生长和转移过程中不可或缺的重要组成部分,沉默HOTAIR基因可以抑制HBMVEC增殖、迁移以及成管能力。在后续研究中,笔者将从胶质瘤细胞培养上清液和患者血浆中分离EVs,并鉴定与HOTAIR相关的EVs的类型,进一步研究HOTAIR在胶质瘤血管生成中的作用及其可能的机制。
[1] Li J,Zhang M,An G,et al.LncRNA TUG1 acts as a tumor suppressorin human gliomabypromotingcell apoptosis[J].Exp Biol Med(Maywood),2016,241(6):644-649.
[2] Wei J,Gabrusiewicz K,Heimberger A.The controversial role of microglia in malignant gliomas[J].Clin Dev Immunol,2013,2013:285246.
[3] Matsuda K,Sato A,Okada M,et al.Targeting JNK for therapeutic depletion of stem-like glioblastoma cells[J].Sci Rep,2012,2(7):516.
[4] Stupp R,Hegi M E,Mason W P,et al.Effects of radiotherapy with concomitant and adjuvant temozolomide versus radiotherapy alone on survival in glioblastoma in a randomised phase III study:5-year analysis of the EORTC-NCIC trial[J].Lancet Oncol,2009,10(5):459-466.
[5] Folkman J.Tumor angiogenesis:therapeutic implications[J].N Engl J Med,1971,285(21):1182-1186.
[6]Hanahan D,Weinberg R A.Hallmarks of cancer:the next generation[J].Cell,2011,144(5):646-674.
[7] Folkman J,Cole P,Zimmerman S.Tumor behavior in isolated perfused organs:in vitro growth and metastases of biopsy material in rabbit thyroid and canine intestinal segment[J].Ann Surg,1966,164(3):491-502.
[8] Lopes-Bastos B M,Jiang W G,Cai J.Tumour-endothelialcellcommunications:importantand indispensable mediators of tumour angiogenesis[J].Anticancer Res,2016,36(3):1119-1126.
[9] Gourley M,Williamson J S.Angiogenesis:new targets for the development of anticancer chemotherapies[J].Curr Pharm Des,2000,6(4):417-439.
[10]Todorova D,Simoncini S,Lacroix R,et al.Extracellular vesicles in angiogenesis[J].Circ Res,2017,120(10):1658-1673.
[11]Becker A,Thakur B K,Weiss J M,et al.Extracellular vesicles in cancer:cell-to-cell mediators of metastasis[J].Cancer Cell,2016,30(6):836-848.
[12]Xu R,Greening D W,Zhu H J,et al.Extracellular vesicle isolation and characterization:toward clinical application[J].J Clin Invest,2016,126(4):1152-1162.
[13]Giusti I,Delle Monache S,Di Francesco M,et al.From glioblastoma to endothelialcells through extracellular vesicles:messages for angiogenesis[J].Tumour Biol,2016,37(9):12743-12753.
[14]Beyer S,Fleming J,Meng W,et al.The role of miRNAs in angiogenesis,invasion and metabolism and their therapeutic implications in gliomas[J].Cancers(Basel),2017,9(7):pii:E85.
[15]Lang H L,Hu G W,Chen Y,et al.Gliom a cells promote angiogenesis through the release of exosomes containing long non-coding RNA POU3F3[J].Eur Rev Med Pharmacol Sci,2017,21(5):959-972.
[16]Rinn J L,Kertesz M,Wang J K,et al.Functional demarcation of active and silent chromatin domains in human HOX loci by noncoding RNAs[J].Cell,2007,129(7):1311-1323.
[17]Zhang J X,Han L,Bao Z S,et al.HOTAIR,a cell cycle-associated long noncoding RNA and a strong predictor ofsurvival,ispreferentially expressed in classical and mesenchymal glioma[J].Neuro Oncol,2013,15(12):1595-1603.
[18]Wu Y,Zhang L,Zhang L,et al.Long non-coding RNA HOTAIR promotes tumor cell invasion and metastasis by recruiting EZH2 and repressing E-cadherin in oral squamous cell carcinoma[J].Int J Oncol,2015,46(6):2586-2594.
[19]Tsai MC,Manor O,Wan Y,et al.Long noncoding RNA as modular scaffold of histone modification complexes[J].Science,2010,329(5992):689-693.
[20]Gupta RA,Shah N,Wang K C,et al.Long non-coding RNA HOTAIR reprogramschromatinstatetopromote cancer metastasis[J].Nature,2010,464(7291):1071-1076.
[21]Ponting C P,Oliver P L,Reik W.Evolution and functions of long noncoding RNAs[J].Cell,2009,136(4):629-641.
[22]Wang P,Liu Y H,Yao Y L,et al.Long non-coding RNA CASC2 suppresses malignancy in human gliomas by miR-21[J].Cell Signal,2015,27(2):275-282.
[23]Wu Z J,Li Y,Wu Y Z,et al.Long non-coding RNA CCAT2 promotes the breast cancer growth and metasis by regulating TGF-β signaling pathway[J].Eur Rev Med Pharmacol Sci,2017,21(4):706-714.
[24]Dong R,Liu G B,Liu B H,et al.Targeting long noncoding RNA-TUG1 inhibits tumor growth and angiogenesis in hepatoblastoma[J].Cell Death Dis,2016,7(6):e2278.
[25]Nakagawa T,Endo H,Yokoyama M,et al.Large noncoding RNA HOTAIR enhances aggressive biological behavior and is associated with short disease-free survival in human non-small cell lung cancer[J].Biochem Biophys Res Commun,2013,436(2):319-324.
[26]Sand M,Bechara F G,Sand D,et al.Long-noncoding RNAs in basal cell carcinoma[J].Tumour Biol,2016,37(8):10595-10608.
[27]Pan L,Liang W,Fu M,et al.Exosomes-mediated transfer of long noncoding RNA ZFAS1 promotes gastric cancer progression[J].J Cancer Res Clin Oncol,2017,143(6):991-1004.
[28]Liu Y,Tao Z,Qu J,et al.Long non-coding RNA PCAT7 regulates ELF2 signaling through inhibition of miR-134-5p in nasopharyngeal carcinoma[J].Biochem Biophys Res Commun,2017,491(2):374-381.
[29]Su J,Zhang E,Han L,et al.Long noncoding RNA BLACAT1 indicates a poor prognosis of colorectal cancer and affects cell proliferation by epigenetically silencing of p15[J].Cell Death Dis,2017,8(3):e2665.
[30]Folkman J.Angiogenesis in cancer,vascular,rheumatoid and other disease[J].Nat Med,1995,1(1):27-31.
[31]Jia P,Cai H,Liu X,et al.Long non-coding RNA H19 regulates glioma angiogenesis and the biological behavior of glioma-associated endothelial cells by inhibiting microRNA-29a[J].Cancer Lett,2016,381(2):359-369.
[32]Zhu Y,Zhang X,Qi L,et al.HULC long noncoding RNA silencing suppresses angiogenesis by regulating ESM-1 via the PI3K/Akt/mTOR signaling pathway in human gliomas[J].Oncotarget,2016,7(12):14429-14440.
[33]Yamada N O.Extracellular vesicles:emerging mediators of intercellular communication and tumor angiogenesis[J].Ann Transl Med,2017,5(3):59.
[34]Gezer U, zgür E,Cetinkaya M,et al.Long non-coding RNAs with low expression levels in cells are enriched in secreted exosomes[J].Cell Biol Int,2014,38(9):1076-1079.
[35]Godlewski J,Krichevsky A M,Johnson M D,et al.Belonging to a network-microRNAs,extracellular vesicles,and the glioblastoma microenvironment[J].Neuro-Oncology,2015,17(5):652–662.
[36]van der Vos K E,Balaj L,Skog J,et al.Brain tumor microvesicles:insights into intercellular communication in the nervous system[J].Cell Mol Neurobiol,2011,31(6):949-959.
[37]Chistiakov D A,Chekhonin V P.Extracellular vesicles shed by glioma cells:pathogenic role and clinical value[J].Tumour Biol,2014,35(9):8425-8438.

