蒺藜苜蓿SBP-box转录因子基因家族全基因组分析
李月颖,李 菁,刘长宁
(1.中国科学院西双版纳热带植物园,热带植物资源可持续利用重点实验室,中国 勐仑 666303;2.中国科学院大学,中国 北京 100049)
蒺藜苜蓿SBP-box转录因子基因家族全基因组分析
李月颖1,2,李 菁1,刘长宁1
(1.中国科学院西双版纳热带植物园,热带植物资源可持续利用重点实验室,中国 勐仑 666303;2.中国科学院大学,中国 北京 100049)
从全基因组水平研究蒺藜苜蓿SBP-box转录因子基因家族,系统地鉴定出22个SBP-box家族基因,根据基因结构分析、结构域的差异和系统发育分析将这些SBP-box基因分成5个亚家族.基因比较分析表明SBP-box基因家族扩张主要是通过部分复制及串联重复事件来实现的.基因表达模式分析表明蒺藜苜蓿SBP-box基因具有器官特异性并且参与干旱胁迫反应.这些结果为进一步研究蒺藜苜蓿SBP-box 家族基因的功能与进化提供理论参考.
SBP-box转录因子;进化分析;基因表达模式;蒺藜苜蓿
转录因子(Transcription Factors)也称反式作用因子,是指能够与真核基因启动子区域中顺式作用元件发生特异性相互作用的DNA结合蛋白.它们通过彼此之间以及与其他相关蛋白之间的相互作用,参与决定基因在何种组织与何种发育阶段开始转录,或者参与基因应答外界环境因素所导致的转录,在调节植物防卫病原微生物和响应外界环境胁迫中发挥重要作用[1].SBP-box基因家族编码的SBP转录因子是植物所特有的一类转录因子.这些基因都有编码DNA结合结构域的保守核苷酸序列,又称为SBP盒(SBP-box)基因.SBP-box基因首先是在金鱼草(Antirrhinummajus)中识别并结合MADS-box基因SQUAMOSA启动子的一个体外实验中被发现而得到确认的[2].自此之后,SBP-box基因在拟南芥、玉米、白桦等植物中相继被分离出来[3-5],并且对其功能进行了很多研究.
Birkenbihl等[6]在拟南芥中发现了16个SBP-box基因,将它们命名为SPL1-SPL16.这些基因编码的蛋白质能够特异结合在AP1基因(SQUAMOSA在拟南芥中的同源基因)的启动子区段.SPL3和SPL8在拟南芥中能够分别影响植株的成花转变和花粉囊的发育[7];SPL5和SPL15两个基因能够控制拟南芥的生殖生长;SPL14是该基因家族中序列最长的基因之一,近来被认为参与受到真菌毒素FB1诱导的细胞程序性死亡的过程[8];AtSPL2在拟南芥AtJMT超量表达植株中响应茉莉酸甲酯介导的抗病途径[9].玉米中LIGULELESS1(LG1)基因缺失突变体的叶耳和叶舌不能形成[4].Wang等[10]发现在TGA1基因的启动子和编码序列中几个核苷酸的改变与玉米花序结构有关. 2015年Tan等和2016年Song等的研究也分别表明白菜和菊花中的SBP基因具有响应激素处理及非生物胁迫的功能[11-12].
SBP蛋白虽然在一级结构上很多样化,但是它们都有一个高度保守的DNA结合结构域(全称DBD),该区域有约76个氨基酸残基长,即SBP结构域.这一结构域包括两个典型的C3H和C2HC型的锌指结构(Zn-finger structure)[13]和C末端的一段高度保守的双向核定位信号(Nuclear Localization Signal,NLS)[6].Zn2+和核定位信号在蛋白与DNA结合过程中是必需的,C端的NLS与C2HC型锌指结构序列部分重叠,具有引导SBP-box基因进入细胞核从而对其下游基因的转录表达进行调控的功能.另外,现在发现的SBP基因中,有一部分还拥有高度保守的miR156/miR157识别位点[14].
豆科(Leguminosae)是种子植物第三大科,是人类及动物获取食物和营养来源的重要科属之一[15].蒺藜苜蓿是豆科苜蓿属一年生植物,由于其具有生育期短、二倍体、自花授粉、基因组较小且遗传转化效率高等特点被作为豆科基因组学和生物学的模式植物进行研究[16].蒺藜苜蓿全基因组测序已经完成,使得利用生物信息学手段研究该物种基因家族系统演化及功能成为可能.截至目前,在全基因组水平对蒺藜苜蓿SBP-box基因家族的研究尚未见报道.本研究利用生物信息学手段,从全基因组水平筛选和鉴定蒺藜苜蓿SBP-box基因,并利用生物信息及系统发育分析方法进行比较基因组学研究,以探究蒺藜苜蓿SBP-box基因家族系统进化关系、基因结构及保守基序的差异和变化以及基因家族的复制特点;同时基于对基因家族的进化分析,结合表达谱的数据对蒺藜苜蓿SBP-box家族基因功能进行深入研究,以期阐明蒺藜苜蓿SBP-box家族基因在生长、发育及防御等方面的生物学功能,从而为豆科基因资源的开发和利用提供理论基础,实现豆科经济作物中重要基因的克隆及功能研究并为豆科育种的快速发展和其他遗传研究提供现实可行的依据.
1 材料方法
1.1 数据来源
本研究蒺藜苜蓿蛋白质数据,编码序列数据以及全基因组数据下载于JCVI数据库(Mtruncatula_198;http://ftp.jpi-psf.org/pub/compgen/phytozome/v9.0/Mtruncatula)[17];拟南芥SBP-box家族蛋白序列和基因序列下载于拟南芥全基因组数据库(TAIR 9.0,http://www.arabidopsis.org/)[18];蒺藜苜蓿基因芯片表达数据来自诺贝尔基金会蒺藜苜蓿基因表达图谱数据库(http://mtgea.noble.org/v3/)[19-20].
1.2 蒺藜苜蓿SBP-box家族成员鉴定
拟南芥的SBP-box家族成员共17个蛋白序列作为query sequence执行本地BLASTP[21](e-value设置为1×10-5)搜索蒺藜苜蓿的蛋白序列;同时在Pfam数据库(http://pfam.sanger.ac.uk/search)中下载了SBP-box结构域的HMM文件(PF03110),利用HMMER (v3.1b2,http://hmmer. org)构建隐马尔科夫模型(Hidden Markov Model,HMM,)在蒺藜苜蓿蛋白数据库搜索含有SBP-box结构域的候选序列(E-value设为1).将第一步和第二步的结果合并并且去冗余,去冗余后假定的蒺藜苜蓿的蛋白序列放到SMART[22](http://smart.embl-heidelberg.de/)和NCBI Conserved Domains[23](http://www.ncbi.nlm.nih.gov/Structure/cdd/cdd.shtml)在线工具中进一步检查是否具有SBP结构域,手工去除冗余和不完整读码框的序列,最终得到蒺藜苜蓿的SBP-box基因.利用 ExPASy Proteomics Server[24](http://prosite.expasy.org/)对所有蒺藜苜蓿SBP-box蛋白氨基酸序列进行相对分子质量、等电点预测.利用softberry (http:// linux1.softberry.com/berry.phtml)中的The ProtComp 9.0 进行SBP-box蛋白的亚细胞定位预测.
1.3 SBP家族系统发育树构建
利用ClustalX (v2.1)[25]对拟南芥和蒺藜苜蓿SBP蛋白进行多序列联配比对分析,比对结果使用 MEGA[26](v6.0)进行系统进化分析.发育树构建采用邻接法(The Neighbour Joining,NJ),检验参数Boot-strapping重复1 000次.
1.4 蒺藜苜蓿SBP-box基因结构分析以及SBP-box蛋白保守功能基序预测
利用在线工具 Gene Structure Display Server(GSDS; v2.0; http://gsds.cbi.pku.edu.cn/)[27]分析SBP-box家族基因结构.在线工具Motif Elicitation tool(MEME; v4.11.2; http://meme-suite.org/tools/meme)[28]可以很好地分析基因的基序.本研究将蒺藜苜蓿SBP-box蛋白序列提交到MEME网站预测SBP-box基因的基序.预测基序的数量为10;基序的长度为20~200;基序重复的数量为“any”.
1.5 蒺藜苜蓿SBP-box家族染色体定位及复制分析
利用Phytozome植物全基因组数据库(http://phytozome.jgi.doe.gov/pz/portal.html)[29]确定SBP-box基因在染色体上的位置,并使用MapInspect工具(http://www.plantbreeding.wur.nl/uk/software_mapinspect.html)标出每个蒺藜苜蓿SBP-box基因在染色体组上的位置,从而得到每个SBP-box基因在基因组中的分布情况.利用MCScanx[30]进行基因家族的复制分析.判定基因复制事件的条件基于植物基因组复制数据库(the Plant Genome Duplication Database)[31]的鉴定方法即基因重复事件必须同时满足以下条件[32]:(1)两个基因序列相互匹配部分的长度大于较长序列长度的80%;(2)两个基因序列相互匹配部分的相似性大于80%;(3)紧密相连的基因中,只参与一次复制事件.同时结合基因在染色体上的位置,判断发生了串联复制还是片段复制.
1.6 蒺藜苜蓿SBP-box基因家族表达模式分析
根据蒺藜苜蓿SBP-box基因对应的CDS序列,在蒺藜苜蓿基因芯片平台BLAST搜索其对应的探针,探针的表达量数据包括蒺藜苜蓿的根部(root)、叶柄(petiole)、芽(bud)、茎(stem)、花(flower)、果荚(pod)、种子(seed)的不同发育阶段,以及根和茎中SBP-box基因在干旱胁迫下的表达量.通过R软件对SBP-box基因的表达数据进行正则化并运用Pheatmap程序包对正则化的蒺藜苜蓿SBP-box基因表达数据进行聚类分析.
2 结果与分析
2.1 蒺藜苜蓿SBP-box 基因家族的鉴定以及蛋白质的理化性质分析
为了鉴定蒺藜苜蓿SBP-box基因家族,同时使用BLASTP以及HMM搜索蒺藜苜蓿中的蛋白序列来找出包含SBP-box结构域的蛋白.为了进一步确定结果的可靠性,使用SMART和NCBI Conserved Domains检验SBP-box结构域的存在,删除不具有SBP-box保守结构域的序列.最后在蒺藜苜蓿中共鉴定出23个SBP-box蛋白,这些蛋白隶属22个基因,使用物种名的缩写作为前缀,给每一个SBP-box蛋白命名.系统性地评估了这些SBP-box蛋白的基本性质,包括预测蛋白质的长度、相对分子质量、等电点以及亚细胞定位.这些SBP-box蛋白序列的长度为144到1 026个氨基酸残基;利用在线工具ExPASy对蒺藜苜蓿SBP-box蛋白质相对分子质量、理论等电点系数进行分析发现,23个SBP-box中07D(MtSBP3)相对分子质量最大,为112 971;最小的61D(MtSBP5)为16 950;这些基因的理论等电点变异相对较小,从5.4(MtSBP2B)到9.2(MtSBP6)不等;利用在线工具softberry进行蛋白质的亚细胞定位预测与它的转录因子家族的角色相对应,有13个SBP-box定位到细胞核上(表 1).
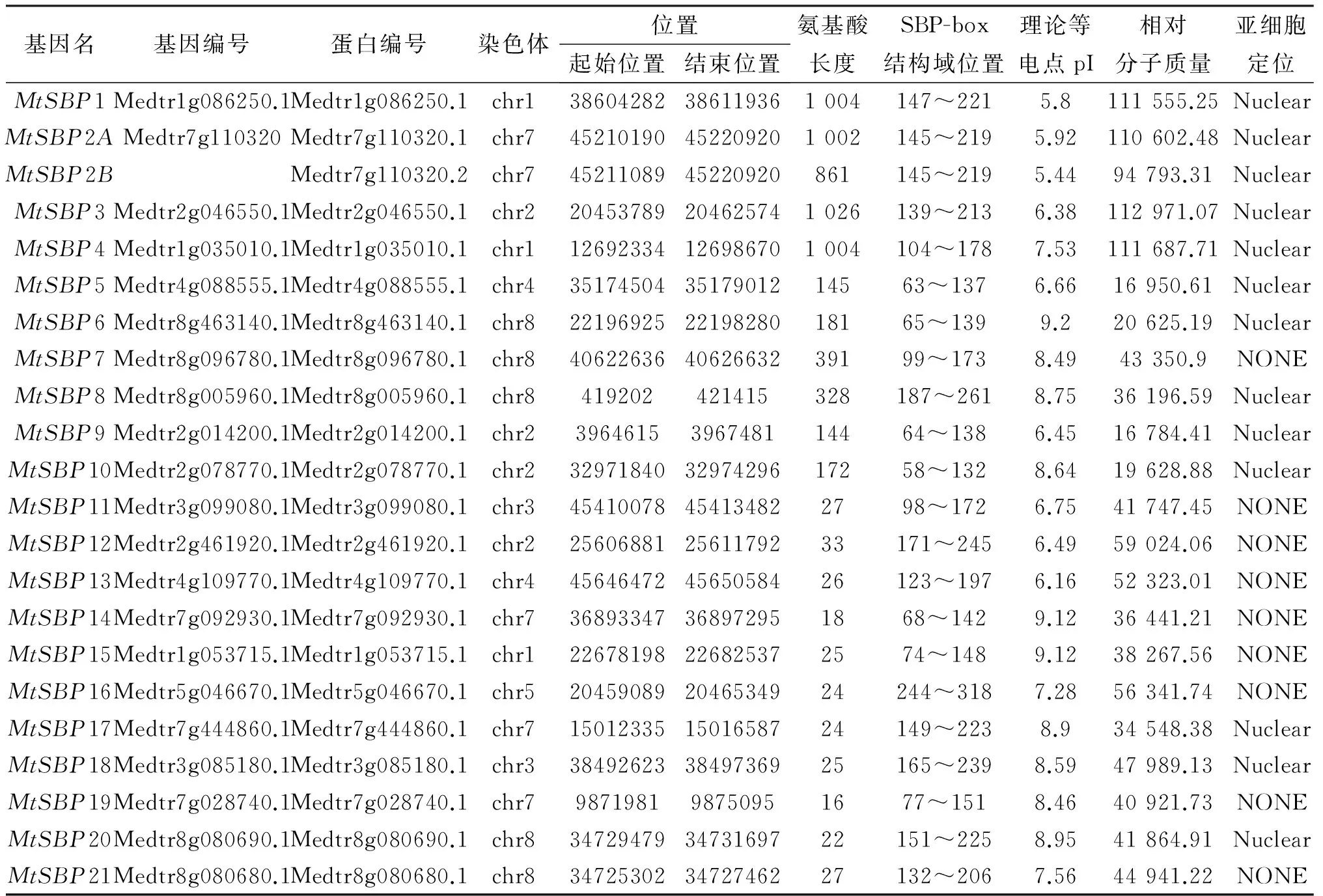
表1 蒺藜苜蓿SBP-box家族信息
2.2 蒺藜苜蓿SBP-box基因家族的进化分析
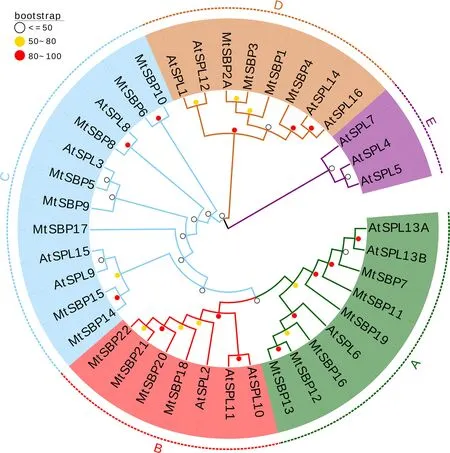
图1 蒺藜苜蓿SBP-box基因家族系统进化树Fig.1 The unrooted dendrogram of all Medicago truncatula SBP-box genes
为了解蒺藜苜蓿SBP-box基因家族成员的系统进化关系,利用蒺藜苜蓿SBP-box蛋白保守结构域构建系统进化树,包括22个蒺藜苜蓿SBP-box蛋白以及17个拟南芥SBP-box蛋白,系统进化分析将这些SBP-box蛋白分为5个亚家族,命名为A,B,C,D以及E(图 1).5个亚家族又可以分为两类,相较于亚家族A,B,C和E,亚家族D的蛋白包含了相对较长的氨基酸序列,这说明这两类之间可能存在一些功能差异性.亚家族 D包含4个蒺藜苜蓿SBP-box蛋白以及4个拟南芥的SBP-box蛋白,其中AtSPL14以及AtSPL16分别为SPL1-Related2 protein (SPL1R2)以及SPL1-Related3 protein(SPL1R3),所以与AtSPL1具有更近的亲缘关系.亚家族A,B,C和E包含19个蒺藜苜蓿SBP-box蛋白以及13个拟南芥SBP-box蛋白,亚家族A,B及C均同时含有蒺藜苜蓿和拟南芥的SBP-box蛋白,亚家族E只含有3个拟南芥的SBP-box蛋白.
2.3 蒺藜苜蓿SBP-box基因家族基因结构以及蛋白质保守基序分析
内含子外显子结构和内含子类型与数量是一个基因家族典型的进化印迹.进一步探究蒺藜苜蓿SBP-box基因家族的22条序列以及拟南芥中SBP-box基因家族17条序列的基因结构,发现系统进化树同一分枝的基因结构比较相似而不同分枝之间的基因结构有所差异.蒺藜苜蓿SBP-box转录因子基因家族的内含子数目为1~10不等(图 2).通过统计发现,在蒺藜苜蓿中,有4个蒺藜苜蓿SBP-box基因家族成员含有1个内含子(比例18.1%);5个蒺藜苜蓿SBP-box基因家族成员含有2个内含子(比例22.7%);7个蒺藜苜蓿SBP-box基因家族成员含有3个内含子(比例31.8%);2个蒺藜苜蓿SBP-box基因家族成员含有4个内含子(比例9.09%);还有4个蒺藜苜蓿SBP-box基因家族成员含有8个以上的内含子(比例18.1%).进一步分析发现,亚家族D的蒺藜苜蓿SBP-box基因家族成员内含子数目大于亚家族A,B,C及E分支,且外显子-内含子结构相似,基因结构比较复杂,MtSBP4含有8个内含子,其余基因含有9个内含子.相反的,A,B,C及E亚家族含有较少的内含子,除了AtSPL7含有9个内含子外,其余的基因仅有1~4个内含子.使用MEME分析工具对所有的蒺藜苜蓿SBP-box蛋白序列进行预测,结果显示10个保守功能基序都具有统计显著性,每个保守基序的e-value都小于1e-300(图3).蒺藜苜蓿SBP-box基因保守基序的预测大部分支持SBP-box转录因子基因家族系统发育分析的分类.这些保守基序的序列特征以及氨基酸长度如图3所示.其中motif2,motif1以及motif4是存在于所有蒺藜苜蓿SBP-box蛋白N端的保守SBP结构域,蒺藜苜蓿的每个亚家族中都有一些基序具有亚家族特异性.motif3,motif7,motif8,motif10,motif5,motif9以及motif6是亚家族D中特有的基序,亚家族D中的蒺藜苜蓿SBP-box蛋白与AtSPL14(SPL1-Related2 protein (SPL1R2)),AtSPL1及AtSPL12比较相似,AtSPL16缺少motif7.与基因结构相对应,A,B,C和E亚家族含有比较统一且简单的蛋白保守基序.亚家族特有的基序可能在亚家族的功能特异性中具有重要作用.
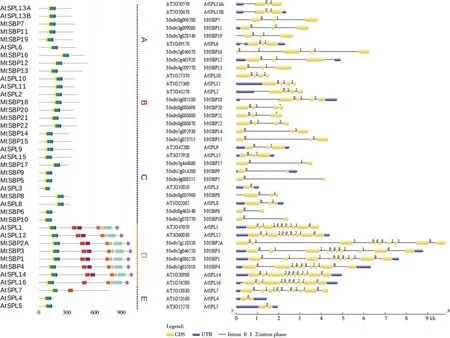
注:黄色框、黑色线条、蓝色框分别表示外显子、内含子和非编码区;不同的保守基序用1~10不同颜色的方框表示图2 蒺藜苜蓿SBP-box家族基因结构以及蛋白保守基序Fig.2 The gene structure and motifs of SBP-box transcription factor

图3 SBP-box家族基序的结构Fig.3 Sequence logo of motifs in SBP-box genes
2.4 蒺藜苜蓿SBP-box基因家族在染色体上的分布与重复
为了探究蒺藜苜蓿SBP-box基因家族的扩增和进化可能的机制,本研究对蒺藜苜蓿SBP-box基因进行染色体定位分析,通过提取蒺藜苜蓿SBP-box基因的染色体定位信息,得到23个SBP-box基因在蒺藜苜蓿染色体上的定位图(图4).23个SBP-box基因不均匀地分布在7条染色体上,每条染色体分布有1~6个SBP-box基因,其中:8号染色体最多,为6个基因;其次是2号和7号染色体,均为4个基因;5号染色体最少,只有1个基因.使用MapInspect软件绘制蒺藜苜蓿SBP-box基因在染色体上的分布,同时检验SBP-box基因家族的复制事件,蒺藜苜蓿SBP-box基因转录因子家族的片段复制和串联重复的基因对分别使用细线以及黄棕色方框进行标示.利用染色体片段、基因组重复区信息,共发现21对发生片段重复和串联重复的同源基因(图4),它们的序列同源性较高,其中19对重复基因对发生片段重复(占所有复制基因对的90.5%),发生片段复制的基因对分布于除亚家族E之外的系统发育分枝的各个亚家族(包括A—D),它们的分布有一定的分支偏好性.大约63.2%(19对片段复制基因中的12对)分布在相同的系统发育亚家族中,这可能是由蒺藜苜蓿基因组的多倍化过程导致;另外两对基因MtSBP20/MtSBP21和MtSBP21/MtSBP22发生了串联复制,且这两对基因均位于亚家族B,串联重复的基因对具有很高的序列相似性,往往高于60%.
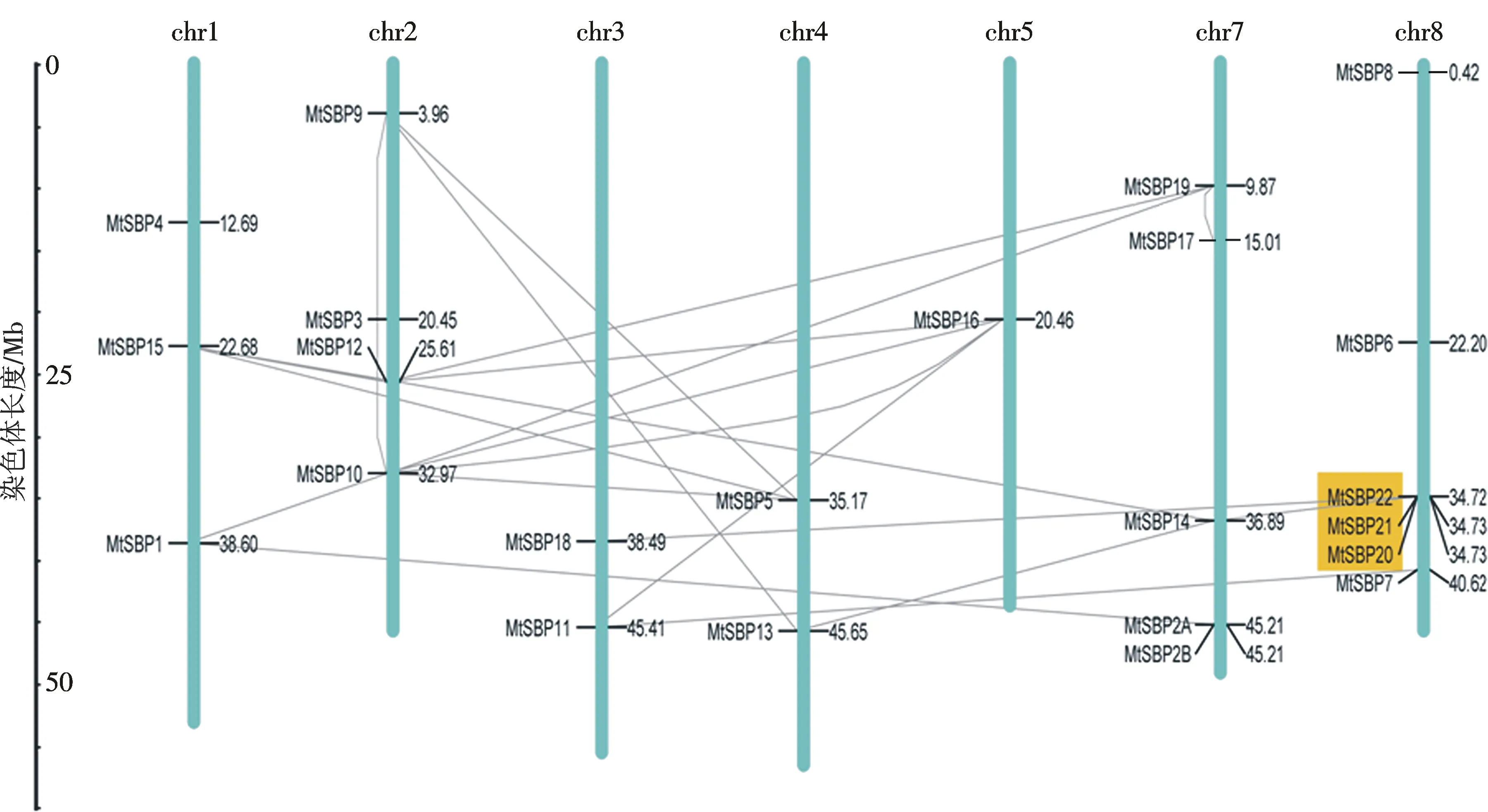
注:片段复制和串联重复的基因对分别用线和方框标明图4 染色体定位以及SBP-box基因家族的基因复制事件Fig.4 Chromosomal locations and gene duplication events of SBP-box genes
2.5 蒺藜苜蓿SBP-box基因家族的表达分析
本研究收集了蒺藜苜蓿不同组织器官(结节、根、茎、叶、花和豆荚以及种子的不同发育阶段)的表达数据.然后使用层次聚类来表现豆科4个物种中的SBP-box基因在不同组织器官的表达模式.如图5(彩图见封三)所示,对于蒺藜苜蓿SBP-box基因,它们被聚类为低表达量(MtSBP17,MtSBP13,MtSBP12以及MtSBP21)、表达量有变化(MtSBP8,MtSBP9,MtSBP14,MtSBP2B和MtSBP16)以及高表达量(MtSBP19,MtSBP1,MtSBP2A,MtSBP3和MtSBP4)3种模式,不同组织器官表达量的分析表明大部分蒺藜苜蓿SBP-box基因家族成员的表达不具有组织特异性,在包括组织器官的生长发育以及种子的生长发育中都发挥重要的作用;但部分基因具有组织特异性如MtSBP19在种子发育的不同阶段表达量差异很大,MtSBP2B/MtSBP14在豆荚中表达量较高,而MtSBP16在根中表达量较高.结合基因复制的结果发现,发生片段复制的基因对,它们倾向于表现出不同的表达模式.在蒺藜苜蓿中总共有19个发生片段复制的基因对,本研究收集到7对的表达数据,其中6对表现出不同的表达模式聚类,例如MtSBP9/MtSBP13与MtSBP17/MtSBP19,所有这些结果表明片段复制的基因参与了植物生长发育的不同过程.同时本研究分析了在干旱胁迫2,3,4,7,10以及14 d豆科SBP-box基因在根和茎中的的表达量变化,通过绘制热图发现,茎中MtSBP9对干旱有显著响应.根中MtSBP2B以及MtSBP16对干旱也有显著响应,表明多个SBP-box基因参与了逆境胁迫应答.
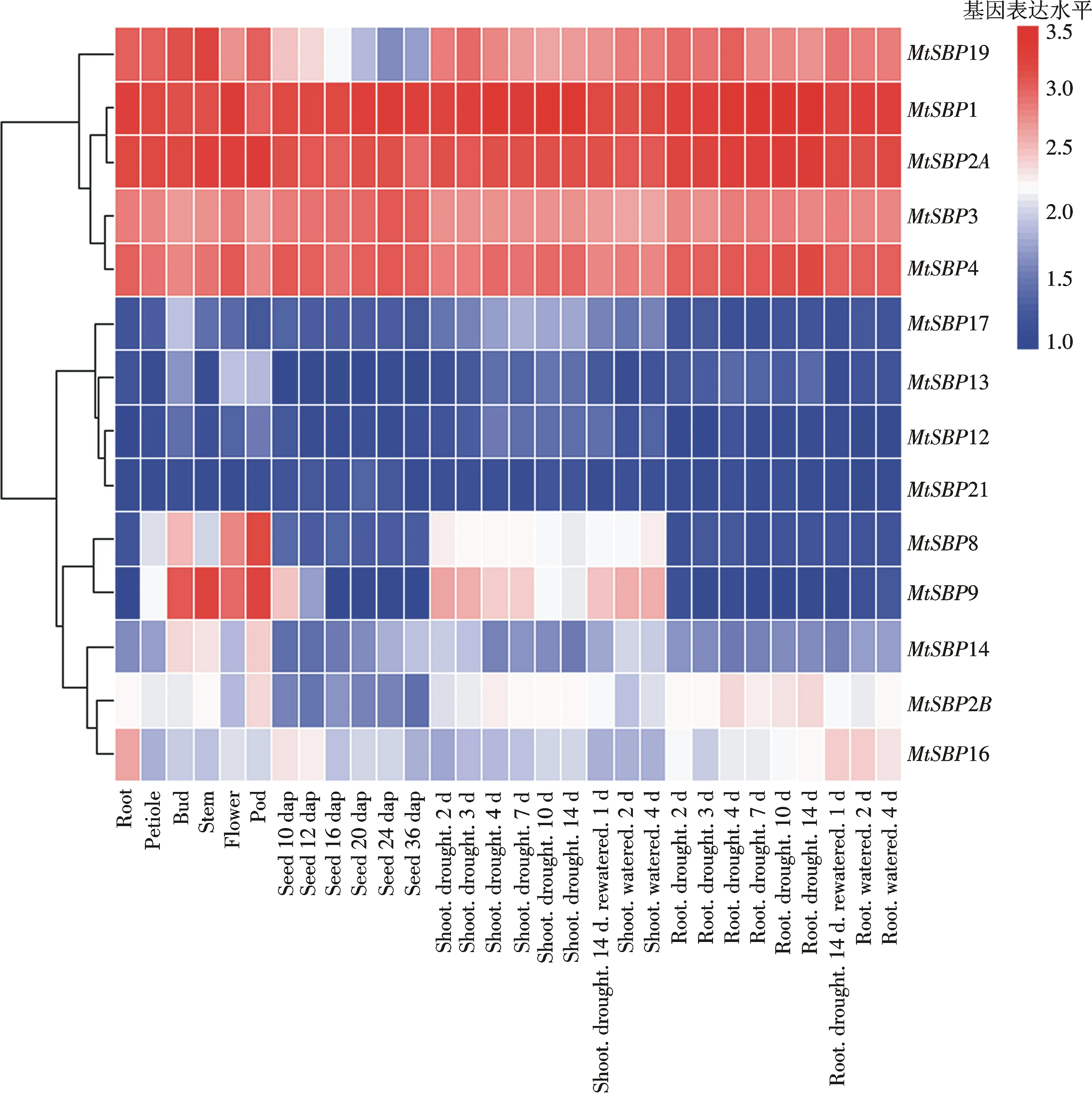
图5 蒺藜苜蓿SBP-box基因表达模式Fig.5 The expression profiles of Medicago truncatula SBP-box genes
3 讨论
SBP-box基因是在研究花形成路径的基因调控网络中发现的,因此被认为和花的发育密切相关,在近几年的研究中发现SBP-box基因有着广泛的生物学功能[33-34].随着基因组测序的发展,SBP-box基因的功能在各种植物中被研究.然而,SBP-box基因在蒺藜苜蓿中的功能至今仍不清楚.本研究运用生物信息学手段,采用结构域搜索的方法,利用已知基因所编码蛋白质中的结构域为检索序列,对蒺藜苜蓿SBP-box基因家族全基因组进行分析,共鉴定出22个蒺藜苜蓿SBP-box基因家族基因,这些基因编码的蛋白质序列中均包含高度保守的SBP结构域.蒺藜苜蓿SBP-box家族成员多于在拟南芥和水稻基因组中鉴定出的17和19个基因[35-36].
通过序列比对和系统进化方法,确定了蒺藜苜蓿SBP-box基因家族的分类和进化关系.根据系统发生树将SBP-box基因家族分成了5个亚家族.通过系统进化树分析得出, SBP-box基因在进化上表现出差异性,暗示其基因功能的多样性;同时每一亚家族的基因间保守性基序的结构和顺序相似,表明同一亚家族中的基因可能具有类似的功能.外显子内含子结构分析表明,处在相同亚家族内的蒺藜苜蓿SBP-box基因大部分具有相同的外显子内含子结构.SBP-box结构域的进化可能与基因结构的多样化有关.蒺藜苜蓿中所有的SBP蛋白都具有完整且高度保守的SBP区(即两个锌指结构和一个双向核定位信号);在拟南芥及蒺藜苜蓿中序列较长的SBP蛋白,尤其是超过1 000个氨基酸残基的SBP蛋白,除了保守的SBP结构域还含有一些其他的区域,如亚家族D中的家族成员,这些区域可以促进SBP蛋白进入核内,进一步通过翻译后修饰而被调控,从而影响它进入核的水平和与DNA结合的水平[6],进一步影响基因在不同生物过程的表达水平.同源性分析是一种相对快速有效了解基因结构、功能和进化的方法[37].因此,蒺藜苜蓿SBP基因的功能可以通过与研究相对通透的模式植物拟南芥进行同源性分析而推测得到.分布在同一亚家族的蒺藜苜蓿以及拟南芥SBP-box基因家族成员可能具有相同的功能.但这些基因的功能需要进一步的试验验证.
基因复制对基因家族的进化具有重要的意义,主要是因为基因复制可为新基因的产生提供最原始的材料,而新基因的产生则促使其产生新的功能[38].植物基因的复制主要有3种方式:片段复制、串联复制、转座事件如逆转录转座和重复转录等.片段复制是最主要的方式,因为大部分植物经历了染色体加倍过程,并在基因组中保留了大量重复的染色体片段[39].本研究分析发现蒺藜苜蓿的SBP-box基因家族主要是通过片段复制进行扩张.之前的研究表明SPL4/SPL5,SPL9/SPL15以及SPL1/SPL12很可能是通过基因组水平重复而产生[40],在本研究中,它们分别分布于亚家族E,C以及D;同时SPL4/SPL5,SPL9/SPL15很可能于芸薹属(Brassica)植物分化之前就已经存在,所以本研究推测亚家族E和C中的蒺藜苜蓿SBP-box基因家族成员存在较早;拟南芥中SPL10/SPL11具有相同的基因结构,且序列高度相似(82.1%),在染色体上彼此相邻,它们可能是通过串联重复机制产生,对应的蒺藜苜蓿中发生串联复制的SBP-box家族成员MtSBP20/MtSBP21以及MtSBP21/MtSBP22也分布在亚家族B中,且具有较高的同源性.拟南芥中SPL01/SPL12,AtSPL14(SPL1-Related2 protein (SPL1R2)),AtSPL16(SPL1-Related3 protein (SPL1R3))和蒺藜苜蓿中的4个家族成员MtSBP1,MtSBP2A,MtSBP3以及MtSBP4构成了亚家族D,这个家族的基因结构比较复杂并且含有除了SBP保守结构域之外的其他的保守基序,这可能为这一家族适应新的功能和进化提供了条件.
本研究利用生物信息学方法进行组织特异性表达模式分析,发现蒺藜苜蓿基因组中重复的基因发生了功能分歧.本研究收集到7对发生片段复制的蒺藜苜蓿SBP-box基因家族成员的表达数据,其中6对表现出不同的表达模式聚类,所有这些结果表明片段复制的基因参与了植物生长发育的不同过程.亚家族D中发生复制的基因对MtSBP1/MtSBP2A具有共同的表达模式,这种表达模式可能意味着其中一个基因的表达已经不占主导地位,或者是生物界常见的冗余现象.也有复制基因对具有不同的表达模式,如位于亚家族A中的MtSBP13以及亚家族B中的MtSBP14,可能意味着基因家族成员正在不断分化.
本研究揭示了蒺藜苜蓿SBP-box基因家族的起源、扩增与进化及可能的功能分化,为后续深入探究蒺藜苜蓿SBP-box基因家族的生物学功能提供了理论参考,并为豆科育种和其他遗传研究提供相关依据.相信随着基因组和转录组数据的积累,对于蒺藜苜蓿SBP-box基因家族的了解将会越来越全面和深入.
[1] JIN J, ZHANG H, KONG L,etal. PlantTFDB 3.0: a portal for the functional and evolutionary study of plant transcription factors[J]. Nucleic Acids Res, 2014,42(D1): D1182-D1187.
[2] KLEIN J, SAEDLER H, HUIJSER P. A new family of DNA binding proteins includes putative transcriptional regulators of theAntirrhinummajusfloral meristem identity gene SQUAMOSA[J]. Mol Gen Genet, 1996,250(1):7-16.
[3] CARDON G H, HÖHMANN S, NETTESHEIM K,etal. Functional analysis of theArabidopsisthalianaSBP-box gene SPL3: A novel gene involved in the floral transition[J]. Plant J, 1997,12(2):367-377.
[4] BECRAFT P W, BONGARD-PIERCE D K, SYLVESTER A W,etal. The liguleless-1 gene acts tissue specifically in maize leaf development[J]. Dev Bio, 1990,141(1):220-232.
[6] BIRKENBIHL R P, JACH G, SAEDLER H,etal. Functional dissection of the plant-specific SBP-domain: overlap of the DNA-binding and nuclear localization domains[J]. J Mol Biol, 2005,352(3):585-596.
[7] SCHMID M, UHLENHAUT N H, GODARD F,etal. Dissection of floral induction pathways using global expression analysis[J]. Development, 2003,130(24):6001-6012.
[8] UNTE U S, SORENSEN A-M, PESARESI P,etal. SPL8, an SBP-box gene that affects pollen sac development in Arabidopsis[J]. Plant Cell, 2003,15(4):1009-1019.
[9] JUNG C, YEU S Y, KOO Y J,etal. Transcript profile of transgenic Arabidopsis constitutively producing methyl jasmonate[J]. J Plant Biol, 2007,50(1):12-17.
[10] WANG H, NUSSBAUM-WAGLER T, LI B,etal. The origin of the naked grains of maize[J]. Nature, 2005,436(7051):714-719.
[11] TAN H W, SONG X M, DUAN W K,etal. Genome-wide analysis of the SBP-box gene family in Chinese cabbage (Brassicarapasubsp.pekinensis)[J]. Genome, 2015,58(11):463-477.
[12] SONG A, GAO T, WU D,etal. Transcriptome-wide identification and expression analysis of chrysanthemum SBP-like transcription factors[J]. Plant Physiol Biochem, 2016,102:10-16.
[13] YAMASAKI K, KIGAWA T, INOUE M,etal. A novel zinc-binding motif revealed by solution structures of DNA-binding domains of Arabidopsis SBP-family transcription factors[J]. J Mol Biol, 2004,337(1):49-63.
[14] MUNUSAMY P, ZOLOTAROV Y, METEIGNIER L V,etal. De novo computational identification of stress-related sequence motifs and microRNA target sites in untranslated regions of a plant translatome[J]. Sci Rep, 2017,7:43861.
[15] AKIBODE S, MAREDIA M K. Global and regional trends in production, trade and consumption of food legume crops: department of agricultural, food, and resource economics[M]. East Lansing: Michigan State University, 2012.
[16] TANG H, KRISHNAKUMAR V, BIDWELL S,etal. An improved genome release (version Mt4. 0) for the model legume Medicago truncatula[J]. BMC Genomics, 2014,15(1):312.
[17] YOUNG N D, DEBELLF, OLDROYD G E,etal. The Medicago genome provides insight into the evolution of rhizobial symbioses[J]. Nature, 2011,480(7378): 520-524.
[18] LAMESCH P, BERARDINI TZ, LI D,etal. The Arabidopsis Information Resource (TAIR): improved gene annotation and new tools[J]. Nucleic Acids Res, 2012,40(D1):D1202-D1210.
[19] BENEDITO V A, TORRES-JEREZ I, MURRAY J D,etal. A gene expression atlas of the model legume[J]. Plant J, 2008,55(3):504-513.
[20] HE J, BENEDITO V A, WANG M,etal. TheMedicagotruncatulagene expression atlas web server[J]. BMC Bioinf, 2009,10:441.
[21] BORATYN G M, CAMACHO C, COOPER P S,etal. BLAST: a more efficient report with usability improvements[J]. Nucleic Acids Res, 2013,41(W1):W29-W33.
[22] LETUNIC I, DOERKS T, BORK P. SMART 7: recent updates to the protein domain annotation resource[J]. Nucleic Acids Res, 2012,40(D1):D302-D305.
[23] MARCHLER-BAUER A, DERBYSHIRE M K, GONZALES N R,etal. CDD: NCBI’s conserved domain database[J]. Nucleic Acids Res, 2015,43(D1):D222-D226.
[24] ARTIMO P, JONNALAGEDDA M, ARNOLD K,etal. ExPASy: SIB bioinformatics resource portal[J]. Nucleic Acids Res, 2012,40(W1):W597-W603.
[25] THOMPSON J D, GIBSON T, HIGGINS D G. Multiple sequence alignment using ClustalW and ClustalX[J]. Curr Protoc Bioinf, 2002,2(3):1-22.
[26] TAMURA K, STECHER G, PETERSON D,etal. MEGA6: molecular evolutionary genetics analysis version 6.0[J]. Mol Biol Evol, 2013,30(12): 2725-2729.
[27] HU B, JIN J, GUO A Y,etal. GSDS 2.0: an upgraded gene feature visualization server[J]. Bioinformatics, 2015,31(8):1296-1297.
[28] BAILEY T L, JOHNSON J, GRANT C E,etal. The MEME suite[J]. Nucleic Acids Res, 2015,43(W1):W39-W49.
[29] GOODSTEIN D M, SHU S, HOWSON R,etal. Phytozome: a comparative platform for green plant genomics[J]. Nucleic Acids Res, 2012,40(D1):D1178-D1186.
[30] WANG Y, TANG H, DEBARRY J D,etal. MCScanX: a toolkit for detection and evolutionary analysis of gene synteny and collinearity[J]. Nucleic Acids Res, 2012,40(7):e49-e49.
[31] LEE T H, TANG H, WANG X,etal. PGDD: a database of gene and genome duplication in plants[J]. Nucleic Acids Res, 2013,41(D1):D1152-D1158.
[32] ZHOU T, WANG Y, CHEN J Q,etal. Genome-wide identification of NBS genes in japonica rice reveals significant expansion of divergent non-TIR NBS-LRR genes[J]. Mol Genet Genomics, 2004,271(4):402-415.
[33] MANNING K, TÖR M, POOLE M,etal. A naturally occurring epigenetic mutation in a gene encoding an SBP-box transcription factor inhibits tomato fruit ripening[J]. Nat Genet, 2006,38(8): 948-952.
[34] WANG Y, HU Z, YANG Y,etal. Function annotation of an SBP-box gene in Arabidopsis based on analysis of co-expression networks and promoters[J]. Int J Mol Sci, 2009,10(1):116-132.
[35] GUO A Y, ZHU Q H, GU X,etal. Genome-wide identification and evolutionary analysis of the plant specific SBP-box transcription factor family[J]. Gene, 2008,418(1):1-8.
[36] YANG Z, WANG X, GU S,etal. Comparative study of SBP-box gene family in Arabidopsis and rice[J]. Gene, 2008,407(1):1-11.
[37] LYONS E, PEDERSEN B, KANE J,etal. Finding and comparing syntenic regions among Arabidopsis and the outgroups papaya, poplar, and grape: CoGe with rosids[J]. Plant Physiol, 2008,148(4):1772-1781.
[38] RENSING S A. Gene duplication as a driver of plant morphogenetic evolution[J]. Curr Opin Plant Biol, 2014,17:43-48.
[39] WANG Y, WANG X, PATERSON A H. Genome and gene duplications and gene expression divergence: a view from plants[J]. Ann N Y Acad Sci, 2012,1256(1):1-14.
[40] ZHANG B, LIU X, ZHAO G,etal. Molecular characterization and expression analysis ofTriticumaestivumsquamosa-promoter binding protein-box genes involved in ear development[J]. J Integr Plant Biol, 2014,56(6):571-581.
Genome-WideAnalysisofSBP-BoxTranscriptionFactorGeneFamilyinMedicagoTruncatula
LIYue-ying1,2,LIJing1,LIUChang-ning1*
(1. Key Laboratory of Tropical Plant Resources and Sustainable Use, Xishuangbanna Tropical Botanical Garden, Chinese Academy of Sciences, Menglun 666303, China; 2. University of Chinese Academy of Sciences, Beijing 100049, China)
This study conducted a genome-wide investigation of the SBP-box gene family inMedicagotruncatula. A total of 22 SBP-box genes were systematically identified fromMedicagotruncatulaand classified into 5 subfamilies according to the gene structure analysis, motif analysis and phylogeny relationship. Further genome comparison analysis revealed that segmental duplication and tandem duplication were two important ways for SBP-box genes to extend in theMedicagotruncatulagenome. Results from expression profile analysis revealed that SBP-box genes have the organ-specificity and were involved in drought stress responses. The data presented here should provide a theoretical foundation for verifying the function and evolution of SBP-box genes inMedicagotruncatula.
SBP-box transcription factors; phylogeny analysis; gene expression patterns;Medicagotruncatula
10.7612/j.issn.1000-2537.2017.06.004
2017-04-26
国家自然科学基金资助项目(31471220)
*通讯作者,E-mail:liuchangning@xtbg.ac.cn
Q349+.13
A
1000-2537(2017)06-0024-10
(编辑 WJ)

