花生种子萌发基因AhSGR的表达分析
陈 静,胡晓辉,苗华荣,杨伟强,张胜忠,石程仁,张智猛
(山东省花生研究所,山东 青岛 266100)
花生种子萌发基因AhSGR的表达分析
陈 静,胡晓辉,苗华荣,杨伟强,张胜忠,石程仁*,张智猛*
(山东省花生研究所,山东 青岛 266100)
种子萌发是一个复杂的多步骤过程,而与花生种子萌发相关基因的研究鲜见报道。本研究以花育52号为试材,根据花生吸胀休眠和休眠释放转录组测序结果获得了一个与花生种子萌发相关的基因AhSGR。AhSGR基因cDNA全长1338 bp,开放阅读框(ORF)为 885 bp,编码295 个氨基酸,该基因编码未知蛋白,在44~122AA处含有DOG1保守结构域,分子量33.39 kD,等电点为5.04,定位于细胞核内,未发现信号肽及其剪切位点,推测AhSGR可能是转录因子。荧光定量检测表明该基因与花生种子萌发密切相关。
花生;种子萌发;AhSGR;基因表达
种子萌发通常是从干燥有活力的种子吸水吸胀开始直到胚轴伸长为止的一系列的生理生化过程,以胚根伸出胚周围的附着物作为可见的发芽标志[1]。种子萌发对于农业生产具有重要的经济意义,首先播种时种子是否正常萌发决定了田间植株群体大小,直接影响作物的产量;其次收获时种子在植株上发芽导致产量损失、品质下降、种用安全等问题,如一些重要的禾谷类作物水稻、小麦和玉米由于长期的栽培选择生产中存在的穗发芽;而花生收获时的籽仁在荚果内萌发也直接影响产量,对花生种用隐患较大。
种子萌发是一个复杂的、涉及多层次调控的过程。花生是地上开花地下结果的油料作物,花生种子萌发机制的探讨聚焦于储藏物质[2]、蛋白质水平的变化[3-5]。花生种子萌发进程中,子叶干物重减少的同时,脂肪和蛋白质含量呈有规律递减;脂肪酸和氨基酸各组分的变化趋向不甚一致,某些组分表现出增加趋势[2]。多胺氧化酶(PAO)的活性变化与花生种子的萌发过程有着密切的关系[3-4]。蛋白质在花生种子萌发过程,胚轴和子叶中41 kD和38.5kD亚基的降解均先于60.5 kD亚基[5];在蛋白质水平上的表达有差异,种子萌发8h与未萌发种子胚蛋白质2-DE图对比,有14个点表达较特异,其中7个蛋白点在萌发的花生胚中表达量下调、7个表达量上调[6]。而目前鲜见关于花生种子萌发分子水平上的研究报道。本研究在转录组分析基础上,发现了一个与花生种子萌发相关的基因,暂定名为AhSGR,利用生物信息学方法对其序列进行了分析,并采用 real-time PCR技术分析了该基因在花生种子萌发过程中的表达情况,为研究该基因在花生种子萌发中的作用奠定基础。
1 材料与方法
1.1 供试材料
供试材料为山东省花生研究所育成的小花生品种花育52号[7]和花育28号[8]。花育52号具有强休眠特性,花育28具有弱休眠特性。以花育52号吸胀24 h(28℃)的休眠种子为对照(CK),用100 mg/L乙烯利处理对照2.5h后取生长不同时间的种子用于转录组测序(附表)。
花育52休眠种子(HY52-D)、花育52室温破除休眠的种子(HY52-ND)、花育28无休眠种子(HY28),取样时间为吸胀0、6、12、18和24 h的种子,以及对照CK、AE1、AE2和AE3(附表)用于验证分析基因的表达差异。

附表 供试材料
注: “—” 种子外在无变化。
Note: “—” indict that there are not superficial variances of seeds.
1.2 花生总RNA的提取及cDNA的合成
花生种子总RNA的提取按照RNA试剂盒(TransGen)说明书进行,用Prime RT Reagent Kit(大连Takara)将提取的总RNA反转录成cDNA。
1.3 荧光定量 PCR
采用LightCycle2.0(rRoche Diagnostics 公司)荧光定量PCR仪。PCR反应条件为: 95℃预变性 30 s;95℃ 变性5 s,60℃退火 20 s,45个循环;PCR后先加热到95℃,然后降至65℃开始缓慢升温(0.1℃/s)至95℃,记录荧光信号的变化,获得扩增产物的溶解曲线。根据AhSGR基因序列设计荧光定量引物,引物为:5'- CAGAAGGCTCAATGAAGG-3'和5'-TCTGATGGTTCCAAAGTTG-3'。内参基因为Actin11,Actin11基因引物为:5'-TTGGAATGGGTCAGAAGGATGC-3'和5'-GCTTCTTACTGAGGCACCACT-3'。实时定量PCR试验SYBR Premix Ex Taq试剂盒(大连TaKaRa),按照说明进行。每个样品重复3次,取平均值,采用2-ΔΔCt的方法。
1.4 花生AhSGR基因生物信息学分析
利用DNA MAN 6.0对AhSGR基因的氨基酸序列、开放阅读框搜索进行分析;利用在线BLAST(http://www.ncbi.nlm.nih.gov/BLAST/)进行序列比对、同源序列搜索;利用TMHMM (http://www.cbs.dtu.dk/services/TMHMM)分析蛋白质跨膜;利用ProtScale (http://web.expasy.org/protscale/)分析蛋白质疏水性;利用SignalP V4.1 http://www.cbs.dtu.dk/services/SignalP/分析蛋白质信号态;利用ProtComp V9.0(http://linux1.softberry.com/berry.phtml?topic=protcomppl&group=programs&subgroup=proloc)进行蛋白亚细胞定位预测。
2 结果与分析
2.1 花生AhSGR基因全长 cDNA 的序列分析
根据转录组结果获得含有DOG1保守结构域的转录本26个, BLASTP比对发现11个为全长,RPMN表达量高于20的仅有4个,与休眠态种子(CK)相比丰度高且上调,表达的一个unigene暂定名为AhSGR并进行分析。
AhSGR基因cDNA 全长为1338 bp,其5'-端有 83 bp 的非编码区(5'-UTR),3'-端有 369 bp 的非编码区(3'-UTR),开放阅读框(ORF)长 885 bp,编码295 个氨基酸(见图1)。
2.2 花生AhSGR推导的氨基酸序列同源性
通过NCBI在线BLASTP分析AhSGR编码氨基酸序列,该基因推导的氨基酸序列与其他植物的转录因子氨基酸序列相似性较高(图2),其中与大豆(transcription factor TGA4-like ,XP_003524316.1)、鹰嘴豆(transcription factor HBP-1b(c38)-like,XP_004503896.1)、蒺藜苜蓿(Transcription factor HBP-1b(c1) XP_003630355.1)的相似性达到66%,与梅(transcription factor TGA2-like XP_008227573.1)的相似性达到59%。推测AhSGR可能是转录因子。
2.3 花生AhSGR编码蛋白的生物信息学分析
2.3.1 AhSGR氨基酸组成分析
利用Protparam预测蛋白质的基本物理化学性质,AhSGR蛋白分子量33.39 kD,等电点5.04,分子式为C1457H2296N406O469S12,不稳定参数55.23,属于不稳定蛋白。该蛋白氨基酸组成包括Leu 11.90%、Glu 10.80%、Ser 8.80%、Ala 7.80%、Asp 6.40%、Thr 6.40%、Gly 5.80%、Arg 5.40%、Lys 5.40%、Gln 4.40%、His 4.10%、Val 4.10%、Ile 3.70%、Met 3.10%、Tyr 2.70%、Phe 2.40%、Pro 2.40%、Asn 1.70%、Trp 1.70%、Cys 1.00%,不含Pyl和Sec (图3)。

图1 花生AhSGR基因全长cDNA的核苷酸序列和推测氨基酸序列 Fig.1 Nucleotide and pupative AA sequences of the AhSGR full length cDNA of the peanut

图2 AhSGR与其他植物的转录因子氨基酸序列比对 Fig.2 Multiple alignment of deduced amino acid sequences of AhSGR and other transcription factor
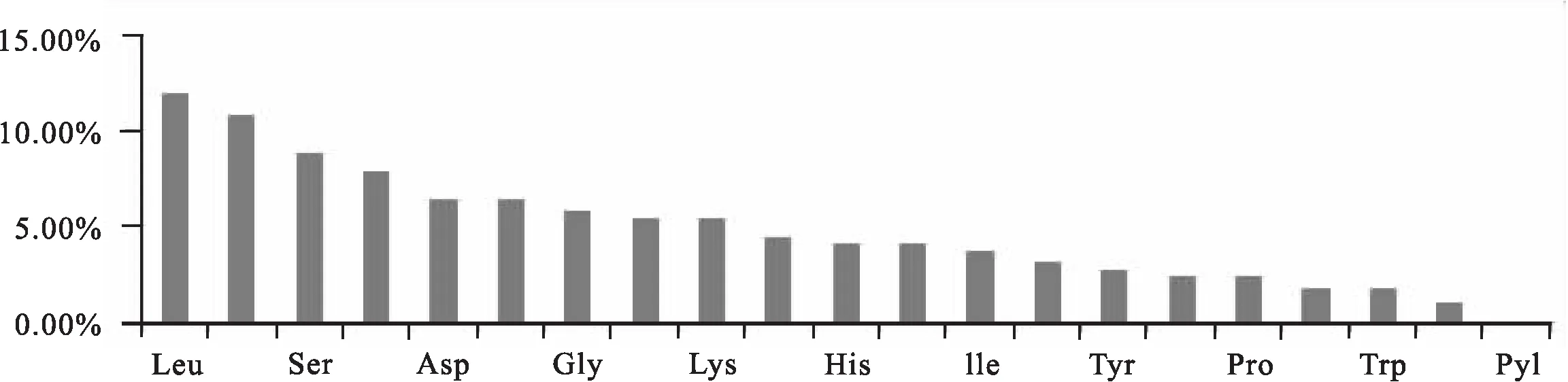
图3 花生AhSGR的核苷酸序列和推测氨基酸序列 Fig.3 Nucleotide and pupative AA sequences of the AhSGR of the peanut
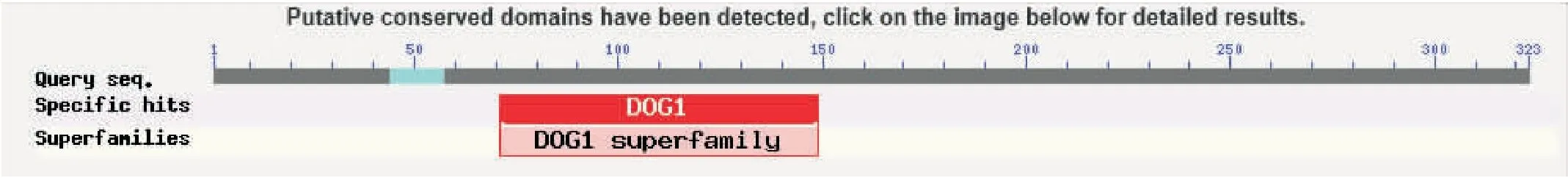
图4 AhSGR编码蛋白保守结构域Fig.4 The conserved domain of AhSGR
2.3.2AhSGR蛋白结构分析
二级结构预测结果表明,AhSGR蛋白含有 57.63%的α-螺旋(Alpha helix),11.53%的延伸链(extended strand), 30.85%的随机卷曲(Random coil)。在 NCBI 上进行保守结构域分析结果表明,AhSGR蛋白具有 DOG1家族的保守结构域(图4)。预测AhSGR蛋白的三级结构如图 5。
2.3.3 AhSGR蛋白跨膜区域预测
通过TMPRED 软件预测分析AhSGR蛋白没有明显的跨膜结构域(图6)。

图5 AhSGR编码蛋白三级结构 图6 AhSGR蛋白的跨膜结构域 Fig. 5 The 3-D structure of AhSGR encoding protein Fig. 6 The transmembrane domains of AhSGR protein
2.3.4 AhSGR蛋白亲水性/疏水性推测
AhSGR蛋白亲水氨基酸含量高于疏水氨基酸含量,是一个水溶性蛋白质(图 7)。
2.3.5 AhSGR亚细胞定位推测以及AhSGR蛋白质信号肽预测
通过ProtComp V9.0分析亚细胞定位结果表明,该蛋白被定位于细胞核中,综合得分8.0。采用 SignalP V4.1软件系统分析,花生AhSGR蛋白没有发现信号肽及其剪切位点(图8)。
2.4 花生AhSGR基因的表达差异
AhSGR基因在不同品种萌发过程中表达有差异(图9)。HY52-D为吸胀休眠态种子,乙烯利处理2.5 h(AE1)时AhSGR基因上调表达,露白(AE2)时达到峰值。对不同品种进行验证表明:HY28吸胀萌发过程中,AhSGR基因在12 h时表达上调,18 h达到峰值,与0 h相比AhSGR在12 h和18 h时表达量上调3倍以上;HY52-ND吸胀萌发过程中,AhSGR基因在18 h时表达量迅速上升,与0 h相比表达上调8倍。种子吸胀相同时间,无休眠种子中AhSGR表达高于休眠种子。
图7 AhSGR蛋白的疏水性/亲水性分析 图8 AhSGR蛋白的信号肽分析 Fig.7 Hydrophobicity/hydrophilicity analysis of AhSGR Fig. 8 Signal peptide analysis of AhSGR
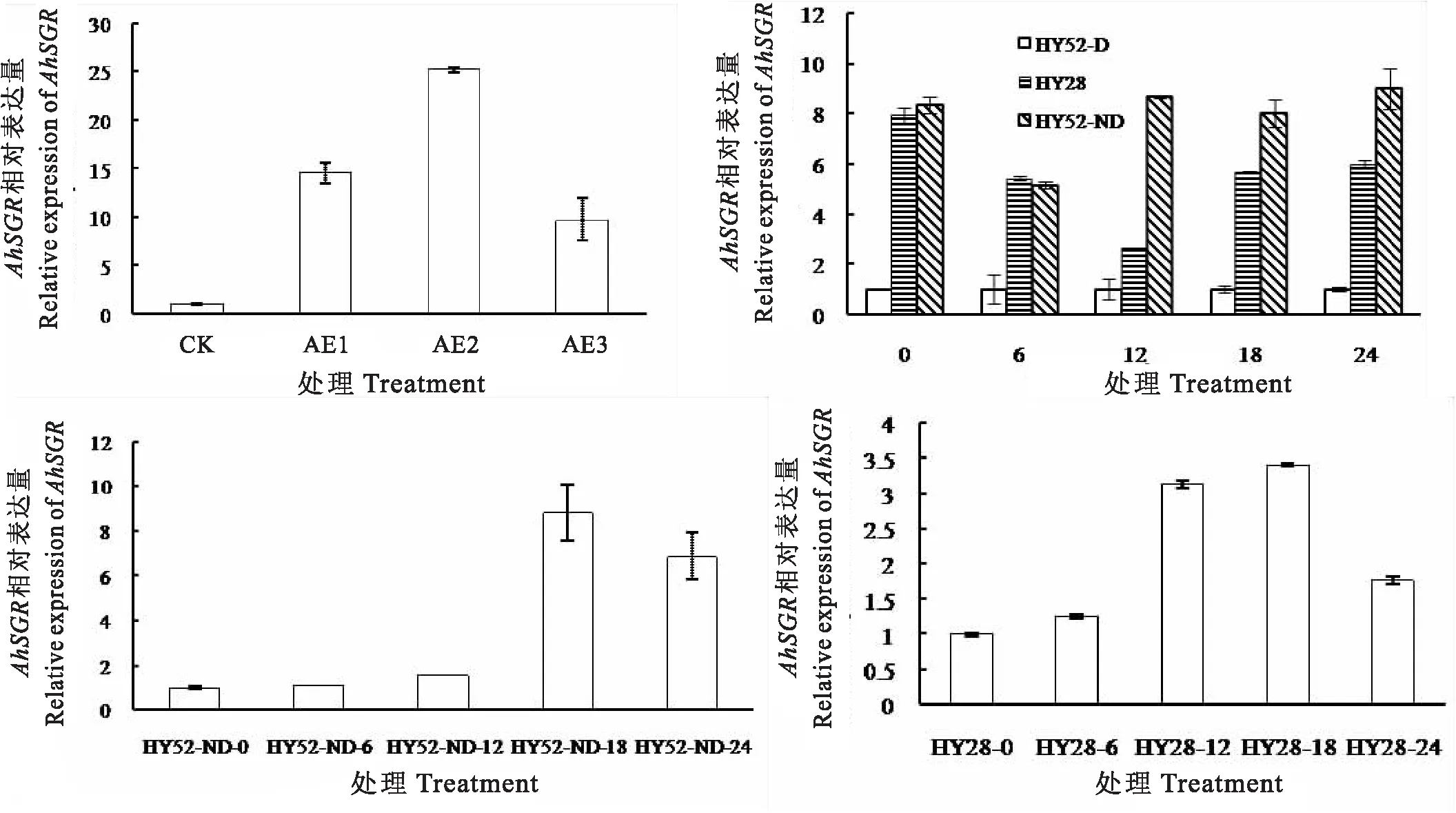
图9 不同品种萌发过程AhSGR基因的相对表达水平 Fig. 9 Relative expression of AhSGR gene at different time in different varieties
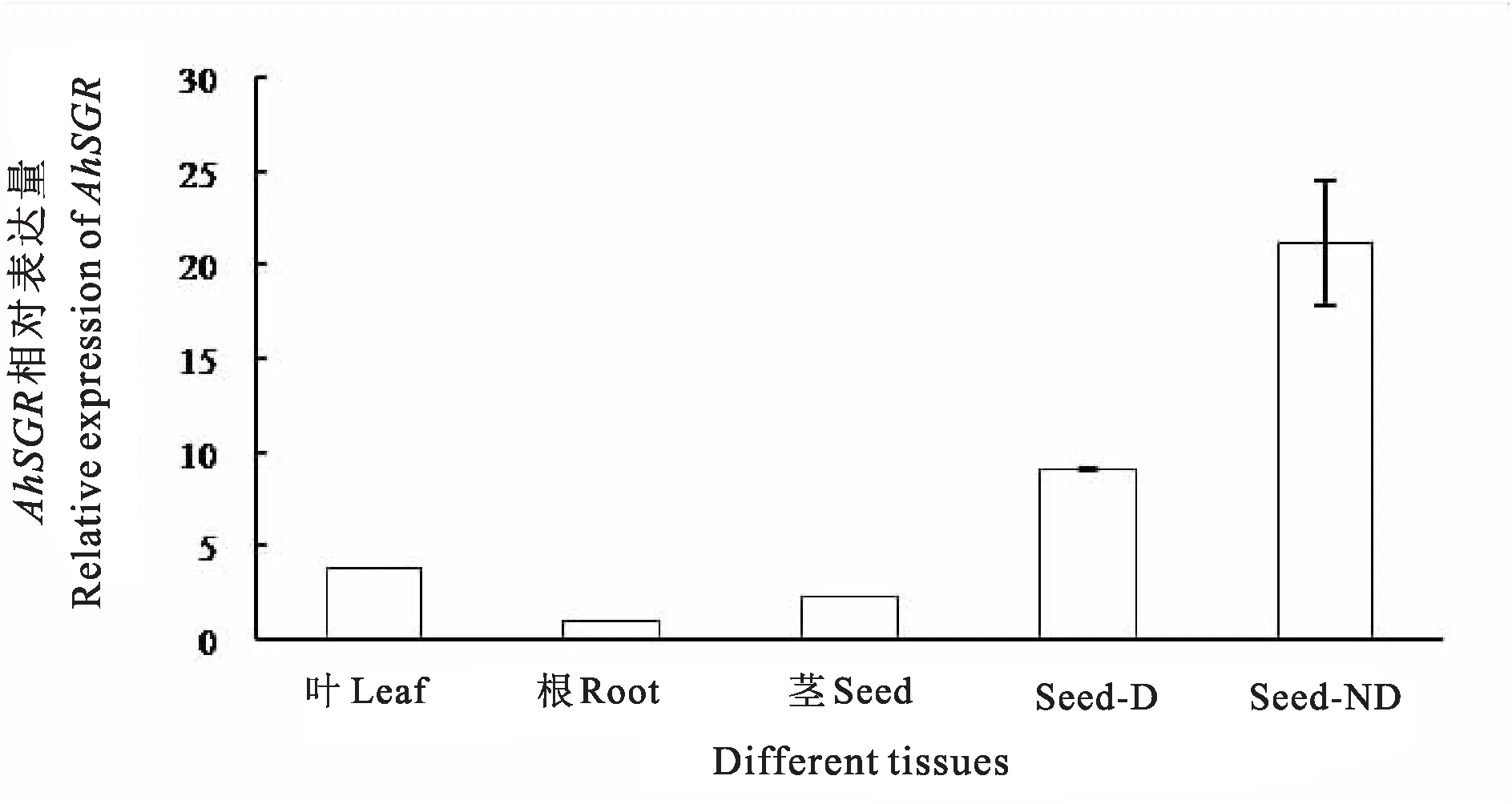
图10 AhSGR基因在不同组织的相对表达水平 Fig. 10 Relative expression of AhSGR in different tissues
2.5 花生不同组织AhSGR基因的荧光定量表达
AhSGR基因在花生叶、根、茎、休眠种子(Seed-D)、无休眠种子(Seed-ND)中都有表达,其中无休眠种子中的相对表达量最高,其次是休眠种子,根中的表达量最低(图10)。
3 讨 论
生物信息学分析表明,AhSGR编码的氨基酸序列与大豆(transcription factor TGA4-like ,XP_003524316.1)、鹰嘴豆(transcription factor HBP-1b(c38)-like,XP_004503896.1)、蒺藜苜蓿(Transcription factor HBP-1b(c1) XP_003630355.1)的相似性达到66%,推测AhSGR可能是转录因子。该基因编码未知蛋白,在44~122 bp处含有DOG1保守结构域;AhSGR基因编码蛋白位于细胞核,分子量33.39 kD,等电点为5.04,在该蛋白中没有发现信号肽及其剪切位点。不同品种验证AhSGR基因表达表明,AhSGR基因在HY28种子吸胀萌发12h时上调表达、在HY52-ND种子吸胀萌发18 h时上调表达。种子吸胀相同时间,无休眠种子中AhSGR表达高于休眠种子。推测AhSGR基因可能与种子萌发密切相关。
DOG1是通过QTL方法揭示的新的休眠基因,该基因是克隆的第一个休眠QTL,编码未知功能蛋白[9],通过dog1突变体证实DOG1是控制种子休眠的重要调控子。dog1突变体与ABA合成信号途径突变体在休眠表现方面相似,但已有研究表明DOG1和ABA是独立的途径。DOG1调控休眠需要ABA信号途径参与[10],通过增强ABI4基因的表达,DOG1能够提高种子对ABA和糖的敏感性,从而有利于维持或增强种子的休眠。小麦和大麦中找到了与拟南芥休眠基因DOG1同源的基因TaDOG1L1和HvDOG1L1,把这两个候选基因转入拟南芥,转基因拟南芥种子休眠性显著增强[11]。AhSGR基因编码未知蛋白,在44~122 bp处含有DOG1保守结构域,但其序列与DOG1不具备相似性,荧光定量表达分析其与种子萌发密切相关,而与种子休眠没有相关性。
[1] Bewley J D, Black M. Seeds: Physiology of development and germination [M]. New York: Plenum Publishing Corporation, 1994: 199-267.
[2] 徐宜民,时焦. 花生种子萌发中主要贮藏物质的变化[J]. 中国油料,1993(2):35-37.
[3] 何生根,黄学林,傅家瑞.花生种子萌发过程中胚轴多胺氧化酶的活性变化[J]. 植物学通报,1998(5):63-65.
[4] 廖斌, 卢春斌,王蕾,等. 花生种子发育和萌发过程中贮藏蛋白的合成和降解[J]. 植物生理与分子生物学学报,2004,30(1):115-118.
[5] 王颖. 花生(ArachishypogaeaL.)种子萌发生长阶段多胺氧化酶的时空特点及调控研究[M]. 重庆:西南师范大学,2003.
[6] 王通,李玲,梁炫强.花生种子萌发状态下胚蛋白质变化初步研究[J]. 广东农业科学,2008(增刊):50-52.
[7] 苗华荣,崔凤高,胡晓辉,等.高油酸系列花生新品种的选育及特性[J]. 花生学报,2015,4(1):64-65.
[8] 苗华荣,吴兰荣,崔凤高,等. 早熟高产油用小花生花育28号选育及其高产栽培技术[J]. 山东农业科学,2011(11):107-108.
[9] Bentsink L, Jowett J, Hanhart C J, et al. Cloning ofDOG1, a quantitative trait locus controlling seed dormancy inArabidopsis[J]. PNAS, 2006, 103(45): 17042-17047.
[10] Nakabayashi K, Bartsch M, Xiang Y, et al. The time required for dormancy release inArabidopsisis determined by DELAY OF GERMINATION1 protein levels in freshly harvested seeds [J]. Plant Cell, 2012, 24(7):2826-2838.
[11] Ashikawa I, Abe F, Nakamura S. Ectopic expression of wheat and barleyDOG1-like genes promotes seed dormancy inArabidopsis[J]. Plant Sci., 2010, 179: 536-542.
ExpressionAnalysisofSeedGerminationRelatedGeneAhSGRinPeanut(ArachishypogaeaL.)
CHEN Jing, HU Xiao-hui, MIAO Hua-rong, YANG Wei-qiang,ZHANG Sheng-zhong, SHI Cheng-ren*, ZHANG Zhi-meng*
(ShandongPeanutResearchInstitute,Qingdao266100,China)
Seed germination is a complex and multistep process. There are little reports on genes related to peanut seed germination. To research important functional genes on control of seed germination, the full-length cDNA sequence of a gene related to seed germination (AhSGR) was screened from seed of peanut cultivar Huayu52 by RNA-seq. The whole sequence ofAhSGRwas 1338bp and its open reading frame was 885bp, encoding a 295 amino acid residuces.AhSGRencoded unknown protein which had DOG1 conserved domain in 44-122AA, a molecular weight of 33.39 kD and pI of 5.04, which was located in nucleus.It had no signal peptide and splice site. According to allignment similarity by Blastp, We speculated thatAhSGRmay be a transcription factor. The results of real-time quantitative PCR showed that the expression ofAhSGRgene was closely correlated to the seed germination.
peanut; seed germination;AhSGR; gene expression
10.14001/j.issn.1002-4093.2017.03.003
Q786; S565.2
A
2017-09-03
山东省农业科学院创新工程(CXGC2016-02);山东省重点研发计划(2016ZDJS10A02);山东省农业科学院重大成果培育(2015CGPY03)
陈静(1976-),女,山东兖州人,山东省花生研究所副研究员,博士,主要从事花生遗传育种研究。
*通讯作者:张智猛(1963-),博士,研究员,主要从事花生逆境生理研究。E-mail: qinhdao@163.com 石程仁(1985-) ,硕士,助理研究员,从事花生经济管理及技术推广工作。E-mail: shchre@foxmail.com

