1株H6N6亚型禽流感病毒分子遗传特性分析
祁思敏,宋勇春,崔仑标,焦永军,祁贤
1. 南京大学生命科学学院,南京 210093; 2. 江苏省疾病预防控制中心,南京 210009; 3. 浙江省中医药大学药学院,杭州 310053
·论著·
1株H6N6亚型禽流感病毒分子遗传特性分析
祁思敏3,宋勇春1,崔仑标2,焦永军2,祁贤2
1. 南京大学生命科学学院,南京 210093; 2. 江苏省疾病预防控制中心,南京 210009; 3. 浙江省中医药大学药学院,杭州 310053
本研究采用无特定病原体(specific pathogen free,SPF)鸡胚,从某活禽市场环境中分离出1株H6N6亚型禽流感病毒(A/environment/Zhenjiang/zj18/2013,en/zj18)。通过二代测序技术进行全基因组测序,通过BLASTn 进行同源性检索,并采用MEGA5.0软件构建系统发生树。基因进化树分析表明,分离株en/zj18的所有8个基因节段(PB2、PB1、PA、HA、NP、NA、M和NS)均与近年来中国华东地区流行的H6N6亚型禽流感病毒的相应基因位于同一进化分支,与参考株的核苷酸同源性达96.7%~99.6%。分离株en/zj18的HA蛋白裂解位点为PQIETR↓GL,是低致病性禽流感病毒的分子特征。HA蛋白上关键受体结合位点190和228位(按H3亚型的HA蛋白序列排序)氨基酸分别是E和G,理论上更易与α2,3-半乳糖苷唾液酸受体结合。结果提示,需加强活禽市场禽流感病毒的持续监测,从而为有效应对禽流感病毒对公共卫生的持续威胁提供科学依据。
禽流感病毒;H6N6亚型;病毒分离;基因组测序
甲型流感病毒(influenza A virus)属正黏病毒科,基因组由8个节段的单股负链RNA组成。RNA可编码的蛋白包括RNA聚合酶(PB1、PB2和PA)、血凝素(hemagglutinin,HA)、神经氨酸酶(neuraminidase,NA)、核蛋白(nuclear protein,NP)、基质蛋白(M1和M2)、非结构蛋白(NS1、NS2/NEP、PB1-F2等)。HA和NA是病毒粒子的两个重要表面糖蛋白,根据其抗原性差异,甲型流感病毒可分为不同亚型。作为甲型流感病毒的主要自然储存宿主,野生水禽中已发现16种HA亚型和9种NA亚型。近年来,在中美洲蝙蝠体内发现新的HA亚型(H17和H18)和NA亚型(N10和N11)[1]。
突破种属障碍实现跨种感染或传播,是甲型流感病毒进化的一个显著特点。由于宿主限制性因素的制约,禽流感病毒跨种感染人的概率很低。但自1996年H5N1亚型禽流感病毒在东亚出现以来,人感染禽流感病毒的病例频繁发生,禽流感成为持续引人注目的重大公共卫生事件。目前报道能感染人的禽流感病毒主要有H5、H9、H7和H10等亚型[2-5]。
2013年,中国台湾地区首次报道1例人感染H6N1亚型禽流感病毒[6-7],表明H6亚型禽流感病毒能跨种感染人,对人类健康和公共卫生的潜在威胁不容忽视。2013年,本研究从江苏省镇江市某活禽市场的环境样本中分离到1株H6N6亚型禽流感病毒,在全基因组测序基础上对其基因起源和遗传特征进行了分析。
1 材料与方法
1.1 材料
1.1.1试剂病毒RNA提取试剂盒购自德国Qiagen公司;ExTaq聚合酶、dNTP、DL2000 DNA Marker、RNA酶抑制剂(ribonuclease inhibitor,RANsin)和随机引物购自宝生物工程(大连)有限公司; Nextera XT DNA Sample Preparation Kit及Illumina MiSeq测序系统购自美国Illumina公司;Qubit®dsDNA HS Assay Kit购自美国Invitrogen公司;High Sensitivity DNA Assay Kit购自美国Agilent Technologies公司。
1.1.2引物根据甲型流感病毒基因片段的核苷酸保守序列设计病毒通用引物UFLUA-IF和UFLUA-IR用于扩增病毒的全基因组,反转录引物为Uni12(M)[8],由南京金斯瑞生物科技有限公司合成。
1.2 方法
1.2.1病毒分离2013年在江苏省镇江市某菜市场活禽销售摊位,用病毒采样拭子擦拭笼具表面,置于采样液中。将标本进行常规无菌处理,取200 μL接种9~11日龄无特定病原体(specific pathogen free,SPF)鸡胚,37 ℃培养72 h,每天观察鸡胚生长情况。上述所有操作均在生物安全三级(biosafety level 3,BSL-3)实验室内完成。
1.2.2血凝试验用pH 7.4的磷酸盐缓冲液(phosphate buffered saline,PBS)配制1%鸡红细胞,按常规方法于96孔微量血凝板上进行血凝试验。
1.2.3实时反转录-聚合酶链反应(reversetranscriptase-polymerasechainreaction,RT-PCR)检测采用病毒RNA提取试剂盒(Qiagen公司)提取病毒RNA,-70 ℃保存。采用One-step RT-PCR 试剂盒(Qiagen公司),实时RT-PCR检测分离株,靶基因为甲型流感病毒的M基因。引物探针序列为:FluA-Forward:5′-GACCRATCCTGTC-ACCTCTGAC-3′;FluA-Reverse: 5′-GGGCATT-YTGGACAAAKCGTCTACG-3′;FluA-probe:5′-FAMTGCAGTCCTCGCTCACTGGGCACGBHQ-3′。反应参数:50 ℃ 30 min;95 ℃ 2 s;95 ℃ 15 s,55 ℃ 30 s,40 个循环。
1.2.4测序提取病毒基因组RNA,以U12反转录引物生成单链cDNA,采用甲型流感病毒通用引物一步法扩增流感病毒基因组8个节段。将纯化的PCR产物定量后,稀释至0.2 ng/μL,按Nextera XT DNA Sample Preparation Kit说明书构建测序文库,步骤包括DNA Tagmentation、PCR扩增、纯化、文库标化及混合。取800 μL混合液加入MiSeq 测序试剂样品孔,进行全基因序列测定,采用CLC Genomics Workbench (CLC Bio)对测序结果进行拼接整理。
1.2.5进化树构建将拼接完整的全基因组序列用基本局部比对搜索工具(Basic Local Alignment Search Tool,BLAST)进行同源性检索,MEGA5.0软件进行多序列比对,Neighbor-Joining法构建系统发生树,用Bootstrap重复抽样1 000次对进化树进行验证。
2 结果
2.1 病毒分离
将处理好的标本接种SPF鸡胚,24~72 h后接种的鸡胚全部死亡,收集尿囊液,贮存于-70 ℃备用。尿囊液血凝效价为29。实时RT-PCR检测尿囊液甲型流感病毒M基因核酸阳性。分离株命名为A/environment/Zhenjiang/zj18/2013(en/zj18)。
2.2 基因来源分析
通过二代测序平台,在标本SPF鸡胚接种物中获得分离株en/zj18的全基因组序列(8个基因的GenBank序列号为KJ938655~KJ938662)。通过BLASTn软件在线分析,初步确定分离株en/zj18为H6N6亚型。为探究病毒的基因来源,将分离株en/zj18的8个基因片段序列与参考株的核苷酸同源性进行比较分析,确认8个片段中核苷酸最大相似性的参考株(表1)。分离株en/zj18 的8个基因片段(PB2、PB1、PA、HA、NP、NA、M和NS)的核苷酸与近年来中国南方地区流行的H6N6禽流感病毒〔如A/chicken/Zhejiang/727023/2014 (H6N6)等〕的同源性高达96.7%~99.6%。
表1分离株en/zj18与参考株基因组核苷酸同源性
Tab.1Nucleotidesequenceidentitybetweenen/zj18andreferencestrainsavailableinGenBank

GenesegmentClosestinfluenzavirusrelativeNucleotideidentity(%)PB2A/chicken/Hunan/S3003/2009(H6N6)99.6PB1A/chicken/Zhejiang/727023/2014(H6N6)96.7PAA/chicken/Zhejiang/727023/2014(H6N6)98.8HAA/chicken/Zhejiang/727023/2014(H6N6)96.9NPA/chicken/Zhejiang/727023/2014(H6N6)97.9NAA/chicken/Zhejiang/727023/2014(H6N6)97.7MA/chicken/Zhejiang/727023/2014(H6N6))99.0NSA/chicken/Zhejiang/727023/2014(H6N6)98.5
为进一步明确分离株en/zj18的基因进化来源,用邻位法构建分离株en/zj18的8基因进化树。在HA和NA基因进化树上,分离株en/zj18与中国浙江流行的H6N6毒株处于一个分支(图1、图2);其他6个片段(PB2、PB1、PA、NP、M和NS)则与近年来流行的H6N6毒株处于一个进化分支(资料未显示),进一步证实分离株en/zj18属于近年来流行于中国南方地区的H6N6亚型病毒。
2.3 编码蛋白的分子特征
通过分析毒株en/zj18全基因组序列,对其编码蛋白的关键氨基酸位点进行分析,这些位点主要涉及病毒的毒力、跨种传播能力和耐药性等。结果显示,毒株en/zj18的HA基因编码566个氨基酸组成的蛋白,N端为由15个氨基酸组成的信号肽。在体内蛋白酶作用下,HA蛋白裂解为HA1(328个氨基酸)和HA2(222个氨基酸)两部分,裂解位点由1个碱性氨基酸R连接(裂解位点氨基酸序列为PQIETR↓GL),符合低致病性禽流感病毒的分子特性。理论上,HA蛋白有7个糖基化位点,其中5个位于HA1,2个位于HA2。对受体结合特性有重要影响的氨基酸位点是S137、P186、E190、Q226和G228(以H3亚型HA蛋白序列排序),理论上更易与α2,3-半乳糖苷唾液酸(sialic acid α2,3-galactose,SAα2-3Gal)受体结合。
NA蛋白由470个氨基酸组成,有8个可能的糖基化位点,没有出现氨基酸缺失。H276 和R294 位点未发生突变,表明病毒对NA抑制剂敏感。PB2蛋白的Q591、E627和D701位点未发生适应感染哺乳动物的突变。由于2个密码子突变为终止密码子,PB1-F2蛋白分别在35和88位出现断裂。NS1蛋白开放读码框(open reading frame,ORF)652~654位核苷酸突变为终止密码子,NS1蛋白包含217个氨基酸,失去C端ESEV基序。

图1分离株en/zj18的HA基因进化树分析
Fig.1ThephylogenetictreeofHAgeneofen/zj18
3 讨论
HA蛋白是甲型流感病毒的重要表面糖蛋白,通过位于HA1的受体结合位点(receptor binding site,RBS)与宿主细胞表面糖蛋白或糖脂上的唾液酸受体结合,完成受体介导的胞吞过程,病毒进入细胞内[9]。HA蛋白的唾液酸受体有两类:禽类消化道黏膜上皮细胞表达的SAα2,3Gal和人上呼吸道黏膜上皮细胞表达的SAα2,6Gal。HA受体结合特性由SAα2,3Gal亲嗜性向SAα2,6Gal亲嗜性的转变,被认为是禽流感病毒跨种感染人的一个重要前提[2,9]。病毒RBS位于HA1头部,由3个较为保守的二级结构域组成:130-loop、220-loop和190-helix,其中一些氨基酸残基在受体亲嗜性方面起关键作用,包括220-loop上的225、226和228位残基,以及190-helix上的190位残基[9]。研究发现,在不同亚型的甲型流感病毒中,这些关键位点的突变对受体结合特性的影响有差异。对H1亚型病毒来说,E190D和G225D位点的突变保证了病毒由SAα2,3Gal向SAα2,6Gal亲嗜性的转变[10]; 而位点Q226L和G228S的突变对H2和H3亚型病毒是关键[11-12]。针对H6亚型病毒,Wang等研究认为,E190V和G228S突变是H6亚型病毒获得SAα2,6Gal受体结合能力的关键,而P186L突变能减弱病毒对SAα2,3Gal 受体的结合力[13]。Ni等认为,S137N/E190V/G228S三位点组合突变是病毒结合人受体的分子基础[14]。上述4个位点的突变在中国台湾地区报道的感染人病毒株A/Taiwan/2/2013(H6N1)中均有体现[13]。但Tzarum等和Yang等研究[15-16]认为,与禽分离株一样,H6N1人分离株仍保留对禽类受体较强的结合能力;与其他亚型不同,H6亚型HA蛋白可能有其他未知位点对受体转变有贡献。由此可见,对于H6亚型来说,受体结合特性转变的分子机制尚需进一步研究。本研究中,分离株HA位点S137、P186、E190、Q226和G228均未突变,理论上对SAα2,6Gal受体的结合能力较弱,是典型的禽受体结合特征。此外,分离株en/zj18的PB2蛋白Q591、E627和D701位点未发生突变,提示该病毒在人体内复制能力较差。虽然分离株en/zj18不具备感染人的某些分子特征,但其进化变异趋势值得关注,持续的点突变也可能增强H6N6对人体的感染力,从而产生引起人类流感大流行的新毒株。
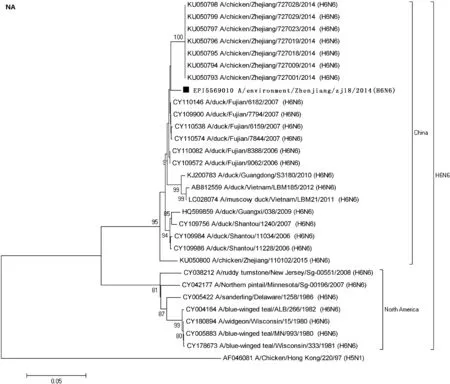
图2分离株en/zj18的NA基因进化树分析
Fig.2ThephylogenetictreeofNAgeneofen/zj18
1965年,H6亚型病毒首次从美国火鸡中分离获得,目前在全球野生水禽中广泛分布[17-18]。中国南方地区家禽养殖密度大,鸡、鸭、鹅等混养现象普遍。除H5、H9和H7亚型外,H6也是禽类中的常见流行亚型。国内研究表明,2000年以来我国H6亚型在宿主分布和亚型进化中呈动态变化[19-20]。H6亚型最初主要在家鸭中流行,然后逐渐跨种感染鹅、鸡、鹌鹑等家禽;进化过程中,H6N6逐渐替代H6N2成为优势亚型,目前出现H6N1、H6N2、H6N5、H6N6和H6N8亚型共同流行的局面。中国南方地区气候湿润,水网密布,是东亚候鸟迁徙的重要中转站,为流感病毒在野禽与家禽间的基因交换提供了便利。此外,混合养殖和活禽市场消费模式也极大促进了病毒通过基因重配的快速进化。
分离株en/zj18的HA蛋白裂解位点仅含1个碱性氨基酸,不符合高致病性禽流感病毒的分子特征。美国和中国台湾地区的研究显示,H6亚型病毒对家禽有一定的致病性,包括产蛋下降和引起上呼吸道症状等[21-22]。国内分离的H6亚型病毒大多从无临床症状的禽类中获得[19-20]。H5和H9亚型禽流感疫苗在大陆的广泛应用,能否使家禽对H6亚型病毒产生部分交叉免疫保护,目前尚不清楚。因此,国内基因型复杂的H6亚型禽流感病毒对家禽的致病性还需进一步研究。除感染人的报道外,H6亚型病毒也可感染小鼠、雪貂和豚鼠,并引发疾病[23-25]。此外,H6亚型病毒还可自然感染猪[26-27],对哺乳动物的适应力在增强[25],其公共卫生意义不容忽视。
[1] Tong S, Zhu X, Li Y, Shi M, Zhang J, Bourgeois M, Yang H, Chen X, Recuenco S, Gomez J, Chen LM, Johnson A, Tao Y, Dreyfus C, Yu W, McBride R, Carney PJ, Gilbert AT, Chang J, Guo Z, Davis CT, Paulson JC, Stevens J, Rupprecht CE, Holmes EC, Wilson IA, Donis RO. New world bats harbor diverse influenza A viruses [J]. PLoS Pathog, 2013, 9(10): e1003657.
[2] Writing Committee of the Second World Health Organization Consultation on Clinical Aspects of Human Infection with Avian Influenza A (H5N1) Virus; Abdel-Ghafar AN, Chotpitayasunondh T, Gao Z, Hayden FG, Nguyen DH, de Jong MD, Naghdaliyev A, Peiris JS, Shindo N, Soeroso S, Uyeki TM. Update on avian influenza A (H5N1) virus infection in humans [J]. N Engl J Med, 2008, 358(3): 261-273.
[3] Peiris M, Yuen KY, Leung CW, Chan KH, Ip PL, Lai RW, Orr WK, Shortridge KF. Human infection with influenza H9N2 [J]. Lancet, 1999, 354(9182): 916-917.
[4] Gao R, Cao B, Hu Y, Feng Z, Wang D, Hu W, Chen J, Jie Z, Qiu H, Xu K, Xu X, Lu H, Zhu W, Gao Z, Xiang N, Shen Y, He Z, Gu Y, Zhang Z, Yang Y, Zhao X, Zhou L, Li X, Zou S, Zhang Y, Li X, Yang L, Guo J, Dong J, Li Q, Dong L, Zhu Y, Bai T, Wang S, Hao P, Yang W, Zhang Y, Han J, Yu H, Li D, Gao GF, Wu G, Wang Y, Yuan Z, Shu Y. Human infection with a novel avian-origin influenza A (H7N9) virus [J]. N Engl J Med, 2013, 368(20): 1888-1897.
[5] Chen H, Yuan H, Gao R, Zhang J, Wang D, Xiong Y, Fan G, Yang F, Li X, Zhou J, Zou S, Yang L, Chen T, Dong L, Bo H, Zhao X, Zhang Y, Lan Y, Bai T, Dong J, Li Q, Wang S, Zhang Y, Li H, Gong T, Shi Y, Ni X, Li J, Zhou J, Fan J, Wu J, Zhou X, Hu M, Wan J, Yang W, Li D, Wu G, Feng Z, Gao GF, Wang Y, Jin Q, Liu M, Shu Y. Clinical and epidemiological characteristics of a fatal case of avian influenza A H10N8 virus infection: a descriptive study [J]. Lancet, 2014, 383(9918): 714-721.
[6] Shi W, Shi Y, Wu Y, Liu D, Gao GF. Origin and molecular characterization of the human-infecting H6N1 influenza virus in Taiwan [J]. Protein Cell, 2013, 4(11): 846-853.
[7] Yuan J, Zhang L, Kan X, Jiang L, Yang J, Guo Z, Ren Q. Origin and molecular characteristics of a novel 2013 avian influenza A (H6N1) virus causing human infection in Taiwan [J]. Clin Infect Dis, 2013, 57(9): 1367-1368.
[8] Hoffmann E, Stech J, Guan Y, Webster RG, Perez DR. Universal primer set for the full-length amplification of all influenza A viruses [J]. Arch Virol, 2001, 146(12): 2275-2289.
[9] Skehel JJ, Wiley DC. Receptor binding and membrane fusion in virus entry: the influenza hemagglutinin [J]. Annu Rev Biochem, 2000, 69: 531-569.
[10] Tumpey TM, Maines TR, van Hoeven N, Glaser L, Solórzano A, Pappas C, Cox NJ, Swayne DE, Palese P, Katz JM, García-Sastre A. A two-amino acid change in the hemagglutinin of the 1918 influenza virus abolishes transmission [J]. Science, 2007, 315(5812): 655-659.
[11] Connor RJ, Kawaoka Y, Webster RG, Paulson JC. Receptor specificity in human, avian, and equine H2 and H3 influenza virus isolates [J]. Virology, 1994, 205(1): 17-23.
[12] Matrosovich M, Tuzikov A, Bovin N, Gambaryan A, Klimov A, Castrucci MR, Donatelli I, Kawaoka Y. Early alterations of the receptor-binding properties of H1, H2, and H3 avian influenza virus hemagglutinins after their introduction into mammals [J]. J Virol, 2000, 74(18): 8502-8512.
[13] Wang F, Qi J, Bi Y, Zhang W, Wang M, Zhang B, Wang M, Liu J, Yan J, Shi Y, Gao GF. Adaptation of avian influenza A (H6N1) virus from avian to human receptor-binding preference [J]. EMBO J, 2015, 34(12): 1661-1673.
[14] Ni F, Kondrashkina E, Wang Q. Structural and functional studies of influenza virus A/H6 hemagglutinin [J]. PLoS One, 2015, 10(7): e0134576.
[15] Tzarum N, de Vries RP, Zhu X, Yu W, McBride R, Paulson JC, Wilson IA. Structure and receptor binding of the hemagglutinin from a human H6N1 influenza virus [J]. Cell Host Microbe, 2015, 17(3): 369-376.
[16] Yang H, Carney PJ, Chang JC, Villanueva JM, Stevens J. Structure and receptor binding preferences of recombinant hemagglutinins from avian and human H6 and H10 influenza A virus subtypes [J]. J Virol, 2015, 89(8): 4612-4623.
[17] Munster VJ, Baas C, Lexmond P, Waldenström J, Wallensten A, Fransson T, Rimmelzwaan GF, Beyer WE, Schutten M, Olsen B, Osterhaus AD, Fouchier RA. Spatial, temporal, and species variation in prevalence of influenza A viruses in wild migratory birds [J]. PLoS Pathog, 2007, 3(5): e61.
[18] Deng G, Tan D, Shi J, Cui P, Jiang Y, Liu L, Tian G, Kawaoka Y, Li C, Chen H. Complex reassortment of multiple subtypes of avian influenza viruses in domestic ducks at the Dongting Lake Region of China [J]. J Virol, 2013, 87(17): 9452-9462.
[19] Huang K, Zhu H, Fan X, Wang J, Cheung CL, Duan L, Hong W, Liu Y, Li L, Smith DK, Chen H, Webster RG, Webby RJ, Peiris M, Guan Y. Establishment and lineage replacement of H6 influenza viruses in domestic ducks in southern China [J]. J Virol, 2012, 86(11): 6075-6083.
[20] Zhao G, Lu X, Gu X, Zhao K, Song Q, Pan J, Xu Q, Duan Z, Peng D, Hu S, Wang X, Liu X. Molecular evolution of the H6 subtype influenza A viruses from poultry in eastern China from 2002 to 2010 [J]. Virol J, 2011. doi: 10.1186/1743-422X-8-470.
[21] Webby RJ, Woolcock PR, Krauss SL, Webster RG. Reassortment and interspecies transmission of North American H6N2 influenza viruses [J]. Virology, 2002, 295(1): 44-53.
[22] Lee MS, Chang PC, Shien JH, Cheng MC, Chen CL, Shieh HK. Genetic and pathogenic characterization of H6N1 avian influenza viruses isolated in Taiwan between 1972 and 2005 [J]. Avian Dis, 2006, 50(4): 561-571.
[23] Gillim-Ross L, Santos C, Chen Z, Aspelund A, Yang CF, Ye D, Jin H, Kemble G, Subbarao K. Avian influenza H6 viruses productively infect and cause illness in mice and ferrets [J]. J Virol, 2008, 82(21): 10854-10863.
[24] Nam JH, Kim EH, Song D, Choi YK, Kim JK, Poo H. Emergence of mammalian species-infectious and -pathogenic avian influenza H6N5 virus with no evidence of adaptation [J]. J Virol, 2011, 85(24): 13271-13277.
[25] Wang G, Deng G, Shi J, Luo W, Zhang G, Zhang Q, Liu L, Jiang Y, Li C, Sriwilaijaroen N, Hiramatsu H, Suzuki Y, Kawaoka Y, Chen H. H6 influenza viruses pose a potential threat to human health [J]. J Virol, 2014, 88(8): 3953-3964.
[26] Zhang G, Kong W, Qi W, Long LP, Cao Z, Huang L, Qi H, Cao N, Wang W, Zhao F, Ning Z, Liao M, Wan XF. Identification of an H6N6 swine influenza virus in southern China [J]. Infect Genet Evol, 2011, 11(5): 1174-1177.
[27] Zhao G, Chen C, Huang J, Wang Y, Peng D, Liu X. Characterisation of one H6N6 influenza virus isolated from swine in China [J]. Res Vet Sci, 2013, 95(2): 434-436.
s. SONG Yongchun, E-mail: songyc@nju.edu.cn; QI Xian, E-mail: qixiansyc@163.com
MolecularcharacterizationofoneH6N6subtypeavianinfluenzavirusisolatedfromalivepoultrymarketinZhenjiang
QI Simin3, SONG Yongchun1, CUI Lunbiao2, JIAO Yongjun2, QI Xian2
1.SchoolofLifeSciences,NanjingUniversity,Nanjing210093,China; 2.JiangsuProvincialCenterforDiseaseControlandPrevention,Nanjing210009,China; 3.CollegeofPharmaceuticalScience,ZhejiangUniversityofTraditionalChineseMedicine,Hangzhou310053,China
One H6N6 subtype avian influenza virus 〔A/environment/Zhenjiang/zj18/2013 (en/zj18)〕 was isolated from a live poultry market in Zhenjiang in an epidemiological surveillance and was subjected to genome sequencing and further analysis. Next-generation sequencing (NGS) by Illumina MiSeq Platform was used to obtain the complete genome sequence from the isolated virus. MEGA5.0 software was used to align the eight fragments respectively. The phylogenetic and molecular characteristics of the isolate were analyzed by Neighbor-Joining method. BLAST searches demonstrated that the 8 genes (PB2, PB1, PA, HA, NP, NA, M and NS) had 96.7%-99.6% nucleotide identity with the ones on the H6N6 subtype reference viruses circulating in southeast China. The enzyme cleavage site on HA was PQIETR↓GL, which was the typical characteristic of the low pathogenic avian influenza virus (LPAIV). The key HA receptor binding sites were E190 and G228, indicating a preference binding for α2-3-linked sialic acids. The results suggest that it is necessary to strengthen the continuous surveillance of avian influenza A viruses.
Avian influenza virus; H6N6 subtype; Virus isolation; Genome sequencing
国家自然科学基金(31370079),江苏省自然科学基金(BK2013145)
宋勇春,祁贤
2017-02-10)

