棉花BZR基因家族的全基因组鉴定及表达分析
郭新磊,路普,王园园,蔡小彦,王星星,周忠丽,王玉红,王春英,王坤波*,刘方*
棉花BZR基因家族的全基因组鉴定及表达分析
郭新磊1#,路普1#,王园园2,蔡小彦1,王星星1,周忠丽1,王玉红1,王春英1,王坤波1*,刘方1*
(1.中国农业科学院棉花研究所/棉花生物学国家重点实验室,河南安阳455000;2.河南科技学院/现代生物育种河南省协同创新中心,河南新乡453003)
【目的】油菜素唑抗性因子 (Brassinazole resistant transcription factor,BZR)是植物油菜素内酯(Brassinosteroid,BR)信号通路中的1类重要转录因子,通过调控相关基因的表达,参与植物生长发育过程。本研究旨在探索棉花中BZR基因的数量及其功能。【方法】利用生物信息学方法,对雷蒙德氏棉、亚洲棉和陆地棉中的BZR基因进行全基因组鉴定及表达模式分析。【结果】雷蒙德氏棉、亚洲棉和陆地棉中分别有7、7和14个BZR基因;通过序列特征分析和系统发育分析可将棉花BZR基因分为2组(a组和b组)。内含子/外显子结构分析发现:大部分BZR基因具有2个外显子,a组成员中均具有较长的内含子序列,b组成员的内含子序列较短,同组BZR成员具有相似的基因结构。基因复制分析表明:片段复制是棉花BZR基因家族扩增的主要方式,棉花BZR基因在进化过程中经历了严格的纯化选择。转录组数据分析表明:14个GhBZR基因在陆地棉的7个组织中特异表达,大多数GhBZR基因在茎、叶、花瓣和雌蕊中高表达;许多GhBZR基因的表达受非生物胁迫诱导。【结论】推测GhBZR基因可能参与棉花抗逆及纤维发育。本研究结果可为棉花BZR基因的生物学功能研究提供信息。
棉花;BZR基因;系统发育分析;表达分析
Abstract:[Objective]Brassinazole resistant transcription factors(BZR)play important roles in Brassinosteroid(BR)signal transduction in plants.The BZR gene family isinvolved in plant developmental processes by regulating the expression of related genes.The aim of this study isto characterize the number,evolutionary characteristics,and biological function of BZR genes in cotton.[Method]We conducted genome-wide identification and expression analysis of the BZR gene family in Gossypium raimondii,G.arboreum,and G.hirsutum.[Result]In this study,seven,seven,and 14 BZR genes were identified from G.raimondii,G.arboreum,and G.hirsutum,respectively.These BZR genes were grouped into two groups,a and b,based on their amino acid sequences and phylogenetic analysis.Gene structure analysis showed that most BZR genes contain two exons.Additionally,BZR genes in"a"group had a longer intron than those in"b"group;within the same group,BZR genes had a similar gene structure.Analysis of gene duplication revealed that segmental duplications played crucial roles in BZR gene family expansion,with cotton BZR genes experiencing purifying selection during evolution.Transcriptome data analysis showed that 14 GhBZR s had differing expression patterns in different organs;most GhBZR s were highly expressed in stem,leaf,petal,and pistil tissue,and were induced or repressed by abiotic stress.[Conclusion]These results suggest that GhBZR genes may beinvolved in stressresistance and fiber development in cotton.Furthermore,they provide information for further analysis aimed at uncovering the biological functions of BZR genes in cotton.
Keywords:cotton;BZR genes;phylogenetic analysis;expression analysis
油菜素内酯(Brassinosteroid,BR)是植物中的1类多羟基类固醇,参与发育和多种生理过程,包括细胞伸长、细胞分裂、衰老、维管分化、繁殖和光形态建成[1-4]。此外,BR作为应激缓解剂广泛分布于植物中,保护植物免受各种环境胁迫的伤害,包括热激、低温、干旱、盐碱、除草剂药害和病虫害[5-8]。油菜素唑抗性因子(Brassinazole resistant transcription factor,BZR)是植物特有的转录因子,广泛分布于植物各种组织中,如根、茎、叶和花[9],此类转录因子通过调控响应BR基因的表达在BR信号通路中发挥重要作用[10-11]。
研究发现BZR蛋白质具有高度保守的N-末端结构域,该结构域在体外和体内均具有DNA结合活性[10-11]。BZR1的DNA结合结构域由BZR基因的第1个外显子编码,是BZR家族蛋白最保守的区域,因此命名为BZR结构域[11]。拟南芥中的BZR1和BZR2/BR不敏感蛋白1-MES抑制子 1(BR insensitive 1-EMS-suppressor 1,BES1)是BR信号途径中研究比较透彻的2个转录因子。在BZR1和BES1的N末端均有1个非典型的螺旋 -环 -螺旋(Basic helix-loop-helix,bHLH)的DNA结合基序,它们分别能与E-box(CANNTG)和 BRRE(CGTGT/CG)元件结合[10-11]。这些蛋白质的中部含有22~24个氨基酸的糖原合成酶激酶3(Glycogen synthase kinase,GSK3)家族磷酸化位点。另外,部分BZR成员还含有富含脯氨酸(Proline,P)、谷氨酸(Glutamic acid,E)、丝氨 酸(Serine,S)和苏氨酸(Threonine,T)的序列(PEST基序),该基序控制蛋白质的稳定性[10]。
BR信号通路中,BR结合并激活受体激酶BR 不敏感蛋白 1(BRinsensitive1,BRI1),激活的BRI1与BRI1相关受体激酶(BRI1-associated receptor kinase,BAK1)相互作用,激活 BR 信号[12-13],而激活的BRI1又可磷酸化BR信号激酶(BR-signaling kinase1,BSK1), 磷酸化的 BSK1激活磷酸酶 BRI1抑制子 (BRI1 suppressor 1,BSU1),进而使BR感应蛋白(Brassinosteroid-insensitive 2,BIN2)激酶脱磷酸化而失活。失活的BIN2对BZR1转录因子的抑制作用消失,BZR1转录因子在蛋白磷酸酶2A (Protein phosphatase 2A,PP2A)作用下去磷酸化(活化),并在核内富集[14-18]。活化的BZR1和BES1能够与下游基因启动子特定区域结合,激活转录,从而调节BR靶基因的表达[10-11]。在植物BR信号通路中,BZR1和BZR2/BES1调控不同性状基因的表达,参与调控植物的生长发育。Jiang等[19]发现BZR基因通过调控MINISEED3和HAIKU2等基因的表达控制种子的大小和形状。BZR转录因子通过与光敏色素相互作用因子(Phytochrome-interacting factor,PIF)家族基因及DELLA基因互作,参与植物细胞生长和光形态建成[20-22]。此外,异位表达拟南芥BZR1-1D基因可提高番茄中类胡萝卜素、可溶性糖和抗坏血酸含量,从而提升番茄果实的品质[23]。
通过鉴定和研究BZR的目标基因,揭示了BR信号通路中BZR转录因子的调控机制和转录网络[24-27]。许多转录因子家族除调控生长发育外,还参与胁迫应答。但目前有关BZR转录因子与抗逆性关系的研究还极少有报道。鉴于亚洲棉、雷蒙德氏棉和陆地棉基因组测序已完成[28-31],本研究对棉花BZR基因家族成员进行了鉴定,对BZR转录因子的理化特性、相关基因结构、系统进化关系、基因复制方式进行了分析,并对陆地棉中BZR基因的表达模式进行了研究,为进一步克隆和研究棉花BZR基因奠定了基础。
1 材料与方法
1.1 棉花BZR蛋白家族成员的鉴定与分类注释
雷 蒙 德 氏 棉 (Gossypium raimondii,D5,JGI_v2.1)、亚洲棉(G.arboreum,A2,BGI_v1.0)和陆地棉(G.hirsutum,AD1,NBI_v1.1)的基因组数据下载自Cottongen数据库(https://www.cottongen.org/)。 从 Pfam 数据库(http://pfam.sanger.ac.uk/)下载BZR蛋白保守结构域的隐马尔科夫模型文件(PF05687.10)[32],然后以 PF05687.10 为探针,利用HMMER 3.0软件搜索这3个棉种蛋白数据库,参数为默认值。同时,以拟南芥(Arabidopsis thaliana)[10]、水稻(Oryza sativa)[33]、芜菁(Brassica rapa)[34]的BZR家族蛋白序列为探针,利用本地BLASTP程序,搜索这3个棉种蛋白数据库(E值为10-2)。这2种方法所得蛋白序列即为候选的棉花BZR蛋白家族成员。将这些候选蛋白序列分别提交到在线软件Pfam、SMART(http://smart.embl-heidelberg.de)[35]和 National Center for Biotechnology Information(NCBI)保守结构域数据库(Conserved domain database,CDD)(http://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi)[36],验证是否具有BZR保守结构域,若存在BZR保守结构域则属于BZR家族。目前BZR基因家族尚无系统的命名准则,本研究依据BZR家族基因成员所在的染色体以及在染色体上的位置进行命名。
1.2 序列分析与进化分析
根据BZR家族基因的gff注释文件信息,利用基因结构显示系统GSDS(http://gsds.cbi.pku.e-du.cn/)[37]分析基因结构特征,绘制基因结构图。利用在线工具 ProtComp 9.0(http://www.softberry.com/berry.phtml?topic=protcomppl&group=programs&subgroup=proloc)预测BZR家族基因的亚细胞定位。利用在线工具ProtParam(http://web.expasy.org/protparam/)分析蛋白质的相对分子质量(Mr)和等电点(pI)。 利用 DNAMAN 软件对BZR家族蛋白进行多重序列比对,并展示保守位点。此外,利用在线程序MEME(http://meme.sdsc.edu/meme/)鉴定棉花BZR家族基因的保守基序。
采用ClustalW方法对拟南芥、芜菁、水稻、雷蒙德氏棉、亚洲棉和陆地棉的BZR家族蛋白进行多重序列比对,利用MEGA 6.0软件通过Neighbor-joining法构建系统进化树。参数设置:模式采用 “Poisson correction”,数据缺失设置为“Pairwise deletion”, 校验参数为 Bootstrap=1000。
1.3 染色体定位与基因复制分析
根据棉花基因组测序的基因注释信息,利用MapChart 2.2软件绘制棉花BZR基因的染色体定位图,并确定其复制方式:在同一染色体上,如果2个同源BZR基因相邻或间隔1个基因,可认为是通过串联复制产生;利用MCScanX[38-39]软件查找棉花基因组内染色体间的共线性区域(E值设为1×10-10),位于共线性区域的BZR基因看作是通过片段复制产生。使用在线工具PAL2NAL (http://www.bork.embl.de/pal2nal/,是基于PAML软件中的codeml程序)[40]计算复制基因的碱基非同义替换率(Ka)和同义替换率(Ks)及Ka/Ks,若Ka/Ks>1,则认为有正选择效应;如果Ka/Ks=1,则认为存在中性选择;如果 Ka/Ks<1,则认为有纯化选择作用。 利用公式 T=Ks/r(r=7×10-9)估算复制事件发生的时间。
1.4 上游顺式作用元件分析
根据雷蒙德氏棉、亚洲棉和陆地棉基因组数据,利用perl程序(由本实验室编写)提取各BZR基因序列转录起始位点ATG上游1500 bp序列,在PlantCARE数据库(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)中检索,从而鉴定基因上游区域可能存在的顺式作用元件。
1.5 BZR家族基因的表达分析
从 NCBI的 Sequence Read Archive(SRA)数据库下载陆地棉TM-1的转录组数据,登录号为PRJNA248163(http://www.ncbi.nlm.nih.gov/sra/?term=PRJNA248163)[31]。采用有参基因组的转录组分析方法进行分析,利用公式log2(FPKM)标准化FPKM (Fragmentsper kilobase of transcript per million fragments mapped),导入软件Mev 4.8绘制陆地棉BZR基因表达量热图。
2 结果与分析
2.1 棉花BZR家族基因的鉴定与命名
通过BLASTP和HMMER搜索获得的候选氨基酸序列,用Pfam、SMART、CDD工具预测保守结构域,剔除不含完整BZR结构域的冗余序列,从雷蒙德氏棉、亚洲棉和陆地棉中分别鉴定出7、7和14个候选基因。目前该基因家族没有标准的命名准则,本研究依据各基因所在的染色体 (从第1号染色体到第13号染色体再到scaffold)及在染色体上的位置进行命名 (表1),即GrBZR1~GrBZR7,GaBZR1~GaBZR7 和 Gh-BZR1~GhBZR14。亚细胞定位预测发现:雷蒙德氏棉和亚洲棉中的BZR基因均定位于细胞核,
而陆地棉中BZR基因都定位于细胞质和细胞核。

表1 棉花BZR 基因家族信息Table 1 The information of BZR gene family in cotton
蛋白质理化性质分析结果表明:棉花BZR基因编码蛋白质长度为313~349个氨基酸,相对分子质量为33 980.0~37 711.2,理论等电点为7.60~9.12,为碱性。
2.2 序列分析与进化分析
根据棉花BZR基因系统发育分析,棉花BZR基因可分为2组,即a组和b组,分别包含16和12个成员,且各组内成员间具有相似的基因结构,编码相似的结构域(图1)。利用GSDS软件对BZR基因的结构分析发现,绝大多数BZR基因具有2个外显子,仅4个成员具有3个外显子,且a组成员的内含子序列较b组的长。
利用在线工具MEME鉴定棉花BZR家族的保守基序,结果表明:棉花BZR家族中存在10个 motif,所有成员都包含 motif 1、2、7(图 1 和图2)。此外,a组成员还具有 motif 3、5、6,部分成员具有motif 10,部分具有motif 8;b组成员均具有motif 4、9、10。 其中,motif 1 是 N 端的 bHLH 结构,motif 2和motif 7是C端结构,motif 6是丝氨酸磷酸化位点,motif 8是PEST基序,motif 4则包含了丝氨酸磷酸化位点和PEST基序。
利用拟南芥、水稻、芜菁、雷蒙德氏棉、亚洲棉和陆地棉中的BZR蛋白构建系统进化树 (图3),可将BZR家族分为2组,与上面系统发育分析的分组一致,推测这2组成员的不同结构可能赋予了它们不同的功能。其中,第1组中棉花具有16个成员,拟南芥仅有2个成员,在第2组中棉花含有12个成员,拟南芥有4个成员,与拟南芥相比,第1组BZR的编码基因在棉花中发生了倍增。在进化树中,水稻BZR与其他物种BZR亲缘关系较远,同为十字花科的拟南芥和芜菁聚类,3个棉种 (雷蒙德氏棉、亚洲棉和陆地棉)聚类,表明BZR在近缘物种中相似度更高,并可能具有相似的功能。
2.3 基因定位与基因复制
雷蒙德氏棉的7个GrBZR基因分布在6条染色体上;亚洲棉的7个GaBZR基因分布在7条染色体上;陆地棉的14个GhBZR基因,有13个和1个基因分别定位于11条染色体和scaffold1329_A06上。串联复制和片段复制是基因家族扩增的主要方式[41],利用MCScanX对雷蒙德氏棉和亚洲棉基因组中的基因复制事件进行检测表明:雷蒙德氏棉和亚洲棉中分别至少有5个和4个BZR基因经历了片段复制,且这2个棉种均不存在BZR基因的串联复制,表明片段复制是棉花BZR基因家族扩增的主要方式。经分析发现所有Ka/Ks均小于1,最大仅为0.154 8(表2),表明棉花BZR基因在进化过程中经历了严格的纯化选择作用,暗示复制基因在进化中较为保守,结构比较稳定,功能具有一致性。
2.4 BZR基因上游顺式元件分析
对雷蒙德氏棉、亚洲棉和陆地棉BZR基因转录起始位点上游区域1500 bp序列的顺式作用元件分析结果显示,棉花BZR基因上游存在许多植物激素响应元件、环境胁迫响应元件和植物生长发育相关元件(附表1,印刷版省略,参见本刊网站)。BZR基因上游发现的植物激素响应元件包括 ABRE、TGA、ERE、GARE、TATC、P-box、TCA、TGACG 和 CGTCA。 其 中 ,ABRE、TGA、ERE和TCA分别响应脱落酸、生长素、乙烯和水杨酸。GARE、TATC和P-box是赤霉素响应元件,TGACG和CGTCA均响应茉莉酸甲酯(Methyl jasmonate,MeJA)。环境胁迫响应元件有7 种,包括 HSE、LTR、MBS、ARE、TC-rich repeats、Box-W1和GCmotif。HSE、LTR和MBS分别是热、低温和干旱响应元件,ARE和GCmotif是水淹或缺氧响应元件,Box-W1是真菌响应元件,TC-rich repeats是防御和胁迫响应元件。生长发育相关元件有:Skn-1_motif、circadian、GCN4_motif、CAT-box和CCGTCC-box,它们和植物的胚乳发育、分生组织发育及生物节律有关。
2.5 陆地棉BZR基因的表达分析
对GhBZR基因在棉花根、茎、叶、花瓣、雌蕊、雄蕊和副萼中的表达模式分析表明,14个GhBZR基因在这7个组织中都能够特异性表达(图5-A)。其中,GhBZR3和GhBZR9具有相似的表达特征,在7个组织中都高表达,暗示这2个基因可能具有相似的功能。相反,GhBZR4和GhBZR10在这些组织表达水平较低。GhBZR11、GhBZR12、GhBZR2、GhBZR5、GhBZR8 和 GhBZR14具有明显的组织特异性表达特征,它们在茎、叶和雌蕊中高水平表达,而在其他组织中低水平表达。此外,大多数BZR基因在茎、叶、花瓣和雌蕊中都高表达,暗示了BZR基因可能参与棉花茎、叶和雌蕊的发育。

图1 棉花BZR 家族基因的结构与编码的结构域Fig. 1 The gene structures and encoding domains of BZR genes in cotton

图2 雷蒙德氏棉、亚洲棉和陆地棉(TM-1)BZR蛋白的多序列比对分析Fig.2 Alignment of amino acid sequences of BZR proteins in G.raimondii,G.arboreum and G.hirsutum

图3 棉花BZR与其他植物BZR的系统进化分析Fig.3 Phylogenetic analysis of BZRs in cotton and other plants

图4 BZR家族基因在棉花染色体上的分布及复制分析Fig.4 Chromosomal distribution and duplication analysis of BZR genes in cotton
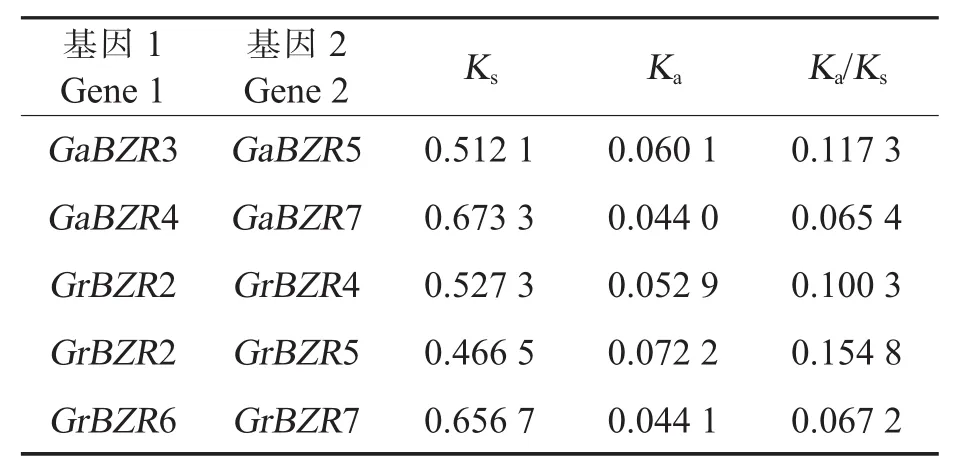
表2 雷蒙德氏棉和亚洲棉中发生复制的BZR基因的碱基替换率Table 2 Synonymous and nonsynonymous substitution rates for the duplication events in GaBZR and GrBZR genes
BZR是1类植物特有的转录因子,在BR信号通路中发挥了重要作用,但有关棉花BZR转录因子抗逆性的研究未见报道。因此,本研究对陆地棉BZR基因在PEG-6000、盐、低温和高温处理下的表达情况进行了研究(图5-B)。PEG和盐处理后BZR基因表现相似的表达特征,一半左右的BZR基因在处理1 h后上调表达,这部分基因全都是a组基因;而另一半基因则在处理6 h后上调,该部分基因主要是b组基因。在高温处理下,大部分基因在处理6 h后上调表达;而在低温下,大部分基因在处理3 h后呈现下调表达。

图5 14个陆地棉BZR家族基因表达模式Fig.5 Expression patterns of 14 GhBZR genes
3 讨论
BZR家族作为BR信号通路中的1类重要转录因子[10-11],其家族基因成员的鉴定对棉花BZR基因的生物学功能分析及BR信号通路的研究具有重要意义。亚洲棉、雷蒙德氏棉和陆地棉基因组测序的完成,为棉花BZR基因家族的预测和分析提供了便利。拟南芥和芜菁分别有6个和15个BZR基因[10,34]。本研究分别从雷蒙德氏棉、亚洲棉和陆地棉中鉴定出7、7和14个BZR转录因子。棉花BZR基因在二倍体棉中数目相等,结构相似(如GrBZR1和GaBZR2),表明BZR基因在雷蒙德氏棉和亚洲棉间高度保守。通过系统发育分析及基因结构分析发现:棉花BZR基因可分为2组,同组基因具有相似的基因结构,且外显子数目几乎一致;a组基因内含子序列较b组的长。结构域分析及序列比对发现,组内BZR基因编码的蛋白具有高度保守的结构域,2组基因编码的蛋白具有不同的保守结构域,推测这2组基因可能具有不同的功能。
棉花BZR基因上游存在环境胁迫响应元件,包括LTR低温响应元件、MBS干旱响应元件等,以适应环境变化。通过构建系统进化树,得到芜菁BZR基因与雷蒙德氏棉、亚洲棉和陆地棉BZR基因的进化关系,有助于推测BZR基因在棉花中的功能。芜菁的2个自交系Chiifu和Kenshin经冷害、冻害胁迫后,BrBZR1-1、BrBES1-3、BrBEH4、BrBEH6、BrBEH7 和 BrBEH8的表达量发生显著变化,经冷害处理的耐低温自交系Chiifu中BrBZR1-1和BrBES1-3的表达量要比冻害处理高,而自交系Kenshin中BrBEH8在2种胁迫下表达量均较高[42],芜菁的BZR基因家族中BrBEH基因在冷害或冻害下的诱导性比BrBZR1、BrBES1更强[43]。芜菁的BrBEH基因与本研究的棉花a组GhBZR基因同源性较高,暗示a组GhBZR基因可能在棉花抵御冷害、冻害胁迫中起重要作用。Sun采用ChIP-chip的方法鉴定出许多BZR的靶标基因,其中包括参与抗旱耐盐的基因RD26、ERD14和STZ以及耐冷相关基因CBF2[25],暗示BZR可能在植物抗旱耐盐等过程具有重要作用。本研究中,GhBZR6、GhBZR1和GhBZR7在PEG及盐处理后的1、3和6 h均上调表达,它们可能参与棉花抗逆过程。
前人研究发现BZR转录因子参与细胞伸长和植物的光形态建成[20-22]。研究发现,无BR信号时,磷酸化的BZR1和14-3-3蛋白结合而滞留在细胞质内,当有BR信号时,BZR1去磷酸化,与14-3-3蛋白分离,进入细胞核中聚集,从而调控植物的生长发育[10,44-45]。目前已从陆地棉中得到8个14-3-3基因,有6个在棉花纤维中优势表达,其中5个的编码产物与BZR蛋白存在互作[46]。周颖等从陆地棉中鉴定出1个BZR转录因子,该转录因子通过保守基序与14-3-3蛋白相互作用,参与棉纤维细胞的伸长发育[45]。本研究发现大多数GhBZR基因在茎、叶、花瓣和雌蕊中都高表达,由此推测BZR转录因子在棉花纤维发育中发挥重要作用。
4 结论
以二倍体雷蒙德氏棉、亚洲棉和四倍体陆地棉为研究对象,分别鉴定得到7、7和14个BZR基因,分析了其位置、结构及编码产物的等电点、相对分子质量和结构域,并通过系统进化分析将棉花BZR家族基因分为2组。一些GhBZR基因的表达具有组织特异性并响应非生物胁迫,推测GhBZR基因可能参与棉花抗逆及纤维发育。棉花BZR基因的全基因组鉴定及表达分析,为挖掘棉花BZR基因潜在的功能奠定了基础。
[1]Clouse SD,Langford M,Mcmorris T C.A brassinosteroid-insensitivemutant in Arabidopsis thaliana exhibits multiple defects in growth and development[J].Plant Physiology,1996,111(3):671-678.
[2]Li J,Chory J.A putative leucine-rich repeat receptor kinase involved in brassinosteroid signal transduction[J].Cell,1997,90(5):929-938.
[3]Ye Qianqian,Zhu Wenjiao,Li Lei,et al.Brassinosteroidscontrol male fertility by regulating the expression of key genes involved in Arabidopsis anther and pollen development[J].Proceedings of the National Academy of Sciencesof the United Statesof America,2010,107(13):6100-6105.
[4]Clouse S D.Brassinosteroid signal transduction:From receptor kinase activation to transcriptional networks regulating plant development[J].The Plant Cell,2011,23(4):1219-1230.
[5]Khripach V,Zhabinskii V,Groot A D.Twenty years of brassinosteroids:steroidal plant hormones warrant better crops for the XXIcentury[J].Annalsof Botany,2000,86(3):441-447.
[6]Bajguz A,Hayat S.Effects of brassinosteroids on the plant responses to environmental stresses[J].Plant Physiology and Biochemistry,2009,47(1):1-8.
[7]Campos M L,de Almeida M,Rossi M L,et al.Brassinosteroids interact negatively with jasmonates in the formation of antiherbivory traitsin tomato[J].Journal of Experimental Botany,2009,60(15):4347-4361.
[8]Yang D H,Hettenhausen C,Baldwin IT,et al.BAK1 regulates the accumulation of jasmonic acid and the levels of trypsin proteinase inhibitors in Nicotiana attenuata's responses to herbivory[J].Journal of Experimental Botany,2010,62(2):641-652.
[9]Clouse SD,Sasse JM.Brassinosteroids:essential regulators of plant growth and development[J].Annual Review of Plant Physiology and Plant Molecular Biology,1998,49:427-451.
[10]Yin Y,Vafeados D,Tao Y,et al.A new class of transcription factors mediates brassinosteroid-regulated gene expression in Arabidopsis[J].Cell,2005,120:249-259.
[11]He JX,Gendron JM,Sun Y,et al.BZR1 is atranscriptional repressor with dual roles in brassinosteroid homeostasis and growth responses[J].Science,2005,307(5715):1634-1638.
[12]Li J,Wen J,Lease K A,et al.BAK1,an Arabidopsis LRR receptor-like protein kinase,interacts with BRI1 and modulates brassinosteroid signalling[J].Cell,2002,110(2):213-222.
[13]Nam K H,Li J.BRI1/BAK1,a receptor kinase pair mediating brassinosteroid signaling[J].Cell,2002,110(2):203-212.
[14]He J X,Gendron J M,Yang Y,et al.The GSK3-like kinase BIN2 phosphorylates and destabilizes BZR1,a positive regulator of the brassinosteroid signaling pathway in Arabidopsis[J].Proceedings of the National Academy of Sciencesof the United Statesof America,2002,99(15):10185-10190.
[15]Li J M,Nam K H,Vafeados D,et al.BIN2,a new brassinosteroid-insensitive locusin Arabidopsis[J/OL].Plant Physiology,2001,127(1):14[2016-12-20].http://www.plantphysiol.org/cgi/reprint/127/14.pdf.DOI:10.1104/pp.127.1.14.
[16]Yin Yanhai,Wang Zhiyong,Mora-Garcia S,et al.BES1 accumulates in the nucleus in response to brassinosteroids to regulate gene expression and promote stem elongation[J].Cell,2002,109(2):181-191.
[17]Zhao J,Peng P,Schmitz R J,et al.Two putative BIN2 substrates are nuclear components of brassinosteroid signaling[J].Plant Physiology,2002,130(3):1221-1229.
[18]Tang Wenqiang,Yuan Min,Wang Ruiju,et al.PP2A activates brassinosteroid-responsive gene expression and plant growth by dephosphorylating BZR1[J].Nature Cell Biology,2011,13(2):124-131.
[19]Jiang Wenbo,Huang Huiya,Hu Yuwei,et al.Brassinosteroid regulates seed size and shapein Arabidopsis[J].Plant Physiology,2013,162(4):1965-1977.
[20]Gallego-BartoloméJ,Minguet E G,Grau-Enguix F,et al.Molecular mechanism for the interaction between gibberellin and brassinosteroid signaling pathways in Arabidopsis[J].Proceedings of the National Academy of Sciences of the United Statesof America,2012,109(33):13446-13451.
[21]Li Qianfeng,Wang Chunming,Jiang Lei,et al.An interaction between BZR1 and DELLAs mediates direct signaling crosstalk between brassinosteroidsand gibberellinsin Arabidopsis[J/OL].Science Signaling,2012,5(244):ra72(2012-10-02)[2016-11-15].http://stke.sciencemag.org/content/5/244/ra72/tab-pdf.DOI:10.1126/scisignal.2002908.
[22]Oh E,Zhu JY,Wang Z Y,et al.Interaction between BZR1 and PIF4 integrates brassinosteroid and environmental responses[J].Nature Cell Biology,2012,14(8):802-809.
[23]Liu Lihong,Jia Chengguo,Zhang Min,et al.Ectopic expression of a BZR1-1D transcription factor in brassinosteroid signalling enhancescarotenoid accumulation and fruit quality attributes in tomato[J].Plant Biotechnology Journal,2014,12(1):105-115.
[24]Luo Xiaomin,Lin Wenhui,Zhu Shengwei,et al.Integration of light-and brassinosteroid-signaling pathways by a GATA transcription factor in Arabidopsis[J].Developmental Cell,2010,19(6):872-883.
[25]Sun Yu,Fan Xiying,Cao Dongmei,et al.Integration of brassinosteroid signal transduction with the transcription network for plant growth regulation in Arabidopsis[J].Developmental Cell,2010,19(5):765-777.
[26]Yu Xiaofei,Li Li,li Lei,et al.Modulation of brassinosteroid-regulated gene expression by Jumonjidomain containing proteins ELF6 and REF6 in Arabidopsis[J].Proceedings of the National Academy of Sciencesof the United Statesof America,2008,105(21):7618-7623.
[27]Yu Xiaofei,Li Lei,Zola J,et al.A brassinosteroid transcriptional network revealed by genome-wideidentification of BES1 target genes in Arabidopsis thaliana[J].The Plant Journal,2011,65(4):634-646.
[28]Paterson A H,Wendel J F,Gundlach H,et al.Repeated polyploidization of Gossypium genomes and the evolution of spinnablecotton fibres[J].Nature,2012,492(7429):423-427.
[29]Wang Kunbo,Wang Zhiwen,Li Fuguang,et al.The draft genomeof adiploid cotton Gossypium raimondii[J].Nature Genetics,2012,44(10):1098-1103.
[30]Li Fuguang,Fan Guangyi,Wang Kunbo,et al.Genome sequence of thecultivated cotton Gossypium arboreum[J].Nature Genetics,2014,46(6):567-572.
[31]Zhang Tianzhen,Hu Yan,Jiang Wenkai,et al.Sequencing of allotetraploid cotton (Gossypium hirsutum L.acc.TM-1)providesa resource for fiber improvement[J].Nature Biotechnology,2015,33(5):531-537.
[32]Finn R D,Mistry J,Tate J,et al.Pfam:The protein family's database[J].Nucleic Acids Research,2010,38(Database):D211-D222.
[33]Bai Mingyi,Zhang Liying,Gampala SS,et al.Functions of Os-BZR1 and 14-3-3 proteins in brassinosteroid signaling in rice[J].Proceedings of the National Academy of Sciencesof the U-nited Statesof America,2007,104(34):13839-13844.
[34]Saha G,Park JI,Jung H J,et al.Molecular characterization of BZR transcription factor family and abiotic stress induced expression profiling in Brassicarapa[J].Plant Physiology and Biochemistry,2015,92(4):92-104.
[35]Letunic I,Doerks T,Bork P.SMART:Recent updates,new developmentsand statusin 2015[J].Nucleic Acids Research,2015,43(Database):D302-D305.
[36]Marchler-Bauer A,Anderson J B,Cherukuri P F,et al.CDD:NCBI'sconserved domain database[J].Nucleic Acids Research,2015,43(Database):D222-D226.
[37]Hu Bo,Jin Jinpu,Guo Anyuan,et al.GSDS 2.0:An upgraded gene feature visualization server[J].Bioinformatics,2015,31(8):1296-1297.
[38]Tang Haibao,Bowers J E,Wang Xiyin,et al.Synteny and collinearity in plant genomes[J].Science,2008,320(5875):486-488.
[39]Wang Yupeng,Tang Haibao,DeBarry JD,et al.MCScanX:A toolkit for detection and evolutionary analysis of gene synteny and collinearity[J/OL].Nucleic Acids Research,2012,40(7):e49(2012-01-04)[2016-11-15].https://doi.org/10.1093/nar/gkr1293.[40]Suyama M,Torrents D,Bork P.PAL2NAL:Robust conversion of protein sequence alignments into the corresponding codon alignments[J].Nucleic Acids Research,2006,34(Web Server issue):609-612.
[41]Cannon S B,Mitra A,Baumgarten A,et al.The roles of segmental and tandem gene duplication in the evolution of large gene families in Arabidopsis thaliana[J/OL].BMCPlant Biology,2004,4(1):10(2004-06-01)[2016-11-15].https://bmcplantbiol.biomedcentral.com/articles/10.1186/1471-2229-4-10.DOI:10.1186/1471-2229-4-10.
[42]Jung H J,Dong Xiangshu,Park J I,et al.Genome-wide transcriptome analysis of two contrasting Brassica rapa doubled haploid lines under cold-stresses using Br135K oligomeric chip[J/OL].PLoSONE,2014,9(8):e106069(2014-08-28)[2016-11-15].https://doi.org/10.1371/journal.pone.0106069.
[43]Dong X,Im S B,Lim Y P,et al.Comparative transcriptome profiling of freezing stress responsiveness in two contrasting Chinese cabbage genotypes,Chiifu and Kenshin[J].Genes and Genomics,2014,36(2):215-227.
[44]Ryu H,Kim K,Cho H,et al.Nucleocytoplasmic shuttling of BZR1 mediated by phosphorylation is essential in Arabidopsis brassinosteroid signaling[J].Plant Cell,2007,19(9):2749-2762.
[45]周颖.14-3-3蛋白参与BR信号调控及其在棉花(Gossypium hirsutum)纤维发育中的功能研究[D].武汉:华中师范大学,2014.Zhou Ying.Functional analysis of 14-3-3 proteins that are involved in regulating BRsignaling in cotton(Gossypium hirsutum)fiber[D].Wuhan:Central China Normal University,2014.
[46]李冰樱.棉花MAD S和BZR基因鉴定及转基因棉花表型分析[D].武汉:华中师范大学,2012.
Li Bingying.Characterization of cotton MADS and BZR Genes and phenotypic analysisof transgenic cotton plants[D].Wuhan:Central China Normal University,2012. ●
Genome-Wide Identification and Expression Analysis of the Gene Family Encoding Brassinazole Resistant Transcription Factors in Cotton
Guo Xinlei1#,Lu Pu1#,Wang Yuanyuan2,Cai Xiaoyan1,Wang Xingxing1,Zhou Zhongli1,Wang Yuhong1,Wang Chunying1,Wang Kunbo1*,Liu Fang1*
(1.Institute of Cotton Research of the Chinese Academy of Agricultural Sciences/State Key Laboratory of Cotton Biology,Anyang,Henan 455000,China;2.Henan Institute of Science and Technology/Collaborative Innovation Center of Modern Biological Breeding,Henan Province,Xinxiang,Henan 453003,China)
S562.01
A
1002-7807(2017)05-415-13
10.11963/1002-7807.gxllf.20170830
2016-12-29 第一作者简介:郭新磊(1989―),男,硕士,guoxlcaas@163.com;路普(1992―),男,硕士研究生,lupu1992@163.com;#同等贡献。*通信作者:刘方,liufcri@163.com;王坤波,wkbcri@163.com
国家自然科学基金(31671745);国家重点研发计划(2016YFD0101401,2016YFD0100203)

