基于寡核苷酸探针套painting的小麦“中国春”非整倍体高清核型及应用
王丹蕊 杜 培 裴自友 庄丽芳,* 亓增军,*南京农业大学作物遗传与种质创新国家重点实验室, 江苏南京 0095;山西省农业科学院作物科学研究所, 山西太原 030030
作物遗传育种·种质资源·分子遗传学
基于寡核苷酸探针套painting的小麦“中国春”非整倍体高清核型及应用
王丹蕊1杜 培1裴自友2庄丽芳1,*亓增军1,*1南京农业大学作物遗传与种质创新国家重点实验室, 江苏南京 210095;2山西省农业科学院作物科学研究所, 山西太原 030030
基于寡核苷酸探针套painting的染色体鉴定技术简单、经济和高效, 可以促进小麦品种及亲缘物种染色体识别和变异体鉴定, 提高染色体工程效率。我们前期开发了寡核苷酸探针套, 包含 pAs1-1、pAs1-3、AFA-4、(GAA)10和pSc119.2-1共5个探针。本研究通过一次荧光原位杂交(FISH), 对源于17个非整倍体的18份材料分析发现, 其中14个染色体组成正确, 可以清晰识别相应的缺体、四体和端体。还构建了基于寡核苷酸探针套涂染的、能准确识别3个基因组和7个部分同源群染色体的高清核型, 发现4个非整倍体发生变异, 其中从N5BT5D中鉴定出一个可能的小片段相互易位系T6AS·6AL-6DL和T6DS·6DL-6AL。进一步对7个地方品种、10个栽培品种(系)和1个人工合成小麦分析, 发现15条染色体存在多态性, 涉及6条B组(除4B)、5条A组(除1A和3A)和4条D组(1D、2D、4D和7D)染色体, 可以清晰识别我国小麦生产上广泛应用的 3种易位类型(T1RS·1BL、T6VS·6AL及相互易位 T1RS·7DL和T7DS·1BL), 省去了基因组原位杂交(GISH)程序。另外, 对5个亲缘物种分析发现, 该探针套可以识别栽培一粒小麦、硬粒小麦Langdon、荆州黑麦、长穗偃麦草(2n=2x=14)全部和中间偃麦草30条染色体, 并构建了这5个物种的核型。本研究结果证实该寡核苷酸探针套可以有效用于小麦及亲缘物种染色体鉴定, 高清晰的中国春非整倍体核型为小麦染色体工程提供了参考标准。
寡核苷酸探针套; 染色体painting; 染色体多样性; 小麦易位系; 非整倍体
Abstract:Oligonucleotide (oligo hereafter) multiplex-based chromosome painting facilitates chromosome identification of both wheat cultivars and its relatives in a simple, easy and high efficient way. In this study, an oligo multiplex containing oligos pAs1-1,pAs1-3, AFA-4, (GAA)10, and pSc119.2-1 developed earlier was used for chromosome painting of 18 accessions from 17 Chinese Spring (CS) aneuploids. The high resolution karyotypes allowed to clearly distinguish individual wheat chromosomes. Fourteen aneuploids had the expected chromosome constitutions whereas the other four had chromosome variations including one with a possible small segmental reciprocal translocation T6AS·6AL-6DL and T6DS·6DL-6AL occurred in N5BT5D. The following analysis on eight landraces, nine cultivars (lines), and one synthetic hexaploid wheat, observed karyotype diversities from 15 chromosomes including six B- (except for 4B), five A- (except for 1A and 3A), and four D-genome (1D, 2D, 4D, and 7D) chromosomes. The three widely-used translocations in China, i.e. T1BL·1RS, T6AL·6VS and the reciprocal translocation T1RS·7DL and T7DS·1BL, were clearly detected after only once fluorescence in situ hybridization (FISH) using the oligo multiplex and without genomic in situ hybridization (GISH). This oligo multiplex also produced rich signals in all chromosomes of Triticummonococum, rye cultivar “Jingzhouheimai”, durum wheat “Langdon”, and Thinopyrum elongatum, and 30 chromosomes of Thinopyrum intermedium. The karyotypes of these five species were thus developed. These results indicate that oligo multiplex-based chromosome painting will play active roles on chromosome identifying, and provide a reference for the standard karyotypes of CS aneuploids.
Keywords:Oligonucleotide multiplex probe; Chromosome painting; Chromosome diversity; Wheat translocations; Aneuploids
小麦是世界上最重要的粮食作物之一, 据联合国粮农组织统计, 2014年全球小麦收获面积约2.204亿公顷, 总产约7.29亿吨(http://www.fao.org/faostat/en/#data/QC)。然而, 随着全球气候变化, 小麦生产受到更多的胁迫[1-2], 不断发掘和利用亲缘物种中的有益基因是拓宽栽培小麦遗传基础和应对这些挑战的重要途径。染色体工程技术的发展为定向转移和利用外源基因和加快育种进程等提供了许多新工具[3]。
染色体工程在小麦育种中取得了显著的成效, 不但育成一些重要的异源易位系在生产上大量应用[4-5],而且创造出一批宝贵的遗传材料广泛应用于遗传学、分子生物学和基因组学研究[6-8]。小麦染色体工程从过去关注较多的黑麦(Secale cereale)[4,9-11]、簇毛麦(Haynaldia villosa)[5,12-13]、长穗偃麦草(Thinopyrum elongatum)[7,14]和中间偃麦草(Thinopyrum intermedium)[15-16]等拓展到更多的物种,例如大麦(Hordeum vulgare)[17]、冰草(Agropyron cristatum)[18-19]、百萨偃麦草(Thinopyrum bessarabicum)[20-21]及山羊草属(Aegilops)[22-24], 甚至探讨关系更远的超远缘杂交[25]。随着染色体诱变技术发展和成熟, 目前在短时间内可以诱致大量染色体变异体, 特别是小片段顶端或插入易位、缺失、小染色体、倒位等[26-28]。虽然多种方法诱导的很多变异体发生在非部分同源染色体之间, 生产上直接利用价值不大, 但这些材料为外源目标基因发掘与定位、染色体物理作图、染色体基因组学和生物学研究提供了重要的遗传工具[7-8,12,18-19,21,29], 同时, 包含目标基因(簇)的小片段易位或渐渗系, 未来有望通过同源重组系统定向转移进优良栽培小麦背景[3], 成为优异基因聚合的重要基因元件[30], 大大减少整条外源染色体或区段导入因遗传累赘造成的负效应。但是, 受染色体鉴定技术的局限, 很多非补偿性的变异体难以得到准确鉴定, 因此限制了这些材料的进一步利用, 开发更有效的染色体鉴定技术具有很大的实用价值。
单链寡核苷酸是一类短的 DNA和 RNA序列,可有效侵入染色体双链DNA而发展成为新的FISH探针, 具有开发简单、容易修饰、成本低、效率高等特点[31-32], 目前已经发展成为生物染色体鉴定的新一代工具, 广泛用于核型分析、染色体多态性分析、染色体易位鉴定、目标染色体或区段特异追踪、部分同源染色体鉴定、物种识别、染色体比较作图等研究[31-44]。本课题组从2010年开始相关研究, 目前成功开发出一批高效寡核苷酸探针(套)用于小麦、黑麦、百萨偃麦草、玉米等染色体研究[38,43-45], 其中,Du等[43]报道的4个寡核苷酸探针套, 通过一次荧光原位杂交(FISH)可以清晰识别全部小麦染色体, 与基因组原位杂交(GISH)结合, 可以识别小麦异染色体系, 为小麦染色体工程提供了简单、经济和高效的工具。为进一步验证上述寡核苷酸探针套及其应用潜力, 本研究利用简化的寡核苷酸探针套#4分析系列中国春非整倍体, 旨在建立准确识别 7个部分同源群染色体的中国春非整倍体高清核型, 并分析我国地方小麦品种、栽培品种、人工合成小麦以及亲缘物种, 探讨该探针套和标准核型在识别不同品种和物种染色体多态性的应用价值, 为小麦染色体工程提供新的工具和参考核型。
1 材料与方法
1.1 植物材料
为源于17个中国春非整倍体的18份材料、7个地方品种、10个栽培品种、1个人工合成小麦和5个小麦亲缘植物(表1)。其中, 中国春非整倍体转引自江苏省农业科学院杨学明研究员和吴纪中研究员, 栽培一粒小麦引自南京农业大学马正强教授, 硬粒小麦和人工合成小麦引自日本鸟取大学 Hisashi Tsujimoto教授。
1.2 细胞遗传分析
1.2.1 根尖有丝分裂中期染色体制片 参照Dolezel等[46-47]描述的方法, 稍作改动。将种子置垫两层滤纸的培养皿, 加水至浸没种子, 在 24℃恒温箱发芽; 种子露白后, 倒掉多余水分, 24℃继续培养至根长约1.2~1.5 cm; 将0.2 μmol L-1甲基氨草磷溶液(amiprophos-methyl, 简称 APM, 溶剂为丙酮)倒入培养皿浸没种子, 24℃继续培养 2 h, 然后倒掉APM, 用清水冲洗种子3次后, 剪取2~3条根置1.5 mL离心管(离心管盖子打好孔), 加1~2滴蒸馏水, 保证根尖湿润; 将离心管放入笑气罐中充笑气, 至压力达0.8~1.2 MPa, 处理1.5 h后取出, 置90%乙酸中在冰上固定10 min后用于制片。

表1 本研究所用的植物材料Table 1 Materials used in this study
制片时, 取出根尖, 在 45%醋酸中解离 3~5 min,切掉根冠, 再切取根尖分生区组织于载玻片, 滴一滴45%醋酸, 盖上盖玻片, 轻敲使细胞分散, 用酒精灯外焰均匀烘烤待雾气散开后压片; 在相差显微镜下观察,将染色体形态好、数目完整和分散均匀的制片放入-70℃冰箱冷冻过夜, 然后揭去盖玻片, 在100%无水乙醇中脱水30 min以上, 取出气干后用于原位杂交。
1.2.2 寡核苷酸FISH 寡核苷酸探针套包含5个寡核苷酸探针(表2), 根据Du等[43]报道的探针套#4略作调整而成, 分别包含 TAMRA修饰的pAs1-1、pAs1-3、AFA-4和 FAM 修饰的 pSc119.2-1和(GAA)10。在同时进行寡核苷酸探针套FISH和GISH时, 改用TAMRA修饰的pSc119.2-1, 以与基因组的绿色信号区别。
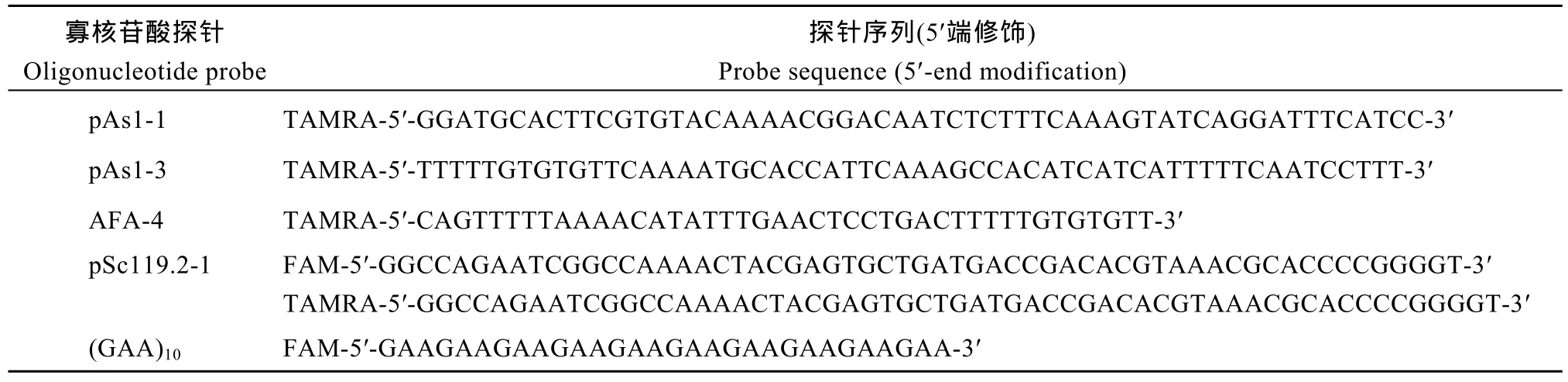
表2 本文所用的寡核苷酸探针Table 2 Oligonucleotide probes used in this study
参考Zhuang等[11]描述的FISH程序, 杂交液体系为 16.6 μL, 包括 7.5 μL dFA, 1.5 μL 20× SSC, 0.5 μL Salmon sperm DNA (10 mg mL-1), pAs1-1 (10 pmol μL-1), pAs1-3 (10 pmol μL-1), AFA-4 (10 pmol μL-1)和 pSc119.2-1 (10 pmol μL-1)各 1 μL, 0.1 μL(GAA)10(10 pmol μL-1), 3 μL dextran sulfate (50%)。在鉴定小麦–黑麦和小麦–簇毛麦易位系时, 除单独采用寡核苷酸探针套分析外, 为确证易位身份还进一步将寡核苷酸探针套与基因组 DNA探针同时进行 GISH/FISH分析, 此时, 杂交液中分别增加黑麦或簇毛麦基因组DNA探针2.5 μL和辉县红基因组DNA 1.7 μL作为封组。采用缺刻平移法制备黑麦和簇毛麦基因组DNA探针, 分别利用簇毛麦和黑麦基因组 DNA为模板, 用 Fluorescein-12-dUTP标记探针。反应液体系 50 μL, 包括 5 μL 10× DNA Polymerase I buffer, 5 μL dNTP, 3~4 μg 模板 DNA, 3 μL DNA Polymerase I (3~6 U μL-1), 1.2 μL Recombinant DNase I (1∶1000), 1 μL Fluorescein-12-dUTP, 30 μL ddH2O。在冰上完成反应液配制, 然后于PCR仪16℃温育2 h, 以-20℃保存备用。
原位杂交时, 混匀杂交液, 在105℃加热块中变性13 min, 取出后迅速置–20℃冰酒精10 min以上;制片置于70%酒精 + NaOH (0.15 mol L-1)中, 24℃变性6 min, 迅速依次置于70%、95%和100%酒精,各脱水3 min, 气干; 将杂交液加在制片上, 于37℃恒温箱杂交6 h以上。
完成杂交后, 揭去盖片, 在室温条件下, 2× SSC洗8 min, 再用清水冲洗一下, 气干。将6.5 μL DAPI胶加在制片上, 盖上盖片, 吸去余胶; 在 Olympus DL72型荧光显微镜下镜检, 用 SPOT CCD (SPOT Cooled Color Digital Camera)摄取图像。分析核型时,观察每个材料 4~15个细胞, 从中选取染色体数完整、分散良好的一个细胞用于核型分析, 少数材料由于染色体重叠或不完整等原因选用 2个细胞进行核型分析。
2 结果与分析
2.1 中国春非整倍体分析与小麦高清核型
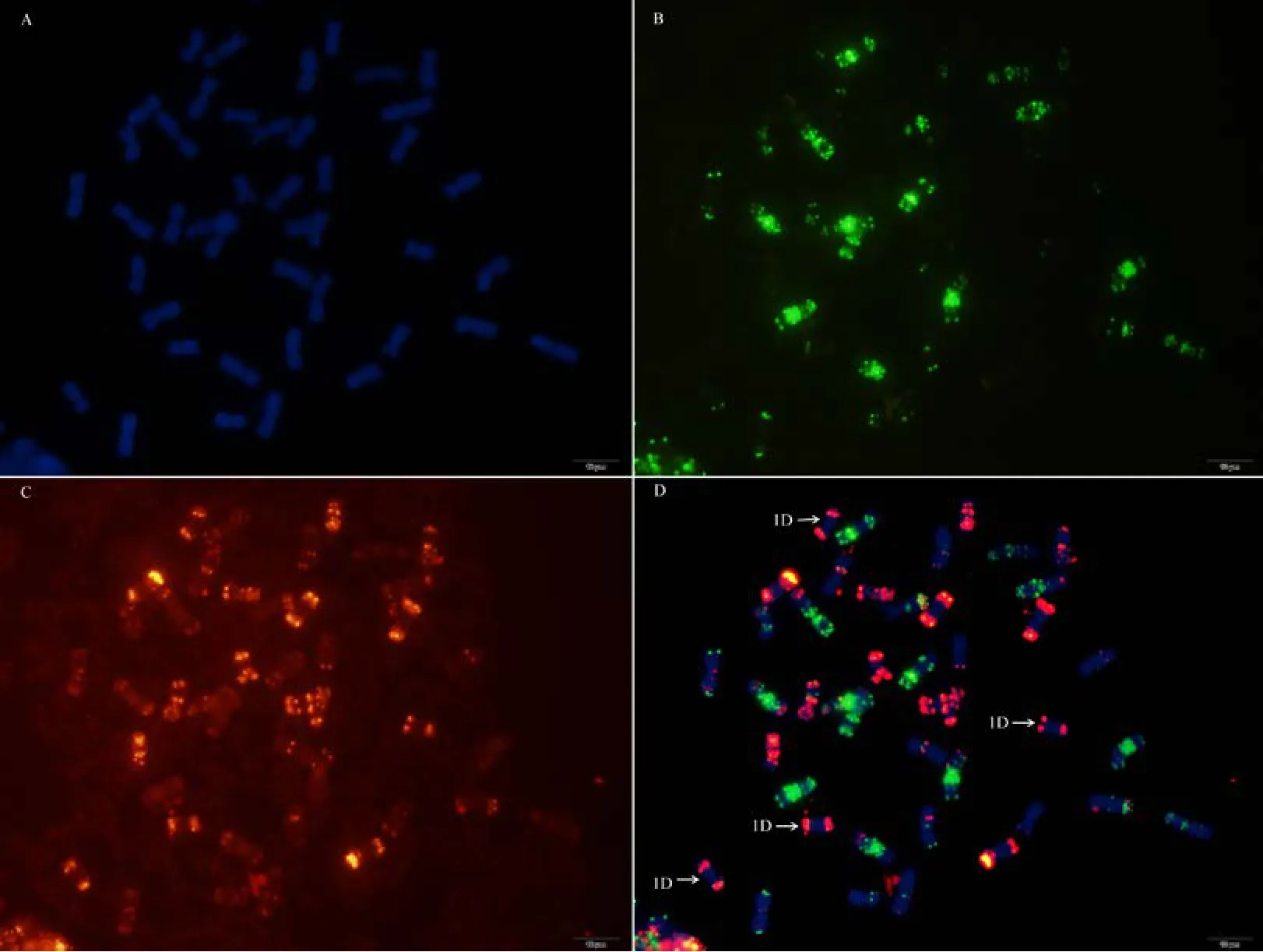
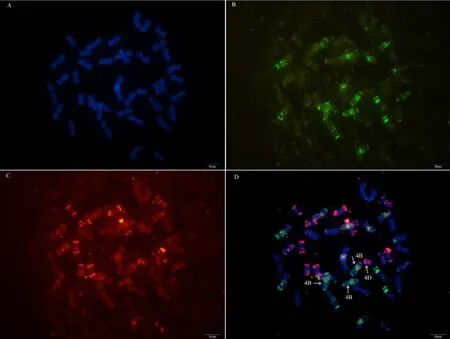
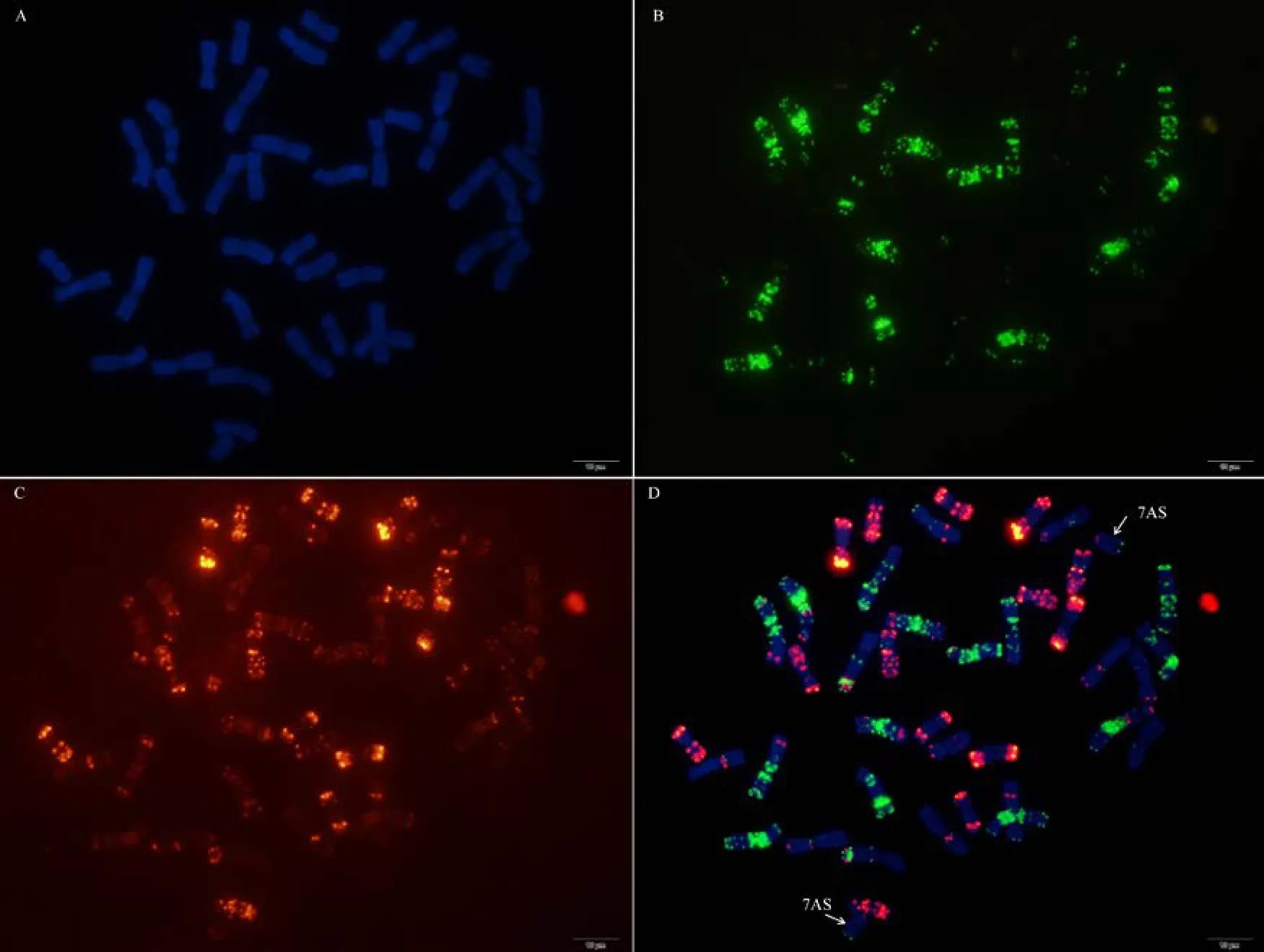
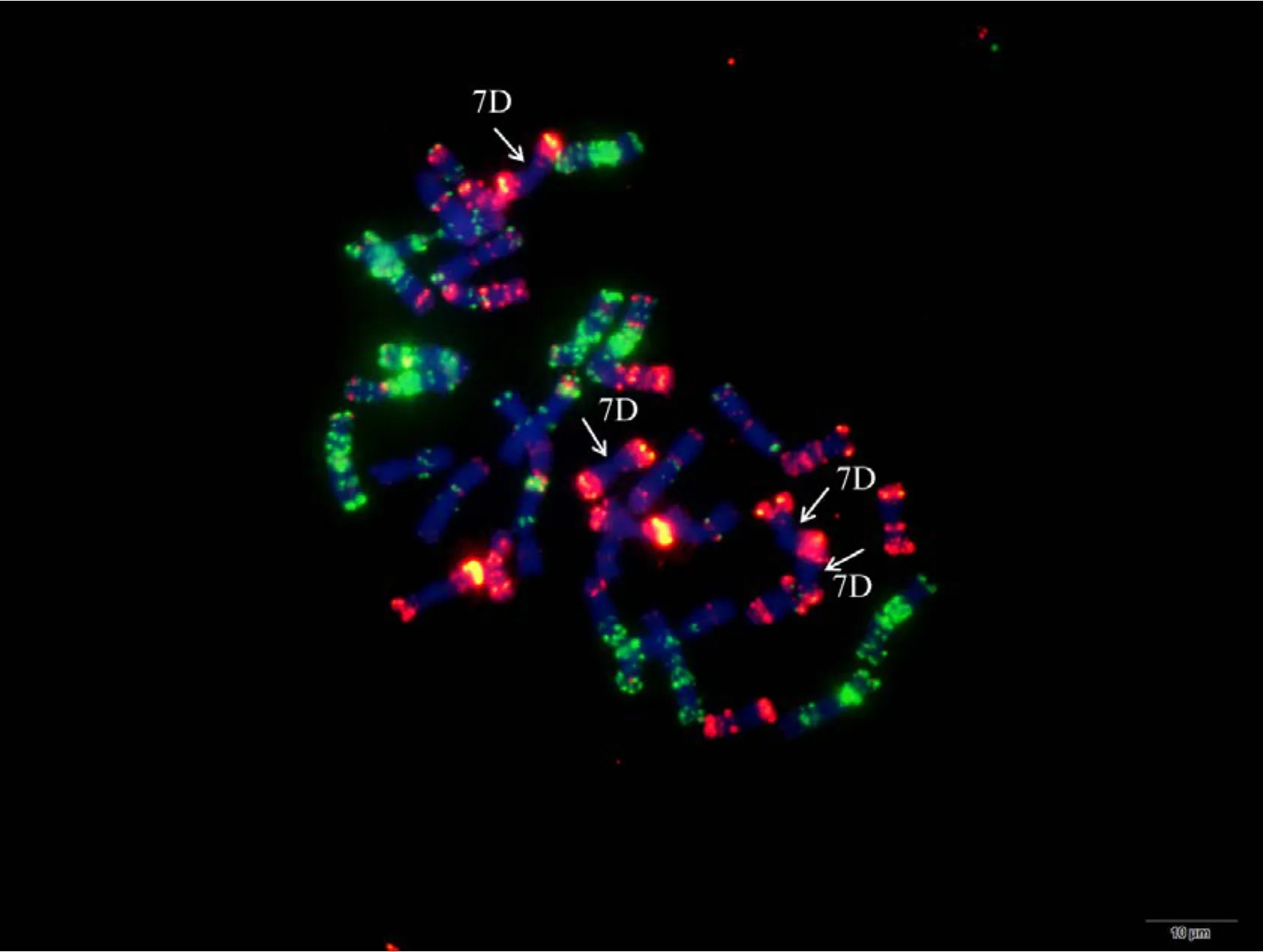
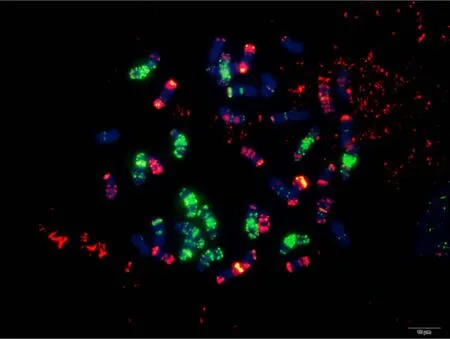
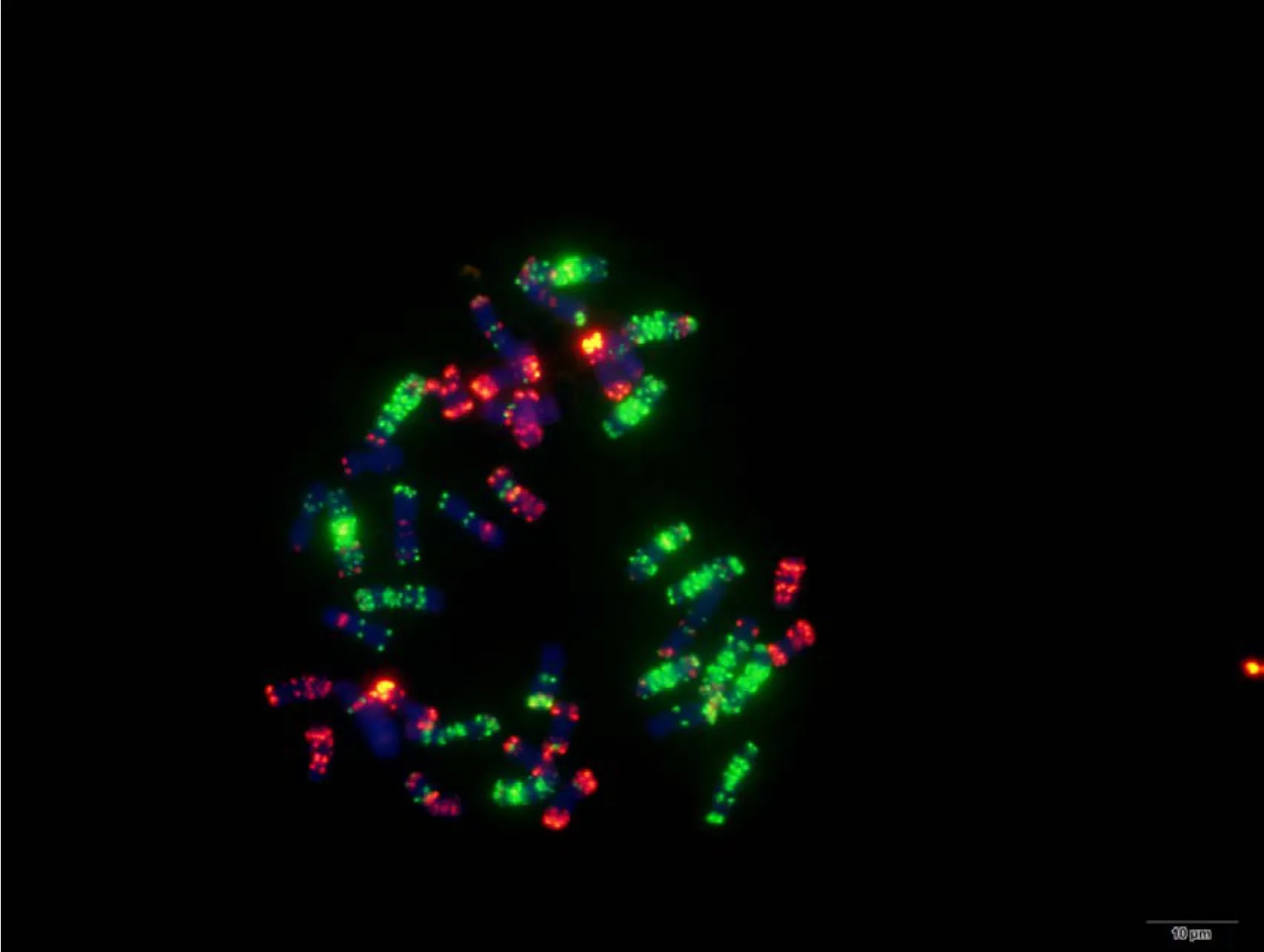
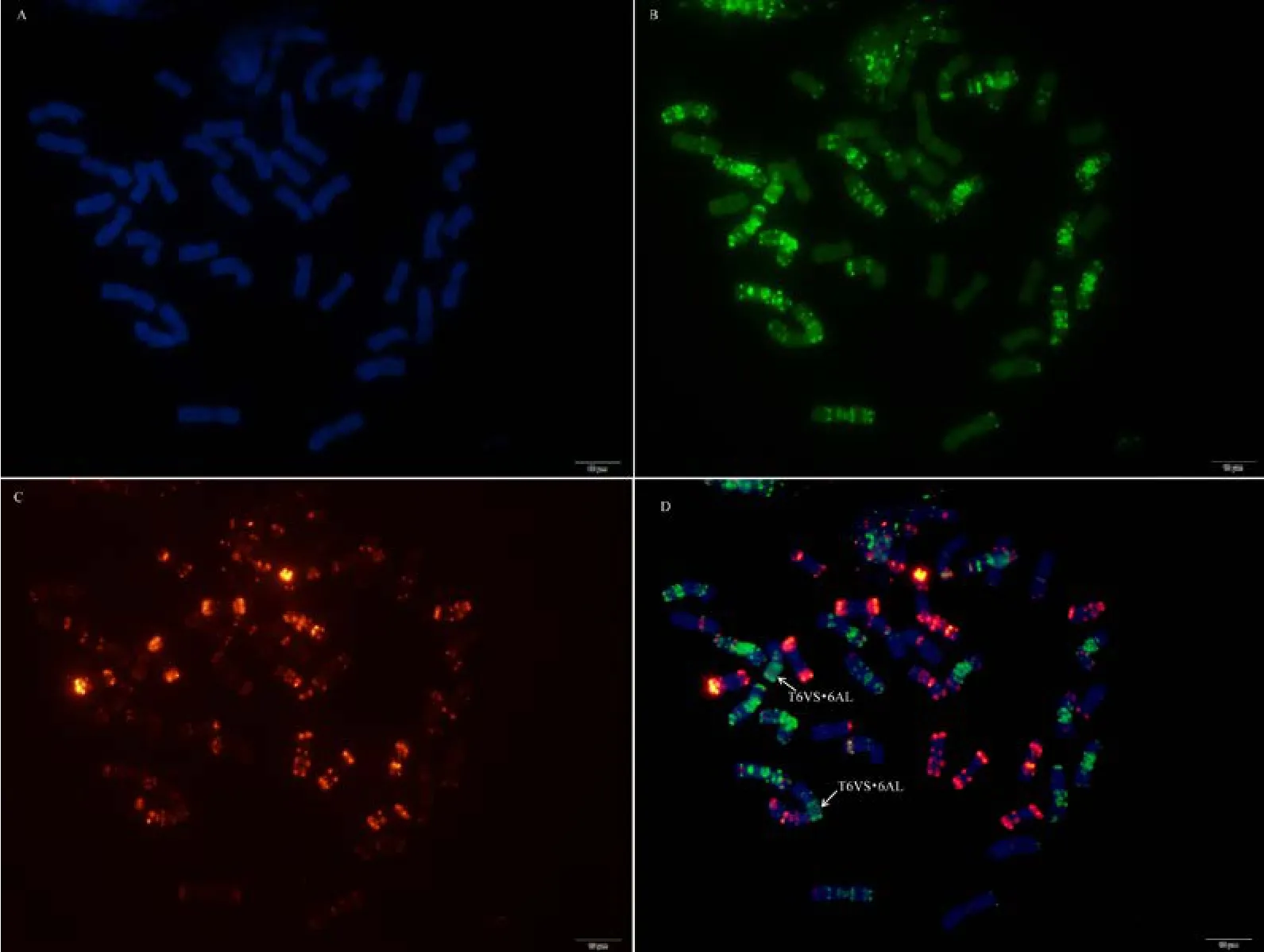
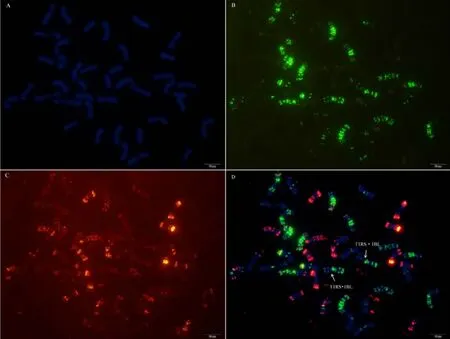
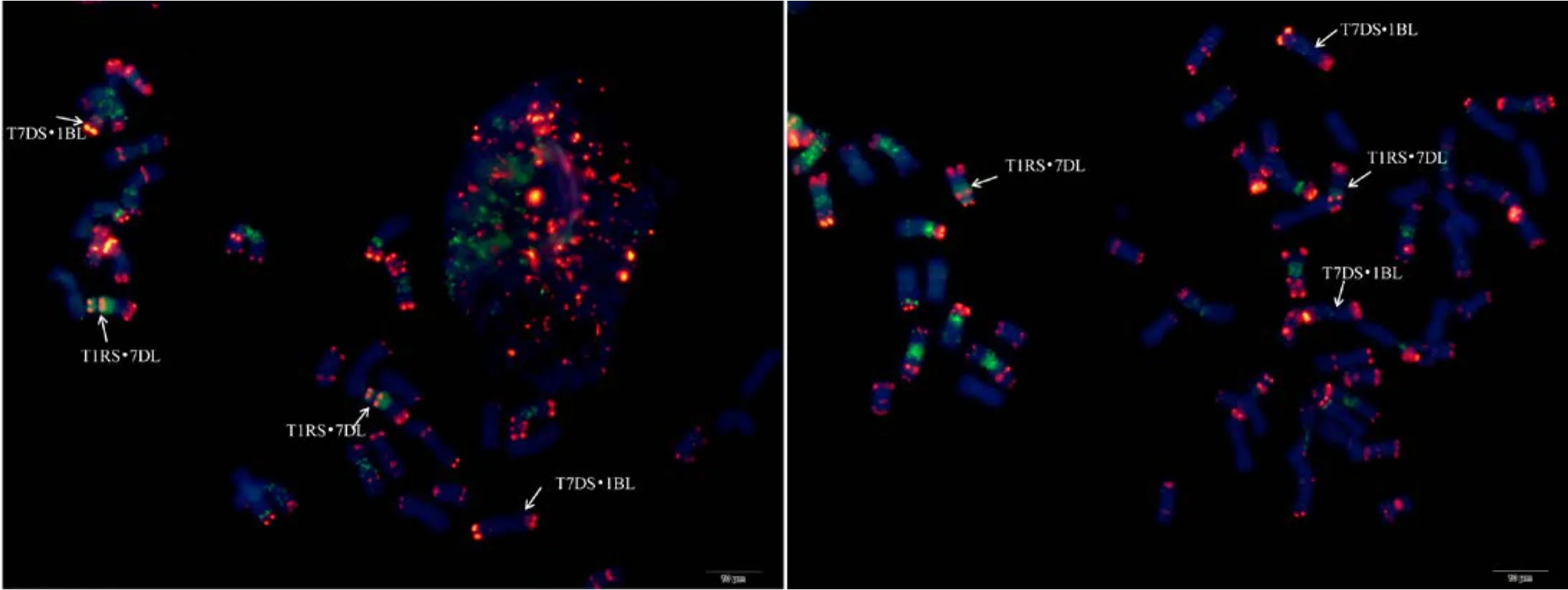
利用寡核苷酸探针套通过一次FISH分析, 对源于17个中国春非整倍体的18份材料(表1)的染色体进行了分析, 与Du等[43]报道的中国春核型比, 由于减少了红色信号探针数目, 使染色体更容易区分,其中在 B组染色体信号最丰富, 且以绿色为主, 其次为D组, 以红色为主, 而A组, 红绿均弱, 据此可以清晰识别3个基因组及全部染色体, 并构建了16个非整倍体高清核型(图 1, 图 2; 附图 1~16)。由图可见, 14个非整倍体染色体组成稳定, 包括N1BT1D、N2BT2D、N3AT3B、N3BT3A、N3DT3B、N4DT4B、N5AT5D、N6AT6D、Dt7AS、N7AT7D、N7BT7D、N7DT7B、Dt7BL (图 2; 附图 1~5, 7~9,11~15)和 N2DT2B (附图 17, 核型未排), 均可以清晰识别相应缺体、四体和端体, 特别是根据 7AS和7BL特征, 将7A和7B长、短臂准确区分; 但是, 4个非整倍体发生变异, 其中 N5BT5D没有缺少 5B,5D也仅有2条, 但发现1对小片段相互易位, 推测为 T6AS·6AL-6DL 和 T6DS·6DL-6AL, 另外, 其 7D长臂顶端也发生变化(图1-F, 图2); 对2份N4DT4B材料的鉴定结果, 其中一份鉴定准确, 而另一份则从中发现1个单株仅包含1条4D但3条4B (图1-E,图2), 明显发生了异交; 从 N6BT6A中鉴定出1个单株虽为6B缺体, 但没有预期6A四体, 而分别多了1条1D和3B, 其2A、2B、5B和7B同时表现明显的多态性(图2); N4AT4B (附图18)中没有发现4A缺体和4B四体, 为正常的整倍体类型。综合非整倍体核型特征, 绘制了中国春高清标准核型, 用于清晰识别小麦3个基因组和7个部分同源群全部染色体(图 2)。
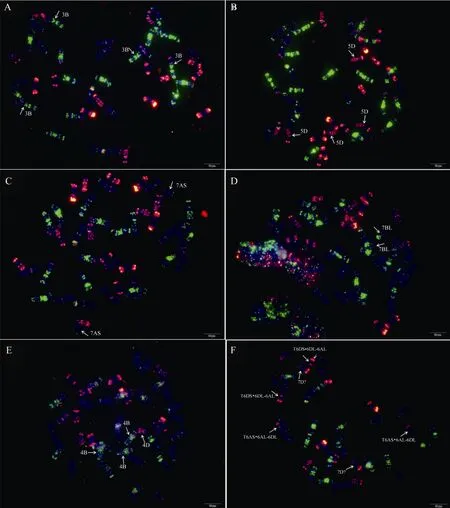
图1 中国春非整倍体寡核苷酸探针套paintingFig. 1 Chromosomes of Chinese Spring aneuploids after oligonucleotide multiplex painting
2.2 地方品种和栽培品种分析
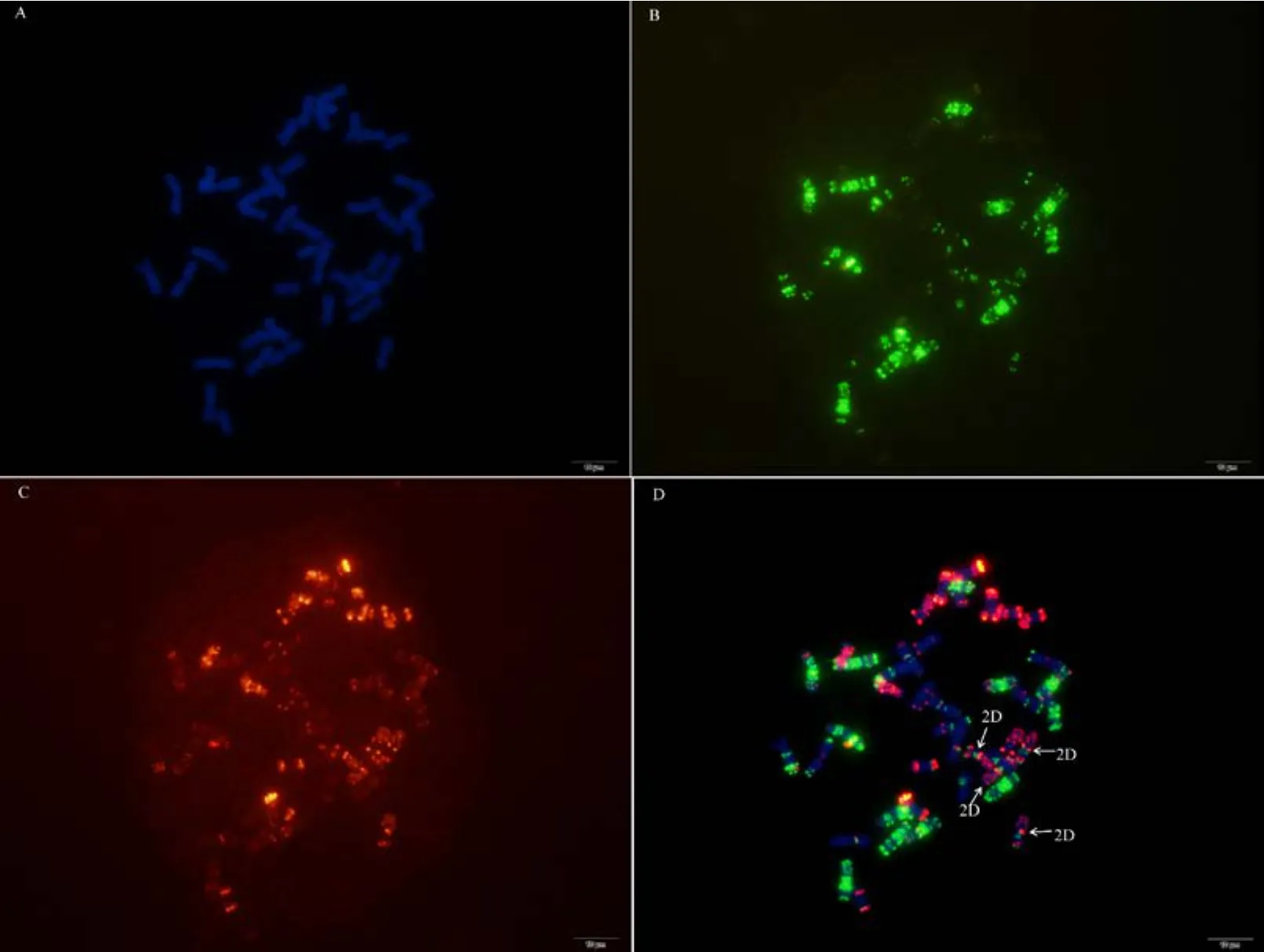
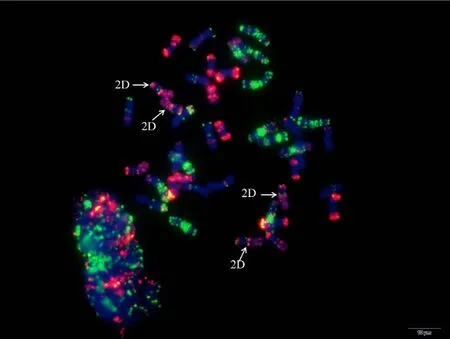
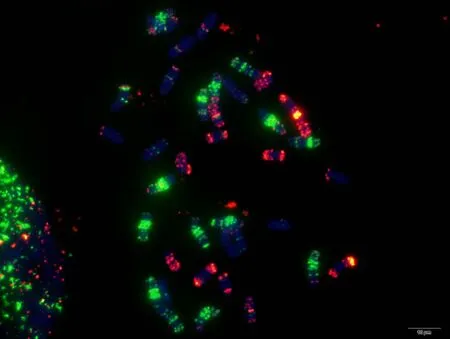
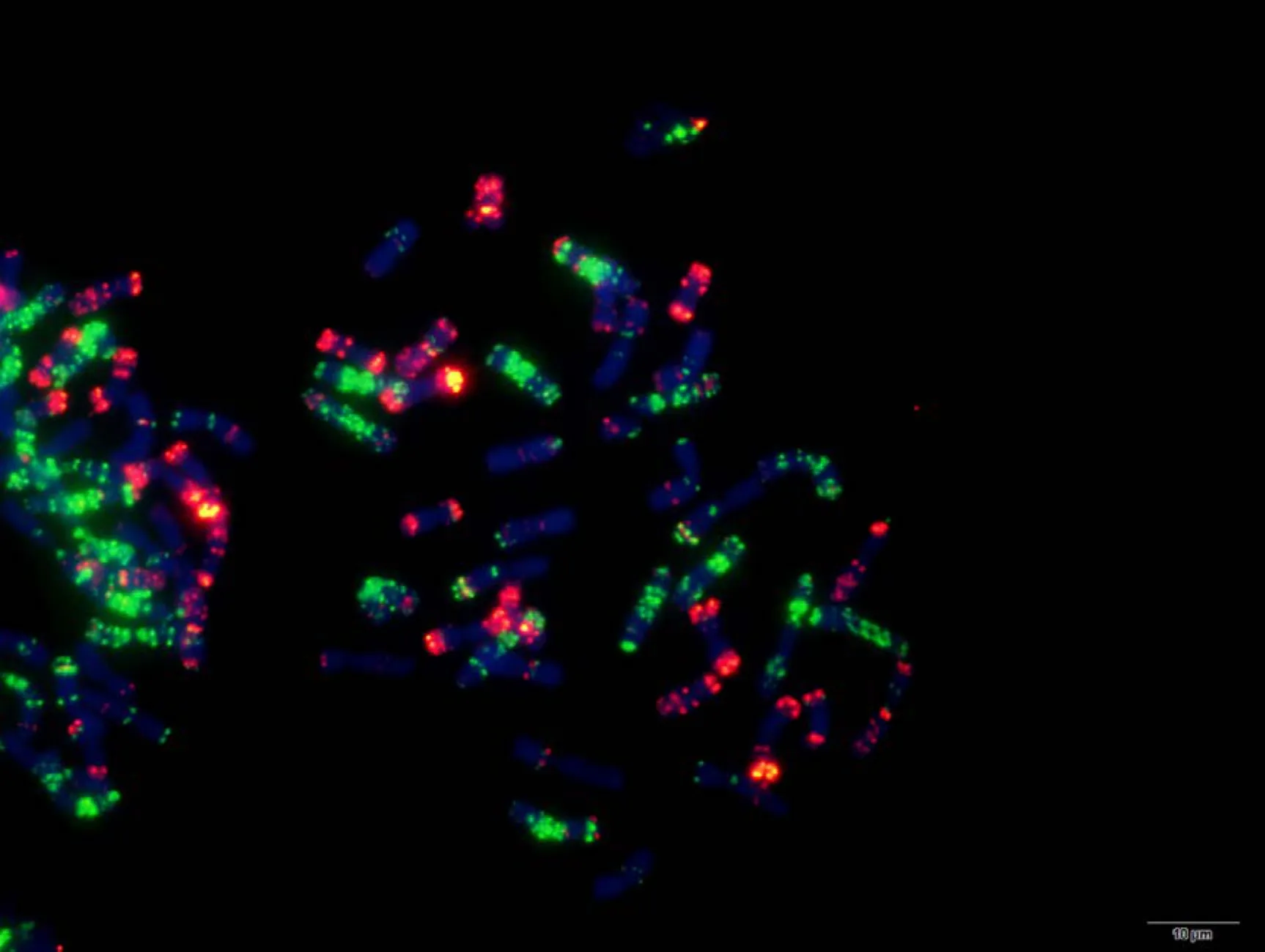
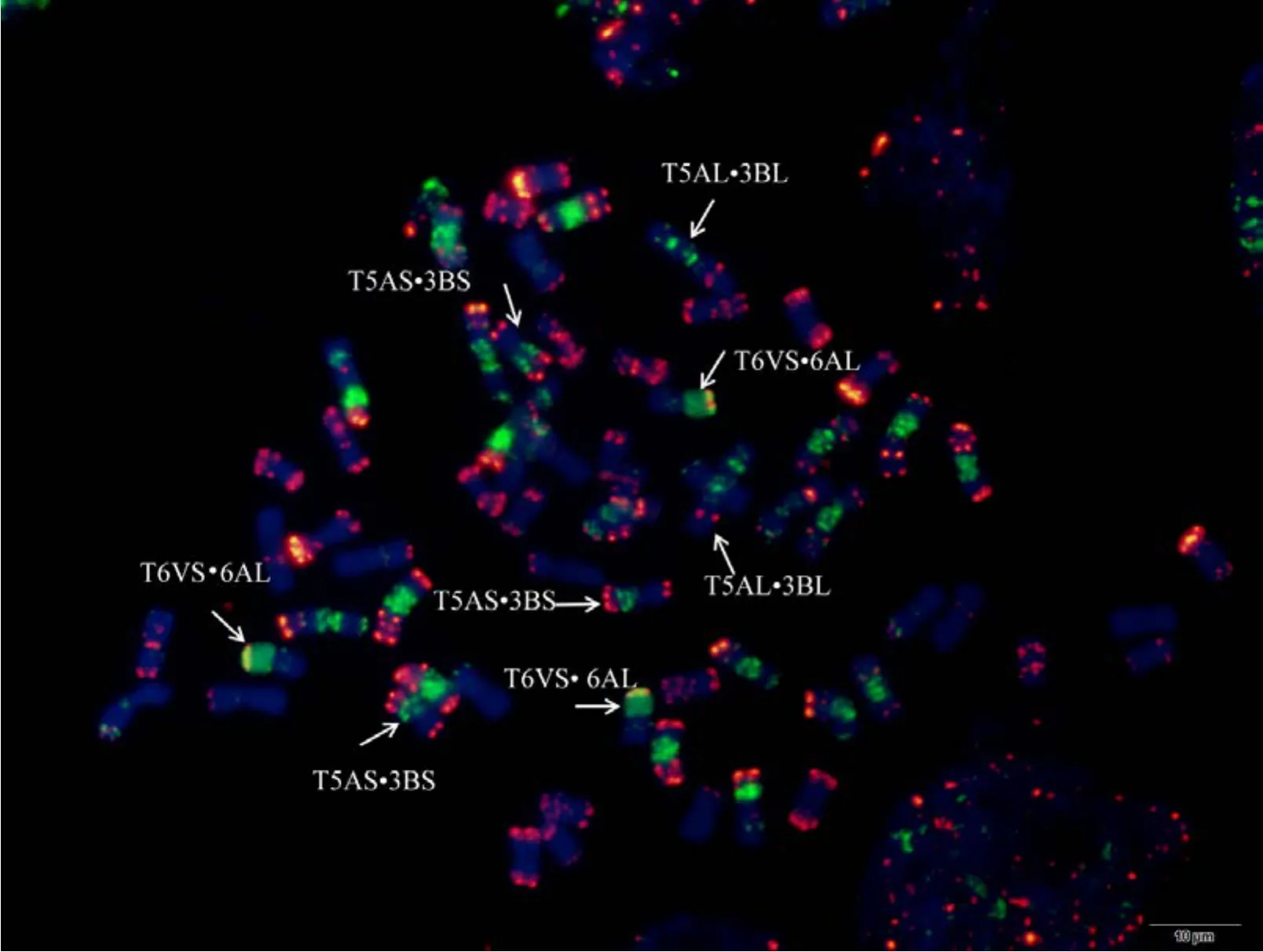
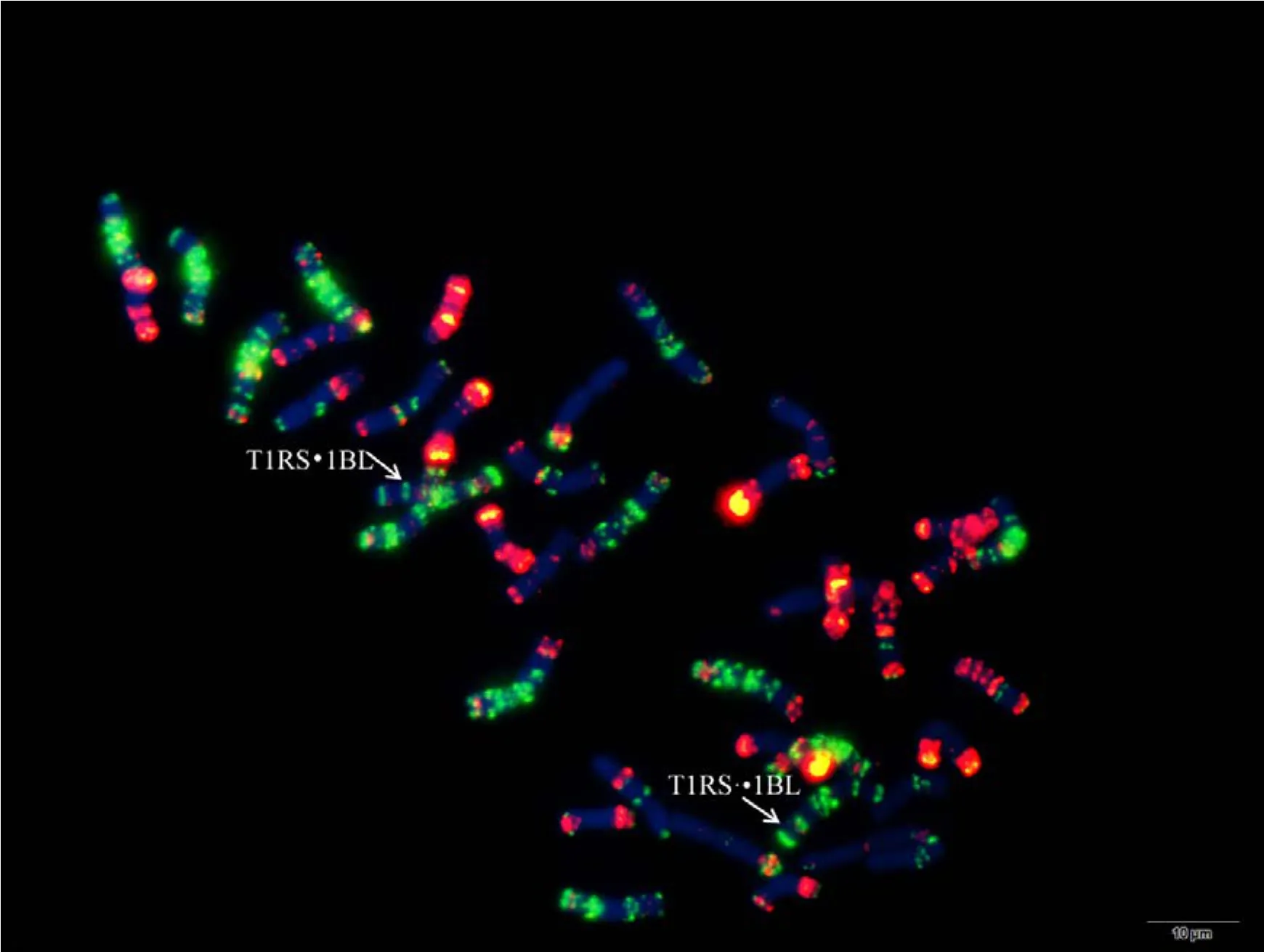
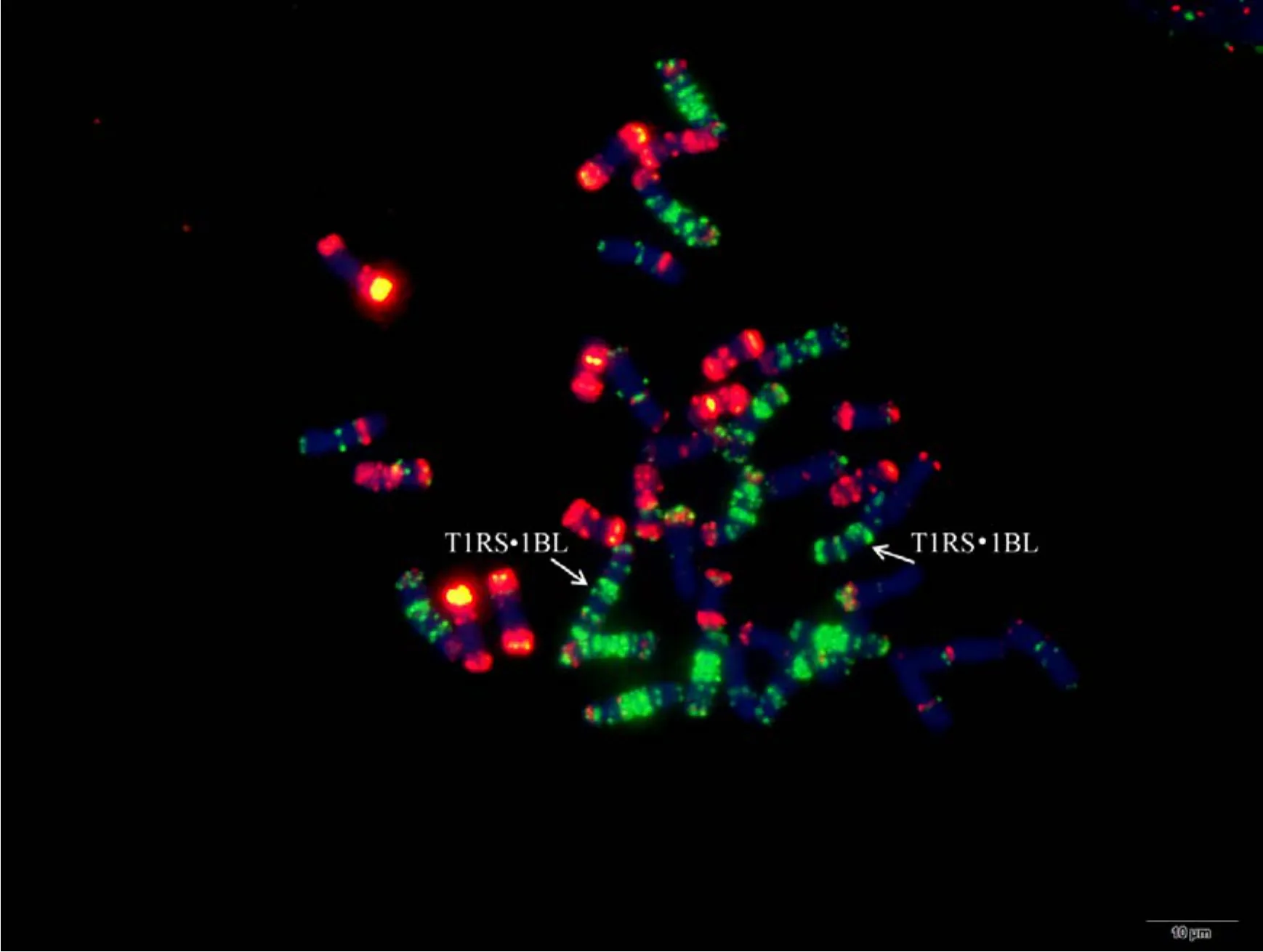
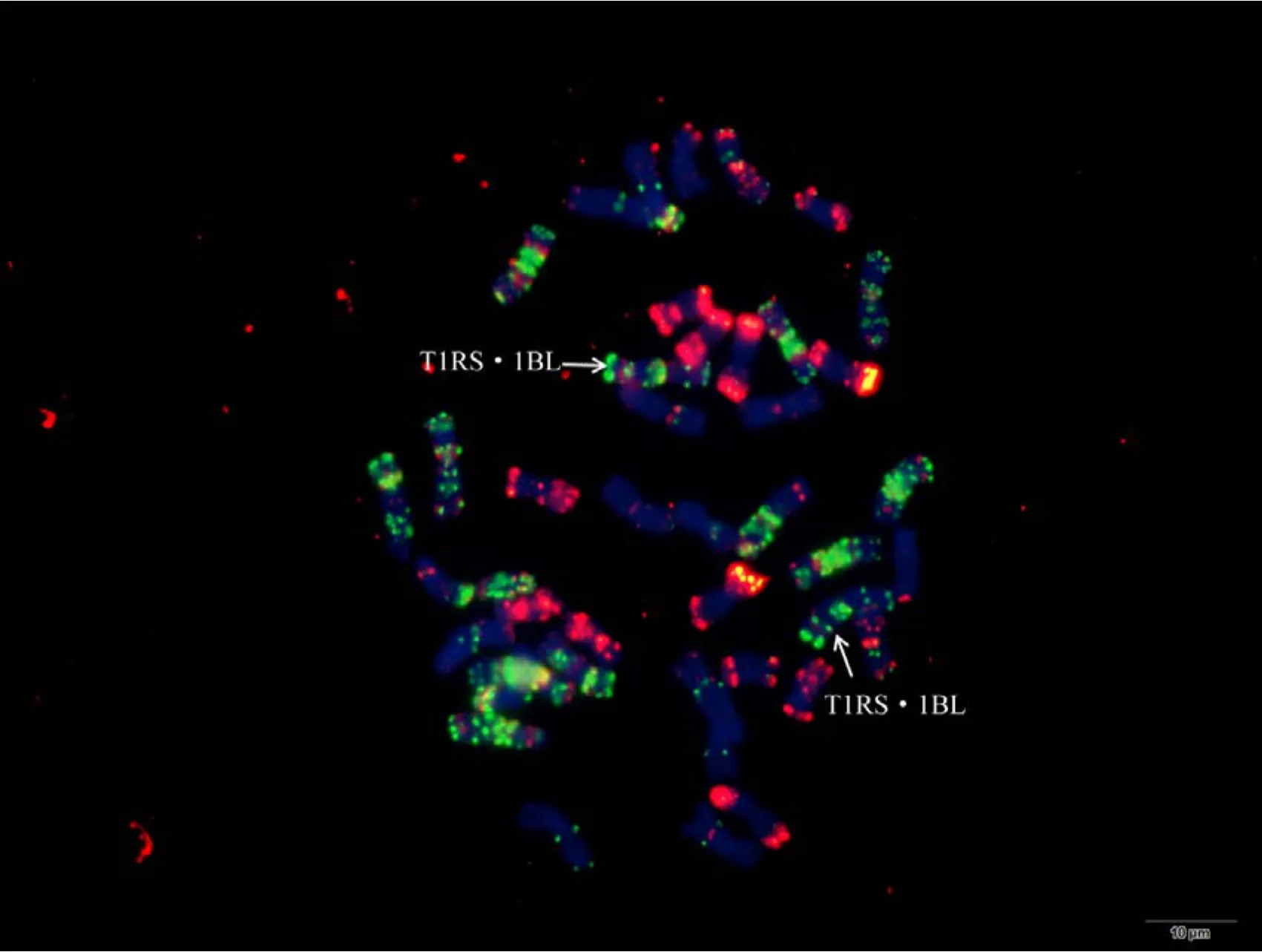
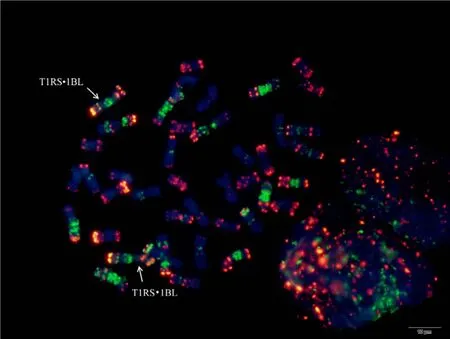
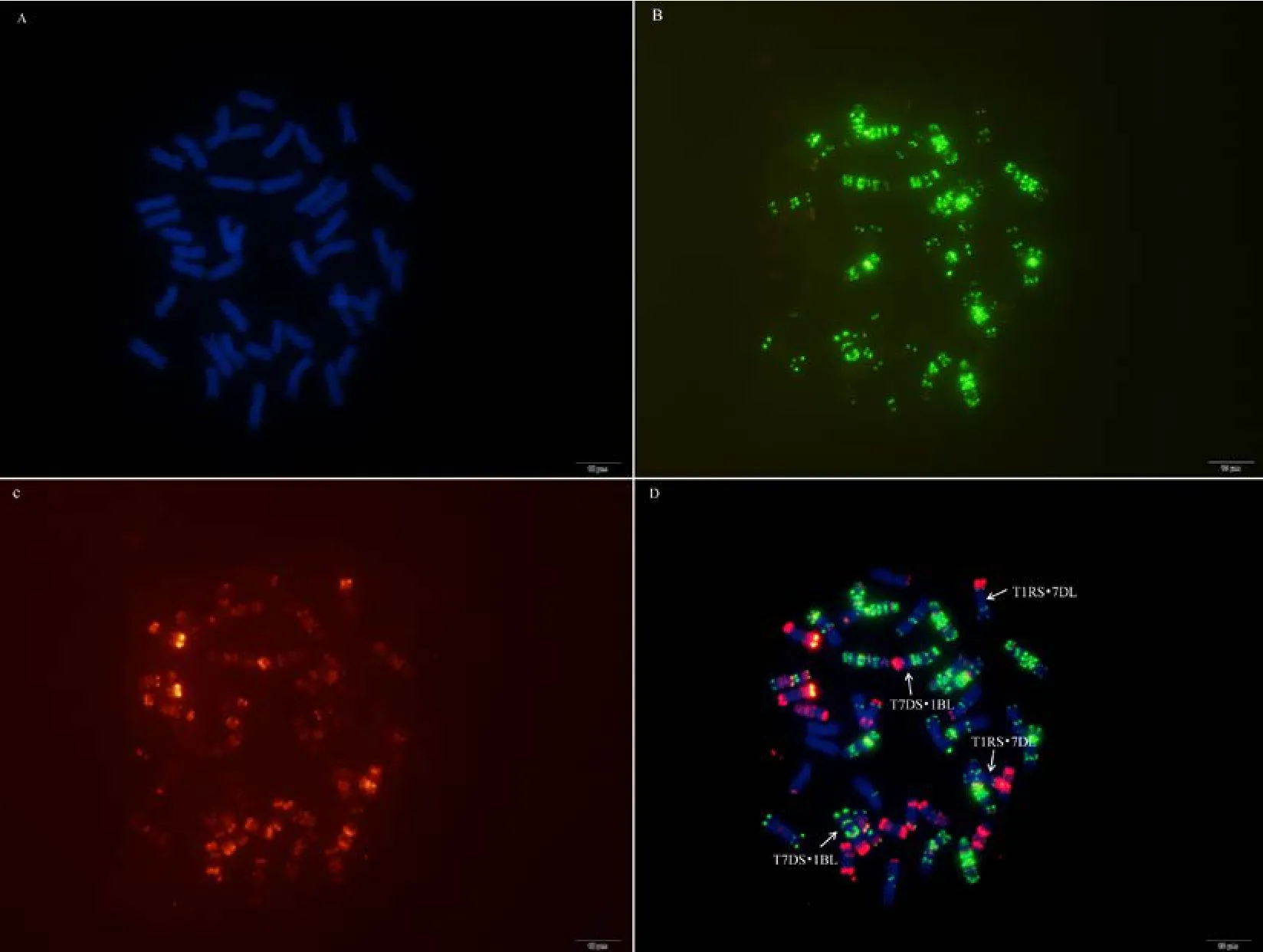
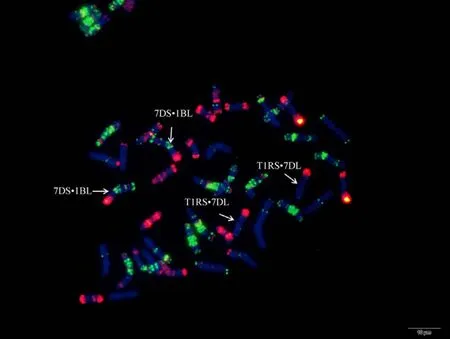
为验证该寡核苷酸探针套及中国春标准核型的应用潜力, 对7个地方品种、10个栽培品种和1个人工合成小麦进行了分析。与中国春相比, 供试材料在15条染色体上具有多态性, 其中B组除4B外6条染色体均具多态, A组5条染色体(2A、4A、5A、6A和7A)具有多态, 而D组4条染色体(1D、2D、4D 和 7D)有多态(图 3, 图 4; 附图 19~36)。我国小麦品种(系)包含的3种易位类型, 分别是T6VS·6AL类型, 如 92R137和南农 1258 (图 3-A, 图 4; 附图31~32), T1RS·1BL类型, 如周麦27、原泛3号和矮孟牛 VII (图 3-C、E, 图 4; 附图 33~35), 以及相互易位 T7DS·1BL 和 T1RS·7DL 类型, 如矮孟牛 IV (图3-G, 图 4; 附图 36), 通过该探针套一次 FISH可以清晰识别, 与GISH分析结果完全一致(图3-B、D、F、H)。经过多次不同颜色探针组合的杂交确证, 在南农1258中除T6VS·6AL外, 还发现1对明显的5A和3B相互易位, 形成T5AS·3BS和5AL·3BL (图3-A、B, 图4; 附图31), 发生易位的3B染色体来自亲本92R137 (图 4, 附图 32)。

图2 基于寡核苷酸探针套painting的中国春非整倍体高清核型Fig. 2 Oligonucleotide multiplex painting-based high resolution standard karyotypes of Chinese Spring aneuploids箭头示染色体多态性。Arrowheads show the polymorphism of chromosomes.
2.3 亲缘物种分析
为了解该探针套在小麦亲缘物种中的应用潜力,对5个物种进行了分析。该探针套在栽培一粒小麦、硬粒小麦“Langdon”、黑麦、长穗偃麦草全部染色体产生清晰信号, 可识别不同染色体(图5, 附图37~附图40), 其中Langdon与普通小麦A、B组染色体相似, 黑麦与Du等[43]报道的结果相似, 但栽培一粒小麦与Langdon、普通小麦A组染色体均存在明显差异; 中间偃麦草30条染色体产生了清晰且丰富的信号, 12条没有明显信号(图5, 附图41), 3个基因组差异明显。
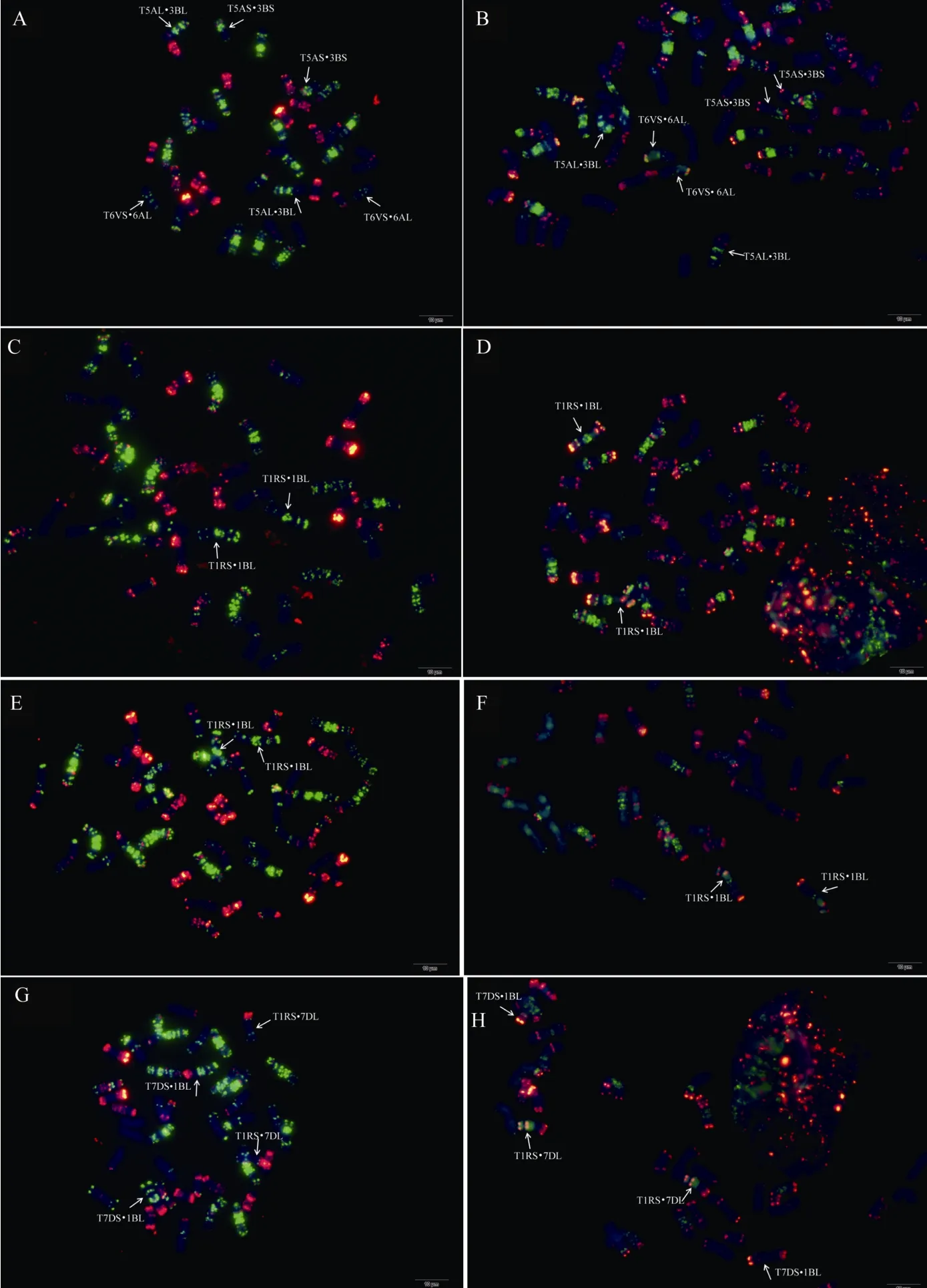
图3 基于寡核苷酸探针套painting和GISH与寡核苷酸探针套painting相结合的小麦品种染色体Fig. 3 Chromosomes of wheat varieties after oligonucleotide multiplex painting and combined GISH oligonucleotide multiplex painting
3 讨论
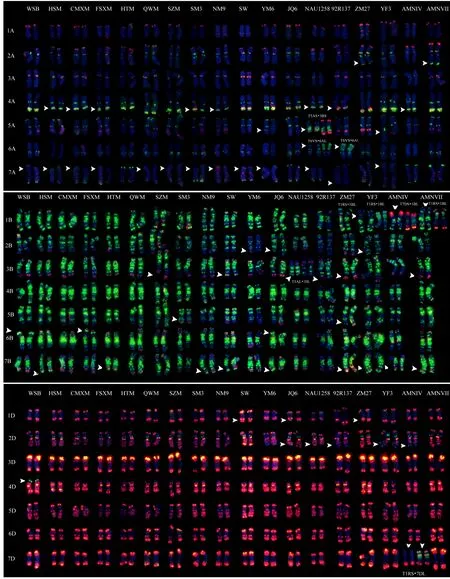
图4 基于寡核苷酸探针套painting的18个小麦品种核型Fig. 4 Oligonucleotide multiplex painting-based karyotypes of 18 wheat varieties
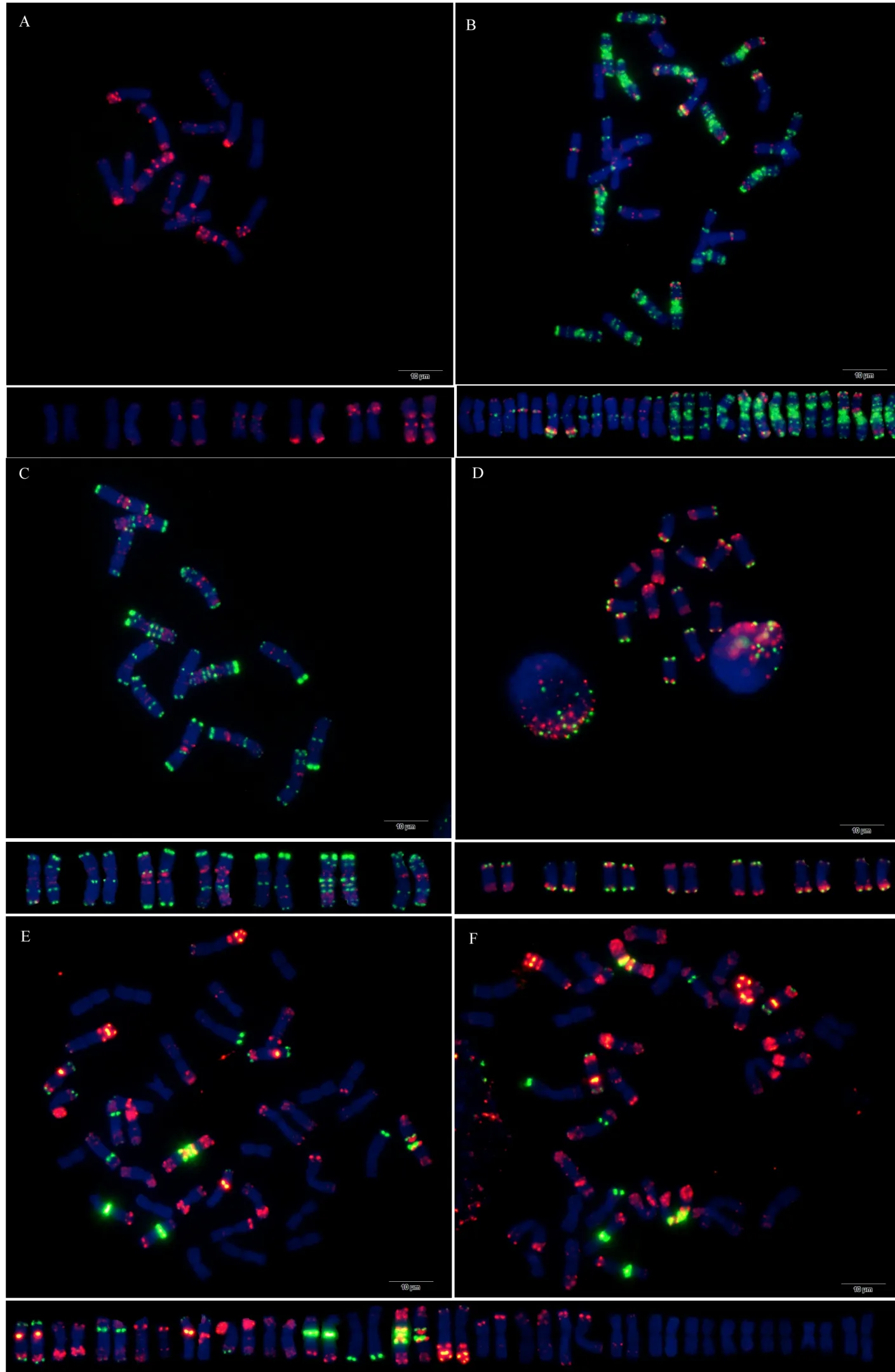
图5 基于寡核苷酸探针套painting的小麦亲缘物种核型Fig. 5 Oligonucleotide multiplex painting-based karyotypes of wheat relatives
FISH技术的开发和应用大大推动了植物染色体工程和细胞遗传学发展[48], 而基于寡核苷酸painting的染色体鉴定技术将迅速发展的基因组学与分子细胞遗传学紧密结合, 不但为染色体精确鉴定和生物学研究提供了新工具, 同时也将推动染色体工程发展。本研究利用我们前期开发的寡核苷酸探针套, 对41份小麦材料进行了迅速鉴定, 明确了这些材料的染色体特征, 构建了多数材料的高清核型。这些带型与曾广泛应用的 C-分带核型[49]相比,不但具有更高的清晰度, 而且 3个基因组染色体颜色和带纹差异明显, 因此更容易识别不同基因组和染色体。利用这些核型, 不但可以发现供试材料间明显的染色体多态性, 而且仅通过一次 FISH, 不通过GISH分析, 即可直接区分生产上常用的3种异源易位系, 甚至揭示发生在6A和6D之间的小片段相互易位, 充分表明这种方法的应用潜力。该法可用于分析更多的小麦品种及亲缘物种, 以揭示其中包含的染色体变异, 特别是自发的相互易位、倒位和小片段易位, 为构建准确的遗传图谱以及骨干亲本基因组重测序后的序列组装提供参考。同时还可以揭示不同多态性染色体的地理分布特征以及骨干亲本变异类型与传递特点, 从染色体水平上解释某些骨干亲本优良变异的聚合与优势传递。
供试18份非整倍体中, 14个保存完好, 未检测出明显的变化, 为进一步利用这些材料提供了信息,但是4份材料发生了明显的变异, 其中N6BT6D的一个单株同时包含3条1D、3条3B, 缺少6B, 其他染色体包括 6D均是完整的, 但多条染色体表现出多态性(图2)。推测该材料因为异交而造成了复杂的染色体变化。进一步鉴定应该可以发现更多的变异类型, 但由于引进的种子有限, 尚未进一步分析。更有趣的是, 从N5BT5D中发现含有完整的5B和5D,但也发现一对明显发生在6A和6D长臂顶端的小片段相互易位。推测早期的 5B缺体由于缺少 Ph1基因而导致部分同源染色体重组产生, 7D染色体长臂顶端的变化可能也归因于此。这说明5B缺体诱导部分同源染色体配对的效应是明显的, 值得在染色体工程中的利用; 但在引进和保存 N5BT5D时要特别注意及时鉴定和严格套袋自交。
从供试栽培品种和地方品种中共发现15条染色体具有多态性, 其中 B组最多, 其次为 A, 最后为D, 这与Du等[43]的分析结果相似, 说明我国小麦D组染色体多态性仍需要继续拓宽。本文鉴定的合成小麦 1D染色体存在明显多态(图 4), 在所有鉴定的品种中均未发现, 说明人工合成小麦可以作为我国小麦 D基因组改良的重要供体, 需要继续进行鉴定和利用。另外, 通过对栽培一粒小麦的鉴定发现, 其A组染色体与普通小麦和硬粒小麦均存在很大多态性, 因此是拓宽 A基因组遗传基础的供体。研究还发现, 该探针套可以通过一次FISH清晰区分小麦生产上广泛应用的3种易位系, T1RS·1BL、T6VS·6AL及相互易位系T1RS·7DL和T7DS·1BL, 而对这些易位系的准确鉴定过去需要采用多种手段[50-51]。另外,发现于“矮孟牛”中的相互易位 T1RS·7DL和T7DS·1BL 是继 T1BL·1RS、T1AL·1RS 在小麦生产上大量应用的第3种小麦–黑麦易位系, 其在把黑麦多个有利基因传递的同时, 由于相互易位杂合体基因重组率下降而使 3条小麦染色体臂上的有利基因也总以集团的形式传递给后代, 因此这种易位在人工选择的过程中可能会有较高的出现频率。相互易位 T1RS·7DL和 T7DS·1BL的育成和大量使用给我们继续培育新的骨干亲本提供了借鉴, 采用染色体工程将优良亲本的有利基因组合以相互易位或倒位的方式加以固定起来以降低基因重组率、保持优良的基因单倍体型应该引起人们的足够重视, 一些新的染色体工程技术(例如同源重组系统或其他基因组编辑系统)可以加速实现这一梦想[3]。值得注意的是, 我们通过矮孟牛V (包含相互易位T1RS·7DL和T7DS·1BL)和 92R137 (T6VS·6AL)杂交育成的小麦新品系南农1258[52]因较好地结合了抗病性和农艺性状, 已被多个单位用作育种亲本, 但是本文鉴定发现, 该品系除含有T6VS·6AL外, 还有1对新的相互易位染色体T5AS·3BS和T5AL·3BL, 很可能由于南农1258双亲涉及多条易位染色体引起的配对异常造成。由此可以推测, 复杂易位的利用可以诱导产生更多新易位, 因此为创造更多相互易位以减少目标区段的重组率和保留优异亲本中的优异基因组合提供了可能。
黑麦、长穗偃麦草和中间偃麦草是我国小麦染色体工程中广泛应用的亲本, 我国科学家已经育成了大量相关小麦异染色体系, 本文建立的核型为鉴定这些材料的染色体组成提供了重要信息。目前,我们已经开发出 20多个小麦、百萨偃麦草寡核苷酸探针, 本文所用的探针套根据 Du等[43]开发的oligonucleotide multiplex#4简化而成, 仅包含5个寡核苷酸探针, 其中红色探针3个(pAs1-1、pAs1-3和AFA-4), 绿色探针2个[pSc119.2-1和(GAA)10],每次合成(每个探针5 OD)仅需要约1500元, 可以分析至少 750张染色体制片, 因此每张成本仅需要约2元, 而且省去了原先质粒的引进、繁殖、保存、质粒提取、检测和标记等程序, 大大简化了实验程序, 适合高通量分析。另外, 我们已经开发出可以循环利用的寡核苷酸染液, 将会进一步简化程序, 为小麦染色体自动化分析提供可能。值得一提的是, 我们开发的寡核苷酸探针完全可以根据在不同物种上的分布特征重新组配, 优化出针对不同物种的特异探针套, 例如, 在鉴定小麦异染色体系的过程中, 由于 pSc119.2-1在很多物种上也能产生杂交信号, 因此为了与GISH信号相区别,可以将FAM修饰改用TAMRA修饰, 以更好地识别不同染色体。同理, 对其他寡核苷酸探针也可以根据实际需要改变修饰物。所有这些都需要对待鉴定物种进行单核苷酸探针的顺序FISH分析, 明确不同探针的信号分布特征和位置, 特别是对于那些前期缺乏遗传研究的小麦亲缘物种, 从而更好地转移和利用亲缘物种的有利基因, 提高染色体工程效率。
4 结论
基于寡核苷酸探针的染色体 panting技术在中国春非整倍体上产生了更加清晰稳定的多色带纹,从而可以将小麦3个基因组和7个部分同源群染色体全部准确区分, 为小麦染色体工程提供了新的参考标准; 我国小麦地方品种和栽培品种, 虽然15条染色体具有多态性, 但是大部分品种具有相似的染色体特征, 缺乏足够的多态性; 同时, 该探针套可以不通过GISH分析清晰区分广泛应用的3种异源易位系, 即 T1RS·1BL、T6VS·6AL 和相互易位系T1RS·7DL 和 T7DS·1BL; 除硬粒小麦“Langdon”外,其他4个亲缘物种均表现出丰富的染色体多态性, 是拓宽栽培小麦遗传基础的重要基因资源。本文报道的寡核苷酸探针套及中国春非整倍体核型为小麦染色体工程提供了新的工具, 值得进一步研究和利用。
致谢:本研究部分植物材料转引自南京农业大学马正强教授、江苏省农业科学院杨学明研究员和吴纪中研究员、日本鸟取大学Hisashi Tsujimoto教授, 特此致谢。
[1]Tack J, Barkley A, Nalley L L. Effect of warming temperatures on US wheat yields. Proc Natl Acad Sci USA, 2015, 112:6931–6936
[2]Liu B, Asseng S, Müller C, Ewert F, Elliott J, Lobell D B, Martre P, Ruane A C, Wallach D, Jones J W, Rosenzweig C, Aggarwal P K, Alderman P D, Anothai J, Basso B, Biernath C, Cammarano D,Challinor A, Deryng D, De Sanctis G, Doltra J, Fereres E, Folberth C, Garcia-Vila M, Gayler S, Hoogenboom G, Hunt L A,Izaurralde R C, Jabloun M, Jones C D, Kersebaum K C, Kimball B A, Koehler A K, Kumar S N, Nendel C, O’Leary G J, Olesen J E, Ottman M J, Palosuo T, Vara Prasad P V, Priesack E, Pugh T A M, Reynolds M, Rezaei E E, Rötter R P, Schmid E, Semenov M A, Shcherbak I, Stehfest E, Stöckle C O, Stratonovitch P, Streck T,Supit I, Tao F, Thorburn P, Waha K, Wall G W, Wang E, White J W, Wolf J, Zhao Z, Zhu Y. Similar estimates of temperature impacts on global wheat yield by three independent methods. Nat Climat Change, 2016, 6: 1130–1136
[3]Chan Simon W L. Chromosome engineering: power tools for plant genetics. Trends Biotechnol, 2010, 28: 605–610
[4]Jiang J M, Friebe B, Gill B S. Recent advances in alien gene transfer in wheat. Euphytica, 1994, 73: 199–212
[5]Cao A Z, Xing L P, Wang X Y, Yang X M, Wang W, Sun Y L,Qian C, Ni J L, Chen Y P, Liu D J, Wang X, Chen P D.Serine/threonine kinase gene Stpk-V, a key member of powdery mildew resistance gene Pm21, confers powdery mildew resistance in wheat. Proc Natl Acad Sci USA, 2011, 108:7727–7732
[6]Endo T R, Gill B S. The deletion stocks of common wheat. J Hered, 1996, 87: 295–307
[7]Zheng Q, Li B, Mu S M, Zhou H P, Li Z S. Physical mapping of the blue-grained gene(s) from Thinopyrum ponticum by GISH and FISH in a set of translocation lines with different seed colors in wheat. Genome, 2006, 49: 1109–1114
[8]Nasuda S, Hudakova S, Schubert I, Endo T R. Stable barley chromosomes without centromeric repeats. Proc Natl Acad Sci USA, 2005, 102: 9842–9847
[9]Friebe B, Kynast R G, Gill B S. Gametocidal factor-induced structural rearrangements in rye chromosomes added to common wheat. Chrom Res, 2000, 8: 501–511
[10]Gyawali Y P, Nasuda S, Endo T R. Cytological dissection and molecular characterization of chromosome 1R derived from‘Burgas 2’ common wheat. Genes Genet Syst, 2009, 84: 407–416
[11]Zhuang L F, Liu P, Liu Z Q, Chen T T, Wu N, Sun L, Qi Z J.Multiple structural aberrations and physical mapping of rye chromosome 2R introgressed into wheat. Mol Breed, 2015, 35:133
[12]Chen P D, You C F, Hu Y, Chen S W, Zhou B, Cao A Z, Wang X.Radiation-induced translocations with reduced Haynaldia villosa chromatin at the Pm21 locus for powdery mildew resistance in wheat. Mol Breed, 2013, 31: 477–484
[13]Zhang R Q, Hou F, Feng Y G, Zhang W, Zhang M Y, Chen P D.Characterization of a Triticum aestivum–Dasypyrum villosum T2VS·2DL translocation line expressing a longer spike and more kernels traits. Theor Appl Genet, 2015, 128: 2415–2425
[14]Sepsi A, Molnár I, Szalay D, Molnár-Láng M. Characterization of a leaf rust-resistant wheat–Thinopyrum ponticum partial amphiploid BE-1, using sequential multicolor GISH and FISH. Theor Appl Genet, 2008, 116: 825–834
[15]Mahelka V, Kopeck D, Baum B R. Contrasting patterns of evolution of 45S and 5S rDNA families uncover new aspects in the genome constitution of the agronomically important grass Thinopyrum intermedium (Triticeae). Mol Biol Evol, 2013, 30:2065–2086
[16]Danilova T V, Zhang G, Liu W X, Friebe B, Gill B S. Homoeologous recombination-based transfer and molecular cytogenetic mapping of a wheat streak mosaic virus and Triticum mosaic virus resistance gene Wsm3 from Thinopyrum intermedium to wheat. Theor Appl Genet, 2017, 130: 549–556
[17]Rey M D, Calderón M C, Prieto P. The use of the ph1b mutant to induce recombination between the chromosomes of wheat and barley. Front Plant Sci, 2015, 6: 160
[18]Song L Q, Lu Y Q, Zhang J P, Pan C L, Yang X M, Li X Q, Liu W H, Li L H. Physical mapping of Agropyron cristatum chromosome 6P using deletion lines in common wheat background.Theor Appl Genet, 2016, 129: 1023–1034
[19]Li H H, Jiang B, Wang J C, Lu Y Q, Zhang J P, Pan C L, Yang X M, Li X Q, Liu W H, Li L H. Mapping of novel powdery mildew resistance gene(s) from Agropyron cristatum chromosome 2P.Theor Appl Genet, 2017, 130: 109–121
[20]Qi Z J, Du P, Qian B L, Zhuang L F, Chen H F, Chen T T, Shen J,Guo J, Feng Y G, Pei Z Y. Characterization of a wheat–Thinopyrum bessarabicum (T2JS-2BS·2BL) translocation line.Theor Appl Genet, 2010, 121: 589–597
[21]Pu J, Wang Q, Shen Y F, Zhuang L F, Li C X, Tan M F, Bie T D,Chu C G, Qi Z J. Physical mapping of chromosome 4J of Thinopyrum bessarabicum using gamma radiation-induced aberrations.Theor Appl Genet, 2015, 128: 1319–1328
[22]Badaeva E D, Dedkova O S, Koenig J, Bernard S, Bernard M.Analysis of introgression of Aegilops ventricosa Tausch. genetic material in a common wheat background using C-banding. Theor Appl Genet, 2008, 117: 803–811
[23]Niu Z X, Klindworth D L, Friesen T L, Chao S M, Jin Y, Cai X W,Xu S S. Targeted introgression of a wheat stem rust resistance gene by DNA marker-assisted chromosome engineering. Genetics,2011, 187: 1011–1021
[24]Liu W X, Koo D H, Xia Q, Li C X, Bai F Q, Song Y L, Friebe B,Gill B S. Homoeologous recombination-based transfer and molecular cytogenetic mapping of powdery mildew-resistant gene Pm57 from Aegilops searsii into wheat. Theor Appl Genet, 2017,DOI 10.1007/s00122-017-2855-y
[25]Ishii T, Ueda T, Tanaka H, Hisashi T. Chromosome elimination by wide hybridization between Triticeae or oat plant and pearl millet: pearl millet chromosome dynamics in hybrid embryo cells.Chrom Res, 2010, 18: 821–831
[26]Endo T R. The gametocidal chromosome as a tool for chromosome manipulation in wheat. Chrom Res, 2007, 15: 67–75
[27]Bie T D, Cao Y P, Chen P D. Mass production of intergeneric chromosomal translocations through pollen irradiation of Triticum durum–Haynaldia villosa amphiploid. J Integr Plant Biol,2007, 49: 1619–1626
[28]Ma X H, Wang Q, Wang Y Z, Ma J Y, Wu N, Ni S, Luo T X,Zhuang L F, Chu C G, Cho S W, Tsujimoto H, Qi Z J. Chromosome aberrations induced by Zebularine in triticale. Genome,2016, 59: 485–492
[29]Tiwari V K, Heesacker A, Riera-Lizarazu O, Gunn H, Wang S C,Wang Y, Gu Y Q, Paux E, Koo D H, Kumar A, Luo M C, Lazo G,Zemetra R, Akhunov E, Friebe B, Poland J, Gill B S, Kianian S,Leonard J M. A whole-genome, radiation hybrid mapping resource of hexaploid wheat. Plant J, 2016, 86: 195–207
[30]张学勇, 马琳, 郑军. 作物驯化和品种改良所选择的关键基因及其特点. 作物学报, 2017, 43: 157–170 Zhang X Y, Ma L, Zheng J. Characteristics of genes selected by domestication and intensive breeding in crop plants. Acta Agron Sin, 2017, 43: 171–178 (in Chinese with English abstract)
[31]Cuadrado Á, Golczyk H, Jouve N. A novel, simple and rapid nondenaturing FISH (ND-FISH) technique for the detection of plant telomeres. Potential use and possible target structures detected. Chrom Res, 2009, 17: 755–762
[32]Cuadrado Á, Jouve N. Chromosomal detection of simple sequence repeats (SSRs) using nondenaturing FISH (ND-FISH).Chromosoma, 2010, 119: 495–503
[33]Matera A G, Ward D C. Oligonucleotide probes for the analysis of specific repetitive DNA sequences by fluorescence in situ hybridization. Hum Mol Genet, 1992, 1: 535–539
[34]Moodie S L, Thornton J M. A study into the effects of protein binding on nucleotide conformation. Nucl Acids Res, 1993, 21:1369–1380
[35]Cuadrado A, Cardoso M, Jouve N. Physical organisation of simple sequence repeats (SSRs) in Triticeae: structural, functional and evolutionary implications. Cytogenet Genome Res, 2008, 120:210–219
[36]Beliveau B J, Joyce E F, Apostolopoulos N, Yilmaz F, Fonseka C Y, McCole R B, Chang Y, Li J B, Senaratne T N, Williams B R,Rouillard J M, Wu C T. Versatile design and synthesis platform for visualizing genomes with oligopaint FISH probes. Proc Natl Acad Sci USA, 2012, 109: 21301-21306
[37]Miks-Krajnik M, Babuchowski A. 16S rRNA-targeted oligonucleotide probes for direct detection of Propionibacterium freudenreichii in presence of Lactococcus lactis with multicolour fluorescence in situ hybridization. Lett Appl Microbiol, 2014, 59:320–327
[38]王艳芝. 百萨偃麦草染色体小片段易位的诱致、鉴定与基因定位分析. 南京农业大学硕士学位论文, 江苏南京, 2013 Wang Y Z. Development and Characterization of Small Segment Translocations of Thinopyrum bessarabicum and Cytological Mapping of Interest Genes. MS Thesis of Nanjing Agricultural University, Nanjing, China, 2013 (in Chinese with English abstract)
[39]Tang Z X, Yang Z J, Fu S L. Oligonucleotides replacing the roles of repetitive sequences pAs1, pSc119.2, pTa-535, pTa71, CCS1,and pAWRC.1 for FISH analysis. J Appl Genet, 2014, 55:313–318
[40]Han Y H, Zhang T, Thammapichai P, Weng Y Q, Jiang J M.Chromosome-specific painting in Cucumis species using bulked oligonucleotides. Genetics, 2015, 200: 771–779
[41]Fu S L, Chen L, Wang Y Y, Li M, Yang Z J, Qiu L, Yan B J, Ren Z L, Tang Z X. Oligonucleotide probes for ND-FISH analysis to identify rye and wheat chromosomes. Sci Rep, 2015, 5: 10552
[42]Tang S Y, Qiu L, Xiao Z Q, Fu S L, Tang Z X. New oligonucleotide probes for ND-FISH analysis to identify barley chromosomes and to investigate polymorphisms of wheat chromosomes.Genes, 2016, 7: 118
[43]Du P, Zhuang L F, Wang Y Z, Yuan L, Wang Q, Wang D R,Dawadondup, Tan L J, Shen J, Xu H B, Zhao H, Chu C G, Qi Z J.Development of oligonucleotides and multiplex probes for quick and accurate identification of wheat and Thinopyrum bessarabicum chromosomes. Genome, 2017, 60: 93–103
[44]Zhu M Q, Du P, Zhuang L F, Chu C G, Zhao H, Qi Z J. A simple and efficient non-denaturing FISH method for maize chromosome differentiation using single-strand oligonucleotide probes.Genome, 2017, 60: 657–664
[45]刘振乾. 荆州黑麦染色体变异体的诱致与鉴定. 南京农业大学硕士学位论文, 江苏南京, 2012 Liu Z Q. Development and Identification of Chromosome Variations of Secale cereale cv. Jingzhouheimai. MS Thesis of Nanjing Agricultural University, Nanjing, China, 2012 (in Chinese with English abstract)
[46]Dolezel J, Cihalikova J, Lucretti S. A high yield procedure for isolation of metaphase chromosomes from root tips of Vicia faba.Planta, 1992, 188: 93–98
[47]Dolezel J, Cihalikova J, Weiserova J, Lucretti S. Cell cycle synchronization in plant root meristems. Methods Cell Sci, 1999, 21:95–107
[48]Jiang J M, Gill B S. Current status and the future of fluorescence in situ hybridization (FISH) in plant genome research. Genome,2006, 49: 1057–1068
[49]Gill B S, Friebe B, Endo T R. Standard karyotype and nomenclature system for description of chromosome bands and structural aberration in wheat (Triticum aestivum). Genome, 1991, 34:830–839
[50]Chen P D, Qi L L, Zhou B, Zhang S Z, Liu D J. Development and molecular cytogenetic analysis of wheat–H. villosa 6VS/6AL translocation lines specifying resistance to powdery mildew.Theor Appl Genet, 1995, 91: 1125–1128
[51]Qi Z J, Chen P D, Liu D J, Li Q Q. A new secondary reciprocal translocation discovered in Chinese wheat. Euphytica, 2004, 137:333–338
[52]庄丽芳, 亓增军, 孙玲, 李爱霞, 陈华锋, 王从磊, 达瓦顿珠,冯祎 高, 裴自友. 衍生于“矮孟牛Ⅴ”与 92R137的小麦新品系南农1258的系统鉴定. 麦类作物学报, 2008, 28: 387–392 Zhuang L F, Qi Z J, Sun L, Li A X, Chen H F, Wang C L,Dawadondup, Feng Y G, Pei Z Y. Identification of a new wheat line Nannong 1258 derived from wheat germplasm Aimengniu V and 92R137. J Triticeae Crops, 2008, 28: 387–392 (in Chinese with English abstract)
Development and Application of High Resolution Karyotypes of Wheat “Chinese Spring” Aneuploids
WANG Dan-Rui1, DU Pei1, PEI Zi-You2, ZHUANG Li-Fang1,*, and QI Zeng-Jun1,*1State Key Laboratory of Crop Genetics and Germplasm Enhancement, Nanjing Agricultural University, Nanjing 210095, China;2Crop Science Institute, Shanxi Academy of Agricultural Sciences, Taiyuan 030031, China
附图1 N1BT1D Fig. S1 N1BT1D
附图2.1 N2BT2D Fig. S2.1 N2BT2D
附图2.2 N2BT2D Fig. S2.2 N2BT2D
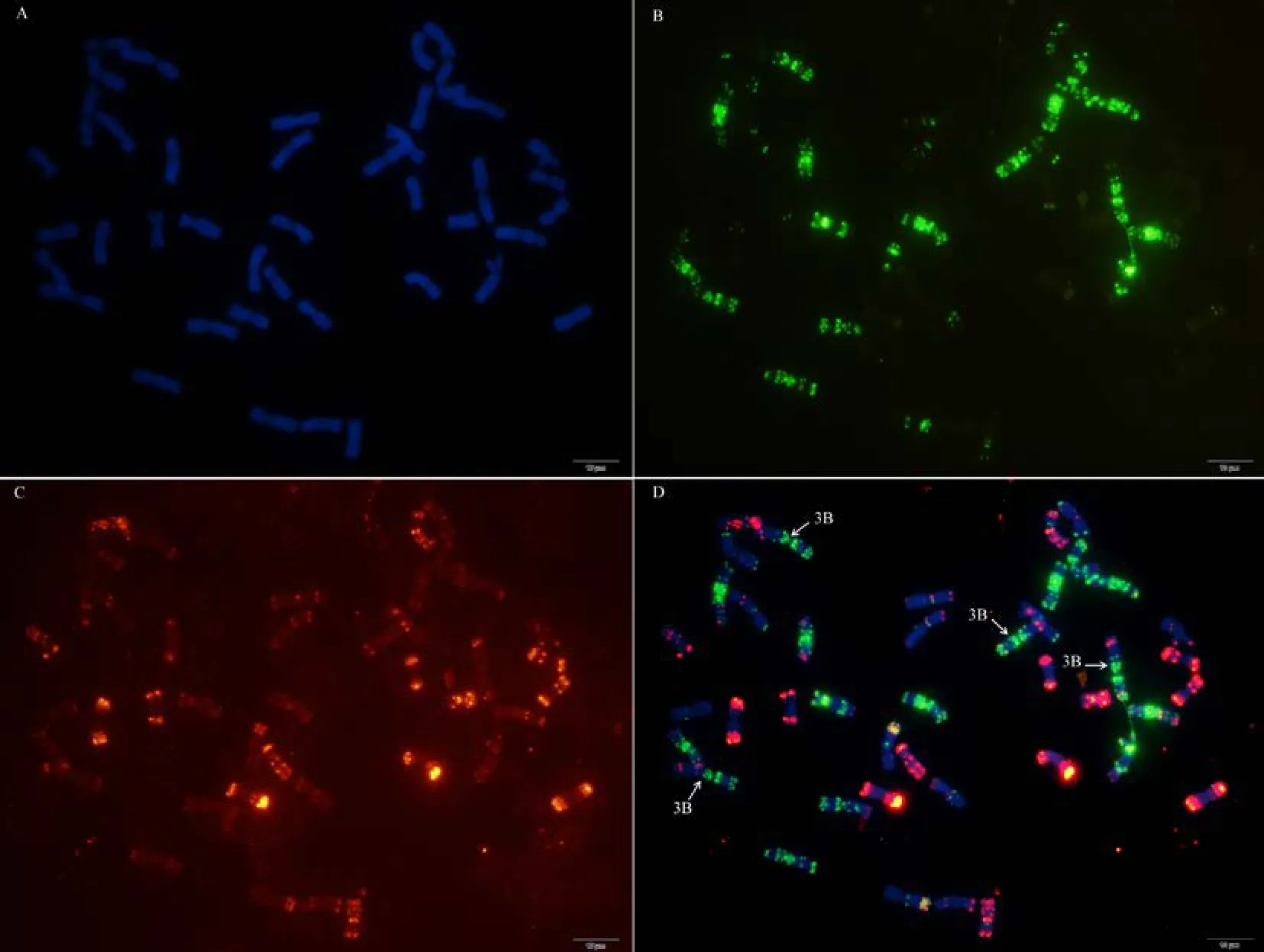
附图3 N3AT3B Fig. S3 N3AT3B
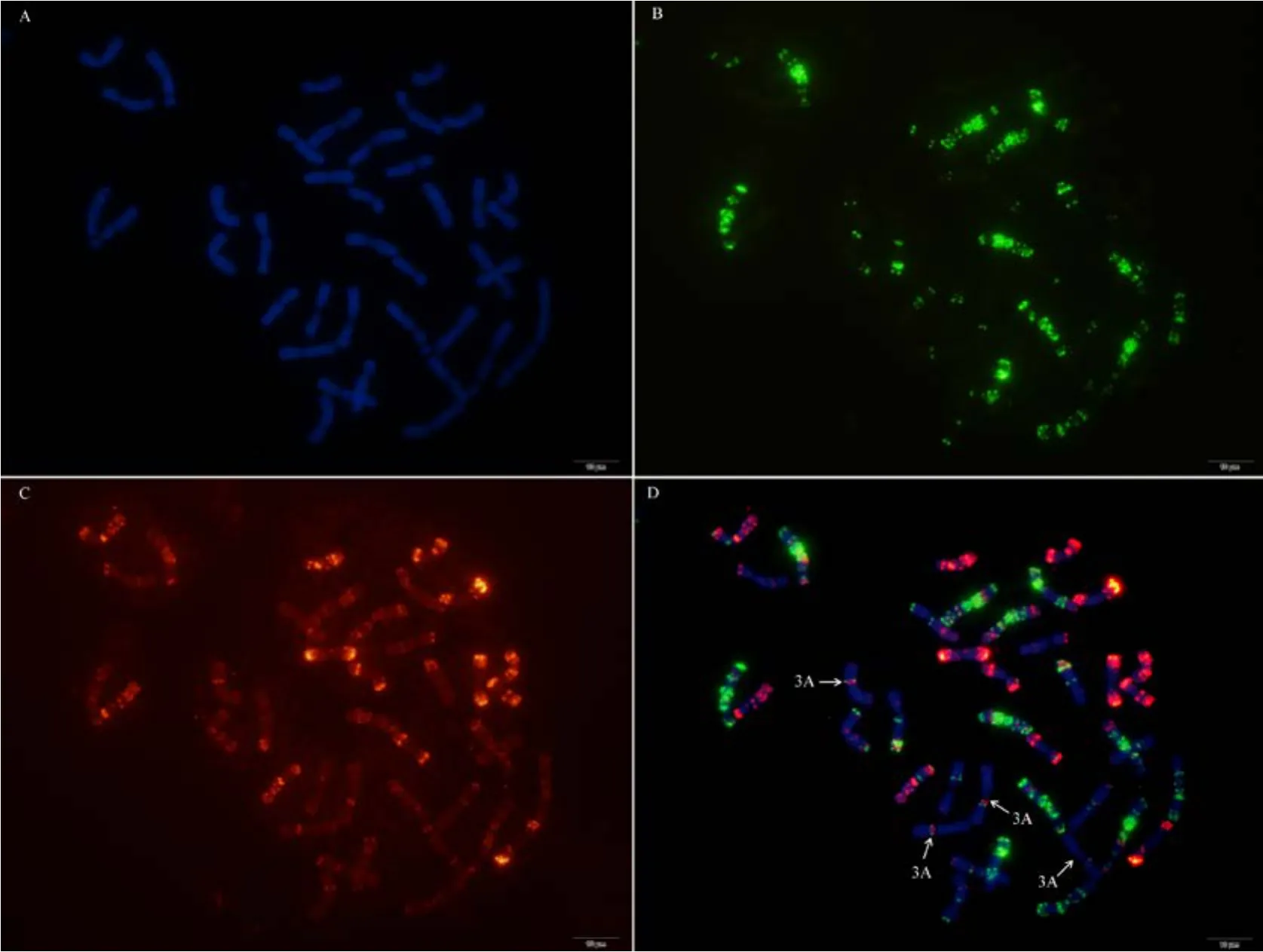
附图4 N3BT3A Fig. S4 N3BT3A
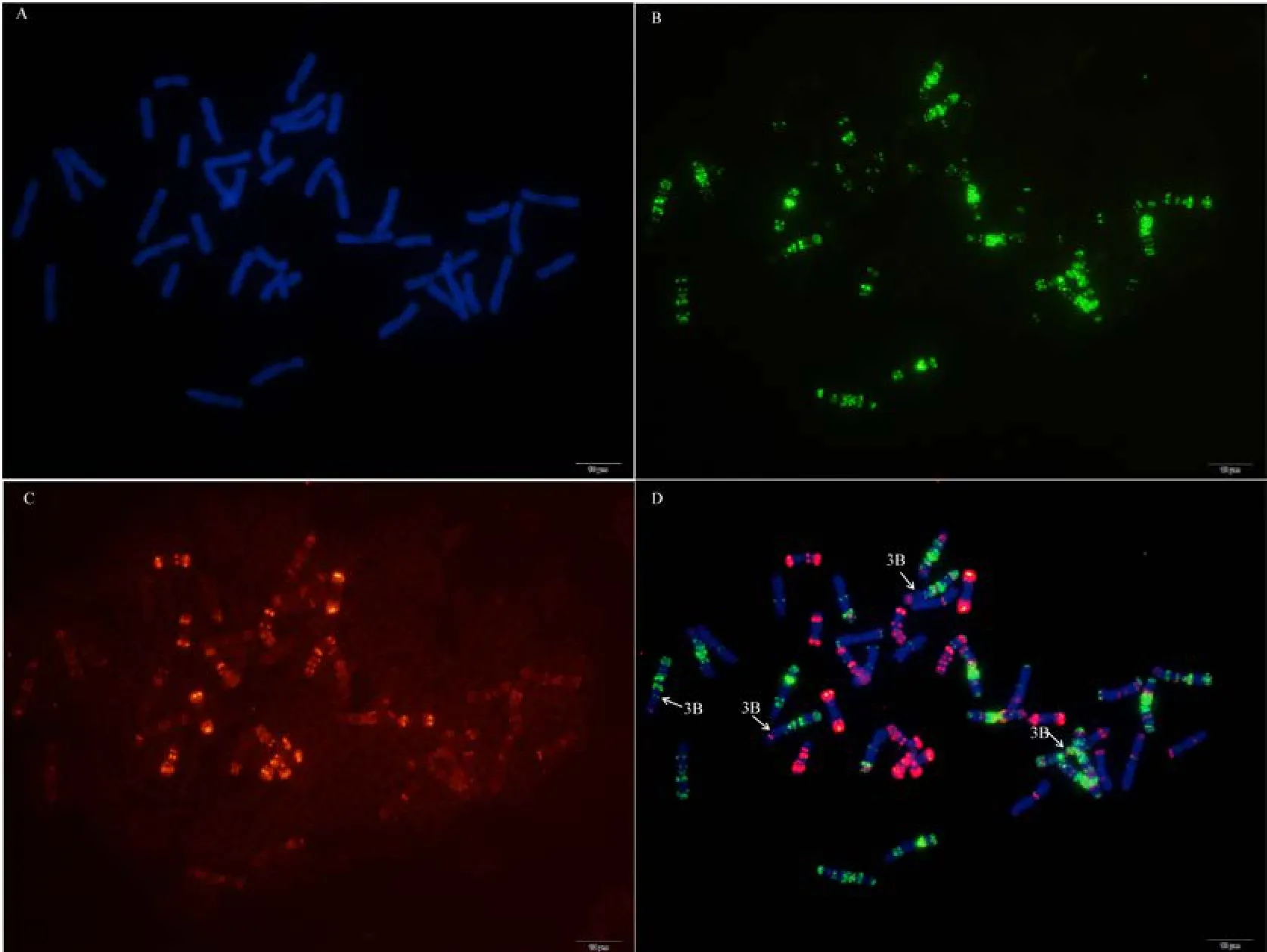
附图5 N3DT3B Fig. S5 N3DT3B
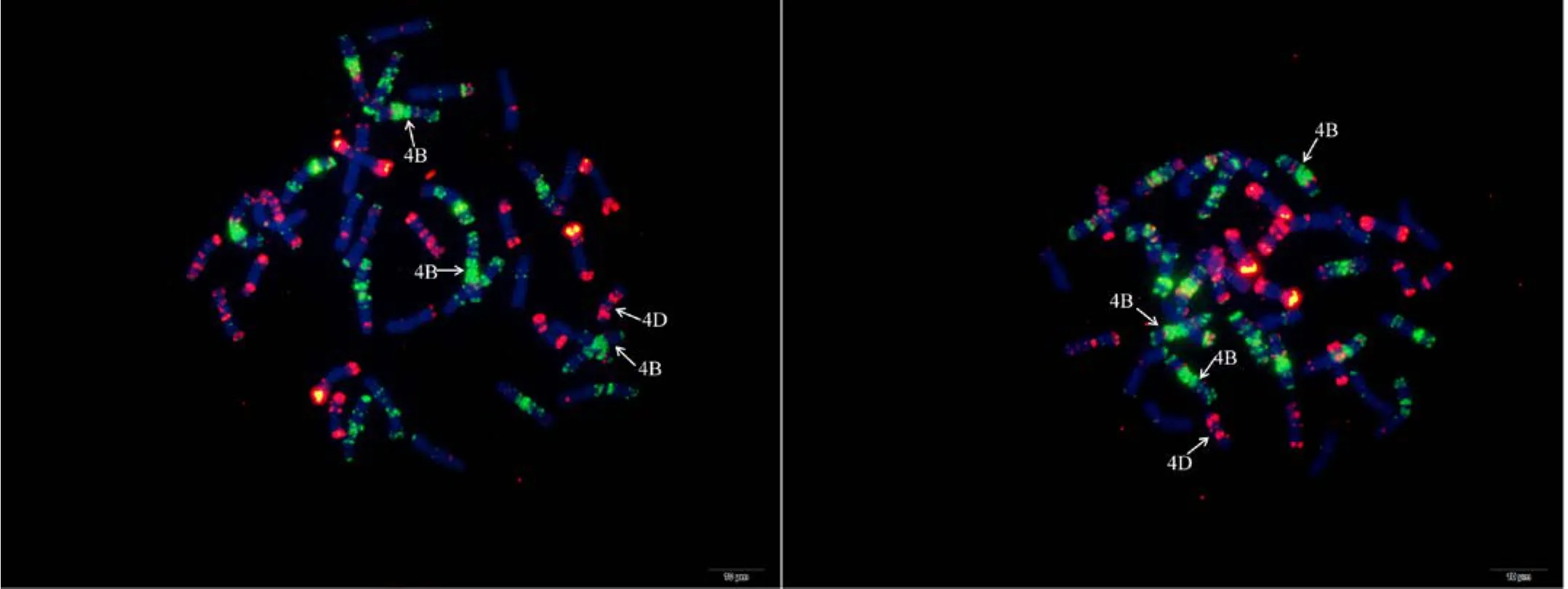
附图6.1 N4DT4B (包含1条4D和3条4B) Fig. S6.1 N4DT4B (with one 4D and three 4B)
附图6.2 N4DT4B (包含1条4D和3条4B) Fig. S6.2 N4DT4B (with one 4D and three 4B)
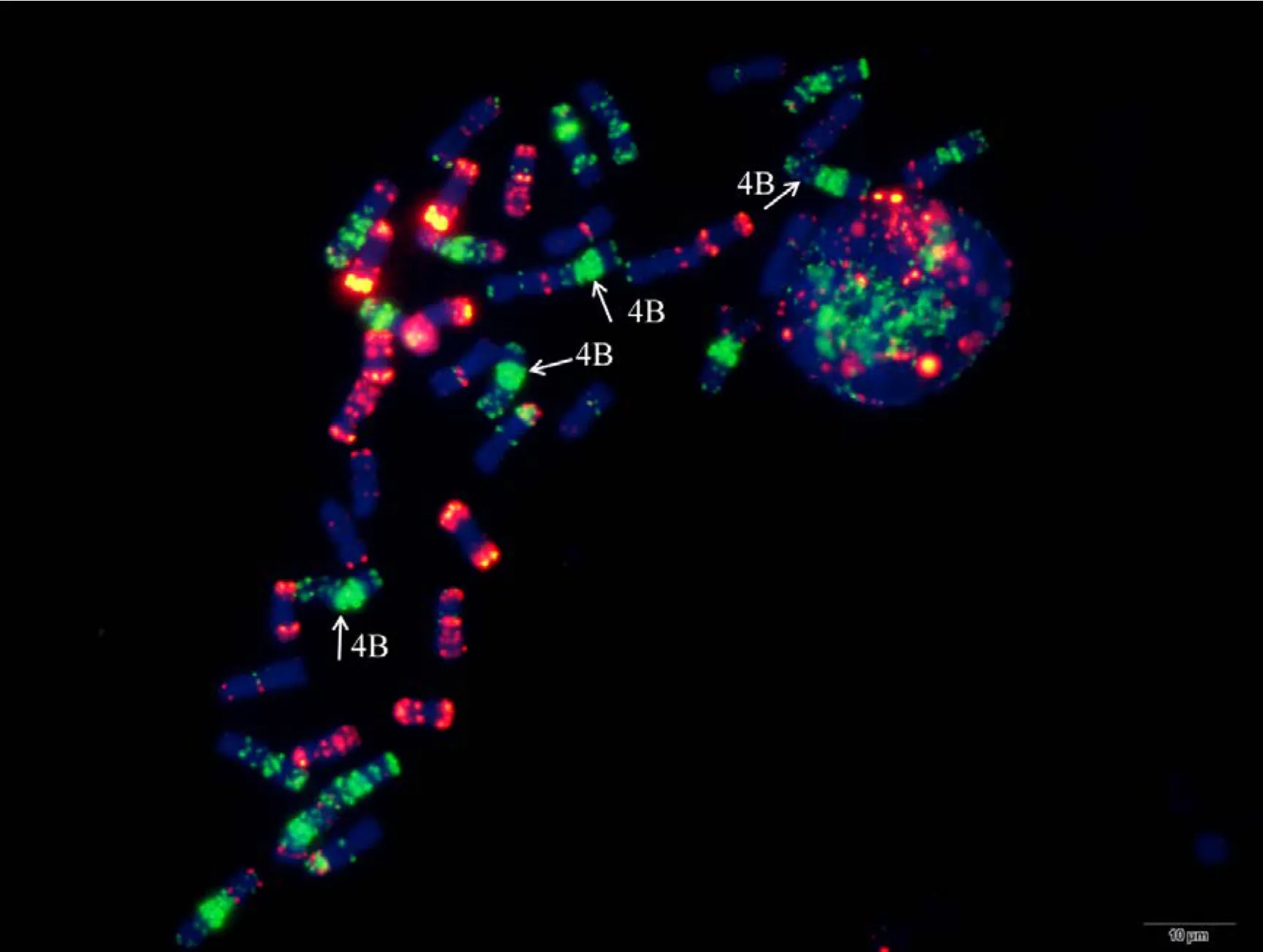
附图7 N4DT4B Fig. S7 N4DT4B
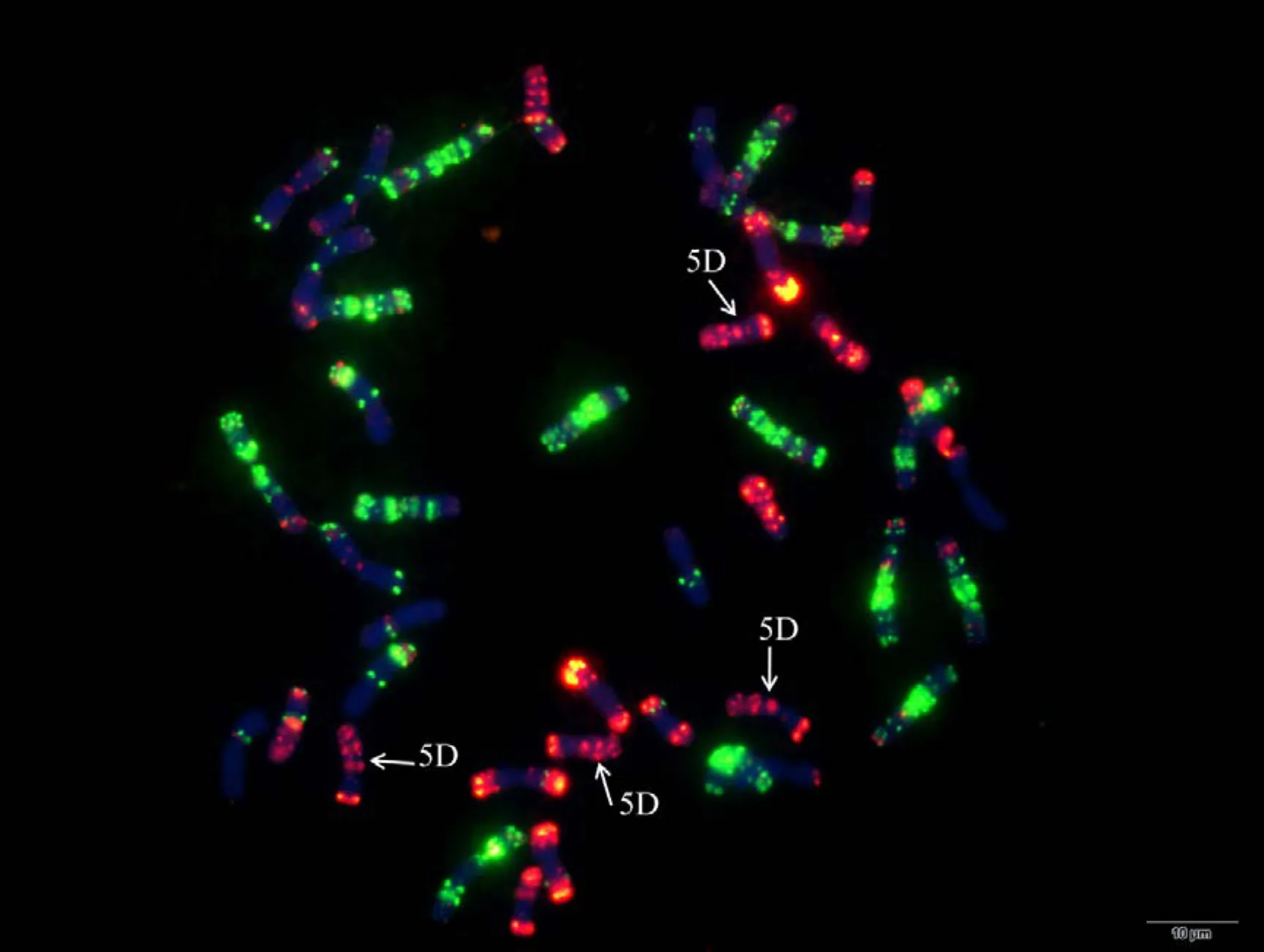
附图8 N5AT5D Fig. S8 N5AT5D
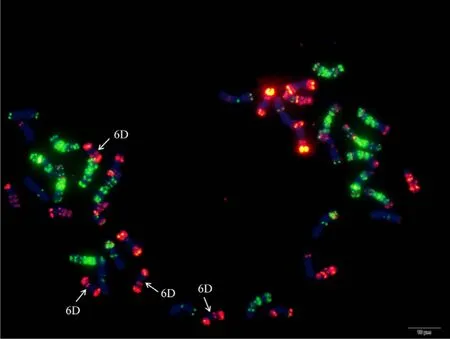
附图9 N6AT6D Fig. S9 N6AT6D
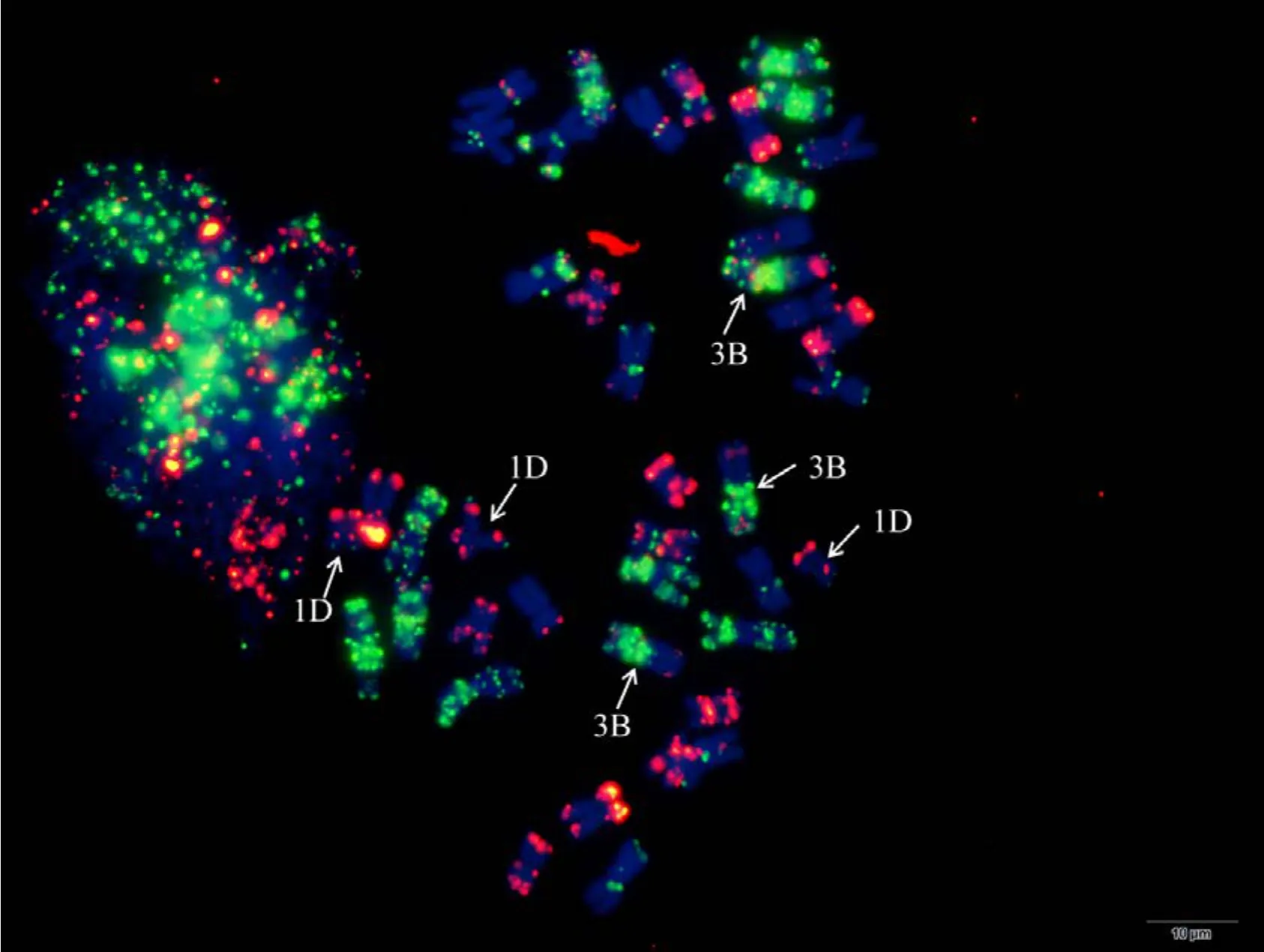
附图10 N6BT6A (缺2条6B、有3条1D和3条3B) Fig. S10 N6BT6A (absence of two 6B, with one 1D and three 3B)
附图11 Dt7AS Fig. S11 Dt7AS
附图12 N7AT7D Fig. S12 N7AT7D
附图13.1 N7BT7D Fig. S13.1 N7BT7D
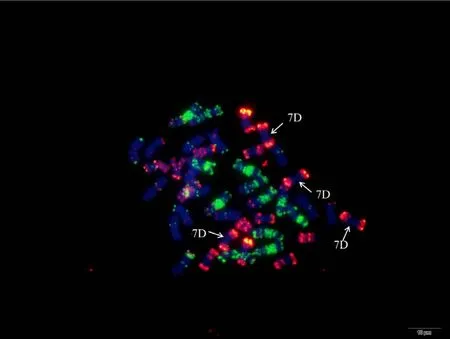
附图13.2 N7BT7D Fig. S13.2 N7BT7D

附图14 N7DT7B Fig. S14 N7DT7B
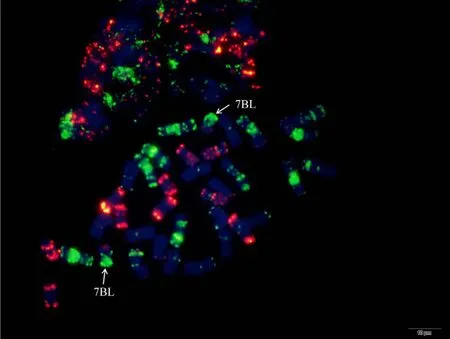
附图15.1 Dt7BL Fig. S15.1 Dt7BL
附图15.2 Dt7BL Fig. S15.2 Dt7BL
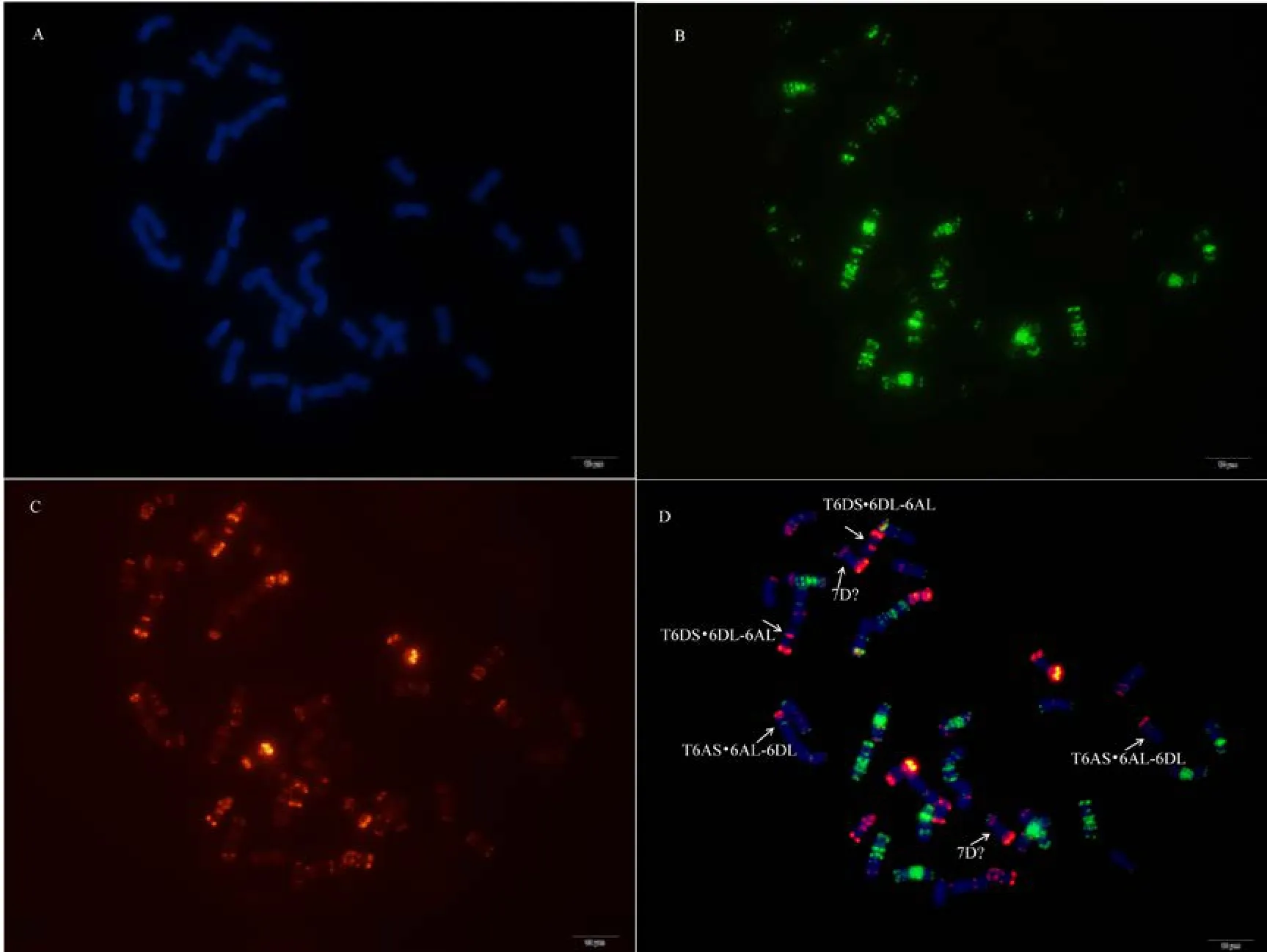
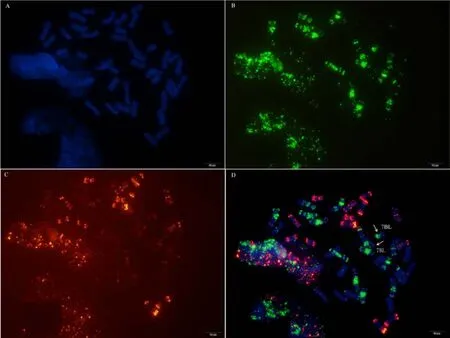
附图16.1 N5BT5D变异为相互易位T6AS·6AL-6DL和T6DS·6DL-6ALFig. S16.1 N5BT5D, a small segmental reciprocal translocation T6AS·6AL 6DL and T6DS·6DL-6AL occurred
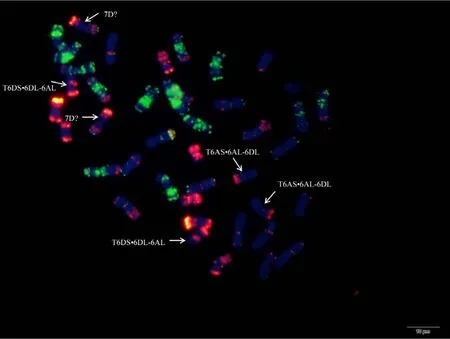
附图 16.2 N5BT5D (T6AS·6AL-6DL 和 T6DS·6DL-6AL)Fig. S16.2 N5BT5D (T6AS·6AL-6DL and T6DS·6DL-6AL)
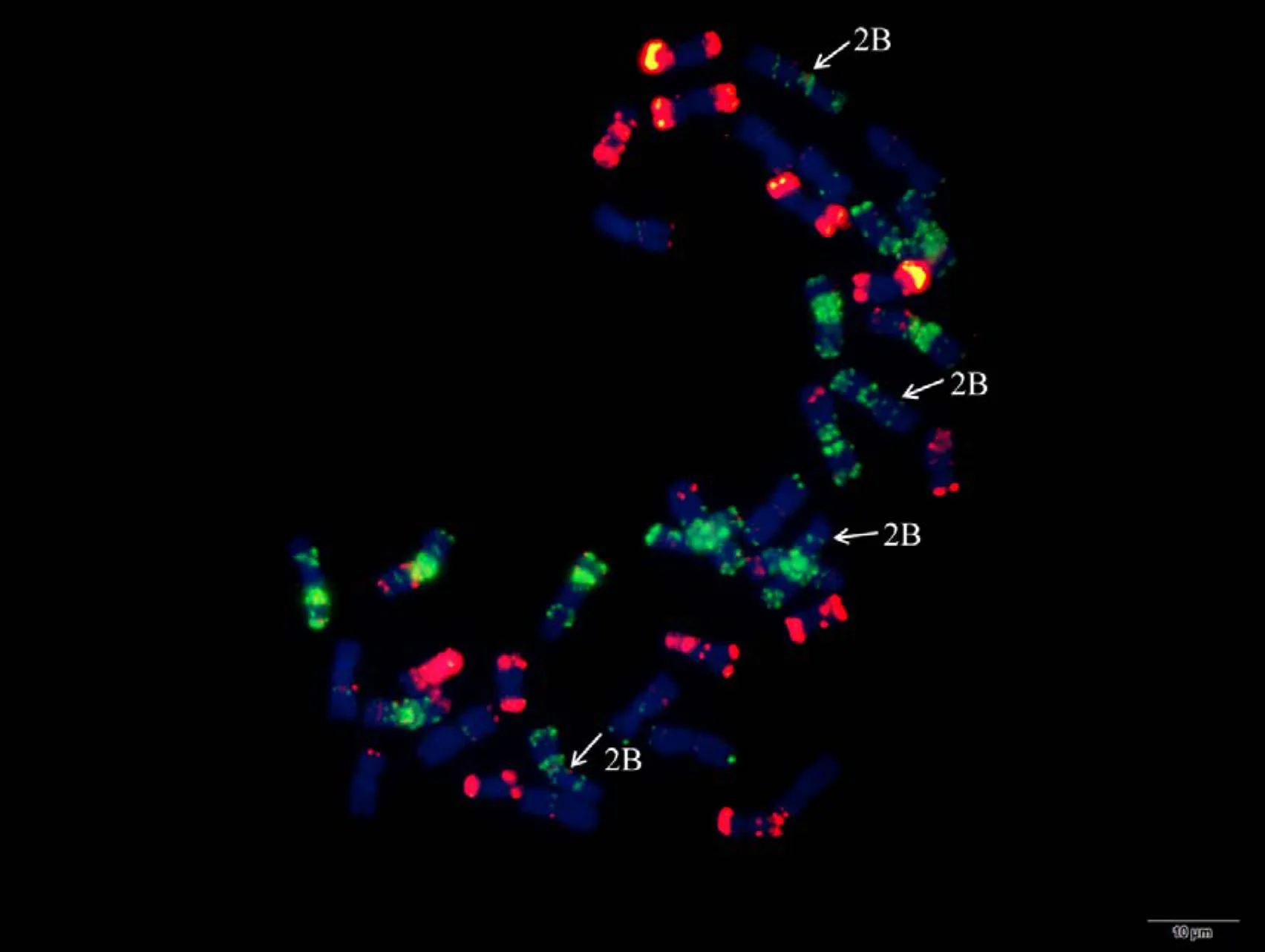
附图17 N2DT2B Fig. S18 N2DT2B
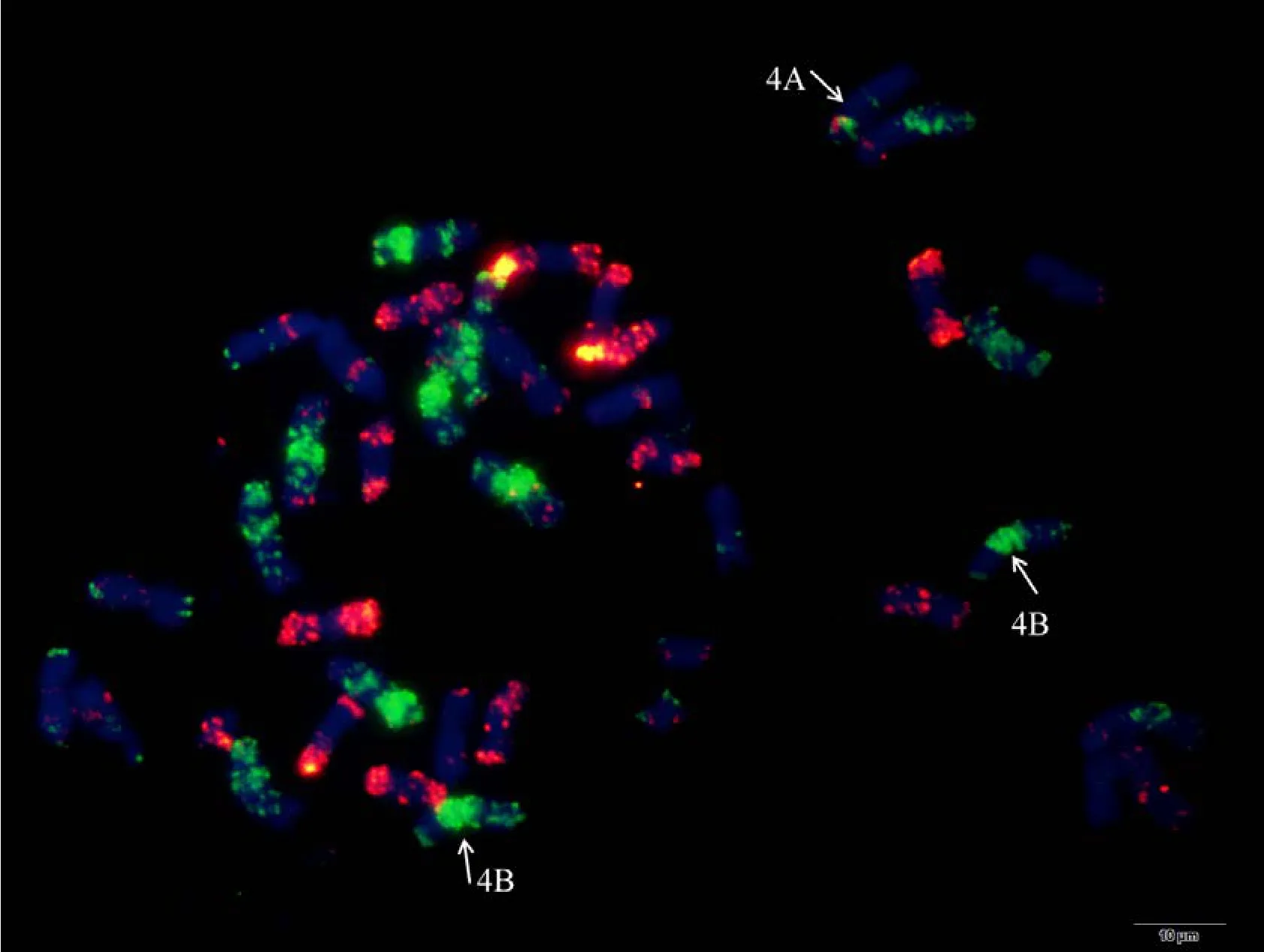
附图18.1 N4AT4B (无4A缺体和4B四体) Fig. S18.1 N4AT4B (nullisomic 4A and tetrasomic 4B not found)
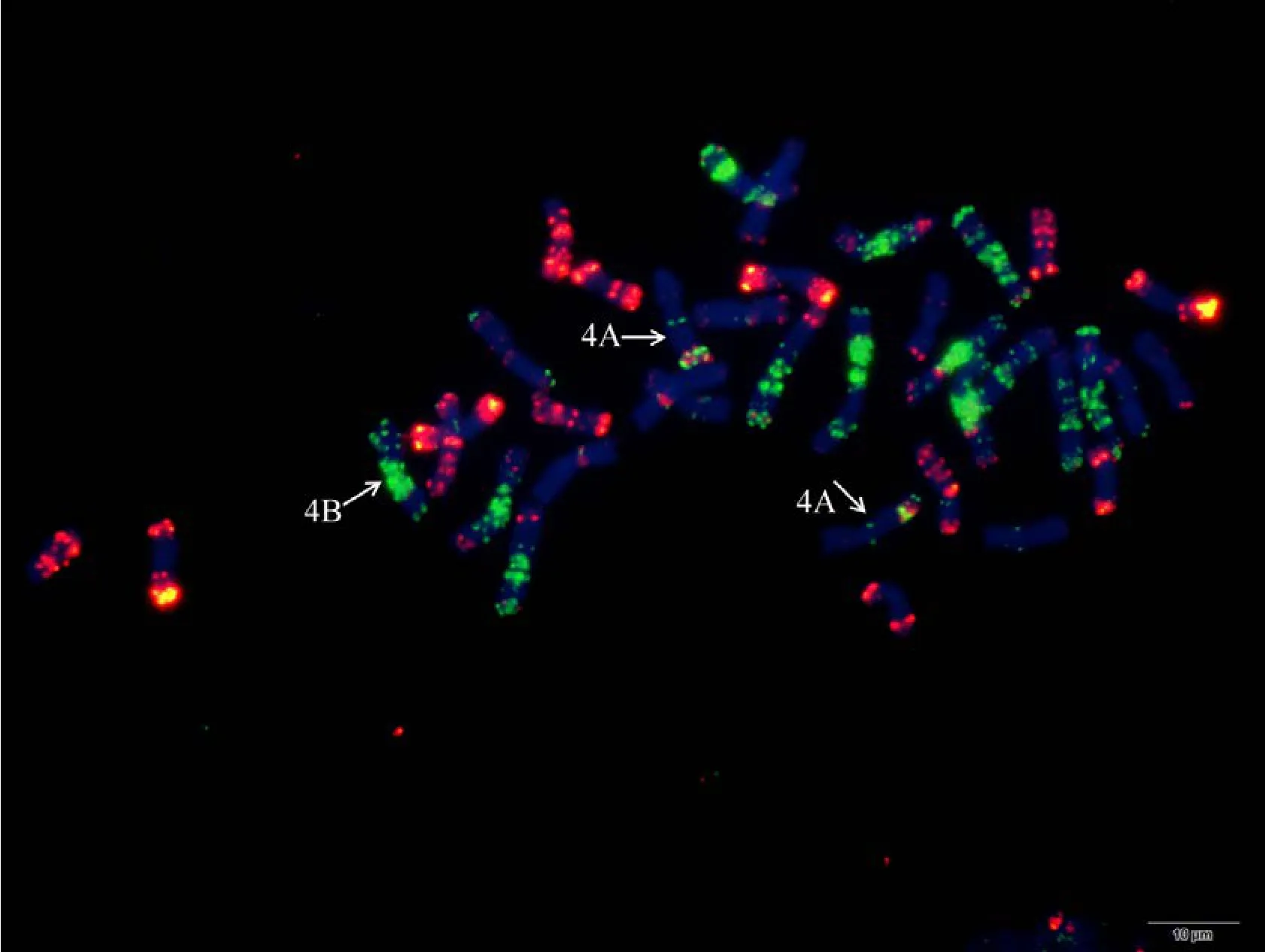
附图18.2 N4AT4B (无4A缺体和4B四体) Fig. S18.2 N4AT4B (nullisomic 4A and tetrasomic 4B not found)
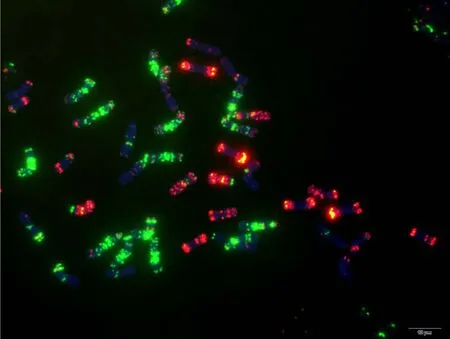
附图19 望水白 Fig. S19 Wangshuibai
附图20 和尚麦 Fig. S20 Heshangmai
附图21.1 赤面小麦 Fig. S21.1 Chimianxiaomai
附图21.2 赤面小麦 Fig. S21.2 Chimianxiaomai
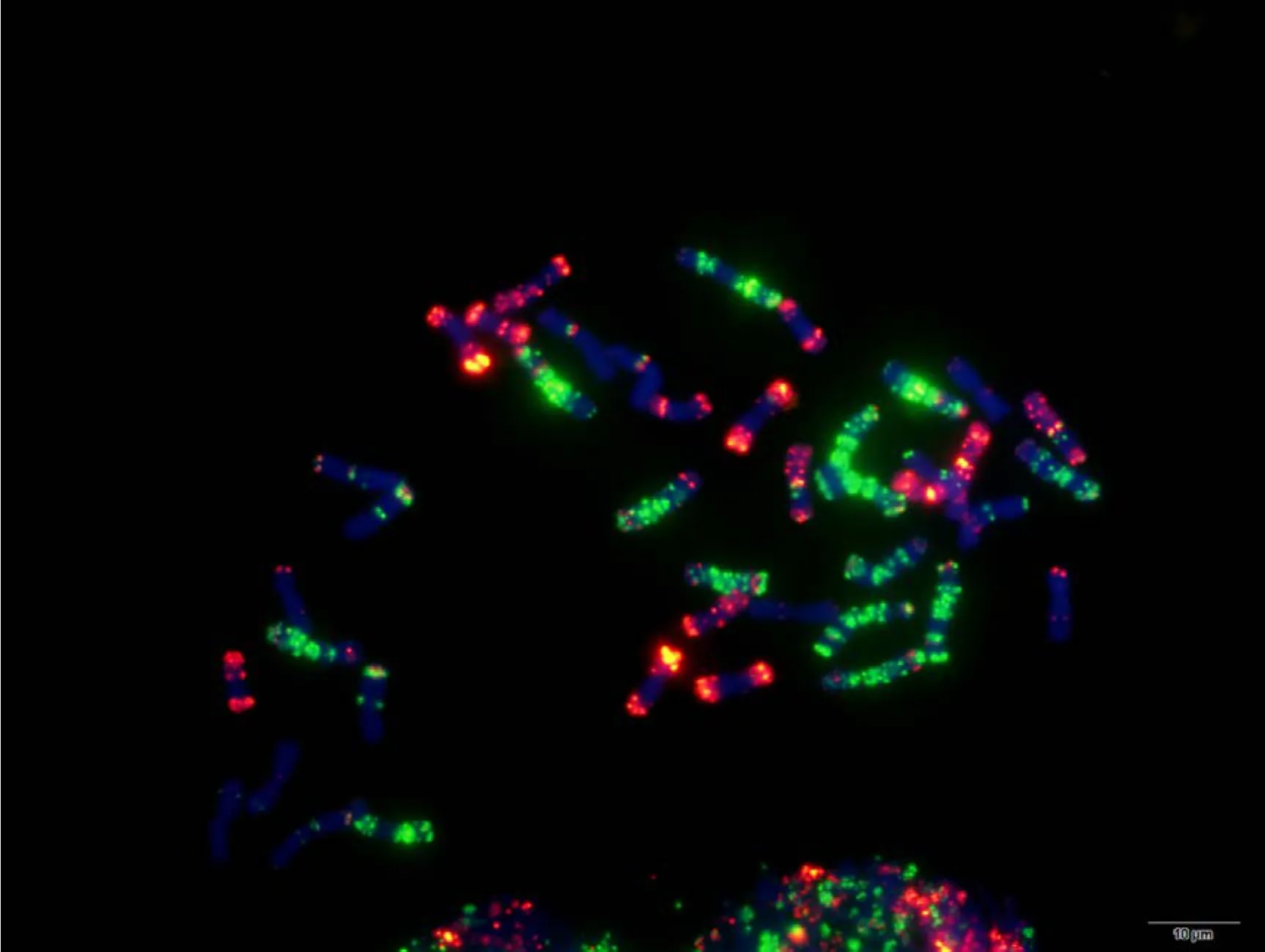
附图22.1 翻山小麦 Fig. S 22.1 Fanshanxiaomai
附图22.2 翻山小麦 Fig. S22.2 Fanshanxiaomai
附图23.1 红头麦 Fig. S23.1 Hongtoumai
附图23.2 红头麦 Fig. S23.2 Hongtoumai
附图24 蔷薇麦 Fig. S24 Qiangweimai
附图25.1 丝籽麦 Fig. S25.1 Sizimai
附图25.2 丝籽麦 Fig. S25.2 Sizimai
附图26.1 苏麦3号 Fig. S26.1 Sumai 3
附图26.2 苏麦3号 Fig. S26.2 Sumai 3
附图27.1 宁麦9号 Fig. S27.1 Ningmai 9
附图28.1 人工合成小麦 Fig. S28.1 Langdon/KU-2088 synthetic wheat
附图28.2 人工合成小麦 Fig. S28.2 Langdon/KU-2088 synthetic wheat
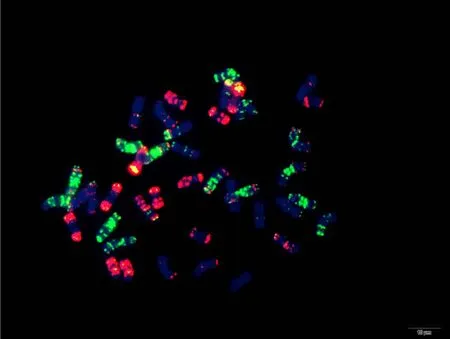
附图29.1 扬麦6号 Fig. S29.1 Yangmai 6
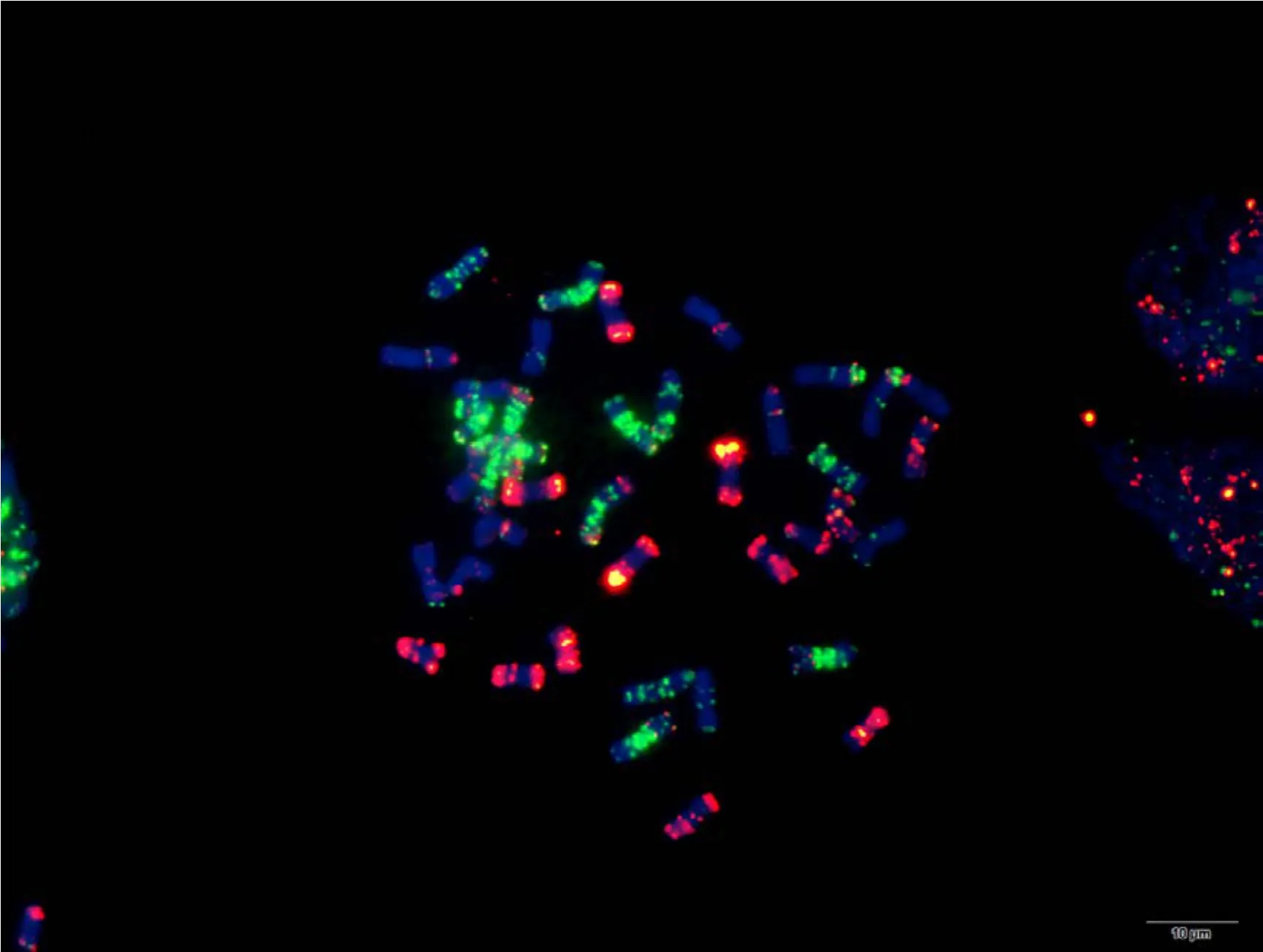
附图29.2 扬麦6号 Fig. S29.2 Yangmai 6
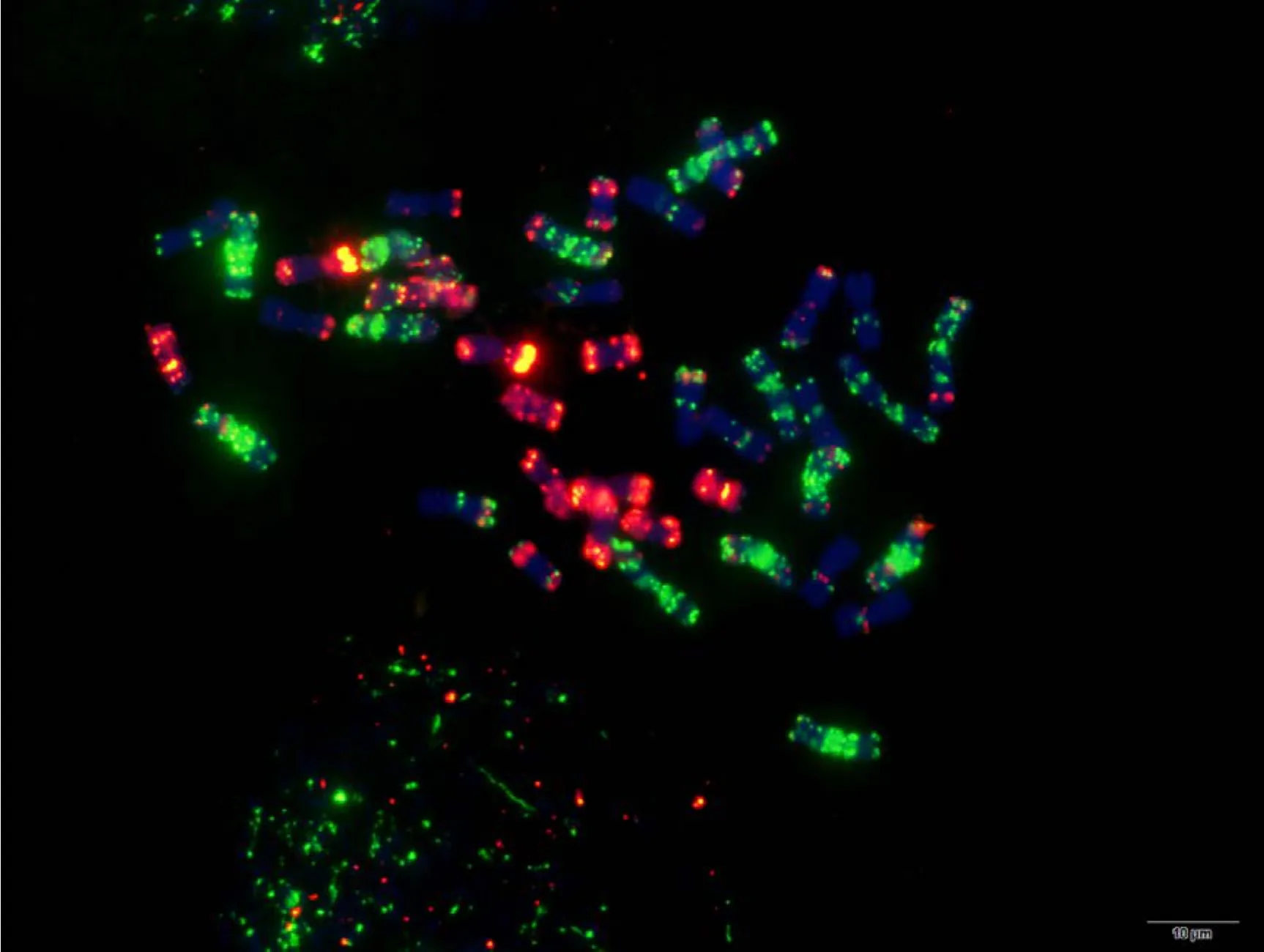
附图30.1 津强6号 Fig. S30.1 Jinqiang 6
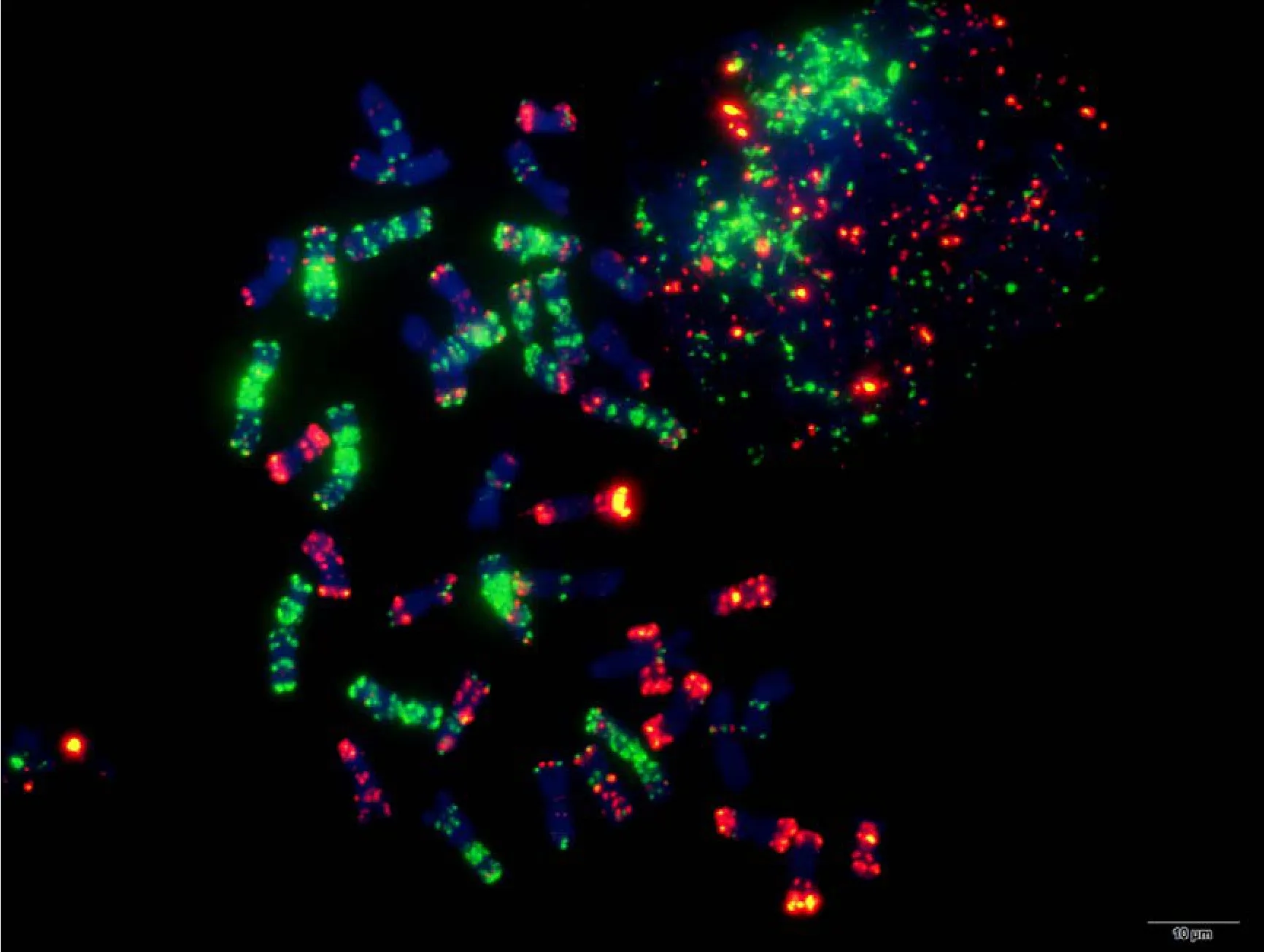
附图30.2 津强6号 Fig. S30.2 Jinqiang 6
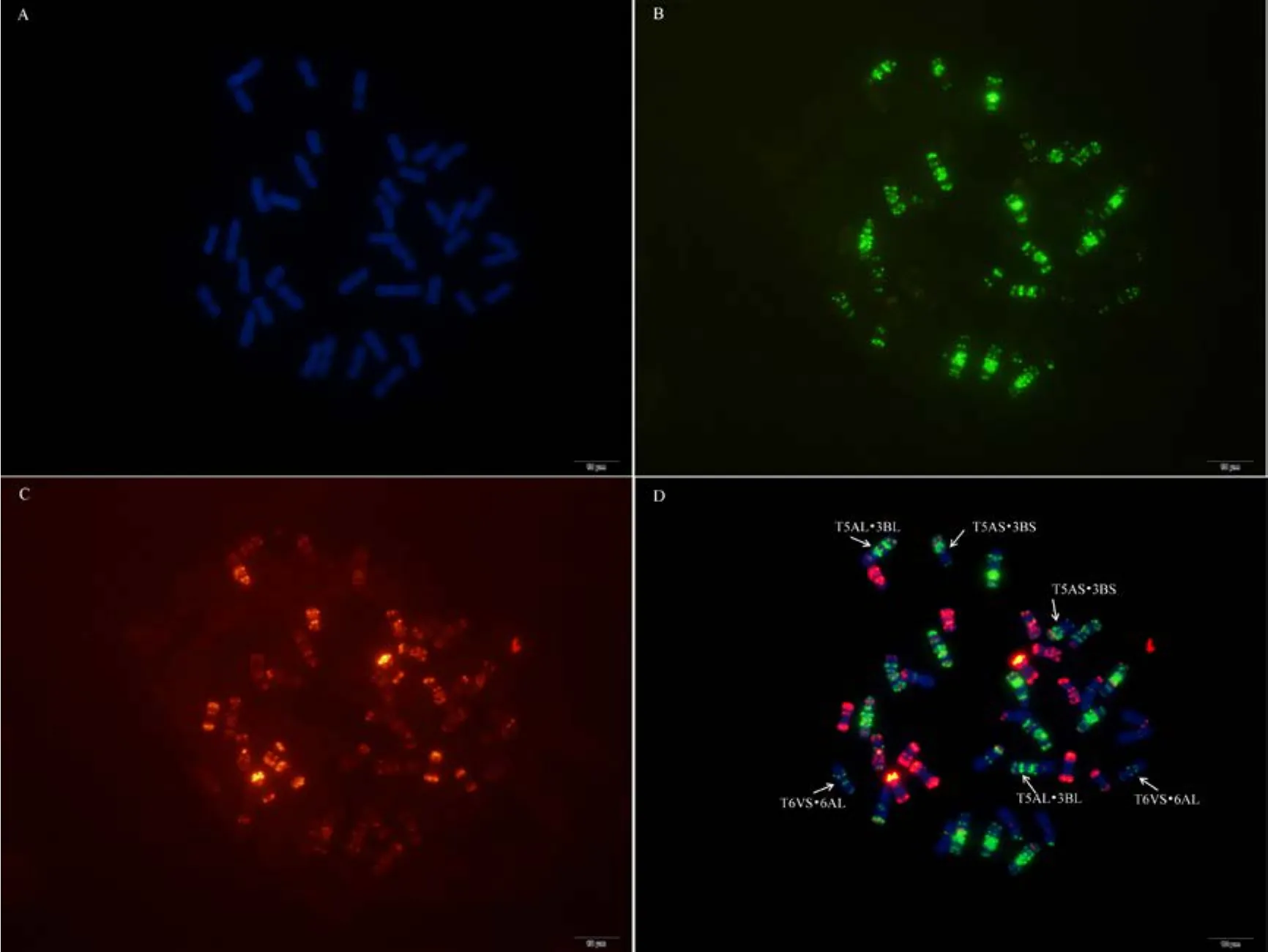
附图31.1 南农1258 Fig. S31.1 Nannong 1258
附图31.2 南农1258 Fig. S31.2 Nannong 1258
附图31.3 南农1258 Fig. S31.3 Nannong 1258
附图31.4 南农1258 Fig. S31.4 Nannong 1258
附图32.1 92R137 Fig. S32.1 92R137
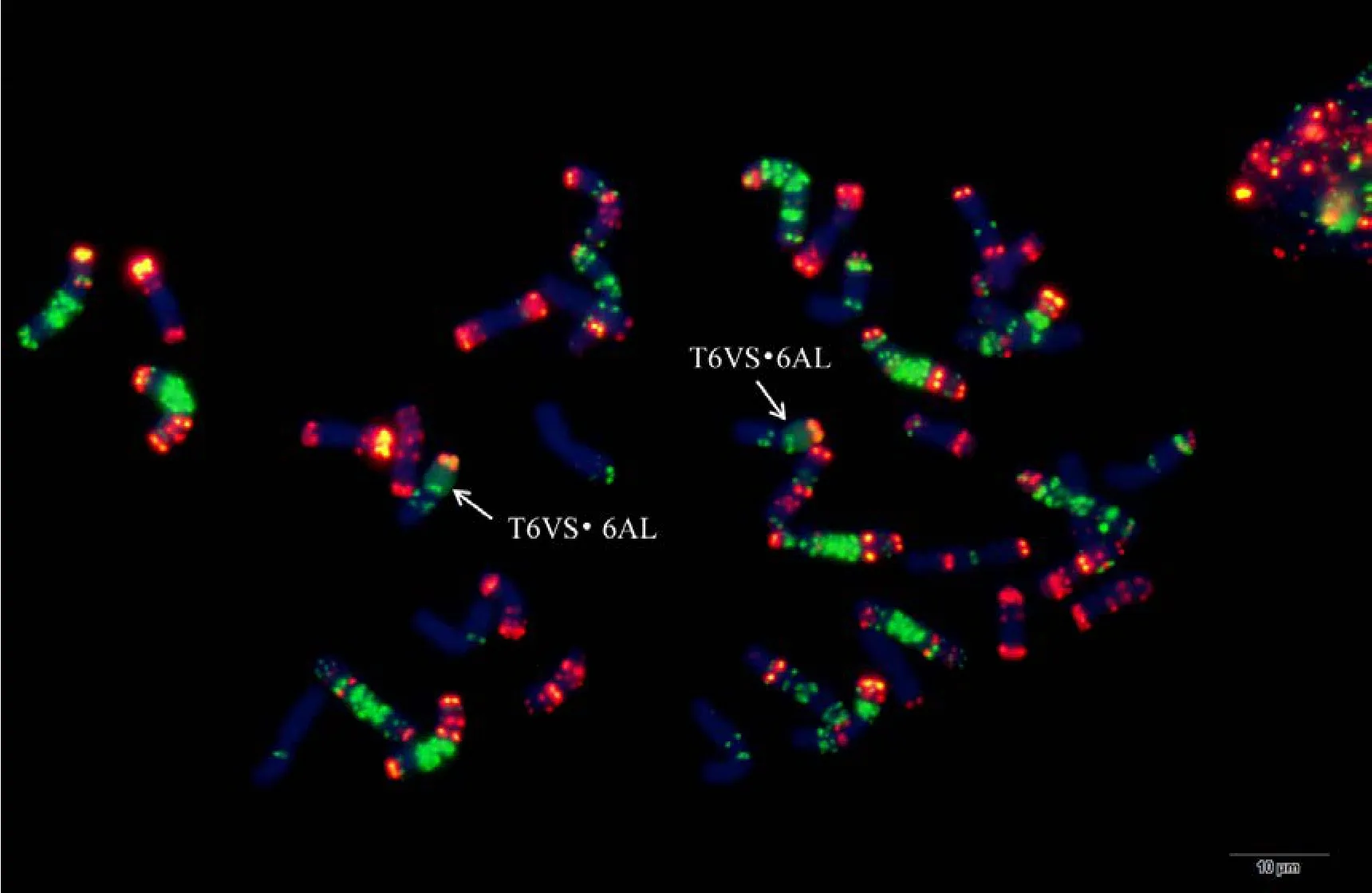
图32 .2 92R137 Fig. S32.2 92R137
附图33.1 周麦27 Fig. S33.1 Zhoumai 27
附图33.2 周麦27 Fig. S32.2 Zhoumai 27
附图34.1 原泛3号 Fig. S33.1 Yuanfan 3
附图34.2 原泛3号 Fig. S33.2 Yuanfan 3
附图34.3 原泛3号 Fig. S34.3 Yuanfan 3
附图34.4 原泛3号 Fig. S34.4 Yuanfan 3
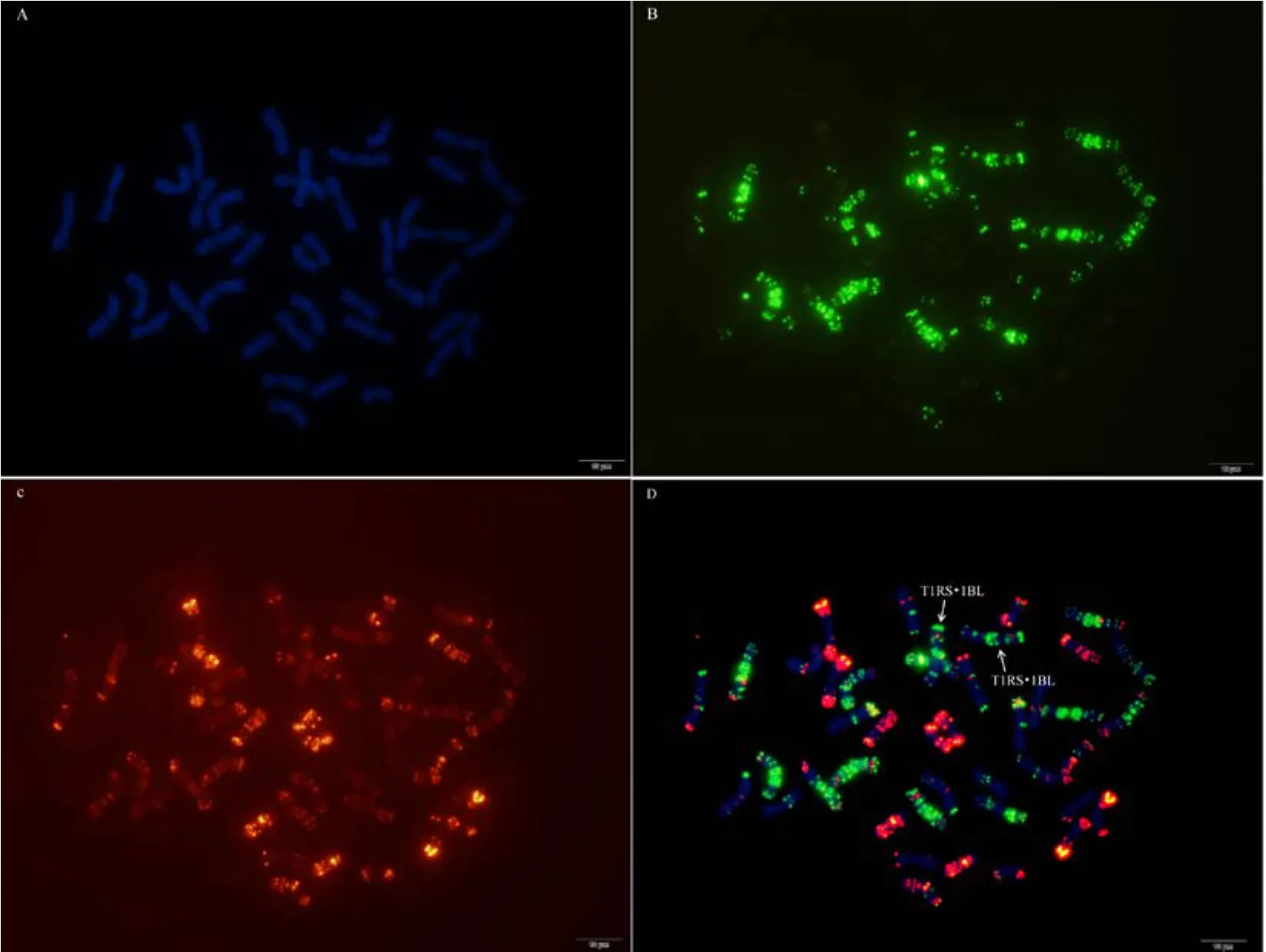
附图35.1 矮孟牛VII Fig. S35.1 Aimengniu VII
附图35.2 矮孟牛VII Fig. S35.2 Aimengniu VII
附图35.3 矮孟牛VII Fig. S35.3 Aimengniu VII
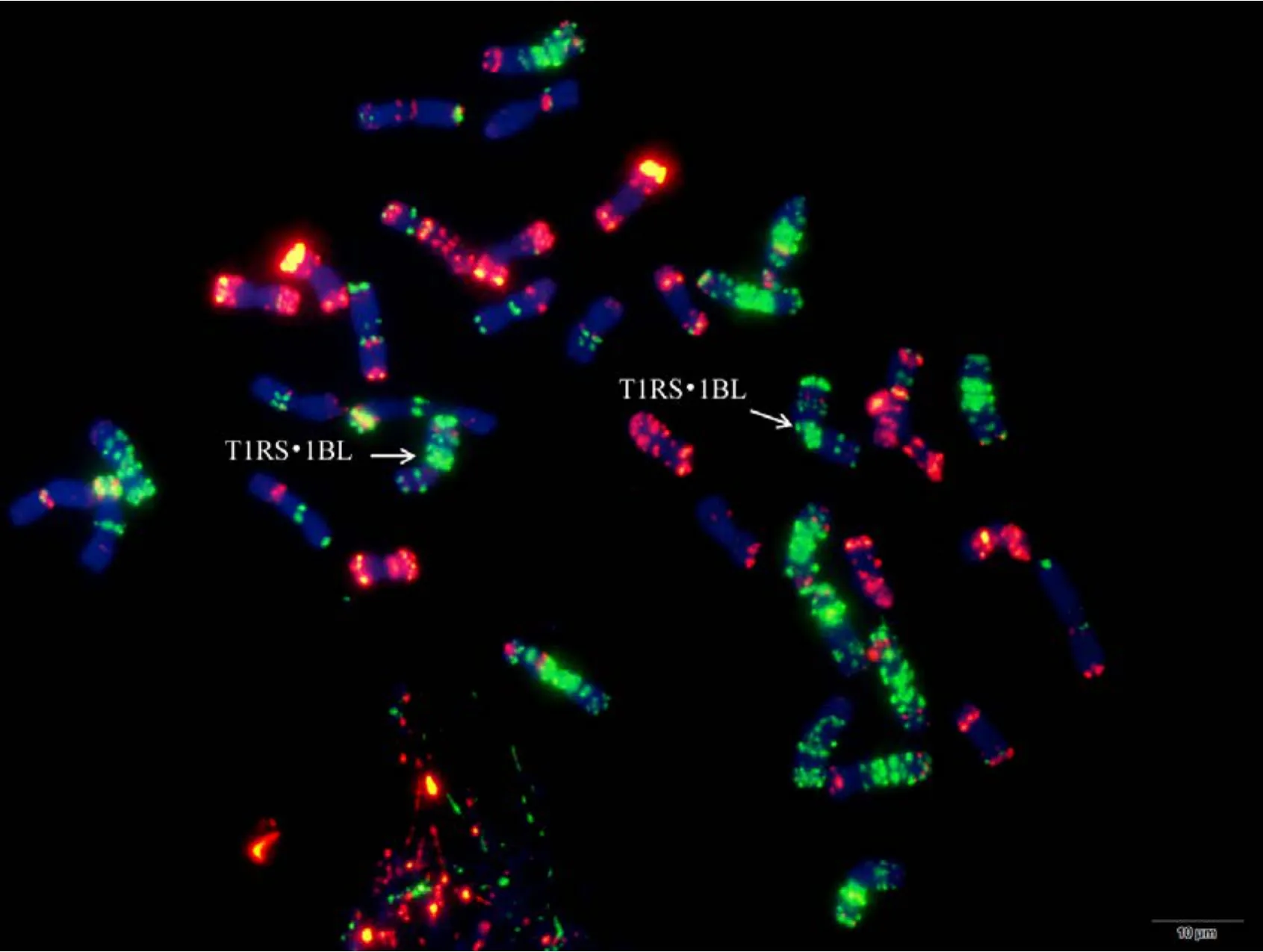
附图36.1 矮孟牛IV Fig. S36.1 Aimengniu IV
附图36.2 矮孟牛IV Fig. S36.2 Aimengniu IV
附图36.3 矮孟牛IV Fig. S36.3 Aimengniu IV
附图37.1 栽培一粒小麦 Fig. S37.1 Triticum monococcum
附图37.2 栽培一粒小麦 Fig. S37.2 Triticum monococcum
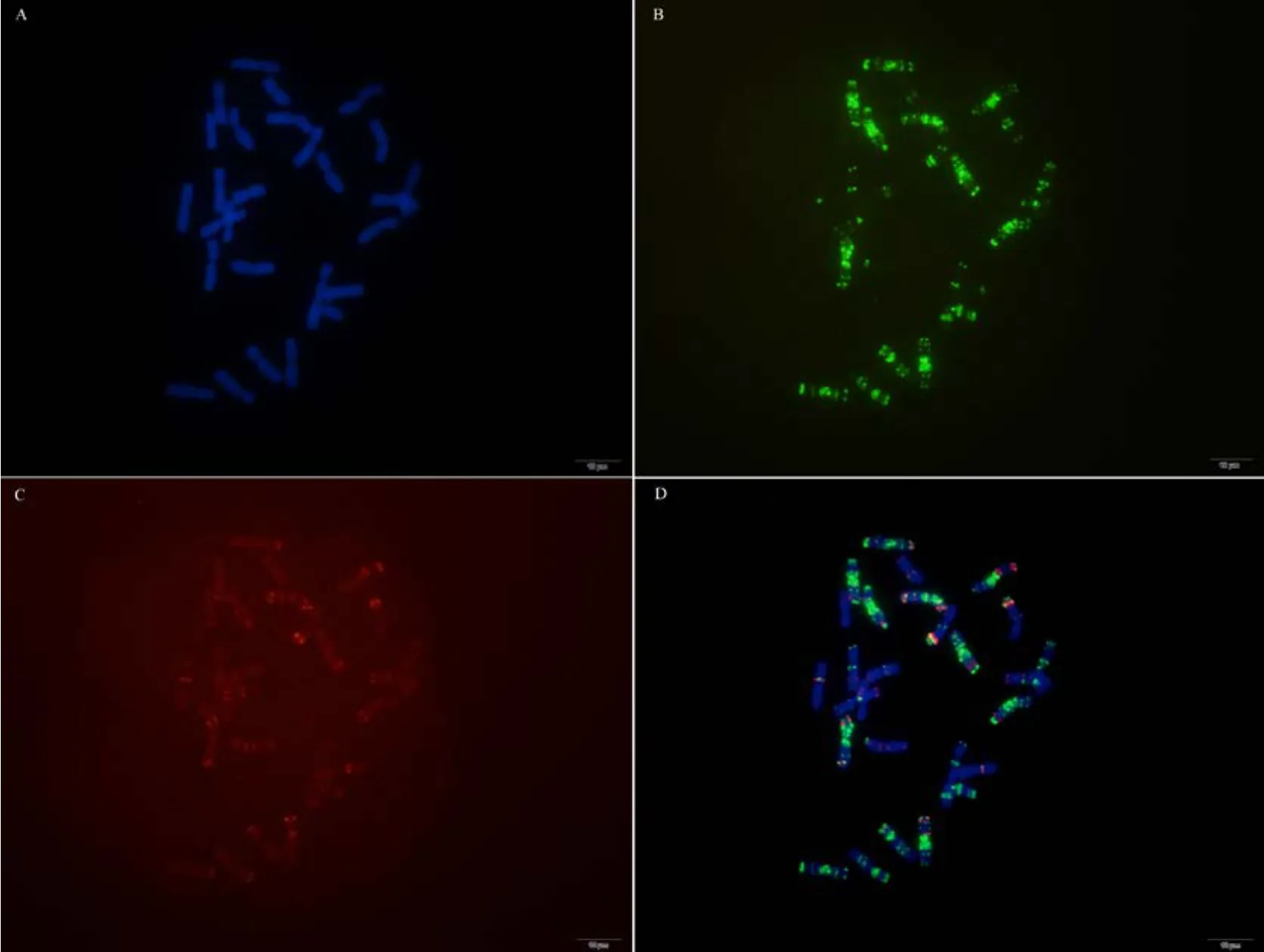
附图38 硬粒小麦Langdon Fig. S38 Langdon Triticum turgidum var. durum
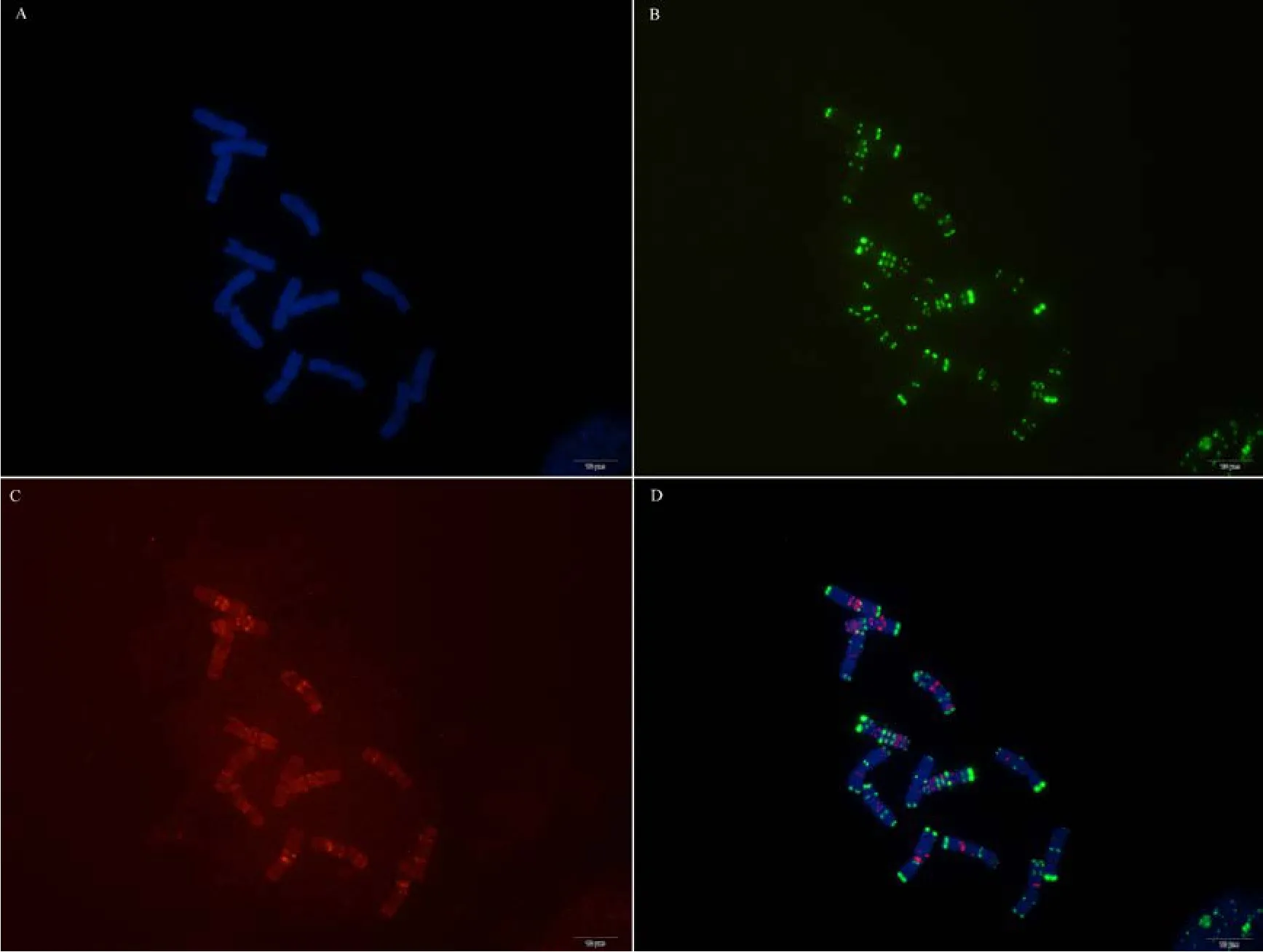
附图39.1 荆州黑麦 Fig. S39.1 Rye cultivar Jingzhouheimai
附图39.2 荆州黑麦 Fig. S39.2 Rye cultivar Jingzhouheimai
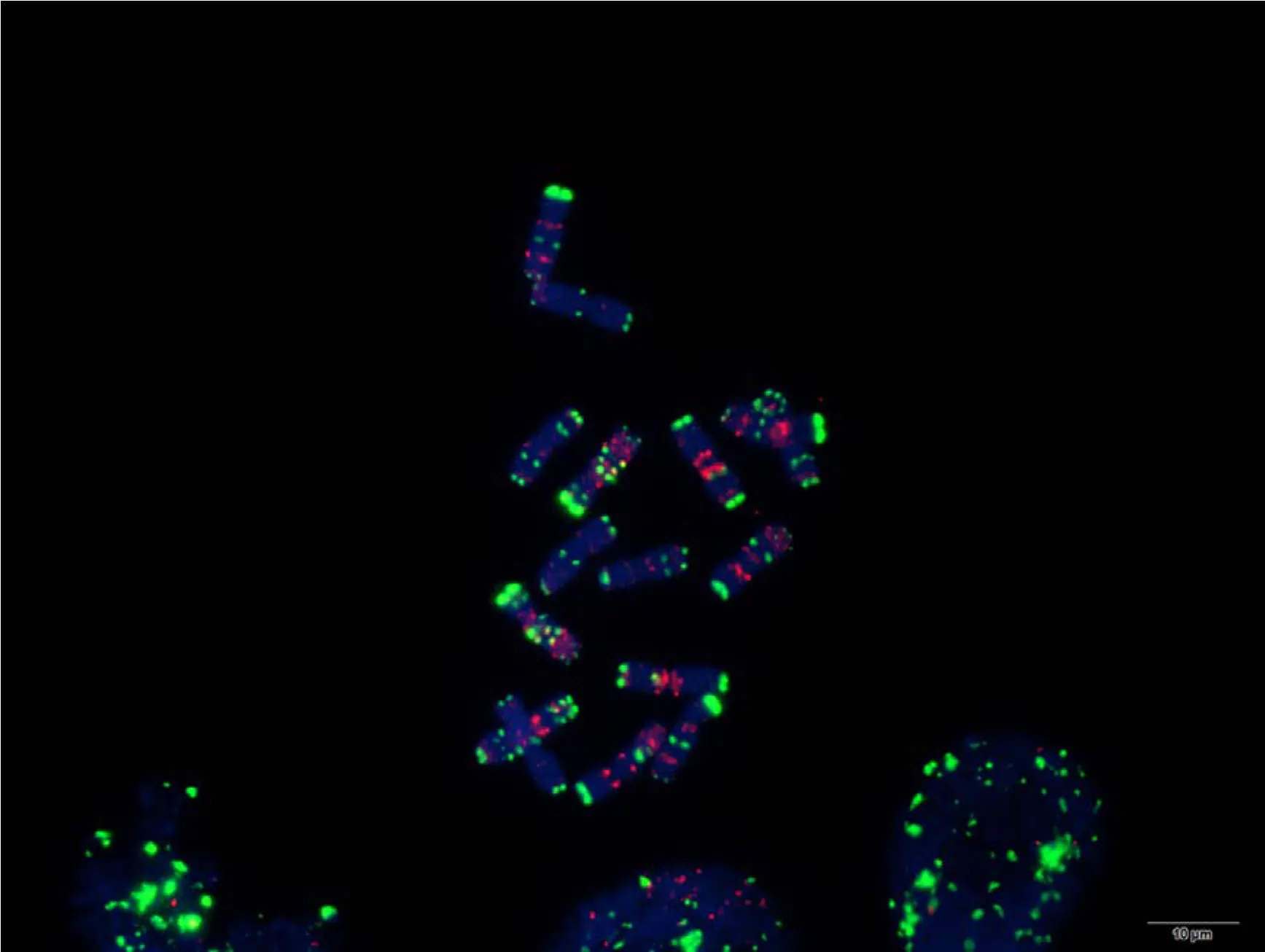
附图40.1 长穗偃麦草(2n = 2x = 14) Fig. S40.1 Thinopyrum elongatum (2n = 2x = 14)
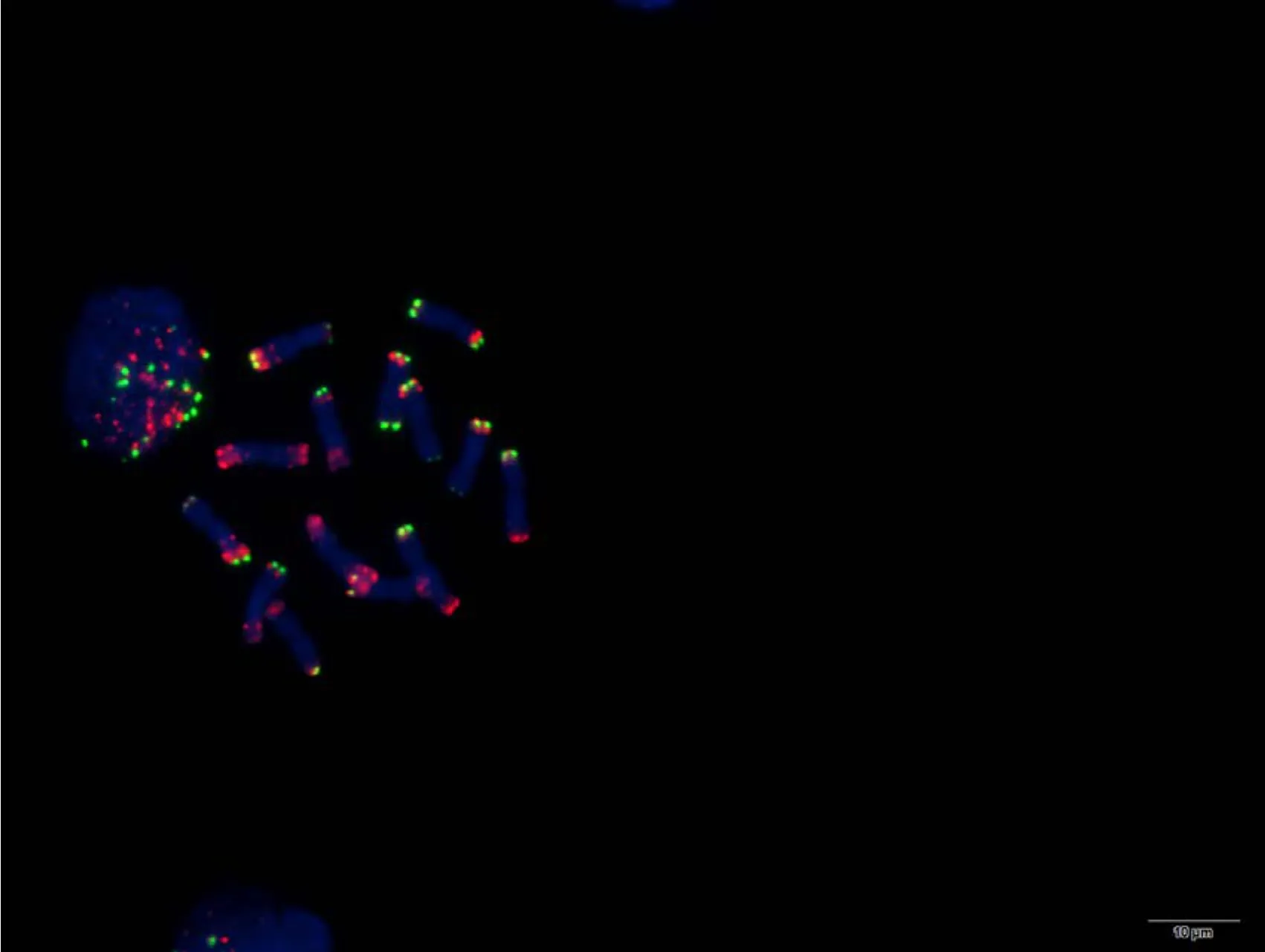
附图40.2 长穗偃麦草(2n = 2x = 14) Fig. S40.2 Thinopyrum elongatum (2n = 2x = 14)
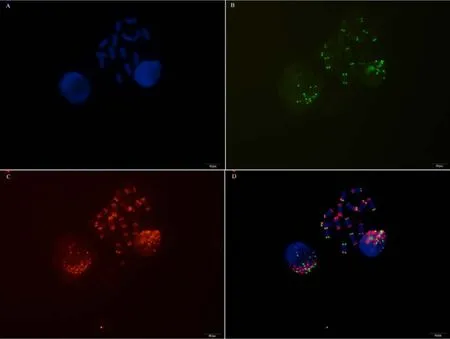
附图41.1 中间偃麦草 Fig. S41.1 Thinopyrum intermedium
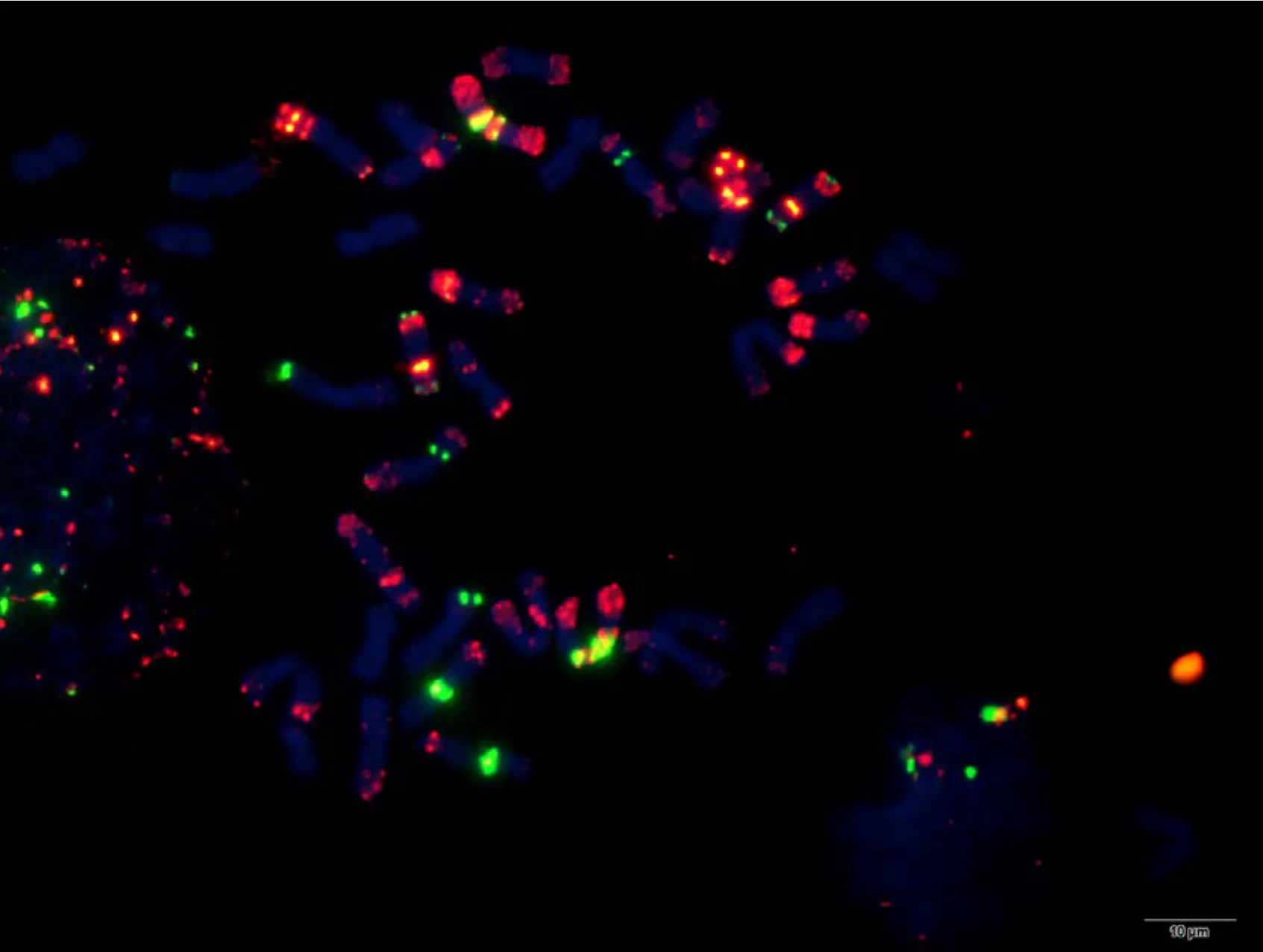
附图41.2 中间偃麦草 Fig. S41.2 Thinopyrum intermedium
10.3724/SP.J.1006.2017.01575
本研究由国家自然科学基金项目(31370385)和山西省农业科学院所长青年引导专项(yydzx09)资助。
This study was supported by the National Natural Science Foundation of China (31370385) and Institute Director Foundation of Shanxi Academy of Agricultural Sciences for Youth (yydzx09).
*通讯作者(Corresponding authors): 亓增军, E-mail: zjqi@njau.edu.cn; 庄丽芳, E-mail: lfzhuang@njau.edu.cn
联系方式: E-mail: 2014101112@njau.edu.cn
): 2017-03-06; Accepted(接受日期): 2017-07-23; Published online(网络出版日期): 2017-08-11.
URL: http://kns.cnki.net/kcms/detail/11.1809.S.20170811.1306.004.html

