不同氮磷钾处理大豆苗期主根长和侧根数的QTL定位分析
梁慧珍,董薇,许兰杰,余永亮,杨红旗,谭政伟,许阳,陈鑫伟
(1河南省农业科学院芝麻研究中心,郑州 450002;2南阳市农业科学院,河南南阳 473083;3商丘市农林科学院,河南商丘 476000)
不同氮磷钾处理大豆苗期主根长和侧根数的QTL定位分析
梁慧珍1,董薇1,许兰杰1,余永亮1,杨红旗1,谭政伟1,许阳2,陈鑫伟3
(1河南省农业科学院芝麻研究中心,郑州 450002;2南阳市农业科学院,河南南阳 473083;3商丘市农林科学院,河南商丘 476000)
【目的】主根长和侧根数是重要的根系性状。通过不同氮磷钾处理,发掘大豆苗期主根长和侧根数的基因资源、了解其遗传机制,定位其主效QTL,分析QTL间的上位性和环境互作效应,对生产提供理论指导。【方法】用以栽培大豆晋豆23为母本、山西农家品种灰布支黑豆(ZDD02315)为父本所衍生的447个RIL作为供试群体,取亲本及447个家系各30粒种子,用灭菌纸包裹后,2015年和2016年分别放置于CK(模拟种植不施肥)、NPK(模拟大田正常配施氮磷钾肥)和1.5NPK(模拟高肥田块)3种生长环境下进行水培试验,每组试验设置3次重复,环境温度20—28℃,幼苗长到V2期,对幼苗期相关根部性状数据进行测量。分别采用WinQTLCart 2.5和QTLNETwork 2.1 2种遗传模型检测QTL,分析QTL间的上位性和环境互作效应。【结果】基于复合区间作图(CIM)共检测到24个影响主根长和侧根数的QTL,分布于第2、3、5、6、7、8、9、10、11、12、13、14、16、17共14条染色体中,单个QTL的贡献率介于8.52%—43.62%,QTL主要表现为加性效应。基于混合线性模型(MCIM)检测到影响主根长和侧根数的QTL各1个,2个QTL均表现出加性效应和环境互作效应。另有2对主根长和2对侧根数均检测出加性×加性上位性互作QTL,主根长和侧根数各有1对表现出主效QTL与非主效QTL加性×加性上位性互作,各有1对表现出非主效QTL与非主效QTL加性×加性上位性互作,2对主根长互作QTL分别解释了1.53%和1.95%的表型变异率,2对侧根数互作QTL分别解释了2.47%和1.13%的表型变异率。2个QTL能在2种分析方法中同时检测到,9个QTL能在3种环境下同时检测到。第6染色体在2015年NPK、1.5NPK和2016年1.5NPK 3个环境下均检测到主根长QTL,第5染色体在2015年NPK和1.5NPK、2016年CK 3个环境下、第17染色体在2015年CK和NPK、2016年NPK 3个环境下均检测到侧根数QTL。【结论】苗期大豆主根长和侧根数对氮磷钾的吸收影响较少,生产中尽可能减少氮磷钾使用量。不同浓度氮磷钾处理苗期主根长和侧根数参数间既有共同的控制基因,也有各自独特的控制基因,多数QTL不能在多个环境下重复检测到,控制其表达的遗传机制较为复杂。加性效应、加性与环境互作和加性×加性上位性互作效应在主根长和侧根数的形成和遗传中发挥着重要作用。主根长和侧根数各有1个QTL能在2种分析方法中同时检测到,Satt442-Satt296和Satt521-GMABABR是共位标记区间。
大豆;氮磷钾;主根长;侧根数;QTL;上位性互作
Abstract:【Objective】Main root length (MRL) and lateral root number (LRN) are important root traits. It is important todevelop the gene resources and reveal the genetic mechanisms of MRL and LRN, and identify quantitative trait loci (QTL) associated with root traits in soybean, including main-effect QTLs, epistatic effects and QTL × environment interactions, meanwhile, map the main-effect QTLs, epistatic effects and QTL × environment interactions in different N, P and K environments. 【Method】A total of 447 RILs derived from a cross between cultivated Jindou23 as the female and native variety HuibuzhiZDD02315 as the male were used as materials. Thirty seeds from each of the RILs and their parents were covered with pasteurized paper, and cultivated in CK (nonfertilized condition), NPK (normal fertilization conditions) and 1.5NPK (high fertilization conditions) at 20-28℃ in 2015 and 2016, and a complete random design with three replications was used in this study. Root traits were measured at V2 stage. Epistatic QTLs and QTL ×environment interactions were performed using WinQTLCart 2.5 and QTLNETwork 2.1. 【Result】Twenty-four QTLs for MRL and LRN were detected on chromosomes 2, 3, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 16 and 17 using CIM method in this study. The variation accounted for by each of these twenty- four QTLs ranged from 8.52% to 43.62%. These QTLs showed additive effect. Two QTLs for MRL and LRN were detected by MCIM, which showed an additive effect. Another two pairs of additive × additive epistatic effects QTLs for MRL and LRN were detected, including one pair of major QTLs and non-major QTL additive × additive epistatic effects, and one pair of non-major QTLs and non-major QTL additive × additive epistatic effects. Two pairs of QTL interaction for MRL explained 1.53% and 1.95% of the phenotypic variation, and two pairs of QTL interaction for LRN explained 2.47% and 1.13% of the phenotypic variation. Two QTLs were simultaneously detected on the same chromosome using two WinQTLCart 2.5 and QTLNETwork 2.1. Nine QTLs were simultaneously detected in three environments. The QTL for MRL was all mapped on chromosome 6 in 2015 (including NPK and 1.5NPK) and 2016 (including 1.5NPK). The QTL for LRN was all detected on chromosome 5 in 2015 (including NPK and 1.5NPK) and 2016 (including CK), another QTL for LRN was all mapped on chromosome 17 in 2015 (including CK and NPK) and 2016 (including NPK). 【Conclusion】MRL and LRN absorb less NPK at seedling stage in soybean, so farmers should minimize the use of NPK in agricultural production. MRL and LRN were controlled by the same controlled gene and specific gene in NPK treatments.Some QTLs were not simultaneously identified in different NPK environments as the related genetic mechanism is comparatively complex. Additive effects, additive × environment interactions and additive × additive epistatic effects are important genetic factors in MRL and LRN formation and inheritance. One each QTL for MRL and LRN was all detected by CIM and MCIM; one stable gene for MRL and LRN existed in interval markers between Satt442-Satt296 and Satt521-GMABABR.
Key words:soybean; nitrogen, phosphorus and potassium; main root length; lateral root number; quantitative trait loci;epistatic effects
0 引言
【研究意义】大豆属于直根系作物,根系主要由主根和侧根组成。大豆根系是其与外界环境之间进行物质交换的主要器官,与植物营养有着密切的关系,对产量和逆境胁迫有着重要影响[1]。根系是数量性状,大量基因控制着根系生长发育和养分的吸收利用[2-3],外部水分和养分环境对大豆根系生长具有重要的影响[4-6]。氮磷钾对植物的生长发育具有重要作用,任何一种元素的缺乏都会对影响到植物的正常生长发育[7-8]。土壤中氮磷钾元素的缺乏或者不均衡,已经严重影响到作物产量的提高[9-11],通过施肥补充土壤中的氮磷钾养分供应是提高大豆产量的重要措施。但是,过度使用化肥会对农业生态环境带来严重的破坏。因此,研究不同氮磷钾处理大豆苗期主根长和侧根数的QTL定位与上位互作分析对开展大豆养分高效利用和分子标记辅助选择育种具有重要意义。【前人研究进展】目前,国内外学者对氮磷钾养分吸收利用和根系性状QTL定位研究主要集中在水稻[12-14]、玉米[15-17]、小麦[18-20]等作物,已经定位出多个根系性状的QTL。以往对大豆根系的研究主要集中在根系形态[21]、生理特性[22]和耐逆性[23-24]等方面,对不同浓度氮磷钾处理根系性状的QTL定位分析研究较少。朱向明等[25]研究了供氮水平对苗期大豆根系吸水特性的影响;ZHANG等[26]以不同磷效率大豆种质组成的自然群体为材料,应用全基因组关联分析,结合大豆基因组信息及低磷胁迫诱导表达,筛选出一个酸性磷酸酶基因(GmACP1);苏辉等[27]利用铁-7555和凤59-15的RIL群体,对相对根系干质量、相对根系磷含量、相对株高和相对叶中磷含量4个性状进行分析,在3和10染色体上共检测到 3个与耐低磷有关的 QTL,可解释表型变异的4.84%—18.2%;BEEBE等[28]在大田 2种磷水平和营养液低磷处理下对根系性状进行了QTL检测,大田2种磷水平下分别检测到8个和6个与磷积累、根长和比根长性状相关的QTL;营养液低磷处理下,检测到12个与根长、主根长、根干重、主根干重和比干重性状相关的 QTL;LIANG等[29]进行了幼苗期大豆根系性状的遗传分析与QTL检测研究,共检测到24个与主根长、侧根数、根重、根体积、茎叶重、下胚轴长和下胚轴重相关的QTL,分别位于第2、3、5、6、7、8、9、10、11、12、13、14、18、19和20连锁群上。【本研究切入点】由于根系生长在地下,生长环境比较复杂,截至目前,对于不同浓度氮磷钾处理条件下,大豆主根长和侧根数的QTL定位分析研究较少。【拟解决的关键问题】以栽培大豆晋豆23和山西农家品种灰布支黑豆(ZDD02315)为母本和父本衍生出的447个RIL群体为试验材料,采用WinQTLCart 2.5[30]中复合区间模型分析方法(http://statgen.ncsu.edu/qtlcart/WQTLCart.htm),对不同氮磷钾处理大豆苗期主根长和侧根数进行QTL定位分析,采用QTLNETwork 2.1[31]混合线性模型分析方法,分析 QTL间的上位性和环境互作效应,为开展大豆养分高效利用和分子标记辅助选择育种提供依据。
1 材料与方法
1.1 供试材料与水培试验
以晋豆23为母本,灰布支黑豆(ZDD02315)为父本所衍生的RIL群体作为供试群体(晋豆23是山西省主栽大豆品种,灰布支黑豆是山西农家品种,2个品种性状差别较大)。参照周蓉等[32]的方法,取亲本及447个家系各30粒种子,用灭菌纸包裹后于2015年5月27日和2016年6月10日分别放置在CK、NPK、1.5NPK各3个环境中进行水培12 d,幼苗长到V2期。根据国家潮土土壤肥力与肥料效益长期监测站(郑州)对不同肥力田块的施肥标准,设置水培试验不同处理的配比(表 1),CK模拟种植不施肥(蒸馏水)、NPK模拟大田正常配施氮磷钾肥、1.5NPK模拟高肥田块,这3种环境更加贴近实际大田施肥情况,研究结果更加有利于解决实际生产施肥问题。每组试验设置3次重复,种子萌发和幼苗生长期间置室外环境,环境温度为20—28℃。

表1 水培试验不同处理的配比Table 1 Different material ratios in the hydroponic experiment
1.2 根系性状测定和表型数据处理
当幼苗生长至V2期时,每个家系均选取5株生长一致的幼苗,洗净吸干后剪取根系部分,分别于2015年6月8日和2016年6月22日对各环境下幼苗期主根长(main root length,MRL)和侧根数(lateral root number,LRN)进行人工测定。用直尺测量每个单株根系的主根长,对侧根数直接计数并计算5株幼苗试验数据的平均值。利用SPSS 19.0软件进行表型数据分析并计算遗传力h2=V(G)/V(P)。
1.3 遗传连锁图谱的构建
采用单粒混传法(single seed multiple descent,SSD)构建作图群体RIL。1998年夏季在山西省以晋豆23为母本,灰布支黑豆(ZDD02315)为父本进行杂交,获得 F1籽粒。种植 F1,收获一株籽粒产量为732粒的F1单株,1998年10月在海南岛单粒种成732个F2,1999年2月单株收获每个F2形成F3株系。于F3开始,每个世代每个株系随机收获10株上的共20个豆荚,每株2个,共约40粒籽粒,随机选取其中的20粒种成20个株行,形成下一个世代,以此类推至高世代。2004年,王珍[33]以晋豆 23×灰布支黑豆(ZDD02315)及其所衍生的F12重组自交系为材料,构建了包含 227个 SSR标记的大豆遗传连锁图谱。2006年,梁慧珍[34]对图谱进行了重新整合与补充研究,图谱全长达到2 047.6 cM,包括27个连锁群,232个标记位点(该图谱在第9、13和16染色体上,均出现了2个间隙,在12染色体出现了1个间隙,因而形成了27个连锁群)。
1.4 QTL定位及上位性互作分析
由于各性状的遗传效应不同,选用不同的统计遗传模型进行研究,能够提高QTL分析的准确性[35]。本研究采用了2种不同的遗传模型来检测主根长和侧根数性状的QTL。利用WinQTLCart 2.5[30]软件中复合区间作图法(CIM)Zmapqtl方法的Model 6,每2 cM对各性状进行全基因组扫描,以确定各性状QTL数目及其在染色体上的位置,通过逐步回归指定解释给定性状最大变异的 5个标记作为余因子(co-factor),模拟运算1 000次,选取临界阈值LOD=2.5,检测每个环境下的QTL效应。当LOD≥2.5时,认为QTL存在;如果临近位点间图距<5 cM,就初步认定是同一个QTL。利用QTL Network 2.1[31]软件中的混合线性模型复合区间作图法(MCIM),选取临界阈值P=0.05,检测每个环境下的QTL、加性效应、加性×加性上位互作效应及环境互作效应。当QTL效应P≤0.05时,认为QTL存在。QTL采取MCCOUCH等[36]命名方式。
2 结果
2.1 亲本及家系根系性状在 RIL群体中的分离及表型分析
观察主根长和侧根数 2个性状在分离群体中的分离并进行遗传力(表2)和方差分析(表3),2个亲本2个性状在后代中表现出较大的分离程度,在不同环境中均存在较大差异。RIL群体的最小值和最大值之间差异明显。各性状遗传力h2为41.56%—50.23%,为QTL分析提供了较好的遗传背景。表2中主根长和侧根数变异系数均较大,侧根数变异系数达到0.75、0.74和0.54,说明该性状在幼苗期稳定性较差,可能同时受基因与环境因素的共同影响。方差分析表明,主根长和侧根数性状在基因、环境和基因与环境的互作间均存在极显著差异(表3),说明这2个性状都是复杂的数量性状,受基因和环境的共同影响。分析 RIL群体的变异范围,2个性状都有超越双亲的株系出现,说明2个性状遗传基因分布在双亲中,可以通过杂交育种得到超亲分离的株系。2个性状的峰度和偏度均较小,变异连续,呈现出近似正态的连续分布(图 1),满足QTL分析要求。
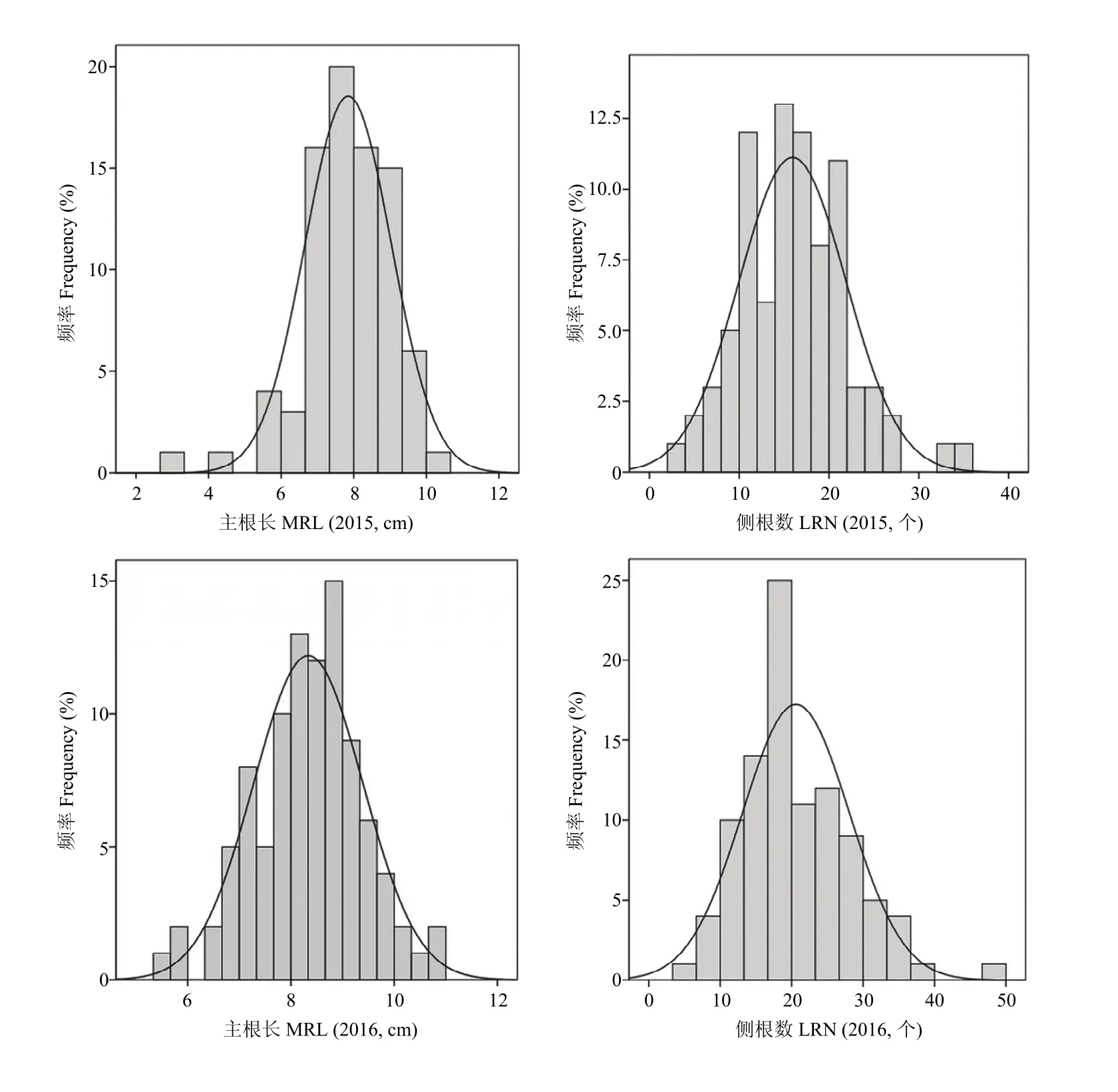
图1 群体主根长和侧根数的频率分布图Fig. 1 Frequency distribution of main root length (MRL) and lateral root number (LRN) of soybean populations in 2015 and 2016
2.2 大豆根系性状之间的相关分析
对2年6个环境下亲本和RIL根系性状进行相关分析(表4),主根长和侧根数均呈现极显著正相关。说明2个性状之间关联性密切,可能同时受基因与环境因素的共同影响。这种即相互影响又相互制约的关系,为不同环境下表型性状选择提供参考。
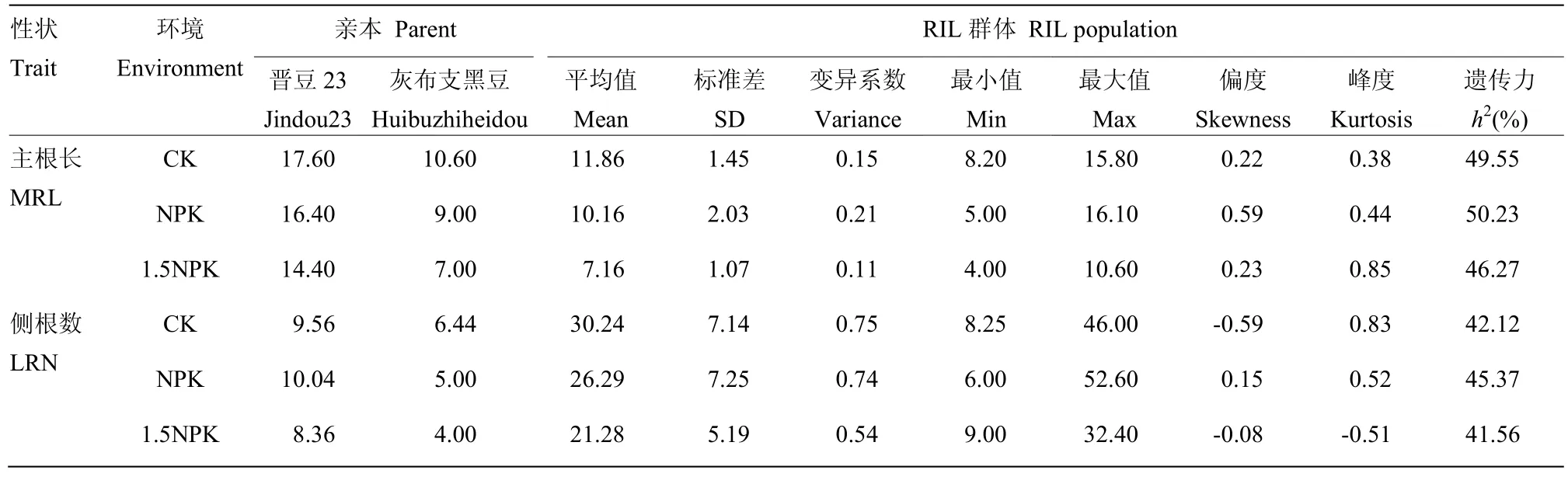
表2 RIL的亲本和家系性状描述统计分析Table 2 Phenotypic variation of all traits of RIL and the parents
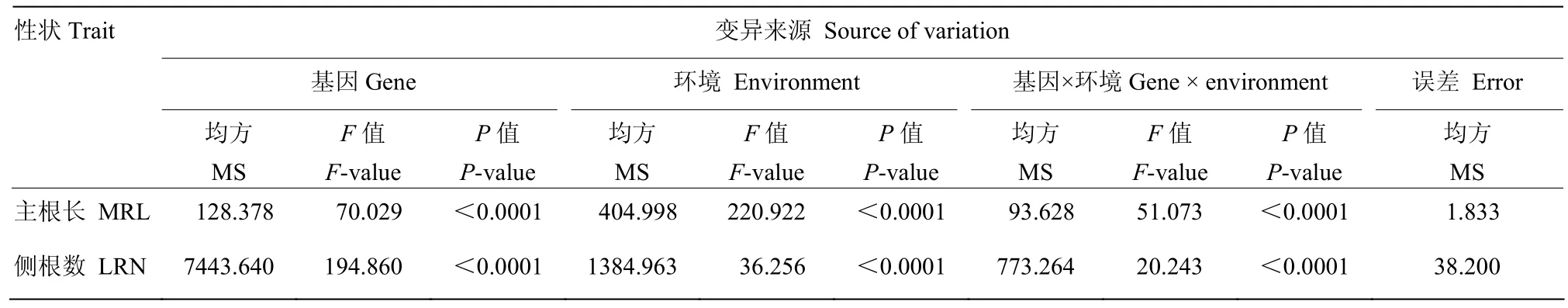
表3 RIL家系主根长和侧根数方差分析Table 3 Variance analysis of MRL and LRN traits of RIL population
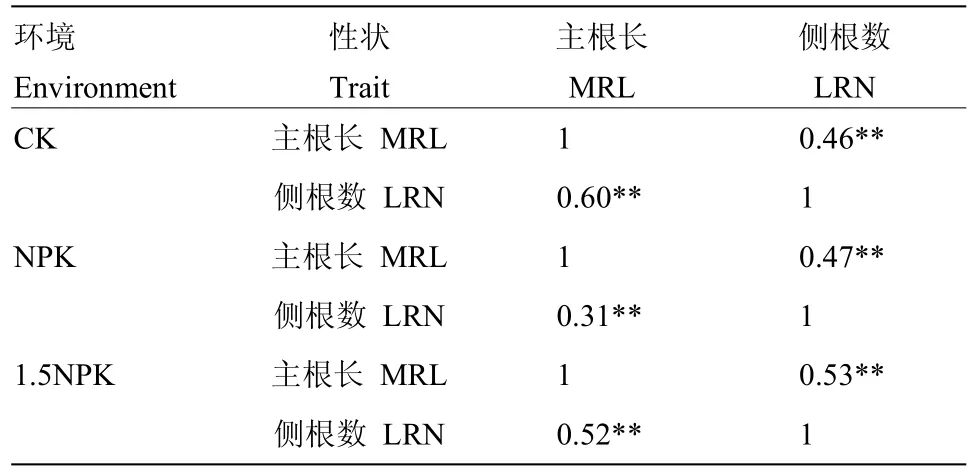
表4 根系性状之间的相关分析Table 4 Correlation analysis of root traits
2.3 主效QTL定位及QE互作效应分析
2种方法对2年6个环境中2个性状共检测到24个主效应QTL,单个性状QTL数分别为9个和15个,分布于第 2、3、5、6、7、8、9、10、11、12、13、14、16、17共14条染色体中(图2、表5、表6),单个QTL的贡献率介于8.52%—43.62%。2个QTL能在2种分析方法中同时检测到,9个QTL能在3种环境下同时检测到。但是,没有检测到在所有环境中均能够稳定表达的QTL。
2.3.1 主根长性状 共检测到9个QTL,分布在第6、7、8、11、12和14染色体上。解释的表型变异范围为8.52%—43.62%,LOD值为2.50—4.20,位于第11染色体Satt430-Satt359区间的qMRL-b1-1,贡献率为43.62%,解释的表型变异最大。加性效应正值和负值都有表现,加性效应为正值,表明增效基因来自母本晋豆23;加性效应为负值,表明增效基因来自父本灰布支黑豆。第6染色体在2015年NPK、1.5NPK和2016年1.5NPK 3个环境下均检测到主根长QTL,其中,qMRL-c2-2与qMRL-c2-3仅相距1.48 cM,LOD分别为3.16和3.23,表型贡献率分别为29.87%和39.02%,均位于Satt305-Satt291标记区间,推测可能是同一个QTL。2种分析方法均在 12染色体上检测到qMRL-h_1-2,加性效应分别为0.067和0.026,MCIM方法检测出加性效应贡献率为 3.08%,加性与环境互作贡献率为 0.33%。互作效应贡献率小于其自身的加性效应贡献率,说明主根长受基因和环境共同作用,基因比环境的影响更加显著。
2.3.2 侧根数性状 共检测到15个QTL,分布在第2、3、5、7、9、10、13、16和17染色体上。解释的表型变异范围为 8.83%—26.37%。LOD值为 2.50—6.36,母本晋豆23和父本灰布支黑豆均有不同的增效基因。位于 17染色体 Satt461-Satt301区间的qLRN-d2-3,表型贡献率为26.37%,解释的表型变异最大。第17染色体在2015年CK和NPK、2016年NPK 3个环境下均检测到侧根数 QTL,其中,qLRN-d2-2和qLRN-d2-3位置相距0.61 cM,均位于Satt488-Satt461区间,解释的表型变异分别为19.60%和 26.37%,推测可能是同一个 QTL。第 5染色体在2015年NPK和1.5NPK、2016年CK 3个环境下均检测到侧根数QTL,其中qLRN-a1-2和qLRN-a1-3位置相距4.37 cM,均位于Satt593-Satt454区间,解释的表型变异分别为17.15%和11.57%, 推测可能是同一个QTL。2种分析方法均在3染色体上检测到qLRN-n-1,加性效应分别为2.751和0.021,MCIM方法检测出加性贡献率为0.87%,加性与环境互作贡献率为2.25%。互作效应贡献率大于其自身的加性效应贡献率,说明侧根数受基因和环境共同作用,环境比基因的影响更加显著。
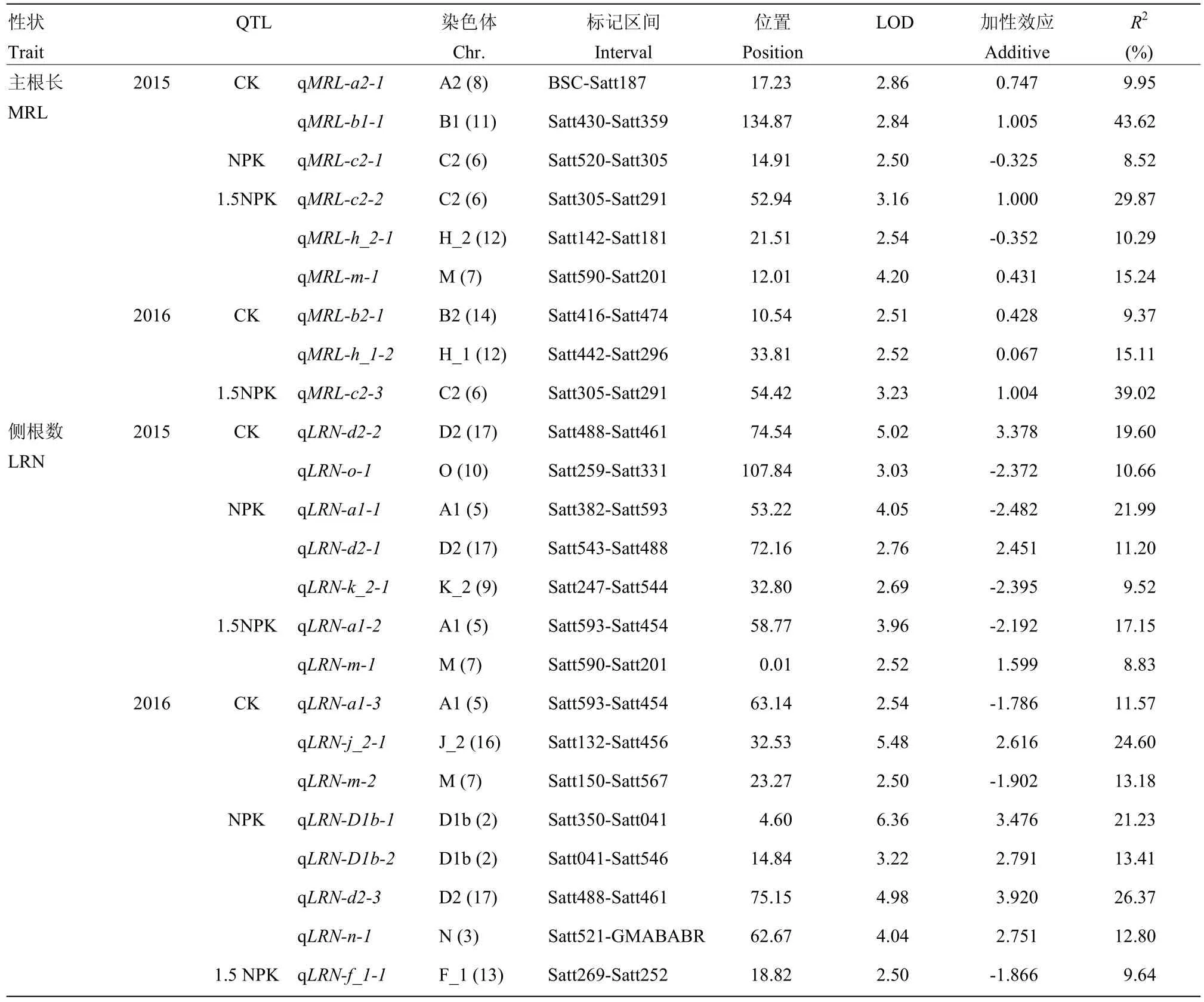
表5 根系性状QTL位置及其参数Table 5 Parameters of QTLs position of root traits
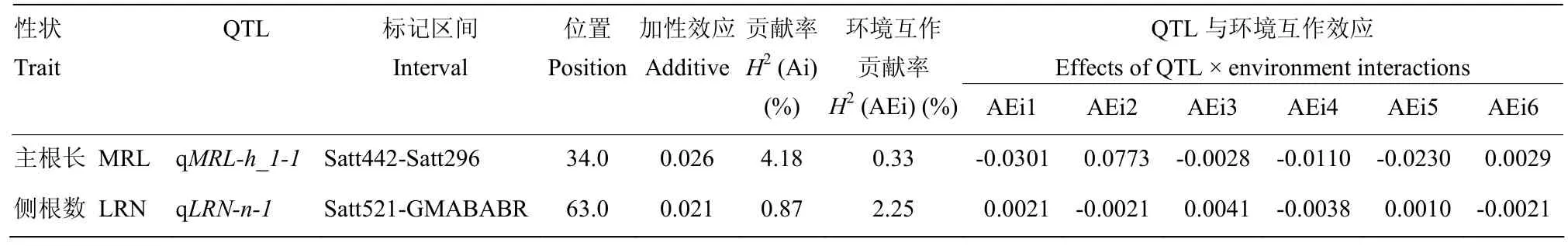
表6 根系性状的QTL及QTL与环境互作Table 6 QTLs and QE interaction of root traits
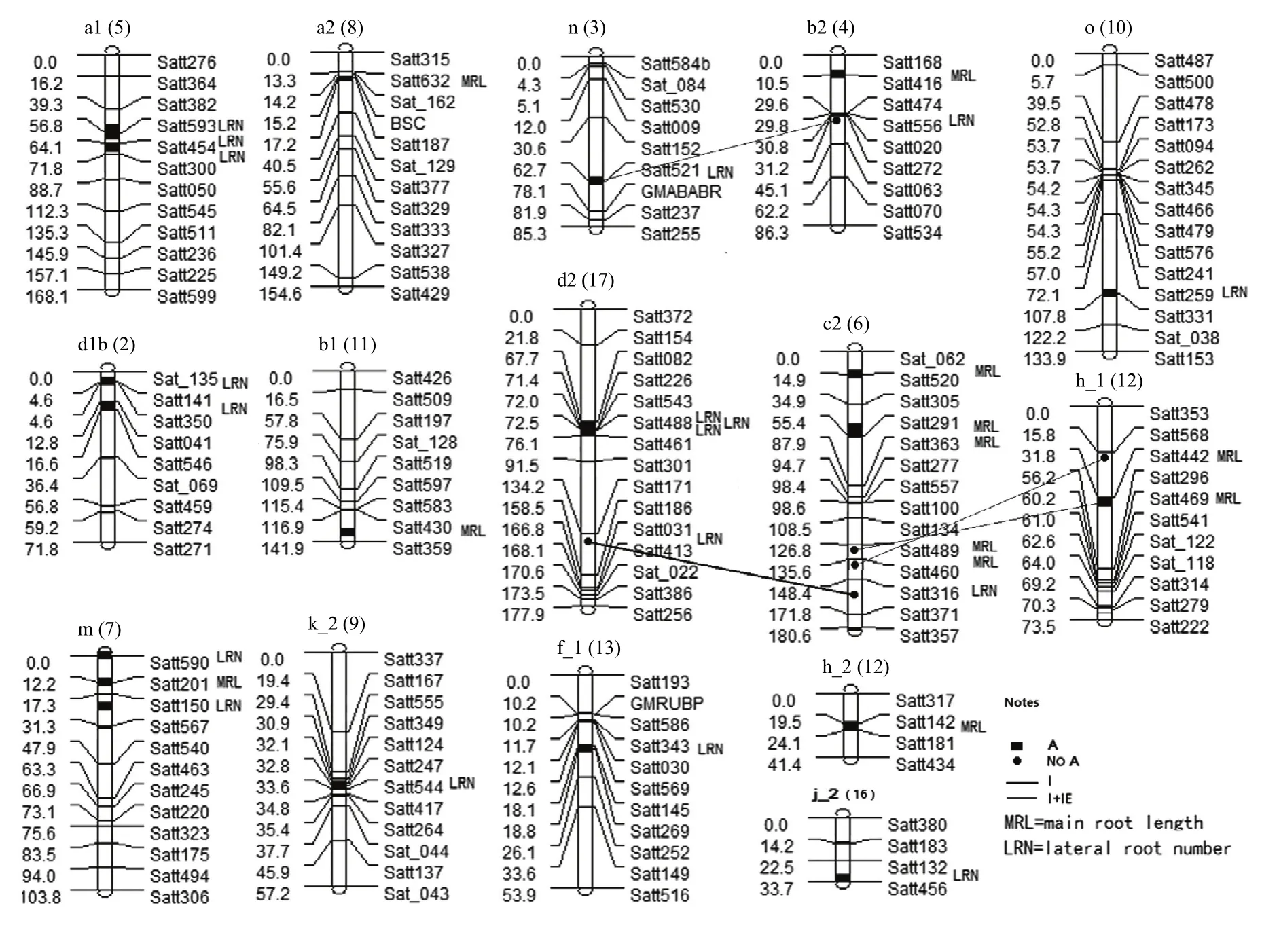
图2 检测到的QTL和加性效应QTL在连锁群上的分布以及根系性状上位性QTL间的遗传结构Fig. 2 Distribution of main QTLs and additive QTLs on linkage groups and graphic presentation of the genetic architecture between epistatic QTLs for root traits
2.4 上位性互作效应
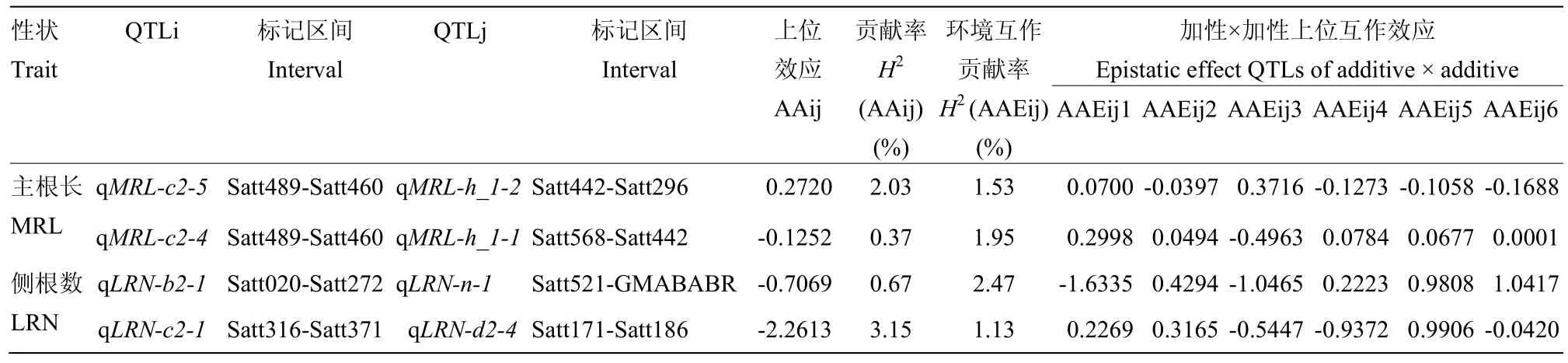
表7 根系性状加性×加性上位互作效应QTLTable 7 Epistatic effect QTLs of additive × additive of root traits
主根长和侧根数共检测到 4对上位性互作 QTL(表7)。检测到2对主根长上位性互作QTL,1对发生在主效 QTL qMRL-h_1-2和非主效 QTL qMRL-c2-5之间,效应值为0.2720,上位效应贡献率2.03%,互作贡献率为1.53%;1对发生在非主效QTL qMRL-h_1-1和非主效QTL qMRL-c2-4之间,效应值为-0.1252,上位互作贡献率为1.95%。检测到2对侧根数上位性互作QTL,1对发生在主效QTL qLRN-n-1和非主效 QTL LRN-b2-1之间,1对发生在非主效QTL q LRN-c2-1和非主效QTL qMRL-d2-4之间,上位效应贡献率分别为 0.67%和 3.15%,互作贡献率分别为2.47%和1.13%,效应值分别为-0.7069和-2.2613,表明该互作为亲本性大于重组型。正向的上位性效应表示两座位间互作基因型与具有正效应的加性基因型方向相同,负向的上位性效应表示两座位间互作基因型与具有正效应的加性基因型方向相反。上位互作贡献率小于(大于)上位效应贡献率,说明上位互作对根系性状遗传的影响小于(大于)上位性的影响。
3 讨论
3.1 主根长和侧根数性状的遗传机制与上位性互作
大豆根系在水分和养分吸收、固定植物以及与根际周围微生物互作等方面具有十分重要的作用。水分和养分环境的改变,对大豆根系生长产生一定程度的影响[37]。本研究中,2年6个环境下,大豆RIL群体中主根长和侧根数的变化趋势均表现出 CK>NPK>1.5NPK,可见幼苗期大豆主根长和侧根数对氮磷钾的吸收较少,在农业生产中,施肥时间应该放到苗期以后更加有效,否则不利于根系的生长发育,该研究结果对于合理施肥和“双减”具有重要的现实意义。
2年6个环境下,表型分析中2016年数据明显低于2015年。2年间数据有差异的原因是:由于观察水培大豆生长时期,只能通过子叶及以上部分观察,处于 V2期大豆虽然地上部分的叶片数一致,但是由于温度、湿度、光照的不同,造成了根系生长的差异,同时遗传上存在明显的互作效应。方差分析结果表明,主根长和侧根数性状在基因、环境和基因与环境的互作间均存在极显著的差异,这2个性状受基因和环境的共同影响。MICM方法分析中,2个性状均检测到存在环境互作效应和上位性互作效应。Network 2.1软件综合考虑了加性、上位性以及与环境互作效应,对研究复杂性状的遗传具有更加重要的意义。同时,对比2个性状RIL群体的变异范围,都有超越双亲的株系出现,说明母本晋豆23和父本灰布支黑豆都具有起到正效作用的等位基因,生产上可以通过杂交育种获得杂种优势。
QTL与环境互作、QTL与QTL上位性互作是作物数量性状遗传的重要组成部分,是作物杂种优势的遗传基础,特别是上位性更适合研究复杂性状的遗传[38]。QTL与环境互作、QTL与QTL上位性互作推进了作物的进化进程,ZHANG等[39]认为上位性比加性QTL更重要。本研究中,上位性贡献率最大为3.15%,加性贡献率最大为 3.08%,主根长主效 QTL qMRL-h_1-2和非主效QTL qMRL-c2-5之间受到上位效应的影响比加性更显著。
LI等[40]认为QTL上位性互作可分为3个类型,一是2个主效应QTL之间互作(typeⅠ);二是1个主效应QTL与1个“背景”位点之间互作(typeⅡ);三是 2个互补位点之间互作(typeⅢ)。本研究检测到的4对互作QTL中,主根长和侧根数各有1对互作QTL qMRL-h_1-2和 qMRL-c2-5、qLRN-n-1和 QTL LRN-b2-1可视为typeⅡ上位性互作,另外2对可视为type III上位性互作。基因的调控网络与表型性状的遗传作用模式相对应,这3种QTL间的上位性互作模式反映的可能正是 QTL所在基因位点间的正向诱导表达或者负向反馈抑制等调控机制[41]。根据本研究检测到的QTL的基因组位置,检索SoyBase数据库中每个QTL附近的功能基因位点后发现,6个QTL中有,标记 Satt489、Satt442分别与影响大豆粒长[42]和大豆叶宽[43]的基因位点位于同一区域,Satt020和Satt316分别与影响大豆脂肪[44]和大豆产量[45]的基因位点位于同一区域。除Satt020外,其余3个位点相应的QTL均为上位性互作QTL。因此这些相应的基因位点是否就是相应互作 QTL的分子基础?相应的基因位点之间是否存在表达调控机制?而这种调控机制是否就是通过QTL间的上位性互作反映出来的?同时,这些在大豆不同性状中发挥作用的位点又如何影响到主根长和侧根数的差异呢?针对这些疑问,需要进行更深入的研究。
3.2 与前人定位结果的比较
进行 QTL定位分析中,建议采用不同的分析方法,优先标定共同发现的QTL[35]。本研究采用2种不同的分析方法共检测到23个主效应QTL,2个QTL能在2种分析方法中同时检测到,分别是主根长QTL qMRL-h_1-2和侧根数QTL qLRN-n-1。参照SoyBase数据库标记信息,这2个QTL均未见报道,全部为新定位出的 QTL,需要在今后加以关注并进行进一步研究。本研究中9个QTL能在3种环境下同时检测到,其中第 6染色体上主根长在 2015年 NPK、1.5NPK和2016年1.5NPK 3个环境下均检测到QTL,分别位于 Satt520-Satt305和 Satt305-Satt291区间。BRENSHA等[46]在第 6染色体上 Satt357-Satt202和Satt239-Sat_105区间也检测到控住主根长的QTL;周蓉等[32]在第6染色体上Satt281-Sat_153也检测到控住主根长的QTL。由于多种研究采取的研究群体、遗传图谱和分析方法不同,尽管定位在同一条染色体上,但区间不统一,这些区间是否有重叠,需要进一步研究。本研究中第5染色体在2015年NPK和1.5NPK、2016年CK 3个环境下均检测到侧根数QTL,作者在前期研究中也在第5染色体上定位出侧根数QTL[29]。本研究中第17染色体在2015年CK和NPK、2016年NPK 3个环境下均检测到侧根数QTL,以往研究中未见报道。同时也发现,本研究与LIAN等[47-48]把主根长分别定位在第11染色体和第8染色体上、LIANG等[29]把侧根数定位在第2染色体上结果一致。除此之外,本研究中检测到的其他QTL,均未见报道。
国内外研究中,因为群体大小和图谱标记密度的不同,QTL定位结果尚不够精细。进一步对大豆主根长和侧根数QTL精细作图,并着重跟踪多环境、多分析方法均能检测到的 QTL区域,并进行功能标记开发,发掘控制主根长和侧根数的功能基因,是下一步研究工作的方向。
4 结论
苗期大豆主根长和侧根数对氮磷钾的吸收影响较少,生产中尽可能减少氮磷钾使用量。不同浓度氮磷钾处理苗期主根长和侧根数参数间既有共同的控制基因,也有各自独特的控制基因,多数QTL不能在多个环境下重复检测到,控制其表达的遗传机制较为复杂。加性效应、加性与环境互作和加性×加性上位性互作效应在主根长和侧根数的形成和遗传中发挥着重要作用。主根长和侧根数各有1个QTL能在2种分析方法中同时检测到,Satt442-Satt296和Satt521-GMABABR是共位标记区间。
致谢:本研究的连锁图谱和群体材料由海南省热带农业资源开发利用研究所方宣钧研究员和山西省农业科学院经济作物研究所刘学义研究员提供,在此表示感谢。
[1]孙广玉, 何庸, 张荣华, 张代平. 大豆根系生长和活性特点的研究.大豆科学, 1996, 15(14): 317-321.SUN G Y, HE Y, ZHANG R H, ZHANG D P. Studies on growth and activities of soybean root. Soybean Science, 1996, 15(14): 317-321.(in Chinese)
[2]JIANG C, GAO X, LIAO L, HARBERD N, FU X. Phosphate starvation root architecture and anthocyanin accumulation responses are modulated by the gibberellins-DELLA signaling pathway in Arabidopsis. Plant Physiology, 2007, 145: 1460-1470.
[3]BI Y M, WANG R L, ZHU T, ROTHSTEIN S J. Global transcription profiling reveals differential responses to chronic nitrogen stress and putative nitrogen regulatory components in Arabidopsis. BMG Genomics, 2007, 8: 281-297.
[4]周蓉, 王贤智, 陈海峰, 张晓娟, 单志慧, 吴学军, 蔡淑平, 邱德珍,周新安, 吴江生. 大豆倒伏性及其相关性状的 QTL分析. 作物学报, 2009, 35(1): 57-65.ZHOU R, WANG X Z, CHEN H F, ZHANG X J, SHAN Z H, WU X J, CAI S P, QIU D Z, ZHOU X A, WU J S. QTL analysis of lodging and related traits in soybean. Acta Agronomica Sinica, 2009, 35(1):57-65. (in Chinese)
[5]TAR’AN B, WARKENTIN T, SOMERS D J, MIRANDA D,VANDENBERG A, BLADE S, WOODS S, BING D, XUE A,DEKOEYER D, PENNER G. Quantitative trait loci for lodging resistance, plant height and partial resistance to mycosphaerella blight in field pea (Pisum sativum L.). Theoretical and Applied Genetics,2003, 107: 1482-1491.
[6]杨秀红, 吴宗璞, 张国栋. 不同年代大豆品种根系性状演化的研究.中国农业科学, 2001, 34(3): 292-295.YANG X H, WU Z P, ZHANG G D. Evolution of root characters of soybean varieties of different ages. Scientia Agricultura Sinica, 2001,34(3): 292-295. (in Chinese)
[7]SANCHEZ P A, SALINAS J G. Low input technology for managing oxisols and ultisols in tropical America. Advances in Agronomy, 1981,34: 279-406.
[8]NOURI M, STEPHEN C M, TOM G, MAX D C. Hybrid and nitrogen influence on pearl millet production in Nebraska: yield, growth, and nitrogen uptake, and nitrogen use efficiency. Agronomy Journal, 1999,91: 737-743.
[9]PRICE A H, STEELE K A, MOORE B J, JONES R G W. Upland rice grown in soil-filled chambers and exposed to contrasting water-deficit regimes: II. Mapping QTL for root morphology and distribution. Field Crops Research, 2002, 76: 25-43.
[10]NARANG R A, BRUENE A, ALTMANN T. Analysis of phosphate acquisition efficiency in different Arabidopsis accessions. Plant Physiology, 2000, 124: 1786-1799.
[11]程琳琳. 中国农田生态系统钾素平衡与钾肥需求[D]. 北京: 中国农业大学, 2007.CHENG L L. Potassium balance and demand of potassium in farmland ecosystem in China[D]. Beijing: China Agricultural University, 2007. (in Chinese)
[12]CHO Y I, JIANG W Z, CHIN J H, PIAO Z Z, CHO Y G, MCCOUCH S R, KOH H J. Identification of QTLs associated with physiologicalnitrogen use efficiency in rice. Molecules and Cells, 2007, 23(1):72-79.
[13]LIAN X M, XING Y Z, YAN H, XU C G, LI X H, ZHANG Q F.QTLs for low nitrogen tolerance at seedling stage identified using a recombinant inbred line population derived from an elite rice hybrid.Theoretical and Applied Genetics, 2005, 112: 85-96.
[14]杨树明, 曾亚文, 王荔, 杜娟, 普晓英, 杨涛. 不同生长环境下水稻氮、磷、钾利用相关性状的QTL定位分析. 植物营养与肥料学报, 2015, 21(4): 823-835.YANG S M, ZENG Y W, WANG L, DU J, PU X Y, YANG T.Identification of QTL traits on N, P and K utilization in rice under different growth environments. Journal of Plant Nutrition and Fertilize, 2015, 21(4): 823-835. (in Chinese)
[15]ZHU J M, MICKELSON S M, KAEPPLER S M, LYNCH J P.Detection of quantitative trait loci for seminal root traits in maize (Zea mays L.) seedlings grown under differential phosphorus levels.Theoretical and Applied Genetics, 2006, 113: 1-10.
[16]苏顺宗, 徐刚, 刘丹, 吴玲, 张啸, 任志勇, 聂治, 林海建, 高世斌.两种磷水平下玉米苗期根系性状的 QTL定位. 玉米科学, 2013,21(4): 33-37.SU S Z, XU G, LIU D, WU L, ZHANG X, REN Z Y, NIE Z, LIN H J,GAO S B. QTL mapping for root traits of maize seedling grown at two phosphorus levels. Journal of Maize Sciences, 2013, 21(4): 33-37.(in Chinese)
[17]蔡红光, 刘建超, 米国华, 袁力行, 陈晓辉, 陈范骏, 张福锁. 田间条件下控制玉米开花前后根系性状的QTL定位. 植物营养与肥料学报, 2011, 17(2): 317-324.CAI H G, LIU J C, MI G H, YUAN L X, CHEN X H, CHEN F J,ZHANG F S. QTL mapping for root traits around flowering stage of maize under field condition. Plant Nutrition and Fertilizer Science,2011, 17(2): 317-324. (in Chinese)
[18]AN D G, SU J Y, LIU Q Y, ZHU Y J, TONG Y P, LI J M, JING R L,LI B, LI Z S. Mapping QTLs for nitrogen uptake in relation to the early growth of wheat (Triticum aestivum L.). Plant and Soil, 2006,284: 73-84.
[19]SU J Y, XIAO Y M, LI M, LIU Q Y, LI B, TONG Y P, JIA J Z, LI Z S.Mapping QTLs for phosphorus-deficiency tolerance at wheat seedling stage. Plant and Soil, 2006, 281: 25-36.
[20]LI Z X, NI Z F, PENG H R, LIU Z Y, NIE X L, XU S B, LIU G, SUN Q X. Molecular mapping QTLs for root response to phosphorus deficiency at seedling stage in wheat (Triticum aestivum L.). Progress in Natural Science, 2007, 17(10): 1177-1184.
[21]刘莹, 盖钧镒, 吕慧能. 大豆根区逆境耐性的种质鉴定及其与根系性状的关系. 作物学报, 2005, 31(9): 1132-1137.LIU Y, GAI J Y, LÜ H N. Identification of rhizosphere abiotic stress tolerance and related root traits in soybean [Glycine max (L.) Merr.].Acta Agronomica Sinica, 2005, 31(9): 1132-1137. (in Chinese)
[22]KING C A, PURCELL L C, BRYE K R. Differential wilting among soybean genotypes in response to water deficit. Crop Science, 2009,49: 290-291.
[23]LEE G J, CARTER J T G, VILLAGARCIA M R, LI Z, ZHOU X,GIBBS M O, BOERMA H R. A major QTL conditioning salt tolerance in S-100 soybean and descendent cultivars. Theoretical and Applied Genetics, 2004, 109(8): 1610-1619.
[24]BIANCHI-HALL C M, ARELLANO C, BOERMA H R, PARROTT W A, HUSSEY R S, ASHLEY D A, BAILEY M A, CARTER T E,RUFTY T W, MIAN M A R. Aluminum tolerance associated with quantitative trait loci derived from soybean PI416937. Crop Science,2000, 40(2): 538-545.
[25]朱向明, 韩秉进. 供氮水平对苗期大豆根系吸水特性的影响. 土壤与作物, 2013, 2(4): 173-176.ZHU X M, HAN B J. Root water uptake characteristics of soybean seedlings under different levels of nitrogen supply. Soil and Crop,2013, 2(4): 173-176. (in Chinese)
[26]ZHANG D, SONG H N, CHENG H, HAO D R, WANG H, KAN G Z,JIN H X, YU D Y. The acid phosphatase-encoding gene GmACP1 contributes to soybean tolerance to low-phosphorus stress.PLoS Genetics, 2014, 10(1): e1004061.
[27]苏辉, 李志刚, 宋书宏. 低磷胁迫下大豆主要农艺性状的 QTL定位. 西北农业学报, 2009, 18(1): 98-101, 116.SU H, LI Z G, SONG S H. Molecular mapping of QTLs major agronomic traits in soybean (Glycine max L.) under phosphorus deficiency stress. Acta Agriculturae Boreali-occidentalis Sinica, 2009,18(1): 98-101, 116. (in Chinese)
[28]BEEBE S E, ROJAS-PIERCE M, YAN X L, BLAIR M W,PEDRAZA F, MUNOZ F M, TOHME J, LYNCH J P. Quantitative trait loci for root architecture traits correlated with phosphorus acquisition in common bean. Crop Science, 2006, 46: 413-423.
[29]LIANG H Z, YU Y L, YANG H Q, XU L J, DONG W, DU H, CUI W W, ZHANG H Y. Inheritance and QTL mapping of related root traits in soybean at the seedling stage. Theoretical and Applied Genetics,2014, 127: 2127-2137.
[30]WANG S C, BASTEN C J, ZENG Z B. Windows QTL Cartographer 2.5 User Manual. Department of Statistics, North Carolina State University, Raleigh, NC, 2005.
[31]YANG J, ZHU J. Predicting superior genotypes in multipleenvironments based on QTL effects. Theoretical and Applied Genetics,2005, 110: 1268-1274.
[32]周蓉, 陈海峰, 王贤智, 伍宝朵, 陈水莲, 张晓娟, 吴学军, 杨中路,邱德珍, 江木兰, 周新安. 大豆幼苗根系性状的QTL分析. 作物学报, 2011, 37(7): 1151-1158.ZHOU R, CHEN H F, WANG X Z, WU B D, CHEN S L, ZHANG X J, WU X J, YANG Z L, QIU D Z, JIANG M L, ZHOU X A. QTL analysis of root traits of soybean at seedling stage. Acta Agronomica Sinica, 2011, 37(7): 1151-1158. (in Chinese)
[33]王珍. 大豆SSR遗传图谱构建及重要农艺性状QTL分析[D]. 南宁:广西大学, 2004.WANG Z. Construction of soybean SSR based map and QTL analysis important agronomic traits [D]. Nanning: Guangxi University, 2004.(in Chinese)
[34]梁慧珍. 大豆子粒性状的遗传及QTL分析[D]. 杨凌: 西北农林科技大学, 2006.LIANG H Z. Genetic analysis and QTL mapping of seed traits in soybean [Glycine max (L.) Merr][D]. Yangling: Northwest A&F University, 2006. (in Chinese)
[35]苏成付, 赵团结, 盖钧镒. 不同统计遗传模型 QTL定位方法应用效果的模拟比较. 作物学报, 2010, 36(7): 1100-1107.SU C F, ZHAO T J, GAI J Y. Simulation comparisons of effectiveness among QTL mapping procedures of different statistical genetic models.Acta Agronomica Sinica, 2010, 36(7): 1100-1107. (in Chinese)
[36]MCCOUCH S R, CHO Y G, YANO M, PAUL E, BLINSTRUB M,MORISHIMA H, KINOSHITA T. Report on QTL nomenclature. Rice Genetics Newsletter, 1997, 14: 11-14.
[37]张志勇, 汤菊香, 王素芳, 王清连. 氮磷钾对植物侧根生长发育的影响及其生理机制. 广东农业科学, 2009, 36(5): 89-92.ZHANG Z Y, TANG J X, WANG S F, WANG Q L. Effects of N, P, K on growth and development of plants and its physiological mechanisms.Guangdong Agricultural Sciences, 2009, 36(5): 89-92. (in Chinese)
[38]王金社, 李海旺, 赵团结, 盖钧镒. 重组自交家系群体 4对主基因加多基因混合遗传模型分离分析方法的建立. 作物学报, 2010,36(2): 191-201.WANG J S, LI H W, ZHAO T J, GAI J Y. Establishment of segregation analysis of mixed inheritance model with four major genes plus polygenes in recombinant inbred lines population. Acta Agronomica Sinica, 2010, 36(2): 191-201. (in Chinese)
[39]ZHANG Z H, YU S B, YU T, HUANG Z, ZHU Y G. Mapping quantitative trait loci (QTLs) for seedling-vigor using recombinant inbred lines of rice (Oryza sativa L.). Field Crops Research, 2005, 91:161-170.
[40]LI Z K, LUO L J, MEI H W, WANG D L, SHU Q Y, TABIEN R,ZHONG D B, YING C S, STANSEL J W, KHUSH G S, PATERSON A H. Overdominant epistatic loci are the primary genetic basis of inbreeding depression and heterosis in rice: I. Biomass and grain yield.Genetics, 2001, 158: 1737-1753.
[41]CARLBORG O, HALEY C S. Epistasis: Too often neglected in complex trait studies. Nature Reviews Genetics, 2004, 5: 618-625.
[42]SALAS P, OYARZO-LLAIPEN J, WANG D, CHASE K, MANSUR L. Genetic mapping of seed shape in three populations of recombinant inbred lines of soybean [Glycine max (L.) Merr.]. Theoretical and Applied Genetics, 2006, 113(8): 1459-1466.
[43]ORF J H, CHASE K, JARVIK T, MANSUR L M, CREGAN P B,ADLER F R, LARK K G. Genetics of soybean agronomic traits: I.Comparison of three related recombinant inbred populations. Crop Science, 1999, 39(6): 1642-1651.
[44]CSANADI G, VOLLMANN J, STIFT G, LELLEY T. Seed quality QTLs identified in a molecular map of early maturing soybean.Theoretical and Applied Genetics, 2001, 103(6/7): 912-919.
[45]REINPRECHT Y, POYSA V, YU K, RAJCAN I, ABLETT G, PAULS K. Seed and agronomic QTL in low linolenic acid, lipoxygenase-free soybean (Glycine max (L.) Merrill) germplasm. Genome, 2006, 49(12):1510-1527.
[46]BRENSHA W, KANTARTZI S, MEKSEM K, GRIER R, BARAKAT A, LIGHTFOOT D, KASSEM M. Genetic analysis of root and shoot traits in the ‘essex’ by ‘forrest’ recombinant inbred line (RIL)population of soybean [Glycine max (L.) Merr.]. Journal of Plant Genome Sciences, 2012, 1(1): 1-9.
[47]LIAN Q, XIAOHUI C, MANTONG M, XIAOLONG Y, HONG L.QTL analysis of root traits as related to phosphorus efficiency in soybean. Annals of Botany, 2010, 106(1): 223-234.
[48]MANAVALAN L, PRINCE S, MUSKET T, CHAKY J, DESHMUKH R. VUONG T, SONG L, CREGAN P, NELSON J, SHANNON J,SPECHT J, NGUYEN H. Identification of novel QTL governing root architectural traits in an interspecific soybean population. PLoS ONE,2015, 10(3): e0120490.
(责任编辑 李莉,岳梅)
QTL Mapping for Main Root Length and Lateral Root Number in Soybean at the Seedling Stage in Different N,P and K Environments
LIANG HuiZhen1, DONG Wei1, XU LanJie1, YU YongLiang1, YANG HongQi1,TAN ZhengWei1, XU Yang2, CHEN XinWei3
(1Sesame Research Center, Henan Academy of Agricultural Sciences, Zhengzhou 450002;2Nanyang Academy of Agricultural Sciences, Nanyang 473083, Henan;3Shangqiu Academy of Agriculture and Forestry Sciences, Shangqiu 476000, Henan)
2017-03-06;接受日期:2017-05-08
河南省农业科技攻关计划(152102110140,172102110088)、农业科研杰出人才及其创新团队项目、河南省农业科学院科研发展专项(201513110)
联系方式:梁慧珍,Tel:0371-65751589;E-mail:lhzh66666@163.com

