菊花几丁质酶基因ChCHI的克隆及表达分析
崔 波, 宋彩霞, 王莹博, 王若斓, 蒋素华, 梁 芳, 叶永忠
(1.河南农业大学生命科学学院,河南 郑州450002; 2.郑州师范学院生物工程研究所,河南 郑州 450044; 3.郑州大学生命科学学院,河南 郑州 450001)
菊花几丁质酶基因ChCHI的克隆及表达分析
崔 波1,2, 宋彩霞1, 王莹博1, 王若斓2,3, 蒋素华2, 梁 芳2, 叶永忠1
(1.河南农业大学生命科学学院,河南 郑州450002; 2.郑州师范学院生物工程研究所,河南 郑州 450044; 3.郑州大学生命科学学院,河南 郑州 450001)
根据GenBank发布的几丁质酶基因的保守序列设计简并引物,采用RT-PCR和RACE技术,克隆到菊花1个几丁质酶基因ChCHI(GenBank登录号为KX881373)。ChCHI基因cDNA全长为1 385 bp,包含960 bp开放阅读框,编码319个氨基酸。ChCHI属于亲水性蛋白,相对分子质量约为35.5 kD,等电点为6.38,为稳定蛋白,二级结构预测表明该蛋白以α螺旋和无规则卷曲为主。蛋白序列比对和进化树分析表明,ChCHI基因编码的蛋白属于糖苷水解酶19家族几丁质酶,与薇甘菊(ACO25187.1)几丁质酶蛋白的亲缘关系最近,序列一致性为87%。qRT-PCR分析结果显示,SA处理可以明显提高贡菊叶片中ChCHI的表达量。菊花ChCHI在对抗环境胁迫的分子调控中起重要作用。
菊花;几丁质酶基因;克隆;表达分析
菊花(Chrysanthemummorifolium)为菊科(Compositae)多年生草本植物。菊花种类繁多、形态多样,是中国十大名花之一,也是世界四大切花之一,被广泛用于切花、盆栽以及园林绿化,栽培历史悠久。菊花富含黄酮类等多种生物活性物质,又具有药用和食用价值,在世界各地广为栽培。随着菊花的消费需求越来越大,为了满足市场的需求,温室栽培条件实现了菊花市场的全年供应。由于连作或温室培养等多种因素的影响,菊花在栽培过程中会感染多种病害,导致其药用性、观赏性和商品性普遍退化,温室栽培虽然打破了季节的局限性,但存在严重病害及耗费能源等问题,严重阻碍了菊花的生产和发展。因此,提高菊花的抗逆性至关重要。几丁质酶(Chitinase)是一种降解几丁质的糖苷酶[1],通过水解病原微生物细胞壁几丁质中的β-1,4糖苷键产生N-乙酰氨基葡萄糖寡聚体或单体而起到抑菌作用。许多细菌[2]、植物[3]和动物[4]都可以产生几丁质酶。正常植物中的几丁质酶基因的表达量很低或者不表达,当植物受到病虫害[5]、真菌[6]、病毒[7]、水杨酸、乙烯利[8]、重金属[9]、高盐、紫外线、低温[10]、干旱[11]、机械损伤[12]等胁迫时,几丁质酶基因的表达量会迅速提高。BROGLIE等[13]最早以菜豆为材料克隆了几丁质酶基因。随着研究的不断深入,研究者至今已从拟南芥[14]、无花果[15]、番茄、马铃薯[16]、凤梨[17]、烟草、大豆等植物中提取和纯化了几丁质酶。转化了几丁质酶基因的多种植物对特定病害的抗性得到增强,如水稻对纹枯病的抗性[18]、番茄对枯萎病和早疫病的抗性[19]、香蕉对黑叶条纹病的抗性[20]、棉花对黄萎病的抗性[21]、柠檬对真菌病害的抗性[22]等。近年来,随着研究者对其结构特点、生化性质、表达调控、作用机理等方面的进一步研究,使几丁质酶在抗病基因工程和分子生物学领域成为研究热点。
水杨酸(SA)作为诱导植物抗性的信号分子[23],也是最早发现的一种化学诱导剂,在植物对逆境的胁迫过程中起重要作用。大量研究结果表明,外源水杨酸可诱导月季[24]、马铃薯[25]、水稻、拟南芥等多种植物病程相关蛋白基因的表达,增强植株的抗病能力。尽管目前在很多植物中都克隆到不同的几丁质酶基因,而在菊花中几丁质酶基因及其研究还未见报道。本研究通过RT-PCR和RACE技术,首次从菊花叶片克隆到一个几丁质酶基因ChCHI,并对其进行了SA诱导的实时荧光定量表达分析,为利用几丁质酶基因进行菊花抗性分子育种奠定理论基础。
1 材料与方法
1.1试验材料与处理
试验材料为贡菊,由郑州师范学院生物工程研究所提供。选取高约20 cm的贡菊幼苗,取经过0.5 mmol·L-1和0.1 mmol·L-1水杨酸处理0、4、8、16、24、48 h的中部叶片组织材料,液氮速冻后保存于-80 ℃超低温冰箱中备用。
1.2方法
1.2.1 菊花叶片总RNA提取和cDNA合成 叶片组织总RNA用TIANGEN公司的多糖多酚植物总RNA提取试剂盒提取,经过1%琼脂糖凝胶电泳检测RNA的完整性,用Q5000核酸蛋白分析仪(美国Quawell)测定提取的RNA的浓度和纯度。用M-MLV反转录酶(TaKaRa)以4 ℃处理不同时间段的叶片混合提取的总RNA为模板,合成单链cDNA,具体操作根据说明书进行。
1.2.2 菊花几丁质酶基因全长的克隆 利用软件DNAMAN和Primer premier 5.0,根据NCBI登录的植物几丁质酶基因序列的保守区,设计一对简并引物,上游引物Ch-CHI-F1(5’-TGTGATCARGGWTGGGAATGT-3’)和下游引物Ch-CHI-R1(5’-AGATTGCRGCTTGGAARGCTA-3’),以反转录合成的cDNA第一链为模板,扩增获得菊花几丁质酶基因的保守片段。PCR扩增总体系(20 μL)为:10×PCR buffer 2 μL,模板cDNA 1 μL,dNTP Mix 1.6 μL,上游引物和下游引物各 1 μL,Taq DNA聚合酶0.2 μL,无菌ddH2O 13.2 μL。PCR反应程序为:94 ℃预变性5 min,94 ℃变性40 s,55 ℃退火40 s,72 ℃延伸40 s,35个循环,72 ℃延伸10 min。用1%的琼脂糖凝胶电泳检测PCR扩增产物,利用凝胶试剂盒(TIANGEN公司)将PCR产物回收纯化后连接到pGEM-T Easy载体上,转化大肠杆菌DH5α感受态细胞挑选阳性克隆测序,并在NCBI中比对分析测序结果。
根据上步获得的几丁质酶基因中间片段序列,设计一对3’端特异性引物,GSP1(5’-CGTTGGGAGTCAAACTTCGT-3’)和GSP2(5’-ACTATGGCTACCTTGGCGACT-3’),一对5’端特异性引物GSP3(5’-GGTAAATGGCAGCAGCAGTAAT-3’)和GSP4(5’-AGTTCCAGAAGACGGGCAAAG-3’),按照试剂盒说明操作,完成反转录反应后进行PCR反应,回收扩增产物,连接到pGEM-T Easy载体上,然后转入大肠杆菌DH5α菌株,挑选阳性克隆测序,并与中间片段进行拼接。
根据拼接序列设计一对引物ChCHI-ORFF(5’- CAAAGAAGAATGGGGACAAAAC -3’)和ChCHI-ORFR(5’- AGGTTAACTCGAAGCCGATTC -3’)用于ORF(开放阅读框)的扩增。采用普通琼脂糖凝胶DNA回收试剂盒(TIANGEN公司)回收目的片段,对目的片段进行转化和克隆,挑选阳性克隆进行测序。引物合成和测序由英潍捷基(上海)贸易有限公司完成 。
1.2.3 菊花几丁质酶基因的生物信息学分析 用Blast软件进行几丁质酶基因的同源序列搜索;用ORF Finder软件预测开放阅读框;在NCBI的蛋白保守区数据库中搜索几丁质酶基因编码蛋白保守结构域及功能域;用ProtParam软件预测编码蛋白基本的理化性质;用SignalP预测蛋白的信号肽;用ProtScale软件预测亲疏水性;用SOPMA软件预测蛋白的二级结构;用Phyre 2预测蛋白的三级结构;用PSORT在线软件对目的蛋白进行亚细胞定位;用MEGA软件构建系统进化树。
1.2.4 菊花几丁质酶基因的表达分析 利用Primer Premier 5.0软件设计菊花几丁质酶基因实时荧光定量PCR引物 qChCHI-F(5’-GGTTGGGAATGTAAAGGATGGTC-3’)和qChCHI-R(5’-GTTGGTAAATGGCAGCAGCAGTA-3’)。以18S rRNA作为内参基因,引物为18S-F(5’-GTCGGGGGCATTCGTATTTC-3’)和18S-R(5’-CGGCATCGTTTATGGTTGAG-3’)。提取菊花经过SA处理的叶片总RNA,使用PrimeScript RT reagent Kit with gDNA Eraser(TaKaRa)试剂盒反转录合成单链cDNA。参照购自宝生物工程(大连)有限公司的SYBR Premix Ex TaqTMⅡ Kit试剂盒说明书进行qRT-PCR反应。使用仪器为Eppendorf Mastercycler ep realplex 2荧光定量PCR仪,反应体系为20 uL,反应程序为95 ℃预变性15 s,58 ℃变性15 s,72 ℃退火15 s,共40个循环。基因相对表达量采用公式2-△△Ct法计算。
2 结果与分析
2.1菊花几丁质酶基因全长cDNA克隆
利用简并引物和0.1 mmol·L-1SA处理叶片8 h的cDNA 进行菊花几丁质酶基因保守片段的PCR扩增,预期得到1条长度约为500 bp的片段,该片段大小与预期目的条带一致,测序结果得到529 bp的特异保守片段。根据该片段序列采用RACE技术,设计RACE引物,利用3’端引物GSP1和GSP2扩增出3’端目的片段,用5’端引物GSP3和GSP4扩增出5’端目的片段。将中间片段、3’端目的片段和5’端目的片段序列拼接,在开放阅读框(ORF)区域设计引物,进行目的片段的扩增,得到了预期的片段,最终获得大小为1 385 bp的菊花几丁质酶基因的全长cDNA序列(图1)。其核苷酸序列在NCBI上Blast分析,发现其与薇甘菊的几丁质酶基因有较高一致性,为83%,将该基因命名为ChCHI,提交GenBank,登录号为KX881373。包含1个19家族几丁质酶结构域,表明该片段为几丁质酶片段。经NCBI在线工具ORF Finder软件查找,该基因具有 960 bp的完整开放阅读框,5’端非编码区为 72 bp,3’端非编码区为353 bp,编码319个氨基酸(图2)。
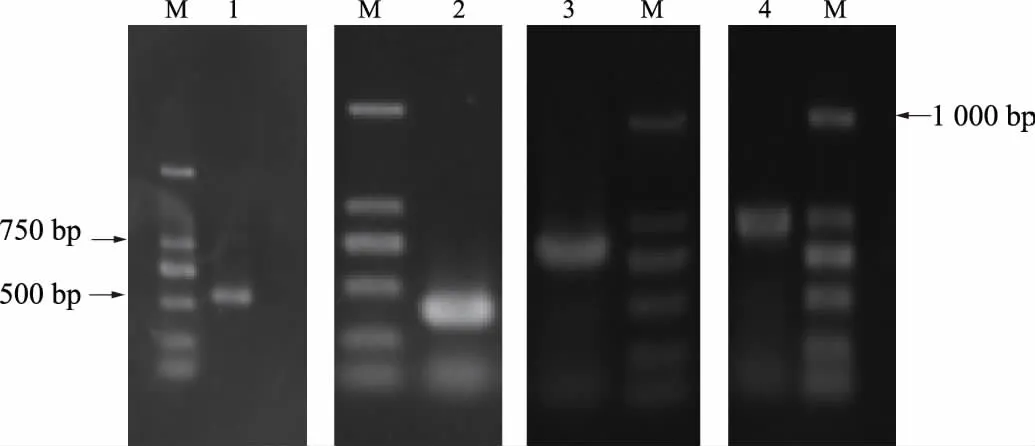
1.保守片段;2.5’-RACE;3.3’-RACE;4.开放阅读框片段;M.DL2000。1.Conserved region; 2.5’-RACE; 3.3’-RACE; 4.ORF; M. DL2000.

ATG.起始密码子;TAA.终止密码子;阴影.保守序列 ATG.Start codon; TAA.Stop codon; Shadow. Conserved Sequence
2.2菊花几丁质酶基因及编码蛋白序列的生物信息学分析
利用NCBI的蛋白保守区数据库对菊花几丁质酶基因编码蛋白的保守区进行预测,结果表明(图3),该蛋白在第64-301氨基酸残基之间有一个几丁质酶糖苷酶19家族结构域,在此结构域内有3个催化残基位点和7个糖结合位点,说明该结构域包含催化区;该结构域还是一个溶菌酶相似超家族基因的保守结构域,推测该蛋白兼具溶菌酶活性。
将菊花的几丁质酶基因与其他植物的几丁质酶基因的氨基酸序列进行同源性比对(图4),发现它们有较高的同源性。一致性分析表明,ChCHI的氨基酸序列与薇甘菊(MikaniamicranthaACO25187.1)、梅(PrunusmumeXP-008222679.1)和芝麻(SesamumindicumXP-011073914.1)的一致性分别为87%、72%和70%,4个物种具有共同的几丁质酶基序。利用PSORT在线软件对目的蛋白进行亚细胞定位,软件分析显示,ChCHI基因编码蛋白分泌到胞外的概率为82%,存在微体或过氧化物体中的概率为16.2%,存在于内质网膜和内质网腔中的概率都为10%,发现该蛋白存在于细胞外的可能性较大。
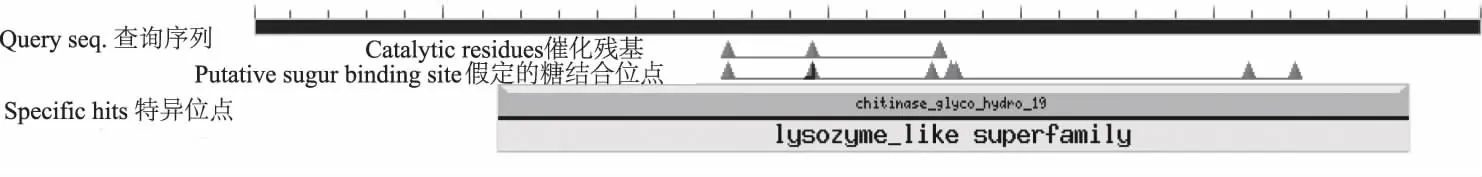
图3 菊花几丁质酶基因预测蛋白的结构域分析Fig.3 Conserved domain analysis of predicted protein of chitinase gene in Chrysanthemum morifolium

图4 ChCHI所编码的氨基酸与其他植物来源的几丁质酶氨基酸序列比对Fig.4 Homology analysis of ChCHI encodes amino acid sequences and those from other plant species
ProtParam软件预测结果表明,菊花几丁质酶基因编码的蛋白相对分子质量约为35.5 kD,理论等电点pI为6.38,不稳定指数为31.25,属稳定蛋白。SignalP的预测结果显示,该蛋白在N-端的第1~23氨基酸残基之间有一信号肽区域,第23位丙氨酸和第24位天冬氨酸之间为信号肽切割位点,从24位氨基酸以后为成熟蛋白。利用ProtScale对ChCHI氨基酸序列的疏水性和亲水性进行预测,结果表明,第148位的Ser具有最低的分值-3.033,亲水性最强;而第12位的Thr具有最高的分值3.400,疏水性最强。整体来看,亲水性氨基酸分布比较均匀,且数量多于疏水氨基酸,可以进一步推测菊花ChCHI蛋白质为亲水性蛋白。
利用SOPMA对菊花ChCHI氨基酸序列进行二级结构预测(图5)。从图5中可以发现,菊花ChCHI由40.75%的无规则卷曲(Random coil),29.47%的α-螺旋(Alpha helix),19.75%的延伸连(Extended strand)和10.03的β-转角(Beta turn)组成。在整个蛋白质结构中,无规则卷曲和α-螺旋是菊花ChCHI最主要的结构元件,而延伸连和β-转角则次之。
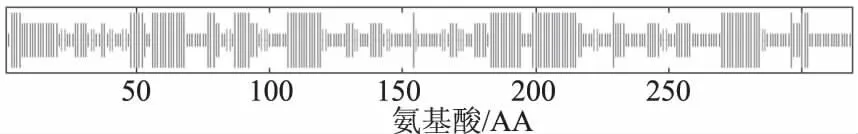
图5 ChCHI蛋白2级结构预测Fig.5 Secondary structure prediction of ChCHI protein
利用Phyre 2 预测ChCHI蛋白的三级结构(图6)。预测结果表明,该蛋白质为一个结构较为松散的球状蛋白,与粳稻的ClassⅠ(模板:c2dkvA)序列一致性为82%,可以看出,该蛋白空间结构主要由α-螺旋和无规则卷曲这种元件构成。
为进一步分析菊花几丁质酶基因与其他物种几丁质酶基因的关系,将NCBI上已登录的16个物种的几丁质酶基因的氨基酸序列进行Blast,利用MEGA4.0软件对其进行进化树分析(图7),结果显示,几丁质酶蛋白分为第19和第18两个家族,菊花的ChCHI蛋白为第19家族, 与19家族中薇甘菊、刺菜蓟(Cynaracardunculusvar.scolymus)的几丁质酶蛋白亲缘关系最近,与陆地棉(Gossypiumhirsutum)、金合欢(Acaciakoa)、美花烟草(Nicotianasylvestris)、葡萄(Vitisvinifera)、芝麻、可可(Theobromacacao)、苹果(Malusdomestica)、梅、草莓(Fragariaxananassa)、川椒(Capsicumannuum)、马铃薯(Solanumtuberosum)等的几丁质酶蛋白具有相对较近的亲缘关系。与18家族的拟南芥(Arabidopsisthaliana)、烟草(Nicotianatabacum)的几丁质酶蛋白亲缘关系相对较远。
2.3菊花几丁质酶基因的表达分析
为了分析菊花几丁质酶基因和SA胁迫的关系,采用实时荧光定量方法检测菊花几丁质酶基因在SA诱导后的表达量。以诱导后的0 h的表达量为对照,用2-△△Ct方法进行分析并作图,以18S rRNA作为内参基因(图8)。结果表明,经过0.5 mmol·L-1和0.1mmol ·L-1水杨酸诱导后,在一定时间内(8 h)较低浓度的水杨酸处理(0.1mmol ·L-1)能使菊花几丁质酶基因的表达量明显升高,总体趋势是在4 h时表达量有所下降,在8 h时几丁质酶基因的表达量最高,随着诱导时间的延长,其表达量呈逐渐下降趋势,比诱导前的表达量低。
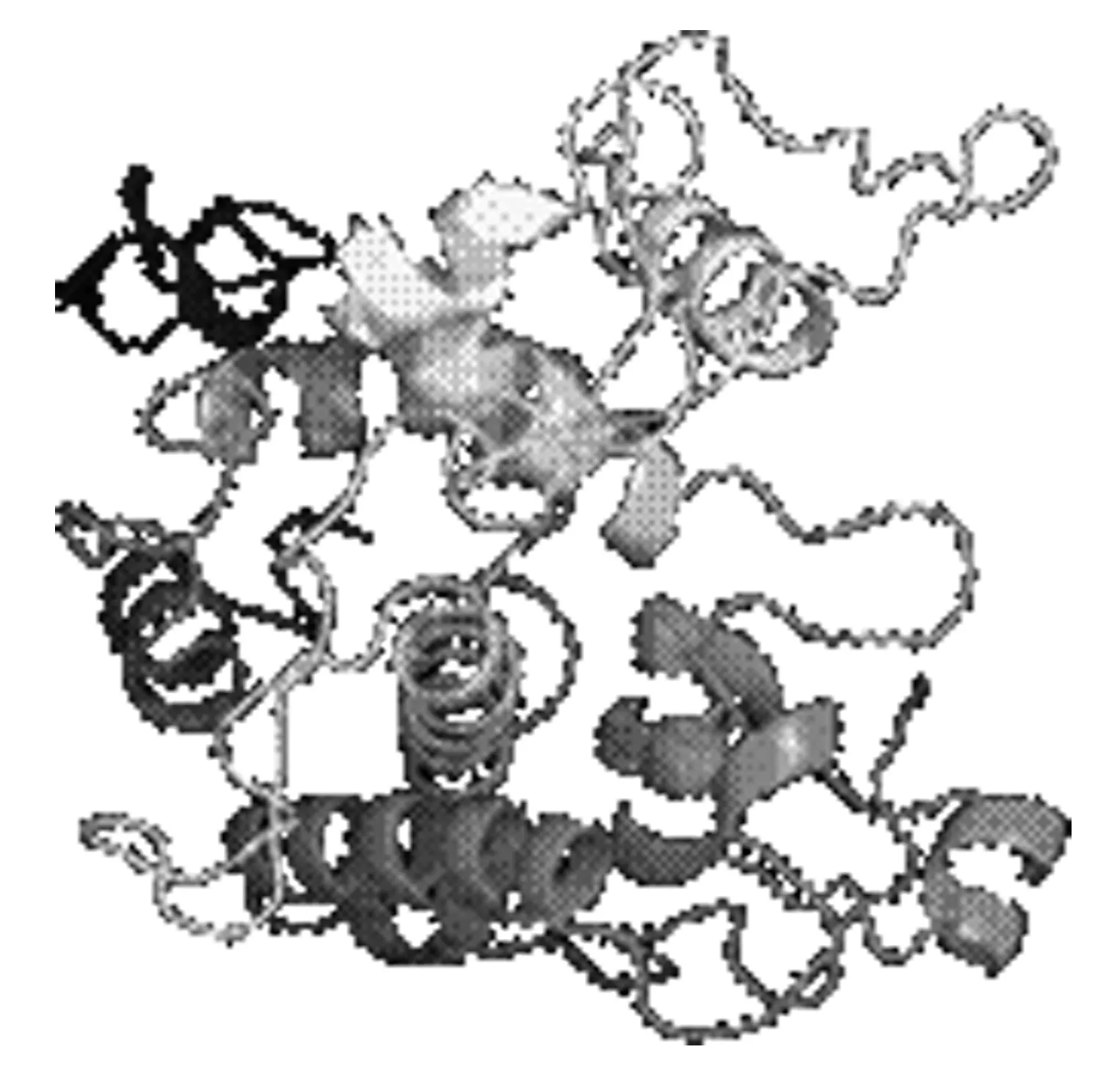
图6 ChCHI蛋白3级结构预测Fig.6 The tertiary structure prediction of ChCHI protein
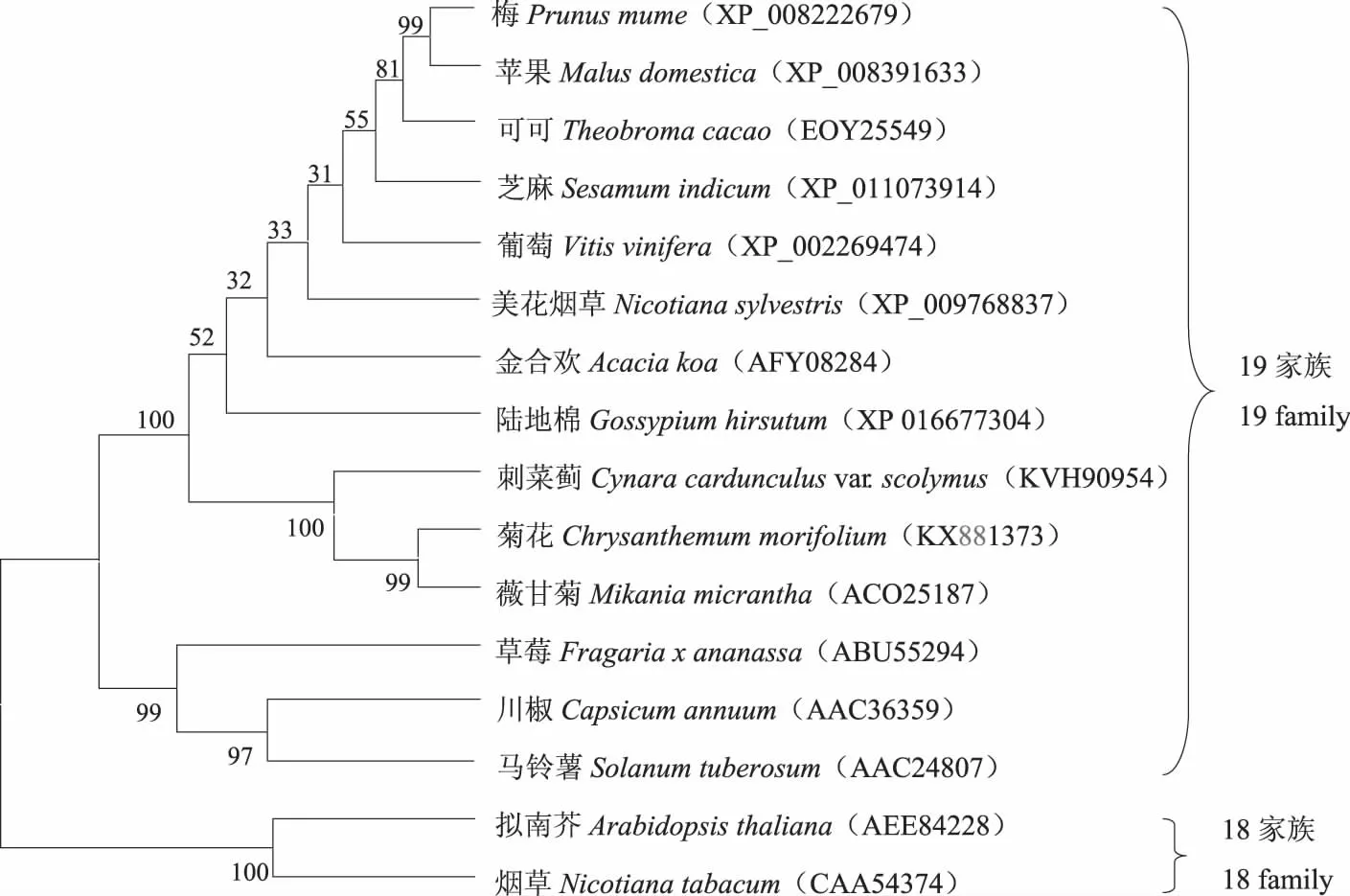
图7 ChCHI与其他植物来源的几丁质酶蛋白的系统进化Fig.7 Phylogenetic tree of the ChCHI and chitinases protein from other plant species
3 结论与讨论
几丁质酶是一种重要的病程相关蛋白,在植物抵御逆境和抵抗真菌病原物方面具有重要的作用,植物体受到伤害时自身几丁质酶基因的表达量会迅速提高。本研究通过同源克隆的方法,首次从菊花叶片中克隆到1个几丁质酶基因的全长cDNA序列ChCHI(GenBank登录号为KX881373),并利用生物信息学方法对ChCHI蛋白的分子特征进行分析和预测。结果表明,ChCHI基因cDNA全长为1 385 bp,包含960 bp开放阅读框,编码319个氨基酸,该基因属于19家族几丁质酶基因,包含1个催化区,在N端有1个信号肽,相对分子质量约为35.5 kD,为稳定蛋白,亲水性强,2级结构表明该蛋白以α螺旋和无规则卷曲为主,属酸性蛋白。 ChCHI蛋白的3级结构预测结果表明,该蛋白质为1个结构较为松散的球状蛋白,与栽培稻粳稻的ClassⅠ序列一致性为82%,可以看出该蛋白空间结构主要由α-螺旋和无规则卷曲这两种元件构成。
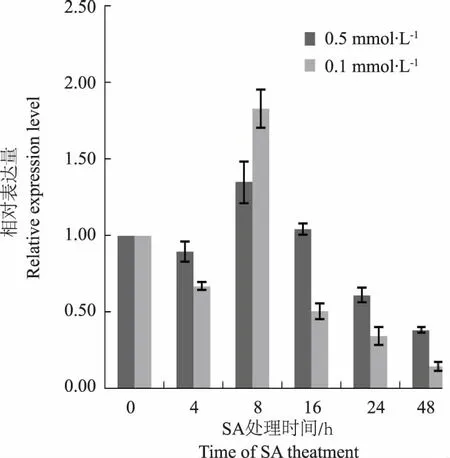
图8 ChCHI的表达特性分析Fig.8 Expression pattern analysis of ChCHI
植物几丁质酶被划分为第18和第19 两个家族。序列比对和系统进化结果表明,这16个物种的几丁质酶分别隶属于18家族和19家族几丁质酶。几丁质酶基因的进化有明显的种属特征,菊花ChCHI蛋白与同为菊科的薇甘菊和刺菜蓟的亲缘关系最近,其一致性达到87%和77.5%,与锦葵科的陆地棉及其他不同科之间的几丁质酶蛋白亲缘关系较远。
有研究表明,多数病原菌是从细胞间隙开始侵染植物的,侵入的病原菌会阻碍或破坏植物组织摄取营养,植物的正常生长将会受到抑制[26]。因此,为了更好地抵御病原菌的侵染,抗真菌蛋白最好在胞间表达。本研究所克隆的ChCHI基因通过亚细胞定位分析,预测其分泌到胞外的概率很大(82%),推断该基因如果在菊花中表达,其所表达的蛋白可能有助于直接参与对菊花真菌病害的防御。本研究为进一步研究菊花ChCHI基因的抗真菌特性提供理论依据。
当植物几丁质酶被发现后,研究者大多关注的是其对几丁质的降解和对多种真菌的抑制作用,本研究通过qRT-PCR分析了经过SA处理后的叶片中ChCHI基因表达量,结果表明,几丁质酶基因在SA处理后的表达量在短时间内快速升高,这与之前报道的水杨酸诱导花生几丁质酶活性显著提高的结果一致[27]。推测几丁质酶不仅可以抵御病原真菌的侵害,在植物其他抗逆过程中可能也发挥了重要作用。因此,ChCHI在SA胁迫条件下表达量短时间内快速升高,表明它参与了植物抵抗逆境的过程,具体的作用机制及基因功能有待下一步试验研究。
[1] HONG J K, HWANG B K. Promoter activation of pepper class: Ⅱ basic chitinase gene,CAChi2, and enhanced bacterial disease resistance and osmotic stress tolerance in theCAChi2-overex-pressingArabidopsis[J]. Planta , 2006, 223(3): 433-448.
[2] 钟万芳,丁少军. 细菌几丁质酶的结构与功能研究进展[J]. 江苏农业科学, 2012, 40(4): 1-3.
[3] ZHU X, ZHANG H, FUKAMIZO T, et al. Properties ofManducasextachitinase and its C-terminal deletions[J]. Insect Biochemistry and Molecular Biology, 2001, 31(12): 1221-1230.
[4] DEBADITYA B,ANAND N, RAJINDER K G. Bacterial chitinases: properties and potential [J]. Critical Reviews in Biotechnology, 2007, 27(1) : 21-28.
[5] PARK Y S, JEON M H, LEE S H, et al. Activation of defense responses in Chinese cabbage by a nonhost pathogen,Pseudomonassyringaepv. tomato[J]. Journal of Biochemistry and Molecular Biology, 2005, 38: 748-754.
[6] DANA M D L M, CUBERO B. Transgenic tobacco plants overexpressing chitinases of fungal origin show enhanced resistance to biotic and abiotic stress agents[J]. Plant Physiology, 2006, 142(2):722-730.
[7] HIRAMATSU S, FUJIE M, USAMI S, et al. Two catalytic domains of chlorella virus CVR2 chitinase[J]. Journal of Bioscience and Bioengineering, 2000, 89 (3): 252-257.
[8] 梁元凯, 陈鹏, 李玉红. 乙烯利诱导黄瓜叶片胞间隙几丁质酶累积的变化[J]. 西北农业学报, 2012, 21(1): 141-145.
[9] WANG L Y, WANG Y S, CHENG H, et al. Cloning of theAegicerascorniculatumclass:Ⅰchitinase gene (AcCHII) and the response of AcCHIⅠ mRNA expression to cadmium stress[J]. Ecotoxicology, 2015. 24(7):1-9.
[10] STRESSMANN M, KITAO S, G RIFFITH M, et al. Calcium interacts with antifreeze proteins and chitinase from cold-acclimated winter rye[J]. Plant Physiol, 2004, 135: 364-376.
[11] HONG J K, HWANG B K. Induction by pathogen, salt and drought of a basic class: Ⅱchitinase mRNA and its in situ localization in pepper (Capsicumannuum)[J]. Physiol Plant, 2002, 114(4): 549-558.
[12] BRAVO J M, CAMPO S, MURILLO I, et al. Fungus-and wound-induced accumulation of mRNA containing a class: Ⅱchitinase of the pathogenesis-related protein 4 (PR-4) family of maize[J]. Plant Molecular Biology, 2003, 52 (4):745-759.
[13] BROGLIE K E, BIDDLE P, GRESSMAN R, et al. Functional analysis of DNA sequences responsible for ethylene regulation of a bean chitinase gene in transgenic tobacco[J]. The Plant Cell, 1989, 1: 599-607.
[14] HERMANS C, PORCO S, VERBRUGGEN N, et al. Chitinase-like protein CTL1 plays a role in altering root system architecture in response to multiple environmental conditions[J]. Plant Physiol. 2010, 152 (2):904-917.
[15] LI Y C, YANG Y C, HSU J S, et al. Cloning and immunolocalization of an antifungal chitinase in jelly fig (ficus awkeot-sang) achenes[J]. Phytochemistry, 2005, 66 (8): 879-886.
[16] SHAH M R, MUKHERIEE P K, EAPEN S. Expression of a fungal endochitinase gene in transgenic tomato and tobacco results in enhanced tolerance to fungal pathogens[J]. Physiol Mol Biol Plants, 2010 , 16 (1): 39-51.
[17] TAIRA T, TOMA N, ISHIHARA M. Purification, characterization, and antifungal activity of chitinase from pineapple (Ananascomosus) leaf [J]. Bioscience, Biotechnology, and Biochemistry, 2005, 69 (1):189-196.
[18] SHAH J M, RAGHUPATHY V, VELUTHAMBI K. Enhanced sheath blight resistance in transgenic rice expressing an endochitinase gene fromTrichodermavirens[J]. Biotechnology Letters, 2009, 31(2): 239-244.
[19] JABEEN N, CHAUDHARY Z, GULFRAZ M, et al. Expression of rice chitinase gene in genetically engineered tomato confers enhanced resistance to fusarium wilt and early blight [J]. Plant Pathol Journal, 2015, 31(3):252-258.
[20] KOVACS G, SAGI L, JACON G, et al. Expression of a rice chitinase gene in transgenic banana (‘Gros Michel’, AAA genome group) confers resistance to black leaf streak disease [J]. Transgenic Res, 2013, 22(1): 117-130.
[21] 朱荷琴, 冯自力, 李志芳, 等. 转几丁质酶和葡聚糖酶双价基因棉花株系对黄萎病的抗性[J]. 棉花学报, 2011, 23(1): 58-63.
[22] GENTILE A, DENG Z, LA M S, et al. Enhanced resistance to phoma tracheiphila and botrytis cinerea in transgenic lemon plants expressing aTrichodermaharzianumchitinase gene[J]. Plant Breeding, 2007, 126(2): 146-151.
[23] 朱伟, 房卫平, 谢德意,等. 水杨酸对NaCl胁迫下抗虫棉幼苗生长和生理特性的影响[J] 河南农业大学学报, 2012, 46(5):515-519.
[24] 金一锋.外源性水杨酸诱导月季对黑斑病抗性的研究[D]. 哈尔滨: 东北农业大学, 2013.
[25] 汤晓莉, 薛红芬, 邓国宾,等. 水杨酸诱导马铃薯疮痂病抗性的生理机制研究[J]. 西南农业学报, 2010, 23(6): 1851-1854.
[26] 张志忠, 吴菁华, 吕柳新,等. 植物几丁质酶及其应用研究进展[J]. 福建农林大学学报(自然科学版), 2005, 34 (4) : 494-499.
[27] 乔利仙, 刘晓琳, 隋烔明, 等. 黄曲霉病和水杨酸对花生几丁质酶诱导的研究[J]. 青岛农业大学学报, 2011, 28(2): 130-132.
(责任编辑:朱秀英)
MolecularcloningandexpressionanalysisofachitinasegeneChCHIfromChrysanthemummorifolium
CUI Bo1,2, SONG Caixia1, WANG Yingbo1, WANG Ruolan2,3, JIANG Suhua2, LIANG Fang2, YE Yongzhong1
(1.College of Life Sciences,Henan Agricultural University,Zhengzhou 450002,China; 2.Institute of Bioengineering,Zhengzhou Normal College,Zhengzhou 450044,China; 3.School of Life Sciences,Zhengzhou University,Zhengzhou 450001,China)
According to the conserved sequences of chitinase gene, the chitinase geneChCHIwas isolated from leaf ofChrysanthemummorifoliumusing RT-PCR and RACE method (The accession number in GenBank: KX881373). The full-length cDNA ofCHCHIwas 1385 bp, containing a 960 bp open reading frame which encodes 319 amino acids. Encoding product ofChCHIgene was a kind of hydrophilic and stable protein with a molecular weight of 33.5 kD and isoelectric point of 6.38. Prediction of secondary structure indicated that the protein was mainly composed of alpha helix and random coil. Sequence alignment and phylogenetic analysis showed that the amino acid sequence ofChCHIbelongs to family 19 of glycoside hydrolase. CHCHI was highly identified with the chitinase protein from Mikania micrantha (ACO25187.1) with the similarity of 87%. The results of qRT-PCR analysis showed that the expression level of theChCHIgene in leaves ofChrysanthemummorifoliumwas significantly increased under the SA treatments. The results implied thatChCHIgene may play an important role in the molecular regulation of resistance to environmental stress inchrysanthemummorifolium.
Chrysanthemummorifolium; chitinase gene; gene clone; expression analysis
Q949.718
:A
2016-10-31
郑州市重点科技攻关项目(141PZDGG189)
崔 波(1962-),男,河南泌阳人,教授,博士,主要从事花卉栽培与分子育种方面的研究。
叶永忠(1957-),男,湖北黄梅人,教授,博士生导师。
1000-2340(2017)02-0218-008

