Rv1635c基因及其编码蛋白质基本特性及抗原表位的生物信息学分析
赵隆麒,李海波,吴启航,王心倩,孙 妍,占卫红,余晓丽
(武汉轻工大学 生物与制药工程学院,湖北 武汉 430023)
Rv1635c基因及其编码蛋白质基本特性及抗原表位的生物信息学分析
赵隆麒,李海波,吴启航,王心倩,孙 妍,占卫红,余晓丽
(武汉轻工大学 生物与制药工程学院,湖北 武汉 430023)
对基因Rv1635c进行生物信息学分析,并对其T、B细胞的抗原表位性进行预测和筛选。用软件ProtParam、SingnalP、TMHMM分别对该基因编码蛋白质的理化性质、信号肽区进行分析预测。以NetMHC、Bimas、NetCTL和SYFPEITHI四个软件来预测分析Rv1635c的T细胞表位。采用IEDB和Bcepred来预测该基因的B细胞表位。分析结果为该基因编码的蛋白质原子总数8573,氨基酸数目556,分子质量60035.23,理论等电点9.84,理论分子式C2772H4323N747O717S14,估计半衰期30 h,不稳定指数39;由SignalP分析得到其不含信号肽;根据TMHMM分析可知Rv1635c为跨膜蛋白;T、B细胞表位预测显示该基因至少含有一个潜在的T、B细胞表位,可为未来疾病的预防、诊断和治疗提供理论基础。
Rv1635c;生物信息学;细胞表位
1 引言
结核病是一种有着长久历史的传染病,自1882年德国著名细菌学家、医学家罗伯特·科赫发现结核分枝杆菌以来,结核病仍然是仅次于艾滋病的第二大致死传染病。结核病疫情虽曾一度得到控制,但随着养殖业、畜牧业的发展,全球经济贸易交流广泛,以及大量的人口流动,使得耐多药及多耐药结核菌再次散布开来,导致结核病疫情在全球范围内卷土重来。近些年来,全球新增加数万例耐多药结核病,三分之一的患者因结核病死亡,其中近半数以上在中国和印度。由此可见我国的结核病疫情非常严峻。结核病治疗的发展经历过一个艰难的过程,从一开始的难以医治,到后来氨基水杨酸,异烟肼,利福平,乙胺丁醇的出现,让结核病的治疗出现很大的转机。而现在耐多药及多耐药结核菌来袭,需要科学家和医学家联手共同研制新型抗结核药物。
细胞壁是细菌表面最外层的部分,细菌在侵入宿主细胞时,需要细胞壁与之接触,因此对细胞壁的成分研究必不可少。脂阿拉伯甘露聚糖(lipoarabinomannan, LAM)是结核分枝杆菌的主要成分,其核心骨架为甘露聚糖,侧链为阿拉伯糖和甘露糖结构[1]。LAM可以介导细胞免疫和体液免疫。有研究表明,LAM能够通过对树突状细胞、T细胞、巨噬细胞的相互作用来介导细胞免疫反应。而LAM含有的9个单克隆抗体表位,具有强免疫原性是其能够介导体液免疫的原因[2]。我国其他科学家通过对甘露糖修饰的脂阿拉伯甘露聚糖研究后发现LAM可以通过在体内与CD1d结合诱导调节性B细胞的产生,同时以B细胞分泌的IL-10来抑制CD4+T细胞介导的免疫反应[3]。种种实验说明LAM作为结核分枝杆菌细胞壁上的主要糖脂,与结核分枝杆菌在宿主体内的存活有很大的关系。
到目前为止,已知的LAM至少有两类,一类是manLAM,来自结核分枝杆菌H37Rv,另一类是araLAM,来自于结核分枝杆菌H37Ra。两者的差别为前者比后者多了甘露糖帽化结构。Rv1635c为结核分枝杆菌H37Rv上的一段基因序列,根据有关资料显示它可能转录翻译成甘露糖基转移酶或与甘露糖基转移酶功能相关的蛋白。
2 实验方法
采用生物信息学分析软件,对Rv1635c的理化性质、信号肽区、跨膜区和T、B细胞表位进行前期预测。
2.1 分析目的基因Rv1635c理化性质
使用在线分析程序ExPASy-Protparam(http://web.expasy.org/protparam/)分析该目的基因所编码的成熟蛋白质的分子质量、氨基酸数目及组成等理化性质。
2.2 预测目的基因Rv1635c信号肽结构
SingnalP(http://www.cbs.dtu.dk/services/SignalP/)用于对目的基因是否含有信号肽的预测。
2.3 预测目的基因Rv1635c跨膜结构
TMHMM(http://www.cbs.dtu.dk/services/TMHMM/)用于分析目的基因是否存在跨膜区。
2.4 预测目的基因Rv1635c T细胞表位
2.4.1 预测目的基因Rv1635c CD4+T细胞表位
打开网页,输入NetMHC[4]的网址http://www.cbs.dtu.dk/services/NetMHCIIpan/。将Rv1635c的氨基酸序列复制粘贴后输入方框中,设置15为aa长度,将等位基因(Select Allele)设置为DRB1_0101、DRB1_0301、DRB1_0401、DRB1_0701、DRBA_0802、DRB1_0901、DRB1_1101、DRB1_1302、DRB1_1501,设置亲和值(Affinity)作为输出结果的排列方式,点击submit开始预测。
2.4.2 预测目的基因Rv1635c CD8+T细胞表位
打开网页,输入NetMHC[5]的网址http://www.cbs.dtu.dk/services/NetMHC/。将Rv1635c的氨基酸序列复制粘贴后输入方框中,设置aa长度为9,选择HLA-A 0201和HLA-A 0301[6]为等位基因。设置亲和值(Affinity)作为输出结果的排列方式,点击submit开始预测。
打开网页,输入Bimas[7]的网址http://www-bimas.cit.nih.gov/molbio/hla_bind/。选择HLA分子为A-0202和A3。设置aa长度为9,输入序列点击submit开始运行。
打开网页,输入NetCTL[8]的网址http://www.cbs.dtu.dk/services/NetCTL/,选择supertype为A2和A3,Sort by score选择为Combined score,输入其氨基酸序列,点击submit开始运行。
打开网页,输入SYFPEITHI的网址http://www.syfpeithi.de/bin/MHCServer.dll/EpitopePrediction.htm ,选择MHC类型为HLA-A-0201和HLA-A-03,设置9为aa长度,输入目的基因氨基酸序列,点击run开始运行。
2.5 预测目的基因Rv1635c B细胞表位
蛋白质所含的某段氨基酸残基能否作为抗原表位在于它是否存在于该蛋白质的表面,根据这个性质我们通过亲水性、表面可及性、柔韧性、抗原性、抗原指数性以及β转角做分析。在一个机体内,疏水基团一般在蛋白质内部,而亲水集团通常位于表面,因此以亲水性作为方案具有可行性。表面可及性表示蛋白质分子所含的氨基酸残基与溶剂分子接触的可能性,可以反映出氨基酸残基在蛋白质分子内外的分布情况。现有的柔韧性分析方法是预测蛋白质某些片段的活动性,进而达到预测表位的可能性。抗原性分析法是氨基酸在抗原区出现频率与氨基酸在蛋白质中的频率的比值,作为抗原性刻度值来对其成为抗原的可能性做预测分析。
这里我们采用的是IEDB和Bcepred两个在线分析软件对Rv1635c B细胞表位进行预测,IEDB采用的是基于上述原理的五种参数,β转角、表面可及性、柔韧性、抗原性以及亲水性。打开IEDB的网站http://tools.immuneepitope.org/bcell/,选取上述五种参数分别进行分析,输入Rv1635c的氨基酸序列,点击Submit。Bcepred选取的是抗原倾向性、暴露表面、柔韧性、β转角、亲水性、表面可及性和极性这七种参数进行分析。打开Bcepred的网站http://www.imtech.res.in/raghava/bcepred/bcepred_submission.html,输入氨基酸序列,同时选择七项参数,然后点击Submit sequence。
3 结果
3.1 分析目的基因Rv1635c理化性质
由ExPASy-Protparam软件分析目的基因Rv1635c后得到,该目的基因编码的蛋白质由556个氨基酸组成,其原子总数为8573,分子质量为60035.23,预测理论等电点为9.84,理论分子式C2772H4323N747O717S14,预测半衰期为30 h,不稳定指数为39。
3.2 预测目的基因Rv1635c信号肽结构
根据软件SingalP的分析,显示目的基因不含信号肽结构。
3.3 预测目的基因Rv1635c跨膜结构
由TMHMM的在线分析预测,显示基因Rv1635c为跨膜蛋白,具有跨膜结构。
3.4 预测目的基因Rv1635c T细胞表位
3.4.1 预测目的基因Rv1635c CD4+T细胞表位
由软件NetMHCIIpan对Rv1635c基因的预测,得到如表1结果。根据表1中的数据,DRB1_0901肽段结合数和Strong Binding数目都是最多的,因此在DRB1_0901中选择细胞表位的可能性较高。32个Strong Binding为362-376PRYLILTAPAAAVIL,363-377RYLILTAPAAAVILA,361-375YPRYLILTAP
AAAVI,448-462GPIRALLATRPAAFR,447-461PGP
IRALLATRPAAF,295-309HRQYFDHSVPFAILA,542-556ERWQFHYSQVVKSTR,449-463PIRALLATRP
AAFRS,296-310RQYFDHSVPFAILAG,364-378YLI
LTAPAAAVILAV,360-374YYPRYLILTAPAAAV,541-555VERWQFHYSQVVKST,446-460RPGPIRALL
ATRPAA,294-308IHRQYFDHSVPFAIL,450-464IRA
LLATRPAAFRSL,297-311QYFDHSVPFAILAGL,
445-459WRPGPIRALLATRPA,540-554IVERWQF
HYSQVVKS,120-134HGWFAIFPPTELWSR,119-133MHGWFAIFPPTELWS,451-465RALLATRPA
AFRSLI,365-379LILTAPAAAVILAVC,393-407VVF
LLAAAAFPNYFF,359-373IYYPRYLILTAPAAA,392-406GVVFLLAAAAFPNYF,118-132LMHGWFAIFP
PTELW,228-242ALLVPAYATMVPLLA,308-322
LAGLIVAAGIAAHLA,310-324GLIVAAGIAAHLA
GA,309-323AGLIVAAGIAAHLAG,121-135GWF
AIFPPTELWSRL,298-312YFDHSVPFAILAGLI。
表1 目的基因Rv1635c与人类白细胞抗原结合表
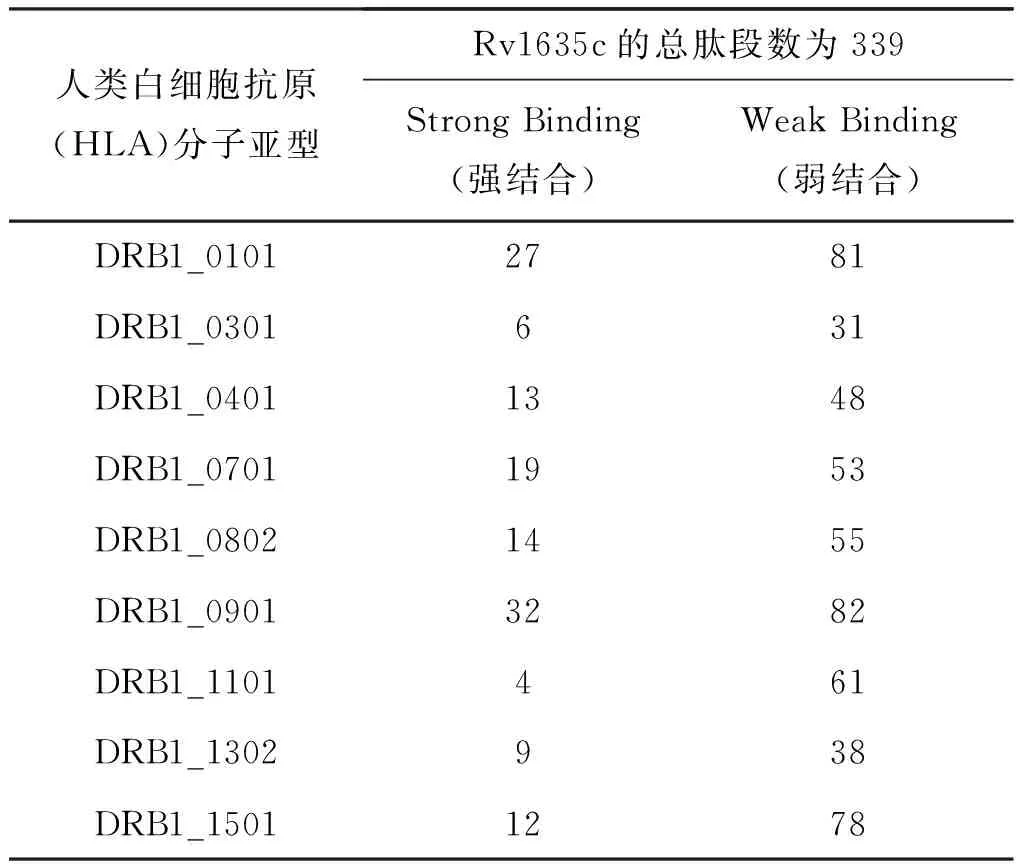
人类白细胞抗原(HLA)分子亚型Rv1635c的总肽段数为339StrongBinding(强结合)WeakBinding(弱结合)DRB1_01012781DRB1_0301631DRB1_04011348DRB1_07011953DRB1_08021455DRB1_09013282DRB1_1101461DRB1_1302938DRB1_15011278
3.4.2 预测目的基因Rv1635c CD8+T细胞表位
把预测目的基因的四个软件NetMHC、Bimas、NetCTL、SYFPEITHI预测的结果进行统计汇总,如表2所示。
表2 四种软件对CD8+T细胞表位预测汇总表
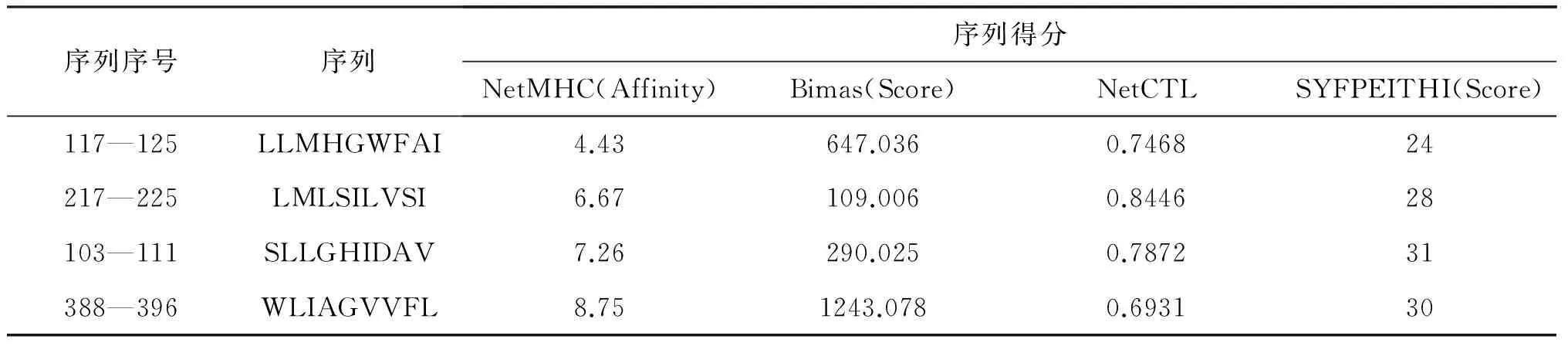
序列序号序列序列得分NetMHC(Affinity)Bimas(Score)NetCTLSYFPEITHI(Score)117—125LLMHGWFAI4.43647.0360.746824217—225LMLSILVSI6.67109.0060.844628103—111SLLGHIDAV7.26290.0250.787231388—396WLIAGVVFL8.751243.0780.693130
3.5 预测目的基因Rv1635c B细胞表位
根据IEDB和Bcepred两个软件的预测,选取两软件中得分较高的几项,最终选择的序列为243—249,504—510,19—25。分别对照三个序列在IEDB和Bcepred中的得分(表3),最终得到最可能的B细胞表位为243—249。
表3 B细胞表位预测结果
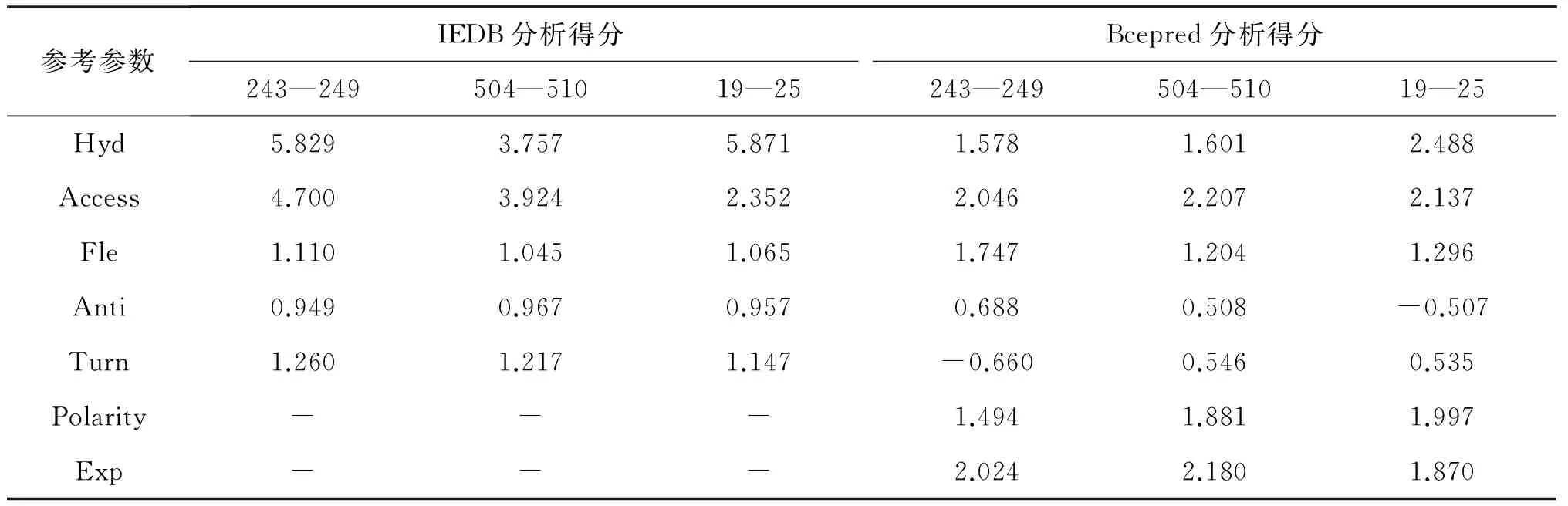
参考参数IEDB分析得分Bcepred分析得分243—249504—51019—25243—249504—51019—25Hyd5.8293.7575.8711.5781.6012.488Access4.7003.9242.3522.0462.2072.137Fle1.1101.0451.0651.7471.2041.296Anti0.9490.9670.9570.6880.508-0.507Turn1.2601.2171.147-0.6600.5460.535Polarity---1.4941.8811.997Exp---2.0242.1801.870
注:Bcepred所取数据为7个数据平均值四舍五入。
4 讨论
目前已有的药物并不能很好地达到治疗结核病的效果,急需有新的药物来对抗耐药型结核病。长久以来科研工作者致力于寻找新的抗原表位,以此来为新药的产生寻找契机,虽然已有很多的特异性抗原表位被发现,但在如此严峻的结核疫情下,需要找到更多新的靶点来制造新药。
结核分枝杆菌细胞壁的主要的成分有脂甘露聚糖,脂阿拉伯甘露聚糖,磷脂酰肌醇甘露糖。磷脂酰肌醇甘露聚糖对细胞的膜的稳定性有一定的作用。脂阿拉伯甘露聚糖是结核分枝杆菌细胞壁上的一种重要的糖脂,并且在含有甘露糖帽结构时能够对巨噬细胞有作用,从而发生免疫调节反应。有研究认为,脂阿拉伯甘露聚糖能够通过抑制吞噬溶酶体合成,从而在吞噬细胞中生存。抑制原因主要是由于甘露糖基和磷脂酰肌醇锚定基与巨噬细胞上的某一部分结合,使吞噬小体的成熟被阻止,达到抑制吞噬溶酶体合成的目的。根据已有的研究表明Rv1635c跟甘露糖基转移酶有一定关系。对于病原菌的抑制通常使用两种方法,一种是通过其细胞上的靶位点对病菌进行直接抑制,另一种是阻断它的重要物质的代谢过程,从而到达抑制整个病菌的目的。Rv1635c与甘露糖基转移酶合成有关,因此对Rv1635c的研究具有一定的意义。
笔者对Rv1635c做的生物信息学分析是对其基本理化性质,以及一些可能结构的预测。对其T细胞表位做预测时,由于CD4+T细胞所选用的软件只有一个,而CD8+T细胞所选用的软件有四个,故因将CD8+T细胞的四个软件预测分析汇总后再与CD4+T细胞的一个预测软件做比较,最终综合得出T细胞表位的预测结果。B细胞表位的上述预测方法都是基于对线性B细胞表位进行预测的。由于是线性的,故B细胞表位所包含的氨基酸数目是没有确定数值的,在此我们选用氨基酸数目为7以便与统计。以上结果均是通过软件进行预测,存在一定的误差。生物信息学只能作为前期寻找方向并不能直观的说明该基因是否一定可作为抗原表位,若想确认还需要进一步实验来支撑。
[1] 彭荣,乐军,韩敏,等. 结核分枝杆菌脂阿拉伯甘露聚糖[J]. 生命的化学,2010,(05):704-707.
[2] 王芳,乐军,彭荣. 胸水中脂阿拉伯甘露聚糖抗原检测对结核性胸膜炎的诊断价值[J]. 检验医学,2012,(03):199-201.
[3] 章晓联. 结核分枝杆菌表面脂糖的免疫抑制作用相关研究[C]//中国免疫学会(Chinese Society for Immunology).第九届全国免疫学学术大会论文集.北京:中国免疫学会(Chinese Society for Immunology):2014:2.
[4] Andreatta M, Karosiene E, Rasmussen M, et al.Accurate pan-specific prediction of peptide-MHC class II binding affinity with improved binding core identification[J]. Immunogenetics,2015,67(11):641-650.
[5] Andreatta M, Nielsen M.Gapped sequence alignment using artificial neural networks: application to the MHC class I system[J].Bioinformatics 2016,32(4):511-7.
[6] 王心倩,孙妍,占卫红,雷航,陈高瞻,余晓丽. 结核分枝杆菌Rv3607c T细胞表位分布情况预测及分析[J]. 武汉轻工大学学报,2016(2) : 36-39.
[7] Parker K C,Bednarek M A,Coligan J E. Scheme for ranking potential HLA-A2 binding peptides based on independent binding of individual peptide side-chains.[J]. Journal of immunology,1994,152(1):163-175.
[8] Larsen Mette V,Lundegaard Claus,Lamberth Kasper,et al.Large-scale validation of methods for cytotoxic T-lymphocyte epitope prediction.[J]. BMC Bioinformatics,2007,8(1):424.
[9] 南文龙,金宁一,鲁会军,等. H5N1亚型禽流感病毒血凝素Th和B细胞表位预测及抗原性分析[J]. 中国免疫学杂志,2009,25(7):630-633.
[10] 叶娟,张舒林,刘文第. 结核分枝杆菌RD12区T细胞表位分布情况预测及分析[J].上海交通大学学报(医学版),2014,34(1) : 7-12.
[11] 刘海灿. 结核分枝杆菌八种蛋白抗原中人T/B细胞抗原表位多态性研究[D].北京:中国疾病预防控制中心,2014.
[12] 顾晓玲. 结核分枝杆菌脂阿拉伯甘露聚糖合成途径相关基因的克隆和功能研究[D].上海:复旦大学,2006.
[13] 杨依丽. α-1,6甘露糖转移酶基因缺陷的毕赤酵母菌在流感疫苗研究中的应用[D].北京:中国人民解放军军事医学科学院,2009.
Bioinformatics analysis to characteristics and antigen epitopes of Rv1635c
ZHAOLong-qi,LIHai-bo,WUQi-hang,WANGXin-qian,SUNYan,ZHANWei-hong,YUXiao-li
(School of Biology and Pharmaceutical Engineering,Wuhan Polytechnic University,Wuhan 430023,China)
Analysising gene Rv1635c by using bioinformatics, predicting and screening T/B cell epitope.ProtParam is used to analysis the physical and chemical propertiesthe.SingnalP is used to analysis the signal peptide.And TMHMM is used to transmembrane region of the gene.The four software NetMHC、Bimas、NetCTL and SYFPEITHI is used to predict and analysis the epitope of T cell.IEDB and Bcepred is used to predict and analysis the epitope of B cell.The result shows that the protein coded by the gene comprises an atomic number of 8573,556 amino acid,a molecular mass of 60035.23, a theoretical isoelectric point of 9.84, a theoretical molecular formula of C2772H4323N747O717S14, an estimated half-life of 30h, an instability index of 39;The SignalP shows that there are not signal peptide in it.The TMHMM shows that the gene Rv1635c is a transmembrane protein.The prediction of T/B cell epitope shows that there is one potential epitope at least,which provide basis for future prevention,diagnosis and treatment.
Rv1635c;bioinformatics;cell epitope
2017-03-29.
赵隆麒(1992-),男,硕士研究生,E-mail:q304451487@vip.qq.com.
余晓丽(1963-),女,教授,E-mail:yxll268@126.com.
2095-7386(2017)02-0031-05
10.3969/j.issn.2095-7386.2017.02.006
Q 93
A

