基于CRISPR/Cas9系统的水稻光敏色素互作因子OsPIL15基因编辑
季新,李飞,晏云,孙红正,张静,李俊周,彭廷,杜彦修,赵全志
基于CRISPR/Cas9系统的水稻光敏色素互作因子基因编辑
季新,李飞,晏云,孙红正,张静,李俊周,彭廷,杜彦修,赵全志
(河南农业大学农学院/河南粮食作物协同创新中心/河南省水稻生物学重点实验室,郑州 450002)
【目的】光作为一种环境信号,可影响植物的基因表达、酶活性和形态建成。光敏色素互作因子在光信号传导过程中起着重要作用。本研究旨在构建水稻光敏色素互作因子的CRISPR/Cas9表达载体,创制突变体,挖掘水稻功能基因,丰富和完善水稻光信号调控分子机制。【方法】依据CRISPR/Cas9技术原理,设计突变靶点。将所设计靶序列在水稻基因组中进行比对,排除非特异性靶位点,同时使该靶序列含有常用酶切位点,方便后期突变体鉴定。化学合成靶位点寡核苷酸序列并与载体pBUN411连接构建CRISPR/Cas9表达载体,利用农杆菌介导法导入粳稻品种日本晴,以除草剂抗性标记筛选获得阳性转基因植株。利用酶切法判断T0代转基因植株是否发生突变,结合测序结果分析突变单株的突变基因型。将靶点序列在水稻全基因组中进行比对分析,选择5个与靶序列同源性较高且错配在4 bp以内的位点作为潜在脱靶位点进行脱靶效应评估,分析所设计靶序列特异性。【结果】所构建表达载体成功实现了对的定向编辑,酶切显示在选取的25株T0代转基因植株中获得15株突变体,其中包括5株纯合突变体、6株双等位突变体和4株杂合突变体,共10种不同突变基因型和11个突变株系。突变类型以单碱基插入或缺失为主,同时也得到2种56和66 bp较大片段缺失株系。对部分纯合突变、双等位突变和杂合突变体的T1代植株进行分析,结果表明,T0代产生的突变基因型绝大部分能稳定遗传给下一代。T0代纯合突变体后代为纯合突变单株,仅在株系14纯合突变体后代中检测到1株未突变单株;T0代双等位突变体后代可得到2种纯合突变型和1种双等位突变型;T0代杂合突变体后代则可得到纯合、杂合及未突变3种类型。对T0代未突变植株的后继世代酶切分析显示,62株T1代转基因植株均未发生突变,表明CRISPR/Cas9在T1代转基因阳性植株中未重新发挥基因编辑作用。对20株突变体的5个潜在脱靶位点进行分析,5个潜在脱靶位点均未检测出脱靶效应,表明所设计靶序列具有较高特异性。对选取的3组不同基因型T1代突变体表型进行初步观察,结果表明,突变体生育期和分蘖数未出现明显变化,株高极显著下降,籽粒粒长极显著增加,最大增幅达5.69%。【结论】CRISPR/Cas9系统能对进行定向编辑,获得的10种不同突变基因型的突变体与野生型相比株高极显著降低、籽粒粒长极显著增大。
水稻;CRISPR/Cas9;基因编辑;;脱靶效应
0 引言
【研究意义】光是作物生长发育所需的最重要生态因子之一,是作物产量形成的基础,同时光也可作为一种重要的环境信号,调节植物基因的表达、影响酶的活性以及植物的形态建成。在光信号途径中,光敏色素互作因子起着承上启下的作用,因此,利用CRISPR/Cas9基因编辑技术创制光敏色素互作因子的突变体,研究其在光信号调控途径中的作用,对挖掘水稻功能基因,丰富和完善水稻光信号调控分子机制具有重要意义。【前人研究进展】基因编辑技术指能够对目标基因进行“编辑”,实现对特定DNA片段的敲除、插入等。过去几年,以锌指核酸酶(zinc fnger nucleases,ZFNs)和类转录激活因子效应物核酸酶(transcription activator-like nucleases,TALENs)为代表的序列特异性核酸酶技术可以高效率的定点编辑基因组,在基础研究、基因治疗和遗传改良等方面发挥了重要作用[1-2]。近年来,一种名为CRISPR/Cas9(clustered regularly interspaced short palindromic repeats/CRISPR-associated nuclease 9,Cas9)的基因组定向编辑技术备受瞩目。2013年初,《Science》两篇文章首次报道了Cas9核酸酶用于人类和小鼠细胞基因组编辑[3-4],其作为第三代基因编辑技术受到人们广泛关注。CRISPR/Cas9技术是利用核酸酶Cas9蛋白与单导向RNA(single guide RNA,sgRNA)形成复合体,sgRNA通过碱基互补配对决定靶序列特异性,Cas9蛋白作为核酸酶切割与sgRNA上的间隔序列(spacers)互补的基因组DNA,造成双链DNA损伤,随后通过体内的NHEJ(non-homologous end joining)修复机制引入基因突变[5]。随着CRISPR/ Cas9系统在人类和动物细胞中建立与应用,CRISPR/ Cas9系统在拟南芥、烟草、水稻、小麦、玉米、高粱、番茄和甜橙等植物中均实现了基因组的定向编辑[6-7]。光敏色素互作因子PIF(phytochrome-interacting factor)或称PIL(phytochrome interacting factor-like)是bHLH转录因子家族中的一类转录因子,其主要特性是能与光敏色素互作直接或间接调控光响应基因。作为bHLH蛋白的一种,所有PIF家族的蛋白都包含在N端与光敏色素互作的APB(active phytochrome B-binding)或APA(active phytochrome A-binding)结构域和C端bHLH-DNA结合结构域及核定位结构域[8-9]。拟南芥PIF能够与靶基因启动子区域的G-box(5′-CACGTG-3′)或PBE-box(PIF- binding E-box, 5′-CACATG-3′)相结合[10],可调控种子萌发[11]、幼苗去黄化[12]、避阴反应[13]和昼夜节律[14]等植物生长发育的各个方面。Nakamura等[15]通过同源性分析在水稻基因组中鉴定了6个PIF转录因子(OsPIL11—OsPIL16),相对于拟南芥而言,PIFs在水稻生长发育过程中的作用研究相对较少。参与红光诱导的幼苗去黄化反应[16];在水稻中超表达后能够促进水稻节间的伸长,反之低表达则抑制其节间伸长[17];超表达的水稻种子在黑暗环境中的萌发受到抑制[18];能够负调控进而调控籽粒大小[19]。【本研究切入点】笔者前期通过高通量测序研究发现,在水稻籽粒抽穗后10、15、21、27和35 d这5个灌浆时期的强势粒和弱势粒中,6个水稻光敏色素互作因子有3个被鉴定出,其中和只在弱势粒某个时期表达,则在强弱势粒的5个灌浆时期均表达[20],而目前有关在水稻籽粒灌浆中的调控作用还不清楚。【拟解决的关键问题】本研究拟利用CRISPR/Cas9系统对水稻光敏色素互作因子进行编辑,转化粳稻品种日本晴,对其突变位点和潜在脱靶位点进行分析鉴定,通过对突变体表型进行观察,了解功能及其调控路径。
1 材料与方法
1.1 试验材料
使用粳稻品种日本晴(sspcv. Nipponbare)作为转基因受体材料,CRISPR/Cas9载体pBUN411由中国农业大学陈其军教授惠赠[21],试验所用引物(表1)和测序分析由苏州金唯智生物科技有限公司完成。

表1 本研究所用引物
引物序列中小写字母为Ⅰ限制性内切酶的黏性末端接头 The lowercase stand for sticky ends ofⅠ
1.2 靶位点设计和CRISPR/Cas9表达载体构建
根据水稻(GenBank登录号AK102252.1)外显子序列设计sgRNA靶点序列。在尽量靠近外显子编码蛋白质的N端,选取PAM(protospacer-adjacent motif)序列(NGG)5′端的一段碱基序列作为靶位点,将该段序列在水稻基因组数据库中进行比对,以排除非特异性靶位点,同时尽可能使PAM 5′端存在常用酶切位点,以方便后期突变体鉴定。
将选取的靶点序列作为正义链,其互补链作为反义链,分别在2条DNA单链5′端添加ggcg和aaac序列作为Ⅰ限制性内切酶的黏性末端接头。使用限制性内切酶Ⅰ酶切CRISPR/Cas9载体pBUN411质粒使其线性化,T4 DNA连接酶连接后用热激法转化大肠杆菌DH5α感受态细胞,使用引物pBUN411- VF和pBUN411-VR进行菌落PCR验证。CRISPR/Cas9表达载体转化农杆菌EHA105,用除草剂(Basta)筛选获得再生苗。
1.3 酶切和测序分析验证突变位点
为了检测靶位点的突变情况,在靶位点两侧设计引物OsPIL15-F和OsPIL15-R,以转基因阳性单株DNA为模板扩增靶位点序列。使用Ⅰ限制性内切酶酶切PCR产物,2%琼脂糖凝胶电泳检测是否发生突变。将突变单株PCR产物测序,对靶位点附近出现套峰的PCR产物进行TA克隆,随机选取单克隆测序,测序结果与野生型序列比对分析确定突变基因型。
1.4 潜在脱靶位点分析
使用The Rice Annotation Project(http://rapdb.dna. affrc.go.jp/)在线BLAST工具,将靶序列在水稻全基因组中进行比对,选取与靶序列同源性较高、错配在4 bp以内且具有PAM序列NGG的位点作为潜在脱靶序列,通过酶切法评估其脱靶效应。
2 结果
2.1靶点设计和表达载体构建
根据CRISPR/Cas9技术原理,在外显子区域靠近起始密码ATG区域选择一段19 bp的序列(5′-GACTTCTTCTCCGAGCTCC-3′)作为靶位点。该序列3′端PAM序列为AGG,Cas9蛋白将在AGG序列上游第3—4 bp处剪切DNA形成平滑末端,该剪切处有一段限制性内切酶Ⅰ识别序列(5′-GAGCT▼C-3′),选择能被限制性内切酶识别并切割的位点进行突变,有利于后续对转基因阳性植株是否发生突变进行筛选。CRISPR/Cas9表达载体的靶序列由启动子驱动,编码Cas9蛋白基因由玉米泛素基因()启动子驱动。
2.2T0代突变体筛选鉴定
使用引物OsPIL15-F和OsPIL15-R扩增25株阳性转基因单株靶点序列(图1-A),野生型扩增片段大小为673 bp,单株1、2、23和24均扩增出片段明显小于野生型的条带,初步表明这4个单株可能发生较大片段缺失,其中单株1和24的PCR产物出现两条带,只有其中一条带明显小于野生型条带,因此二者可能只有一条同源染色体有较大片段缺失;单株2和23的PCR产物只有一条带且明显小于673 bp,可初步判断二者两条同源染色体都发生较大片段缺失。其余21个单株不能根据条带大小判断是否突变,因此,利用酶切法继续进行鉴定。
使用Ⅰ酶切PCR产物,若PCR产物能被完全切开,得到400和273 bp的两条带,表明靶位点未发生突变,为未突变单株;若PCR产物被部分切开,得到400、273和约673 bp三条带,表明一条同源染色体突变,为杂合体;若PCR产物完全不能切开,表明两条同源染色体均发生突变,则可能为2个等位基因发生不同突变的双等位突变体或2个等位基因发生相同突变的纯合体。如图1-B所示,结果表明,单株4、5、6、7、10、12、15、16、18和25未发生突变;单株8、9、11和17的PCR产物被部分酶切,形成三条带,为一条同源染色体突变的杂合体;单株1、2、3、13、14、19、20、21、22、23和24的PCR产物完全不能切开,则可能为双等位突变或纯合突变体。
PCR产物片段大小和酶切PCR产物只能初步定性判断转基因植株靶点是否发生突变,而突变体靶点序列的改变还需测序进行鉴定。由于杂合突变或双等位突变的两条同源染色体突变情况不一致,用PCR产物测序时会在某一位点(一般在突变位点附近)之后出现重叠峰,后续序列无法准确得知,因此,对可能出现重叠峰的突变体进行TA克隆,随机选取单菌斑进行测序。同时对突变基因型一致的单株进行Southern blot分析(电子附图1),检测是否属同一株系,结果表明,1和24、2和23、3和14、19和21分属同一株系。综合分析测序结果和Southern blot结果得到各单株突变情况(图2),共获得5株纯合突变体(2、3、13、14和23)、6株双等位突变体(1、19、20、21、22和24)和4株杂合突变体(8、9、11和17),包含10种不同突变基因型和11个突变株系,多为单碱基的插入或缺失,但也得到了2种56和66 bp较大片段缺失的株系。
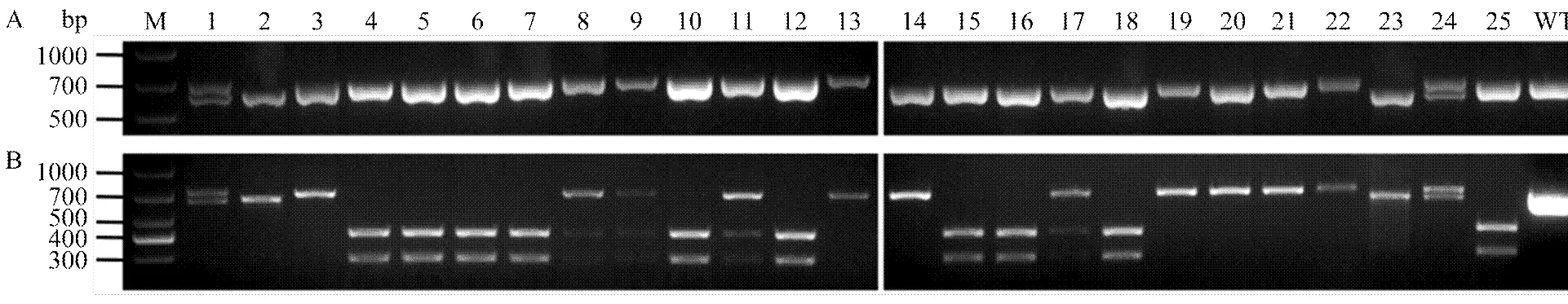
A:PCR产物酶切前;B:PCR产物酶切后。1—25:T0代转基因单株;M:DL1000标记;WT:野生型(日本晴);+RE:使用SacⅠ酶切;黑色三角:SacⅠ酶切后预期片段。下同

蓝色字母:靶位点序列;黄色高亮:PAM序列;红色删除线:缺失碱基;红色小写字母:插入碱基;-示缺失;+示插入;M1/M2:双等位2种突变型
2.3T1代突变体分析鉴定
为验证T0代突变体产生的突变序列能否稳定遗传,选取部分纯合突变、双等位突变和杂合突变株系的下一代植株,对这些T1代植株突变情况进行测序分析(表2),纯合突变体(3和14)后代为纯合突变单株,但株系14的30株T1代植株中有29株纯合突变,发现1株未突变单株;3个双等位突变体(21、22和24)后代既获得了纯合突变体,也获得了双等位突变体;2个杂合突变体(9和17)的T1代单株中,既有纯合突变和杂合突变体,也有未突变的单株。上述结果表明,T0代突变体产生的突变基因型基本上能稳定遗传给下一代,纯合株系后代为纯合体,双等位株系后代产生纯合体和双等位突变体,而杂合株系后代会得到未突变、纯合和杂合3种类型。
为了鉴定T1代单株是否会重新发生基因编辑,随机选取了62株T1代为阳性(含有Cas9基因)且在T0代未发生突变的转基因单株,使用限制性内切酶Ⅰ进行酶切鉴定(图3),62个阳性转基因单株PCR产物均被切开,得到400和273 bp的两条带,因此,判定这62株T1代转基因植株均未发生突变。表明T0代未突变株系,T1代依然未出现突变。

表2 T1代植株分析鉴定
M1/M2示图2中两条同源染色体突变类型
M1/M2 indicates the mutation types of two homologous chromosomes in Fig. 2

62个编号:T0代未突变(4、5、6、7、10、12、15、16、18和25)的T1代阳性转基因单株;-RE:未进行酶切。下同
2.4突变体脱靶效应评估
CRISPR/Cas9系统基因编辑的特异性主要取决于sgRNA的识别序列,但由于高等生物基因组较大且较为复杂,sgRNA可能会与非靶点DNA序列发生局部错配,造成CRISPR/Cas9系统基因编辑的脱靶。因此,需要对的突变体脱靶效应进行评估(表3),选取水稻基因组中与sgRNA序列错配碱基数在4 bp以内且具有PAM序列的5个位点作为潜在脱靶位点,每个潜在脱靶位点均随机选取10株T0代突变单株和10株T1代突变单株,通过酶切法进行检测(图4)。Off-target1位点若被Ⅰ酶切可形成444和179 bp的两条带,Off-target2位点酶切后产生359和173 bp的两条带,Off-target3位点酶切可得到268和152 bp的两条带,Off-target4位点可被酶切为411和153 bp的两条带,Off-target5位点若被切开会得到360和254 bp两条带。所选取的20个突变单株的5个潜在脱靶位点PCR产物均能被Ⅰ酶成功切割,因此,这5个潜在脱靶位点在T0代和T1代均未产生脱靶效应,表明所设计的sgRNA序列具有较高的特异性。

表3 CRISPR/Cas9系统潜在脱靶位点突变检测
灰色标记为错配碱基,下划线标记为PAM序列
mismatching bases are shown by a grey box, the PAM motif is marked by an underline

20个编号:10株T0代突变体和10株T1代突变体
2.5功能初步分析
选择水稻中PIFs家族的6个基因(Os12g0610200)、(Os03g0639300)、(Os03g0782500)、(Os07g0143200)、(Os01g0286100)、(Os05g0139100)和拟南芥中PIFs家族的7个基因/(AT2G20180)、(AT1G09530)、(AT2G43010)、/(AT3G59060)、/(AT3G62090)、(AT5G61270)、(AT4G00050),根据它们的氨基酸序列进行系统进化树分析(图5-A),水稻中光敏色素互作因子OsPIL15和OsPIL16亲缘关系最近,二者蛋白同源性几乎达到了100%。有研究表明,超表达能使水稻籽粒减小[22],OsPIL15和OsPIL16十分相近的亲缘关系表明也有可能调控水稻籽粒大小。因此,选取T1代不同基因型的三组突变体,对其表型进行观察,与野生型相比,突变体生育期没有变化,株高极显著降低,平均降低12.49%;有效分蘖数没有明显差异;而籽粒粒长极显著增加,其中,突变体22增加最高,增幅达5.69%,增幅最小的突变体21也达3.88%(图5-B—图5-E)。

A:OsPIL15蛋白系统进化树分析;B:ospil15突变体株高变化;C:ospil15突变体有效分蘖数变化;D:ospil15突变体籽粒粒长变化;E:ospil15突变体与野生型日本晴籽粒表型比对。**:在0.01水平上差异显著;Bar=0.5 cm
3 讨论
CRISPR/Cas9技术以其突变高效、操作简单、成本低廉等优势在动植物中得到了广泛应用。本研究从靶位点选择,表达载体构建,突变位点分析和脱靶效应评估等方面,详细阐述了利用CRISPR/Cas9系统对水稻的编辑。本研究针对设计了1个靶向位点,有研究认为单靶点在T0代形成的小片段突变,使得T1代植株中sgRNA仍有可能识别靶序列造成二次编辑,后代突变具有不稳定性[23]。本研究在纯合突变体14后代中发现了一株未突变单株,且突变体14仅为1个单碱基插入突变,是否是由于CRISPR/ Cas9系统在T1代再次对靶位点进行编辑形成新的碱基改变还需进一步验证。本试验获得突变体多为单碱基插入或缺失,但也得到了两种分别缺失66和56 bp的较大片段缺失突变体。针对基因编码序列设计多个靶位点有利于形成大片段缺失突变而获得稳定遗传,但靶点越多产生脱靶的可能性越大[24],需综合考虑目标基因靶点位置及数量,尽可能保证其特异性。本试验采用酶切和测序相结合的方法对突变位点进行分析鉴定,但由于杂合突变和双等位突变的PCR产物直接测序会出现重叠峰而无法准确读取,需将PCR产物进行TA克隆后挑选单菌斑测序,工作量较大,成本较高。为此华南农业大学刘耀光教授课题组基于DSD(degenerate sequence decoding)法[25]开发了在线工具DSDecode(http://dsdecode.scgene.com/),可对各突变类型测序文件自动解码[26],但限于不同公司和不同PCR产物测序质量,部分测序结果不能很好地解码,因此,此方法可为测序分析提供参考,具体仍需验证。
CRISPR/Cas9系统成功对植物基因组编辑后,所获得突变基因序列能否稳定遗传给下一代决定了该突变体是否具有应用前景。由于拟南芥多采用蘸花法进行转基因,CRISPR/Cas9系统诱导的突变在T1代中主要发生在体细胞,从而形成嵌合体,较难稳定遗传给下一代,而发生在生殖细胞中的突变可稳定遗传[27]。水稻多采用农杆菌介导愈伤组织,转基因苗多来源于单个细胞或细胞团,因此本试验在T0代即获得了纯合突变体,对纯合突变、双等位突变和杂合突变的T1代植株分析显示这些突变绝大多数均能稳定遗传,有研究证明这些稳定遗传突变体自交后代分离均符合孟德尔遗传定律[28-29]。有报道认为CRISPR/Cas9系统在后代植株当中对野生型依然可行使功能[30],T0代转基因植株靶位点表现为野生型的植株后代仍有可能发生一定比例的突变[27]。本研究对T0代未突变植株的T1代(阳性)靶位点突变情况进行鉴定,未获得新的突变单株。Zhang等[31]认为这可能与CRISPR/Cas9系统插入到基因组的位置有一定关系。本研究仅对部分T1代植株进行了鉴定,CRISPR/Cas9系统是否会在其他后继世代中重新发挥作用还需作进一步验证。
CRISPR/Cas9系统在植物基因编辑中的特异性对于其安全高效的利用具有重要意义,因此有必要对可能出现的脱靶效应进行全面准确地评估。有研究表明,针对人源CLTA基因的4个靶位点均存在脱靶现象,脱靶效率最高可达84%[32];针对鼠源Crygc基因的2个靶点存在40%的脱靶率[33]。而在植物中CRISPR/Cas9系统脱靶效率较低,朱健康教授实验室通过对拟南芥全基因组测序和潜在脱靶位点分析未检测到脱靶效应[27],对水稻中多个与靶位点高度同源序列进行检测,仅在一个碱基不匹配处发现脱靶效应[31],杜彦修等[34]对水稻基因两个潜在脱靶位点进行检测并未发现脱靶。本研究选择了PAM序列5′端同源性较高的5个位点作为潜在脱靶位点,经检测这5个位点均未发生脱靶效应,因此设计的sgRNA序列具有较高特异性。有研究表明,sgRNA的特异性主要由靠近PAM序列的10—12 bp碱基所决定,而远离PAM区的8—10 bp碱基对sgRNA的特异性识别影响不大[3, 35]。本试验只是对最有可能出现脱靶效应的5个位点进行了检测,不排除其他位点出现脱靶的可能性,更有效的方法则是对获得的突变体进行全基因组深度测序分析,但其成本较高且费时费力。为此科学家开发了新的基于Cas9核酸酶消化基因组(Digenome- seq)技术和基于整合酶缺陷型慢病毒载体(IDLV)技术来精确检测CRISPR/Cas9系统脱靶效应[36-37]。通过优化sgRNA序列(长度和GC含量等)、应用双Cas9切口酶[38]和应用fCas9复合体[39]等方法可降低CRISPR/Cas9系统脱靶效应。
本试验利用CRISPR/Cas9技术创制了突变体,并对其T1代表型进行了初步观察,发现突变体株高极显著降低,籽粒粒长极显著增大,与OsPIL15蛋白系统进化分析结果是一致的。后续仍需进行多代鉴定,获得稳定纯合的突变体,同时对不同突变类型的株系进行观察,以确定不同突变类型造成的氨基酸改变而产生的表型是否一致。
4 结论
依据CRISPR/Cas9系统原理创制突变体,成功获得5株纯合突变体、6株双等位突变体和4株杂合突变体。对部分突变体后代突变序列进行分析,发现绝大多数突变均能稳定遗传。62株T0代表现为未突变的T1代阳性植株经酶切鉴定均未发现新的突变,表明CRISPR/Cas9系统在T1代转基因阳性植株中未重新发挥基因编辑作用。5个与靶序列同源性较高且错配在4 bp以内的潜在脱靶位点经评估均未出现脱靶效应,表明所选取sgRNA有较高的特异性。对获得的突变体表型进行初步观察发现,与野生型相比T1代突变体株高极显著降低,分蘖数未发生显著变化,籽粒粒长极显著增大。
References
[1] Carroll D, Morton J J, Beumer K J, Segal D J. Design, construction and in vitro testing of zinc finger nucleases., 2006, 1(3): 1329-1341.
[2] Li T, Liu B, Spalding M H, Weeks D P, Yang B. High- efficiency TALEN-based gene editing produces disease-resistant rice., 2012, 30(5): 390-392.
[3] Cong L, Ran F A, Cox D, Lin S, Barretto R, Habib N, Hsu P D, Wu X, Jiang W, Marraffini L A, Zhang F. Multiplex genome engineering using CRISPR/Cas systems., 2013, 339(6121): 819-823.
[4] Mali P, Yang L, Esvelt K M, Aach J, Guell M, Dicarlo J E, Norville J E, Church G M. RNA-guided human genome engineering via Cas9., 2013, 339(6121): 823-826.
[5] Wiedenheft B, Sternberg S H, Doudna J A. RNA-guided genetic silencing systems in bacteria and archaea., 2012, 482(7385): 331-338.
[6] Doudna J A, Charpentier E. The new frontier of genome engineering with CRISPR-Cas9., 2014, 346(6213): 1258096.
[7] Belhaj K, Chaparro-Garcia A, Kamoun S, Patron N J, Nekrasov V. Editing plant genomes with CRISPR/Cas9., 2015, 32: 76-84.
[8] Khanna R, Huq E, Kikis E A, Al-Sady B, Lanzatella C, Quail P H. A novel molecular recognition motif necessary for targeting photoactivated phytochrome signaling to specific basic helix-loop-helix transcription factors., 2004, 16(11): 3033-3044.
[9] Shen H, Zhu L, Castillon A, Majee M, Downie B, Hu q E. Light-induced phosphorylation and degradation of the negative regulator PHYTOCHROME-INTERACTING FACTOR1 fromdepend upon its direct physical interactions with photoactivated phytochromes., 2008, 20(6): 1586-1602.
[10] Zhang Y, Mayba O, Pfeiffer A, Shi H, Tepperman J M, Speed T P, Quail P H. A quartet of PIF bHLH factors provides a transcriptionally centered signaling hub that regulates seedling morphogenesis through differential expression-patterning of shared target genes in., 2013, 9(1): e1003244.
[11] Oh E, Yamaguchi S, Hu J, Yusuke J, Jung B, Paik I, Lee H, Sun T, Kamiya Y, Choi G. PIL5, a phytochrome-interacting bHLH protein, regulates gibberellin responsiveness by binding directly to theandpromoters inseeds., 2007, 19(4): 1192-1208.
[12] Leivar P, Monte E, Al-Sady B, Carle C, Storer A, Alonso J M, Ecker J R, Quail P H. Thephytochrome-interacting factor PIF7, together with PIF3 and PIF4, regulates responses to prolonged red light by modulating phyB levels., 2008, 20(2): 337-352.
[13] Lorrain S, Allen T, Duek P D, Whitelam G C, Fankhauser C. Phytochrome-mediated inhibition of shade avoidance involves degradation of growth-promoting bHLH transcription factors., 2008, 53(2): 312-323.
[14] Shin J, Anwer M U, Davis S J. Phytochrome-interacting factors (PIFs) as bridges between environmental signals and the circadian clock: diurnal regulation of growth and development., 2013, 6(3): 592-595.
[15] Nakamura Y, Kato T, Yamashino T, Murakami M, Mizuno T. Characterization of a set of phytochrome-interacting factor-like bHLH proteins in., 2007, 71(5): 1183-1191.
[16] 梁卫红, 李莉, 彭威风, 刘婧, 杨慧, 谢先芝. 水稻光敏色素相互作用因子基因的表达模式及其在光信号传导中作用的初步分析. 中国水稻科学, 2012, 26(3): 255-260.
Liang W H, Li L, PENG W F, LIU J, YANG H, XIE X Z. Expression patterns of, a phytochrome interacting factor in rice, and preliminary analysis of its roles in light signal transduction., 2012, 26(3): 255-260. (in Chinese)
[17] Todaka D, Nakashima K, Maruyama K, Kidokoro S, Osakabe Y, Ito Y, Matsukura S, Fujita Y, Yoshiwara K, Ohme-Takagi M. Rice phytochrome-interacting factor-like protein OsPIL1 functions as a key regulator of internode elongation and induces a morphological response to drought stress., 2012, 109(39): 15947-15952.
[18] Zhou J, Liu Q, Zhang F, Wang Y, Zhang S, Cheng H, Yan L, Li L, Chen F, Xie X. Overexpression of, a phytochrome‐interacting factor-like protein gene, represses etiolated seedling growth in rice., 2014, 56(4): 373-387.
[19] Heang D, Sassa H. Antagonistic actions of HLH/bHLH proteins are involved in grain length and weight in rice., 2012, 7(2): e31325.
[20] 杜彦修, 季新, 彭廷, 孙红正, 张静, 李俊周, 赵全志. 水稻灌浆期籽粒中光信号相关基因鉴定与表达分析. 分子植物育种, 2016, 14(7): 1637-1647.
DU Y X, JI X, PENG T, SUN H Z, ZHANG J, LI J Z, ZHAO Q Z. Identification and expressing analysis of light signal genes involved in endosperm development during rice grain filling., 2016, 14(7): 1637-1647. (in Chinese)
[21] Xing H, Dong L, Wang Z, Zhang H, Han C, Liu B, Wang X, Chen Q. A CRISPR/Cas9 toolkit for multiplex genome editing in plants., 2014, 14(1): 327.
[22] Heang D, Sassa H. Overexpression of a basic helix-loop-helix gene(APG) decreases grain length of rice., 2012, 29(1): 65-69.
[23] Xu R, Li H, Qin R, Li J, Qiu C, Yang Y, Ma H, Li L, Wei P, Yang J. Generation of inheritable and “transgene clean” targeted genome-modified rice in later generations using the CRISPR/Cas9 system., 2015, 5: 11491.
[24] Endo M, Mikami M, Toki S. Multigene knockout utilizing off-target mutations of the CRISPR/Cas9 system in rice., 2015, 56(1): 41-47.
[25] Ma X, Chen L, Zhu Q, Chen Y, Liu Y. Rapid decoding of sequence-specific nuclease-induced heterozygous and biallelic mutations by direct sequencing of PCR products., 2015, 8(8): 1285-1287.
[26] Liu W, Xie X, Ma X, Li J, Chen J, Liu Y. DSDecode: A web- based tool for decoding of sequencing chromatograms for genotyping of targeted mutations., 2015, 8(9): 1431-1433.
[27] Feng Z, Mao Y, Xu N, Zhang B, Wei P, Yang D, Wang Z, Zhang Z, Zheng R, Yang L, Zeng L, Liu X, Zhu J. Multigeneration analysis reveals the inheritance, specificity, and patterns of CRISPR/Cas-induced gene modifications in., 2014, 111(12): 4632-4637.
[28] Ishizaki T. CRISPR/Cas9 in rice can induce new mutations in later generations, leading to chimerism and unpredicted segregation of the targeted mutation., 2016, 36(12): 165.
[29] Mao Y, Zhang Z, Feng Z, Wei P, Zhang H, Botella J R, Zhu J. Development of germ-line-specific CRISPR-Cas9 systems to improve the production of heritable gene modifications in., 2016, 14(2): 519-532.
[30] Ma X, Zhang Q, Zhu Q, Liu W, Chen Y, Qiu R, Wang B, Yang Z, Li H, Lin Y, Xie Y, Shen R, Chen S, Wang Z, Chen Y, Guo J, Chen L, Zhao X, Dong Z, Liu Y. A robust CRISPR/Cas9 system for convenient, high-efficiency multiplex genome editing in monocot and dicot plants., 2015, 8(8): 1274-1284.
[31] Zhang H, Zhang J, Wei P, Zhang B, Gou F, Feng Z, Mao Y, Yang L, Zhang H, Xu N, Zhu J. The CRISPR/Cas9 system produces specific and homozygous targeted gene editing in rice in one generation., 2014, 12(6): 797-807.
[32] Pattanayak V, Lin S, Guilinger J P, Ma E, Doudna J A, Liu D R. High-throughput profiling of off-target DNA cleavage reveals RNA-programmed Cas9 nuclease specificity., 2013, 31(9): 839-843.
[33] Wu Y, Liang D, Wang Y, Bai M, Tang W, Bao S, Yan Z, Li D, Li J. Correction of a genetic disease in mouse via use of CRISPR-Cas9., 2013, 13(6): 659-662.
[34] 杜彦修, 季新, 陈会杰, 彭廷, 张静, 李俊周, 孙红正, 赵全志. 基于CRISPR/Cas9系统的基因编辑及其脱靶效应分析. 中国水稻科学, 2016, 30(6): 577-586.
DU Y X, JI X, CHEN H J, PENG T, ZHANG J, LI J Z, SUN H Z, ZHAO Q Z. CRISPR/Cas9 system-based editing ofgene and its off-target effect analysis., 2016, 30(6): 577-586. (in Chinese)
[35] Hsu P D, Scott D A, Weinstein J A, Ran F A, Konermann S, Agarwala V, Li Y, Fine E J, Wu X, Shalem O, Cradick T J, Marraffini L A, Bao G, Zhang F. DNA targeting specificity of RNA-guided Cas9 nucleases., 2013, 31(9): 827-832.
[36] Kim D, Bae S, Park J, Kim E, Kim S, Yu H R, Hwang J, Kim J, Kim J. Digenome-seq: genome-wide profiling of CRISPR-Cas9 off-target effects in human cells., 2015, 12(3): 237-243.
[37] Wang X, Wang Y, Wu X, Wang J, Wang Y, Qiu Z, Chang T, Huang H, Lin R, Yee J. Unbiased detection of off-target cleavage by CRISPR-Cas9 and TALENs using integrase-defective lentiviral vectors., 2015, 33(2): 175-178.
[38] Ran F A, Hsu P D, Lin C, Gootenberg J S, Konermann S, Trevino A E, Scott D A, Inoue A, Matoba S, Zhang Y, Zhang F. Double nicking by RNA-guided CRISPR Cas9 for enhanced genome editing specificity., 2013, 154(6): 1380-1389.
[39] Guilinger J P, Thompson D B, Liu D R. Fusion of catalytically inactive Cas9 to FokI nuclease improves the specificity of genome modification., 2014, 32(6): 577-582.
(责任编辑 李莉)

M:DNA Molecular-Weight Marker;B:Basta探针;1、24、2、23、3、14、19、21:突变单株
M: DNA Molecular-Weight Marker; B: Basta probe; 1, 24, 2, 23, 3, 14, 19, 21: mutant plants
附图1 Southern blot分析突变单株的T-DNA插入
Supplemental Fig. 1 Analysis of T -DNA region by southern blot
CRISPR/Cas9 System-Based Editing of Phytochrome-Interacting Factor
JI Xin, LI Fei, YAN Yun, SUN HongZheng, ZHANG Jing, LI JunZhou, PENG Ting, DU YanXiu, ZHAO QuanZhi
(Agronomy College of Henan Agricultural University/Collaborative Innovation Center of Henan Grain Crops/Henan Key Laboratory of Rice Biology, Zhengzhou 450002)
【Objective】 As an important environmental signal, light can regulate gene expression, affect the activity of enzymes and plant morphogenesis. Phytochrome interacting factors play an important role in the signal transduction of light. Therefore, constructing the expression vector of CRISPR/Cas9 containing rice phytochrome-interacting factorsand creating themutants have important significance to exploit functional genes and enrich light signal regulation of the molecular mechanism of rice. 【Method】 According to the principle of CRISPR/Cas9 technology, the sgRNA was designed. To exclude non-specific target sites, the sgRNA was analyzed by sequence alignment in the rice genome database. At the same time, the target sequence contained the common restriction site to identify mutants. The oligonucleotides of sgRNA were chemically synthesized and inserted into linearized plasmid pBUN411 to construct the expression vector. Transgenic rice plants harboring sgRNA:Cas9 were obtained by-mediated stable transformation and the positive transgenic plants were screened by herbicide resistance. The PCR products of T0transgenic plants were digested by restriction enzyme to judge whether they were mutants. Then, the mutated genotypes of these mutants were analyzed by DNA sequencing. After searching rice genome using sgRNA sequence, five highly identical sites with less 4 mismatching bases were selected to assess off-target efficiency and specificity of sgRNA.【Result】The recombinant vector succeeded in oriented editing of. The restriction enzyme analysis results indicated that 15 mutants from the 25 randomly selected T0transgenic lines were obtained. They included 5 homozygous mutants, 6 biallelic mutants and 4 heterozygous mutants, and a total of 10 different genotypes and 11 mutant lines. The mutant types were mainly insertions or deletions of single base, besides, two mutant types of large deletions with 56 and 66 bp were obtained. Analysis of T1transgenic plants from some T0mutants indicated that the genotypes in T0mutants could descend stably into the next generation. The progeny of homozygous mutants in T0were homozygous mutants. However, only one wild type with no mutation was detected in a homozygous mutant progenies of line 14. Two homozygous mutations and one biallelic mutations were obtained in the progeny of biallelic T0lines. Three mutation types including homozygous mutants, heterozygous mutants and wild type with no mutation were detected in the progenies of heterozygous T0mutants. Restriction enzyme analysis was used to detect the engineered target site of T1positive transgenic plants which had no mutation in T0. No mutation in 62 T1plants was detected. The results showed that CRISPR/Cas9 system played no role in gene editing in T1positive transgenic plants. After searching the rice genome using the target sequence with PAM, five highly identical sites were found. However, any mutations at these sites in T0and T1generations were not observed by restriction enzyme, which indicated the sgRNA was highly specific. Three groups of different genotypes were selected representativemutants in T1generation to observe phenotypes, the investigation result showed that the growth stage and tiller number were not obviously changed, the plant height decreased significantly (<0.01), and the grain length was significantly increased compared with the wild type, up 5.69%. 【Conclusion】CRISPR/Cas9 system succeeded in oriented editing of, themutants with 10 different genotypes were obtained and observed that the plant height decreased significantly and the grain length increased significantly compared with those of wild type.
rice; CRISPR/Cas9; gene editing;; off-target efficiency
2017-01-22;接受日期:2017-04-05
河南省重大科技专项(141100110600)、河南省高等学校重点科研项目(16A210026)
季新,E-mail:jixin1@126.com。李飞,E-mail:LFight123@163.com。季新和李飞为同等贡献作者。通信作者杜彦修,E-mail:dumatt@163.com。通信作者赵全志,E-mail:qzzhaoh@126.com

