猪睾丸组织定量PCR分析中内参基因的选择
彭馥芝,冉茂良,翁波,李智,董莲花,陈斌
猪睾丸组织定量PCR分析中内参基因的选择
彭馥芝,冉茂良,翁波,李智,董莲花,陈斌
(湖南农业大学动物科学技术学院/畜禽遗传改良湖南省重点实验室,长沙 410128)
【目的】采用实时荧光定量PCR技术(qRT-PCR)对基因表达进行分析时,选择适当的内参基因是获得准确分析结果的关键。在猪睾丸分子生物学研究中,表达稳定的蛋白编码内参基因和microRNA(miRNA)内参基因均有哪些目前尚不清楚。本研究旨在筛选出合适的内参基因,为准确定量不同时期猪睾丸组织中目的基因的表达提供可靠依据。【方法】选用8个不同发育时期(E 90、D 1、D 30、D 60、D 90、D 120、D 150、DM)的猪睾丸组织为材料,采用Trizol裂解法提取各样品的总RNA,利用NanDrop ND-2000分光光度计检测总RNA的浓度和纯度。设计并合成已报道的猪其他组织中表达相对稳定的5个编码内参基因(、、、和)以及5个miRNA内参基因(U6、ssc-miR-17-5p、ssc-miR-26a、ssc-miR-27a和ssc-miR-103)的特异性引物序列,逆转录得到cDNA模板后,按1:10梯度稀释为7个浓度梯度进行qRT-PCR反应以构建标准曲线。并对10个候选内参基因在各时期睾丸组织中的表达情况进行实时荧光定量全面检测,采用GeNorm法对定量结果进行综合分析,最后根据内参基因稳定性值(M值)的大小筛选出最稳定的参考基因;M值越小,表明内参基因的稳定性越好,反之则越差。【结果】对、、、、、U6、ssc-miR-17-5p、ssc-miR-26a、ssc-miR-27a和ssc-miR-103 10个候选内参基因的熔解曲线进行分析发现,均无非特异扩增及引物二聚体,说明各内参基因特异性良好,qRT-PCR反应的专一性高;以Ct值为纵坐标,相对拷贝数的对数为横坐标进行标准曲线分析可知,各内参基因在系列稀释的浓度梯度内具有良好的线性关系;通过对10个候选内参基因在不同时期猪睾丸组织中的表达稳定性分析发现,蛋白编码基因中,最稳定的为,最不稳定的是;miRNA候选内参中,最稳定的为U6,最不稳定的是ssc-miR-26a。【结论】成功筛选出猪睾丸组织基因表达分析中稳定表达的内参基因和U6,可作为猪睾丸组织基因表达研究中最佳内参基因候选者。
猪睾丸组织;内参基因;qRT-PCR;miRNA
0 引言
【研究意义】实时荧光定量PCR(real-time fluorescence quantitative PCR, qRT-PCR)是一种通过始点定量和荧光检测系统实时监测累积荧光强度进行定量基因检测的灵敏度高、特异性强、线性关系好、操作简单的定量PCR技术,已成为分子生物学研究领域中分析基因表达的重要工具[1]。内参基因是一种维持细胞最低限度功能不可缺少的基因,理想的内参基因在不同类型细胞和组织中的表达应无显著差异,且不受试验和临床条件的影响。但由于内参基因的表达具有物种及组织特异性,所以其表达的稳定性是相对的。而在使用qRT-PCR 技术检测目的基因表达水平时,为减少检测样本在RNA产量、质量和反转录效率上可能存在的差异,通常需要引入一个表达较为稳定的管家基因作为内参基因进行校正和标准化[2-3]。【前人研究进展】目前,各个研究领域关于内参基因的报道很多,例如,在家蚕不同组织中、的表达相对较稳定[4];陈瑞等[3]以3个阶段的新西兰白兔的3种不同组织作为研究对象,证实新西兰白兔不同的发育时期和组织中,最稳定的内参基因不同;而、和为山羊不同组织和不同时期中最合适的内参基因[5],为波尔山羊10个不同组织中表达水平最稳定的内参基因[6],校正绵羊不同组织qRT-PCR结果时,也需要选择各组织中最适用的内参基因[7];LISOWSKI 等[8]在牛的不同组织中筛选内参基因的结果也表明,不同组织中各内参基因的表达稳定性也有所不同。这些结果对不同试验材料及其在不同时期基因表达研究中内参基因的选择提供了有效参考。而作为生物基因调控网络中重要成员的miRNAs,尽管现在对其的研究取得了很大进展[9-10], 但是对于均一化miRNA表达的内参基因的系统研究还比较匮乏,主要在人类疾病方面的研究比较多。SCHAEFER等[11]对前列腺癌组织样本进行了相关试验,建议在前列腺癌组织中使用miR-103b作为内参基因。WOTSCHOFSKY等[12]的研究结果指出,作为透明细胞肾癌研究的miRNA内参基因应用一种miRNA内参来进行标准化,那么miRNA-28是最佳选择。人类各种组织miRNA内参基因的选择结果显示,miR-191是多种正常组织中最稳定的miRNA,而在肿瘤组织中表达最稳定的是miR-103[13]。【本研究切入点】雄性睾丸是调控雄性生殖发育和精子成熟的重要器官,探索睾丸组织内各基因的表达水平对促进雄性生育及预防生殖方面的疾病具有重要的意义。目前,内参基因的筛选在仔猪心、肝脏、脾脏等不同组织[14]、梅山-大白猪肌肉组织[15]、猪血管内皮细胞[16]、猪骨骼肌组[17]等猪的相关组织中已有报道,但对其在猪睾丸组织中表达的内参基因的筛选还未见系统研究。因此,本研究以不同发育阶段猪睾丸组织样品为材料,综合比较猪其他组织中各内参基因稳定性后,选择、、、和五个蛋白编码基因以及U6、ssc-miR-17-5p、ssc-miR-26a、ssc-miR-27a和ssc-miR-103等5个miRNA为候选内参基因,采用qRT-PCR技术,结合GeNorm分析软件[18],对10个候选基因在不同时期猪睾丸组织中的表达稳定性进行评价。【拟解决的关键问题】获得适用于猪睾丸组织基因表达研究的最佳内参基因,以期为后续相关研究中内参基因的选择提供依据。
1 材料与方法
试验于2016年4—6月在湖南农业大学动物科学技术学院畜禽遗传改良湖南省重点实验室进行。
1.1 材料
试验所用的沙子岭猪睾丸组织样品采于湖南省湘潭市沙子岭猪保种场,通过屠宰的方法采集猪胚胎期睾丸组织样品,采用阉割的方法采集出生后公猪的睾丸组织样品。采集90胚龄(E 90)、1日龄(D 1)、30日龄(D 30)、60日龄(D 60)、90日龄(D 90)、120日龄(D 120)、150日龄(D 150)及180日龄(DM)8个发育阶段的正常健康公猪睾丸组织,剪成小块,放入RNase-free的EP管中,立即置于液氮中速冻,带回实验室,于-80℃超低温冰箱中保存备用。每个阶段采集3头公猪睾丸组织样品,共计24个样品。
1.2 总RNA的提取和cDNA的合成
采用Trizol裂解法提取各样品的总RNA,利用NanDrop ND-2000分光光度计检测总RNA的浓度和纯度。OD260/280和OD260/230的值用来确定所有RNA样品的纯度。只有OD260/280比值介于1.8—2.0之间,OD260/230在2.0以上的RNA样品才能用于后续研究。按照RR047A型PrimeScript™ RT Reagent Kit反转录试剂盒(TaKaRa,日本)说明书反转录合成cDNA,于-80℃超低温冰箱内保存备用。
1.3 引物设计及标准曲线的构建
本研究选取、、、和等5个蛋白编码基因以及U6、ssc-miR-17-5p、ssc-miR-26a、ssc-miR-27a和ssc-miR-103等5个miRNA内参基因作为候选基因。利用miRBase数据库搜索5个miRNA内参基因的成熟序列,查阅文献资料确定5个蛋白编码基因的序列,设计10个候选基因的特异性引物序列(表1),并由生工生物工程有限公司(上海,中国)合成。将cDNA模板按1﹕10梯度稀释为7个浓度梯度后进行qRT-PCR反应以构建标准曲线。

表1 候选基因的引物序列
1.4 实时荧光定量PCR
qRT-PCR反应体系为25 μL:荧光染料SYBR®Premix Ex TaqⅡ12.5 μL,dH2O(灭菌蒸馏水)8.5 μL,PCR Forward Primer 1.0 μL(10 μmol·L-1)、PCR Reverse Primer(10 μmol·L-1)1.0 μL,cDNA模板2.0 μL。反应在CFX Connect(Bio Rad,美国)实时荧光定量PCR仪中进行。实时荧光定量PCR反应条件为:95℃预变性30 s;95℃5 s,60℃30 s扩增,40个循环;55℃—95℃熔解30 s,监测熔解曲线的过程中,每0.5℃进行1次荧光监测。每个阶段睾丸组织均有3个生物学重复,每个样品进行3次qRT-PCR,同时设置阴性对照和空白对照。
1.5 表达数据分析
GeNorm程序的原理是根据基因相对表达水平的几何平均值计算候选内参基因的稳定性值(M值),根据M值的大小筛选出最稳定的参考基因,M值越小,表明内参基因的稳定性越好,反之则越差[19]。数据读取由CFX Connect(Bio Rad,美国)实时荧光定量PCR仪自动完成,通过CFX Manager软件中的GeNorm程序获得内参基因在各时期表达稳定度的平均M值,从而筛选出最稳定的内参基因。
2 结果
2.1 内参基因引物的特异性
对、、、、、U6、ssc-miR-17-5p、ssc-miR-26a、ssc-miR-27a和ssc-miR- 103等10个候选内参基因的熔解曲线进行分析发现,均无非特异扩增及引物二聚体,说明各内参基因特异性良好,qRT-PCR反应的专一性高, 结果准确可靠,满足试验要求(图1,2)。
2.2 标准曲线的构建
以Ct值为纵坐标,相对拷贝数的对数为横坐标进行标准曲线分析,由表2可知,各内参基因在系列稀释的浓度梯度内具有良好的线性关系,满足qRT-PCR的扩增条件。
2.3 内参基因在不同时期表达的稳定性
通过对10个内参基因在不同时期猪睾丸组织中的表达稳定性分析发现,蛋白编码基因中,最稳定的为,最不稳定的是(图3);miRNA中,最稳定的为U6,最不稳定的是ssc-miR-26a(图4)。
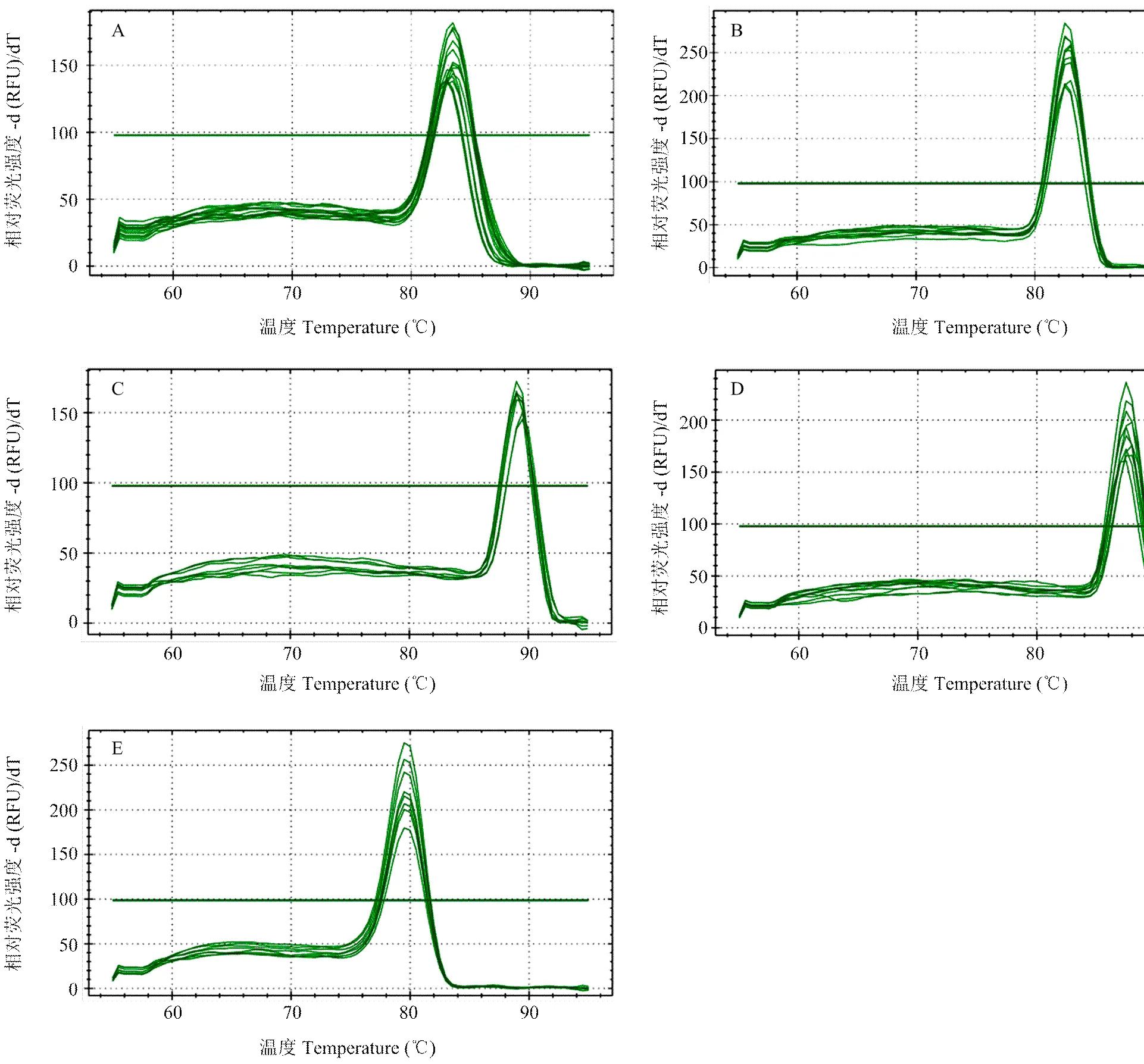
(A) β-actin; (B) TBP; (C) SDHA; (D) GAPDH; (E) B2M
(A) ssc-miR-27a; (B) ssc-miR-17-5p; (C) U6; (D) ssc-miR-26a; (E) ssc-miR-103
图2 猪睾丸组织中5个候选miRNA内参基因Real-time PCR熔解曲线
Fig. 2 Real-time PCR melting curves of 5 candidate miRNA reference genes of porcine testis tissues
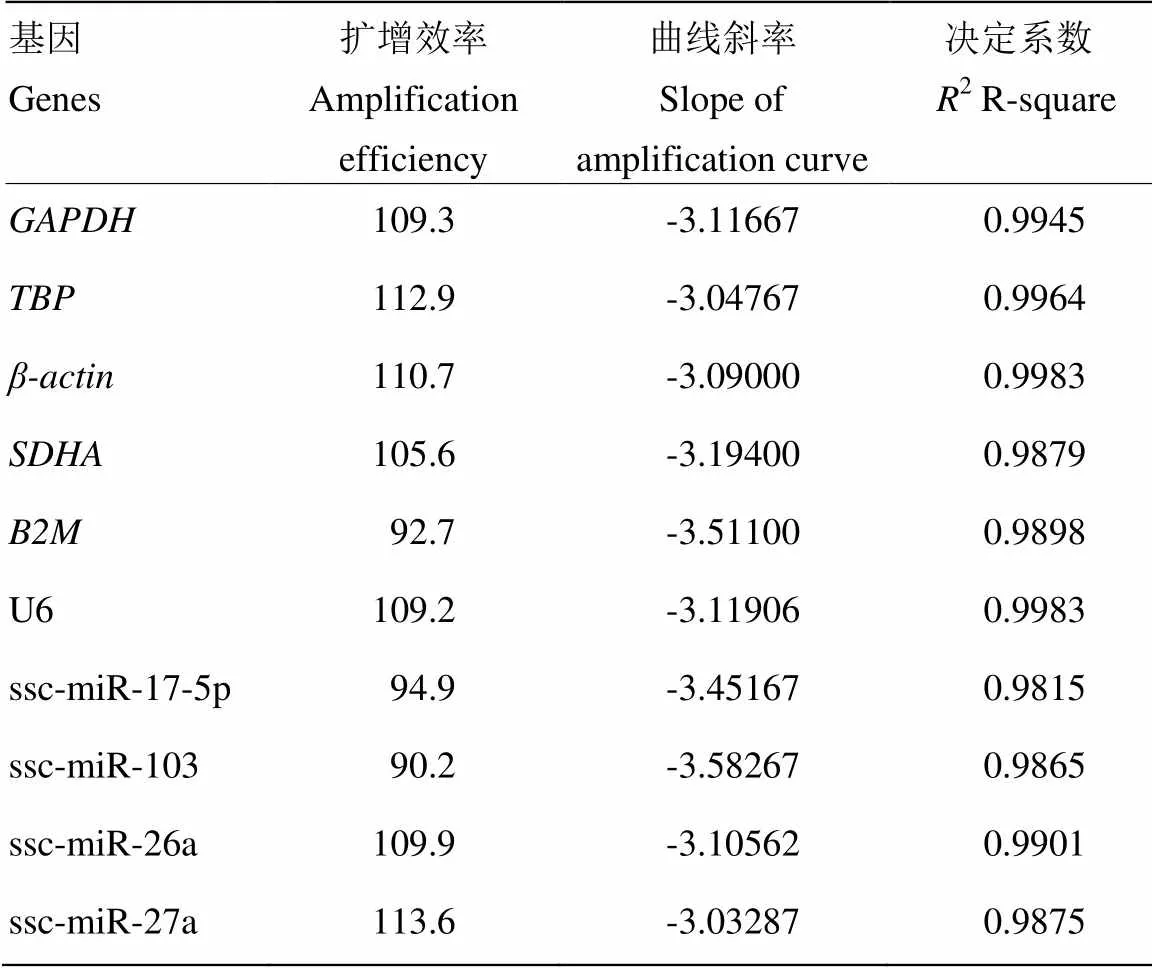
表2 猪睾丸组织10个候选内参基因的qRT-PCR 标准曲线
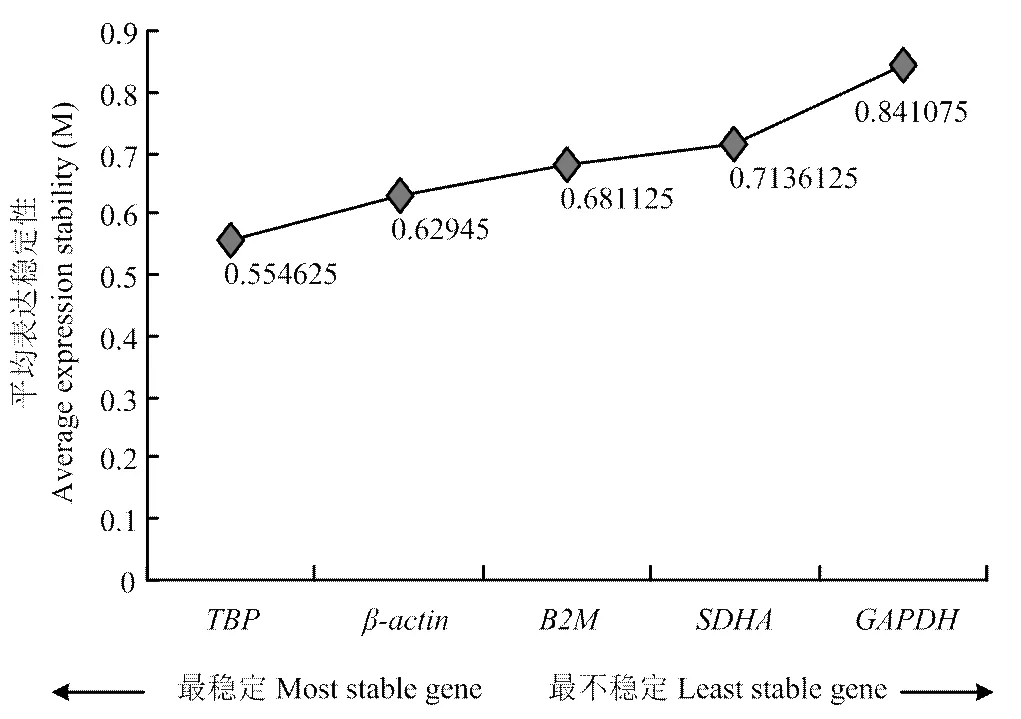
图3 猪睾丸组织中内参基因的平均表达稳定度
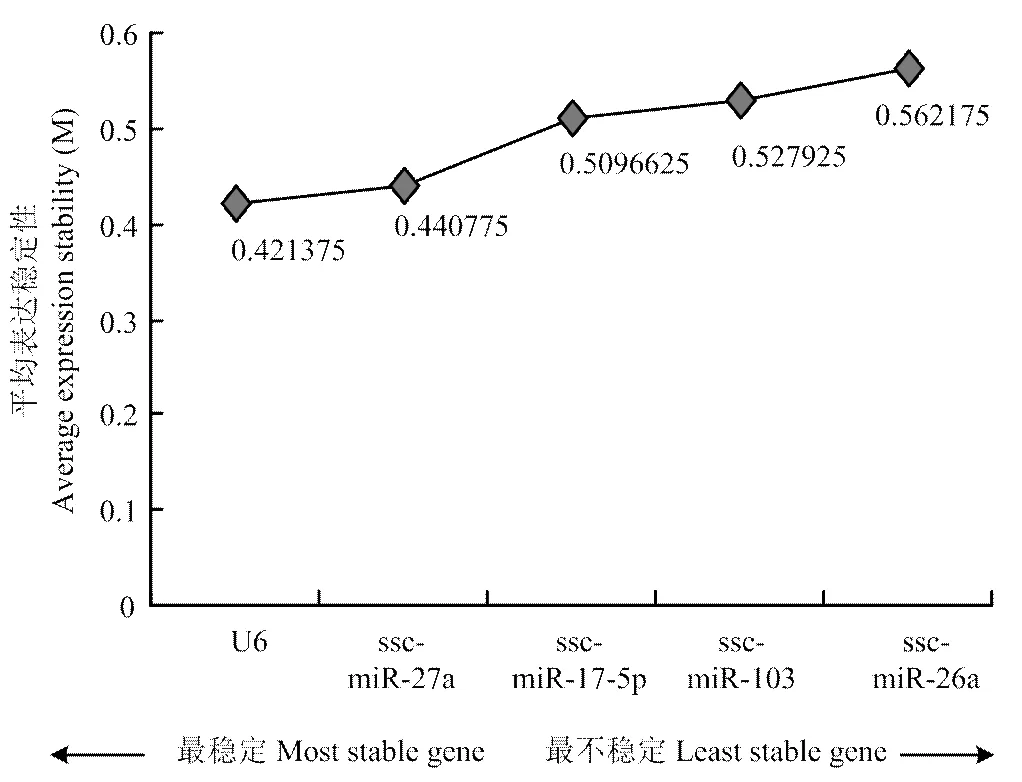
图4 猪睾丸组织中miRNA内参基因的平均表达稳定度
3 讨论
内参基因也叫看家基因,指在检测基因表达水平的试验中,通常用来作为参照物,对试验误差进行校正的基因。理想的内参基因要求在各种试验条件及各组织、细胞和发育时期中的表达均相对恒定[20]。采用稳定的内参基因能对定量试验中qRT-PCR的数据进行标准化,但尚未发现任何一个内参基因能在所有试验条件下均稳定表达,而是在不同的组织或同一组织的不同发育时期均存在差异表达[21-23]。因此在对特定基因表达研究中,筛选出最稳定的基因作为内参基因对数据进行校正,才能保证结果的准确性[24]。本试验首次对猪睾丸组织基因表达分析中适宜的内参基因进行研究。结果表明,猪睾丸组织8个发育时期的10个候选基因中蛋白编码基因和miRNA表达最稳定的内参基因分别是和U6,稳定性最差的分别是和ssc-miR-26a。
是转录起始复合物TFIID 的核心组分,与相关启动子结合后,能识别并特异性结合TATA元件,进而指导RNA 聚合酶和其他转录因子有序装配,形成稳定的转录起始复合物,并直接影响基因的转录水平[25-26]。本研究结果揭示在猪睾丸组织中的表达稳定性与之前在猪各组织中的研究结果一致。例如,ERKENS等[27]研究发现和可作为最合适、表达最稳定的内参基因用于猪的背脂和背肌的基因表达研究。在NYGARD等的研究中,分析了9个内参基因在猪的不同组织中的表达稳定性,结果发现、、和这4个基因的表达最稳定,而表达最不稳定[28]。对猪外周血基因表达分析中适宜的内参基因进行研究,结果表明,6个候选内参基因在全血中稳定表达顺序中稳定性最好的内参基因的是和,最差的是[29]。
U6是剪接体的组成元件之一,参与mRNA前体的加工过程[30]。U6在真核生物各种组织和细胞中广泛表达,而且表达水平恒定,因此,常被选作内参基因用于miRNA的定量表达分析[31-32]。本研究结果也表明,在猪睾丸组织miRNA表达水平的研究中,U6为最稳定的内参基因。但U6作为理想内参基因其稳定性也一直受到质疑。例如,在猪肝脏、脾脏、胰脏、心脏、胃、肺、小肠、大脑、小脑和腿肌的正常组织中筛选出miR-103实时定量PCR检测分析的合适内参时,采用GeNorm 算法和扩增效率试验对miR-196、U6和总RNA[33](以总RNA为标准归一化各候选基因的表达水平)3个候选内参进行了评价,结果表明,miR-196的表达稳定性和扩增效率均优于U6;以miR-196为内参的miR-103 相对定量结果具有更高的准确性[34]。李春枚[35]选择13个在猪的miRNA定量中常被用作内参的基因(5S,Met-rRNA,ssc-miR-16,ssc-miR-17,ssc-miR-23a,ssc-miR-24,ssc-miR-27a,ssc-miR-103,ssc-miR-106a,ssc-miR-107,ssc-miR-186,ssc-miR-221和U6),通过EvaGreenq-PCR法分别比较其在47个不同组织,10个脂肪组织和4个肌肉组织中的表达差异,然后利用GeNorm软件评估这些基因在3个不同样本分类中的表达稳定性。结果显示,通过内参基因的表达稳定性分析,在全部47个组织中,有7个内参基因的表达显示出可靠的稳定性,其中ssc-miR-17和ssc-miR-103是最稳定的两个内参基因。而在3个不同样本分类中,U6的表达都是最不稳定的。由此可见,不同组织中最适用的内参基因并不完全一致,因此有必要针对猪不同组织进行内参基因的优选。
4 结论
本研究成功筛选了猪睾丸组织基因表达分析中稳定表达的内参基因(和U6),为准确定量不同时期猪睾丸组织目的基因的表达提供了可靠的依据。但是本试验结果并不是绝对的,在其他试验条件下,还需要通过系统的试验分析重新确定合适的编码基因或miRNA作为内参基因,盲目地选择内参基因可能会导致试验结果与预期结果出现偏差,甚至得到错误的结论。
References
[1] 蒋春燕, 王泰健, 王琴, 范学政. 实时荧光定量PCR技术. 动物医学进展, 2005, 26(12) : 97-101.
JIANG C Y, WANG T J, WANG Q, FAN X Z. Real-time fluorescence quantitative PCR., 2005, 26(12): 97-101. (in Chinese)
[2] 吴文凯, 刘成前, 周志刚, 卢山. 用于莱茵衣藻荧光定量PCR分析的内参基因选择. 植物生理学通讯, 2009(7): 667-672.
WU W K, LIU C Q, ZHOU Z G, LU S. The selection of reference genes inP. A. Dangeard by real-time quantitative PCR., 2009(7): 667-672. (in Chinese)
[3] 陈瑞, 杨晓农, 曾婉秋, 文娟. 家兔不同发育阶段和组织中内参基因的稳定性分析. 畜牧兽医学报, 2016, 47(3) : 477-483.
CHEN R, YANG X L, ZENG W Q, WEN J. Expression stability analysis of reference genes in different development periods and tissues in., 2016, 47(3): 477-483. (in Chinese)
[4] 彭然.家蚕常用内参基因的稳定性分析及两种实时荧光定量PCR方法比较[D]. 苏州: 苏州大学, 2012.
PENG R. Analysis of reference gene expression for real time PCR based on relative quantitative and dual spike-in strategy in the silkworm[D]. Suzhou: Suzhou University, 2012. (in Chinese)
[5] 陈利, 赵薇, 占思远, 李丹, 李利, 仲涛, 王林杰, 张红平. 山羊不同组织及不同发育时期骨骼肌内参基因的表达稳定性分析. 畜牧兽医学报, 2014, 45(8) : 1228-1236.
CHEN L, ZHAO W, ZHAN S Y, LI D, ZHONG T, WANG L J, ZHANG H P. The expression stability analysis of reference genes in different tissues and skeletal muscle of different development periods in goat., 2014, 45(8): 1228-1236. (in Chinese)
[6] 张晓东. 山羊卵巢microRNA的鉴定及生物学功能分析[D]. 合肥: 安徽农业大学, 2013.
ZHANG X D. Identification and biological role analysis of microRNAs in goat ovaries[D]. Hefei: Anhui Agricultural University, 2013. (in Chinese)
[7] GARCIA-CRESPO D, JUSTE R A, HURTADO A. Selection of ovine housekeeping genes for normalisation by real-time RT-PCR; analysis ofgene expression and genetic susceptibility to scrapie., 2005, 1:3.
[8] LISOWSKI P, PIERZCHALA M, GOSCIK J, PAREEK C S, ZWIERZCHOWSKI L. Evaluation of reference genes for studies of gene expression in the bovine liver, kidney, pituitary, and thyroid., 2008, 49(4) : 367-372.
[9] 冉茂良, 陈斌, 尹杰, 杨岸奇, 蒋明.睾丸发育和精子生成相关miRNA研究进展. 遗传, 2014, 36(7) : 646-654.
RAN M L, CHEN B, YIN J, YANG A Q, JIANG M. Advances in miRNA research related to testis development and spermatogenesis., 2014, 36(7) : 646-654. (in Chinese)
[10] 蔡婷, 刘志红, 王志新, 赵濛, 俎红丽, 李金泉. miRNA在调控皮肤和毛囊发育中的作用. 遗传, 2013, 35(9) : 1087-1094.
CAI T, LIU Z H, WANG Z X, ZHAO M, JU H L, LI J Q. miRNA in regulation of skin and hair follicle development., 2013, 35(9) : 1087-1094. (in Chinese)
[11] SCHAEFER A, JUNG M, MILLER K, LEIN M, KRISTIANSEN G, ERBERSDOBLER A, JUNG K. Suitable reference genes for relative quantification of miRNA expression in prostate cancer., 2010, 42(11) : 749-758.
[12] WOTSCHOFSKY Z, MEYER H A, JUNG M, FENDLER A, WAGNER I, STEPHAN C, BUSCH J, ERBERSDOBLER A, DISCH A C, MOLLENKOPF H J, JUNG K. Reference genes for the relative quantification of microRNAs in renal cell carcinomas and their metastases., 2011, 417(2): 233-241.
[13] 陈旭. miRNA定量检测中内参基因的选择. 国际检验医学杂志, 2012, 33(11): 1338-1340.
CHEN X. The selection of reference genes for miRNA quantitative detection., 2012, 33(11): 1338-1340. (in Chinese)
[14] 台玉磊, 韩立强, 杨国庆, 王艳玲, 常磊, 臧猛, 武宇晓, 王静, 张志强, 杨国宇. 仔猪组织基因表达中实时定量PCR内参基因的选择. 农业生物技术学报, 2010, 18(4): 732-736.
TAI Y L, HAN L Q, YANG G Q, WANG Y L, CHANG L, ZANG M, WU X Y, WANG J, ZHANG Z Q, YANG G Y. Selection of the reference genes of real-time quantitative PCR in the gene expression of piglet tissues., 2010, 18(4): 732-736. (in Chinese)
[15] 冯小婷. 梅山-大白猪肌肉组织差异表达基因的筛选、鉴定及功能研究[D]. 武汉: 华中农业大学, 2011.
FENG X T. Screening, identification and function analysis of genes differentially expressed in porcine skeletal muscle between Meishan and Yorkshire pigs[D]. Wuhan: Huazhong Agricultural University, 2011. (in Chinese)
[16] 朱鑫, 吴巧, 冯晓辉, 汤承, 岳华. H1N1猪流感病毒感染猪血管内皮细胞后内参基因的筛选. 西南民族大学学报:自然科学版, 2014, 40(4): 489-494.
ZHU X, WU Q, FENG X H, TANG C, YUE H. Selection of reference genes in porcine umbilicus vein endothelial cells infected with H1N1 swine influenza virus., 2014, 40(4): 489-494. (in Chinese)
[17] 张晶. 骨骼肌发育中miR-148a功能与qPCR内参基因的研究[D]. 武汉: 华中农业大学, 2012.
ZHANG J. Study on function of miR-148a and the reference genes for qPCR in skeletal muscle development [D]. Wuhan: Huazhong Agricultural University, 2012. (in Chinese)
[18] VANDSOMPELE J, PRETER K D, PATTYN F, POPPE B, ROY N V,
PAEPE A D, SPELEMAN F. Accurate normalization of real-time quantitative RT-PCR data by geometric averaging of multiple internal control genes., 2002, 3(7) : 1-12.
[19] AIROLDI M, AMADORI D, BARNI S, CINIERI S, PLACIDO S D, LEO A D, GENNARI A, IACOBELLI S, IONTA M T, LORUSSO V, LOTRIONTE M, MARCHETTI P, MATTIOLI R, MINOTTI G, PAONZATO P, ROSTI G, TONDINI C A, VERONESI A. Clinical activity and cardiac tolerability of non-pegylated liposomal doxorubicin in breast cancer: a synthetic review., 2012, 97(6): 690-692.
[20] SEHLOTTER Y M, VEENHOF E Z, BRINKHOF B, RUTTEN V P M G, SPEE B, WILLEMSE T, PENNING L C. A GeNorm algorithm-based selection of reference genes for quantitative real-time PCR in skin biopsies of healthy dogs and dogs with atopic dermatitis., 2009, 129(l/2): 115-118.
[21] JAIN M, NIJHAWAN A, TYAGI A K, KHURANA J P. Validation of housekeeping genes as internal control for studying gene expression in rice by quantitative real-time PCR., 2006, 345: 646-651.
[22] FACCI M R, AURAY G, MEURENS F, BUCHANAN R, KESSEL J V, GERDTS V. Stability of expression of reference genes in porcine peripheral blood mononuclear and dendritic cells., 2011, 141(1/2): 11-15.
[23] VANDESOMPELE J, PRETER K D, PATTYN F, POPPE B, ROY N V, PAEPE A D, SPELEMAN F. Accurate normalization of real-time quantitative RT-PCR data by geometric averaging of multiple internal control genes., 2002, 3(7): research0034.1-0034.11.
[24] 胡金川. 如何利用geNorm软件筛选基因表达测定的内参基因. 中华检验医学杂志, 2008, 31(8) : 918.
HU J C. How to screen the gene expression of reference genes determined by geNorm software., 2008, 31(8): 918. (in Chinese)
[25] AKHTAR W, VEENSTRA G C. TBP-related factors: a paradigm of diversity in transcription initiation., 2011(1): 23.
[26] THOMAS M C, CHIANG C M. The general transcription machinery and general cofactors., 2006, 41: 105-178.
[27] ERKENS T, POUCKE M V, VANDESOMPELE J, GOOSSENS K, ZEVEREN A V, PEELMAN L J. Development of a new set of reference genes for normalization of real-time RT-PCR data of porcine backfat and longissimus dorsi muscle, and evaluation with PPARGC1A., 2006, 6: 41.
[28] NYGARD A B, JORGENSEN C B, CIRERA S, FREDHOLM M. Selection of reference genes for gene expression studies in pig tissues using SYBR green qPCR., 2007, 8(1): 67.
[29] 王继英, 王彦平, 郭建凤, 王怀中, 林松, 张印, 武英. 仔猪外周血中内参基因的筛选及细胞因子和受体的表达水平. 中国农业科学, 2015, 48(7): 1437-1444.
WANG J Y, WANG Y P, GUO J F, WANG H Z, LIN S, ZHANG Y, WU Y. Selection of reference genes and determination of cytokines and receptor mRNA expression in peripheral blood of piglets., 2015, 48(7): 1437-1444. (in Chinese)
[30] BESSONOV S, ANOKHINA M, WILL C L, URLAUB H, LUHRMANN R. Isolation of an active step I spliceosome and composition of its RNP core., 2008, 452(7189): 846-850.
[31] 张晓军, 李传民, 赵伟, 于海滨, 赵志辉, 孙博兴. 猪睾丸组织中miR-375的表达及靶基因预测. 中国兽医学报, 2014, 34(11): 1846-1849.
ZHANG X J, LI C M, ZHAO W, YU H B, ZHAO Z H, SUN B X. The expression of miR-375 and its target genes prediction in testicular tissue of junmu NO.1 white pig., 2014, 34(11): 1846-1849. (in Chinese)
[32] 罗志宇, 潘华, 冉雪琴, 王嘉福.香猪睾丸2种miRNA在不同生长发育阶段的变化.畜牧与兽医, 2015, 47(4): 91-96.
LUO Z Y, PAN H, RAN X Q, WANG J F. Changes of 2 kinds of miRNA in the testis of Xiang Pig in different developmental stages., 2015, 47(4): 91-96. (in Chinese)
[33] Esau C, Kang X L, Peralta E, Hanson E, Marcusson E G, Ravichandran L V, Sun Y Q, Koo S, Perera R J, Jain R, Dean N M, Freier S M, Frank Bennett C, Lollo B, Griffey R. MicroRNA-143 regulates adipocyte differentiation.,2004, 279(50): 52361-52365.
[34] 李国喜, 宁小敏, 李新建, 吴宗松, 杨公社.猪组织中miR-103实时定量PCR分析时合适内参的确定. 中国生物化学与分子生物学报, 2009(12): 1149-1154.
LI G X, NING X M, LI X J, WU Z S, YANG G S. Identification of suitable reference for quantitative RT-PCR assays of miR-103 in pig tissues., 2009(12): 1149-1154. (in Chinese)`
[35] 李春枚. 猪microRNA内参基因稳定性的验证[D]. 雅安:四川农业大学, 2011.
LI C M. Identification of suitable endogenous control microRNA genes in normal pig tissues[D]. Yaan: Sichuan Agricultural University, 2011. (in Chinese)
(责任编辑 林鉴非)
Validation of Reference Genes for Quantitative RT-PCR Analysis in Porcine Testis Tissues
PENG FuZhi, RAN MaoLiang, WENG Bo, LI Zhi, DONG LianHua, CHEN Bin
(College of Animal Science & Technology, Hunan Agricultural University / Hunan Provincial Key Laboratory for Genetic Improvement of Domestic Animal, Changsha 410128)
【Objective】Reference gene is the key to obtain the accurate analysis results when apply quantitative RT-PCR (qRT-PCR) technique to identify the gene expression level. However, reference gene and microRNA (miRNA) which are used in researching the molecular biology of porcine testis are not well characterized. Screening suitable reference gene will provide a reliable evidence for the quantitative analysis of target gene in different periods of porcine testis tissues.【Method】Porcine testis tissues at 8 different developmental stages (E 90, D 1, D 30, D 60, D 90, D 120, D 150, and DM) were selected as materials in this study. Trizol method was used to extract the total RNA and the concentration and purity of total RNA were detected by ND-2000 NanDrop spectrophotometer. The primers of 5 commonly used protein coding reference genes (,,,and) and 5 miRNA reference genes (U6, ssc-miR-17-5p, ssc-miR-26a, ssc-miR-27a and ssc-miR-103) were designed and synthesized to reverse transcription. cDNA templates were diluted to 7 concentration gradients with 1:10 serial dilution to construct standard curves. The expression of 10 candidate reference genes in porcine testis tissues was systematically analyzed at specified times, then the Genorm 3.5 was used to analyze the results. The most stable reference genes were picked up according to the stability value which called M value, the smaller the value of M, the better the stability of the reference gene; otherwise, the stability would be worse.【Result】The melting curves of 10 candidate reference genes (U6, ssc-miR-17-5p, ssc-miR-26a, ssc-miR-27a, and ssc-miR-103) indicated that the expression of all the candidate reference genes was of some specificity, no primer-dimers and nonspecific amplification. The relationship between Ct value and the logarithm of relative copy number was coincidence with linear relation according to the standard curves which were made with Ct value as ordinate and the logarithm of relative copy number as abscissas in a series of diluted concentration gradients. Analysis showed that the most stable reference gene of protein encoding gene wasand the most unstable was. U6 was the most suitable gene for miRNA expression analysis and ssc-miR-26a was the most unsuitable. 【Conclusion】This study has screened the most suitable reference genes (and U6) to identify porcine testis gene expression level successfully. The highly ranked reference genes identified from this study can provide a theoretical basis for the selection of the most suitable reference gene to detect differences in expression rates of genes and miRNAs in porcine testis tissues.
porcine testis tissue; reference genes; qRT-PCR; miRNA
2016-07-01;接受日期:2017-06-15
国家现代农业产业技术体系建设专项资金(CARS-36)
彭馥芝,Tel:13117516124;E-mail:pengfuzhi0809@126.com。通信作者陈斌,E-mail:chenbin7586@126.com

