CRIP1生物学功能的定量蛋白质组学分析
杨 超,霍 祎,邓海腾
(清华大学生命科学学院教育部生物信息学重点实验室,北京 100084)
CRIP1生物学功能的定量蛋白质组学分析
杨 超,霍 祎,邓海腾
(清华大学生命科学学院教育部生物信息学重点实验室,北京 100084)
CRIP1(cysteine-rich intestinal protein 1)是含有双锌指结构域的蛋白,在很多肿瘤细胞中高表达,但其在肺癌细胞中的生理学功能尚不明确。本研究应用定量蛋白质组学探究CRIP1过表达对肺癌顺铂耐药细胞(A549/DDP)的影响及作用机制。运用慢病毒载体系统构建了CRIP1过表达的A549/DDP稳转细胞系,利用Western Blotting证实了单克隆细胞系中CRIP1的过表达。发现CRIP1过表达能够加快细胞增殖,增加细胞的耐药性。通过定量蛋白质组学分析,鉴定了CRIP1过表达引起的蛋白质组的变化,并且对上调和下调的蛋白进行聚类分析。结果表明,CRIP1过表达上调了烟酰胺磷酸核糖转移酶(NAMPT)和NAD依赖型氧化还原酶的表达,从而促进细胞的增殖,并且提高了细胞的耐药性。
CRIP1;过表达;生长速率;蛋白质组学;烟酰胺磷酸核糖转移酶(NAMPT)
CRIP1(cysteine-rich intestinal protein 1)属于含有双锌指结构域(即LIM结构域)的蛋白质[1],其分子质量为8.5 ku[2],LIM结构域与心衰和心脏的发育有关[3]。CRIP家族成员在胚胎发育过程中起着重要作用[4],CRIP1会影响锌离子的转运[5]。近期研究发现,CRIP1在许多肿瘤细胞中高表达,是一个潜在的肿瘤标志物。在骨肉瘤中,CRIP1高表达是病症减轻的标志[6]。在乳腺癌的治疗中,CRIP1是预后良好的标志物,也可以作为乳腺癌分期的标志物[7-8]。另外,CRIP1和Galectin-3在子宫内膜癌中共表达,会影响患者预后和生存[9];CRIP1和S100P是前列腺癌中的高表达基因,其表达水平受表观遗传调节[10]。CRIP1在肺癌细胞中的生物学功能尚不清楚。
近年来,蛋白质组学已经成为癌症研究的重要工具[11-12]。串联质谱标签(TMT)是一种常用的蛋白定量技术,通过TMT试剂与多肽N端及赖氨酸残基侧链的胺基反应,标记酶解后的肽段。TMT试剂由反应基团、报告基团和平衡基团组成,不同样本的同一肽段标记后的质荷比在一级谱图中是相同的,而在二级质谱中,报告基团和平衡基团之间的键断裂,产生不同质荷比的报告离子用于定量分析。
本研究拟通过建立CRIP1过表达稳转细胞系,阐明CRIP1对细胞生长和耐药的影响,并且结合定量蛋白质组学分析,揭示CRIP1调控肿瘤细胞生长和耐药的生物学功能。
1 实验部分
1.1仪器
Dionex Ultimate 3000高效液相色谱仪、Orbitrap Fusion Tribrid液相色谱-质谱仪、细胞CO2培养箱、酶标仪:美国Thermo公司产品;台式低温高速离心机:德国Eppendorf公司产品;凝胶成像系统、核酸电泳仪、蛋白转膜装置、蛋白质电泳装置:美国Bio-Rad公司产品。
1.2材料
2×Taq PCR MasterMix、琼脂糖凝胶回收试剂盒、质粒小提试剂盒和DH5α:北京天根生化科技有限公司产品;AceQ qPCR SYBR Green Master Mix:南京诺唯赞生物科技有限公司产品; CRIP1抗体:英国Abcam公司产品;CCK8试剂盒:日本Dojindo公司产品;pcDNA3.1(+)、pLVX-IRES-Zsgreen1和A549/DDP:均为实验室保存;DMEM、RPMI 1640、PBS和双抗:加拿大Wisent公司产品;DTT:德国Merck公司产品;IAM:美国Sigma公司产品;胰酶(质谱级):美国Promega公司产品;TMT试剂和乙腈:美国Thermo公司产品。
1.3实验方法
1.3.1目的基因的获取与载体构建 首先从肺癌顺铂耐药细胞(A549/DDP,下同)中提取总RNA,逆转录成cDNA,以cDNA为模板,所用上游引物为5’-CCCAAGCTTGGGATGGATTACAAGGACGATGACGATAAGCCC-AAGTGTCCCAAGTG-3’,下游引物为5’-CGCGGATCCGCGTTACTTGAAAGTGTG-GCTCTCGG-3’进行聚合酶链式反应(PCR),产物回收。分别用HindIII和BamHI回收的目的片段和pcDNA3.1(+)载体进行双酶切。PCR目的片段与载体连接,进行连接产物转化。
构建pLVX-CRIP1-IRES-Zsgreen1质粒,以pcDNA3.1(+)-CRIP1质粒为模板进行亚克隆设计CRIP1引物,以5’-GGAATTCCATGGATTACAAGGACGATGACGATAAGCC-CAAGTGTCCCAAGTG-3’为上游引物,以5’-GCTCTAGAGCTTACTTGAAAGTGTGGCTCTCGG-3’为下游引物,进行PCR实验和产物回收。用EcoRI和XbaI进行双酶切,依次进行连接、转化和测序。
1.3.2细胞培养与细胞转染 用含10%胎牛血清的DMEM培养基培养293T细胞至70%左右用于慢病毒包装。用3种辅助质粒(pLP1∶pLP2∶VSVG=2∶3∶2),pLVX-CRIP1-IRES-Zsgreen1作为主质粒,对照组用pLVX-IRES-Zsgreen1作为主质粒,分别在293T细胞中进行慢病毒颗粒包装,获得包装完成的慢病毒颗粒并进行浓缩。
用含10%胎牛血清的RPMI 1640培养基培养A549/DDP细胞系用于过表达CRIP1,将A549/DDP铺于24孔板,细胞密度为30%~40%,加入一定量浓缩后的慢病毒颗粒和终浓度7 mg/L的聚凝胺溶液,感染6 h,换成含有10%胎牛血清和1%双抗的RPMI1640培养基继续培养至48 h,可通过荧光显微镜观察是否具有绿色荧光,长满至100 mm平皿。
选取绿色荧光较强的平皿,作为选定的多克隆细胞,一部分用于实时荧光定量核算扩增(qPCR)鉴定,一部分用于分选单克隆,用流式细胞仪分选GFP单细胞于96孔板,培养一段时间后挑选亮度较高的细胞系扩大培养,进一步鉴定。
1.3.3稳转细胞系的鉴定 提取上一步的多克隆细胞系,进行CRIP1过表达qPCR鉴定。用Trizol试剂提取细胞总RNA,配置逆转录PCR反应体系,对以cDNA为模板的CRIP1的qPCR引物进行qPCR检测,采用SYBR Green Master Mix配置反应体系,每组3个平行实验,使用的仪器是Roche,结果用2-ΔΔCt法分析,内参基因为Actin。
利用Western Blotting鉴定单克隆细胞系CRIP1过表达。选取2种荧光显微镜下较亮的过表达单克隆细胞系(CRIP1-OE1和CRIP1-OE2)和1种对照组细胞系(Control)提取全蛋白,用BCA(bicinchoninic acid)法测定并计算蛋白浓度,取等量的蛋白煮沸变性后,进行SDS-PAGE电泳,转PVDF膜,封闭1 h,CRIP1一抗于4 ℃孵育过夜并回收,洗膜,二抗于室温孵育1 h,洗膜。加入ECL发光液反应1 min,置于Bio-rad凝胶成像仪成像。
1.3.4细胞生长曲线的检测 用胰酶将CRIP1-OE和Control细胞从100 mm培养皿消化下来,收取细胞;使用血球计数法将细胞浓度调整为30 000个/毫升,加入96孔板(每孔100 μL),设置4组平行实验;分别在0、12、24、36、48、60、72、84、96 h以10∶1加入CCK8,2 h后测定450 nm吸光度值,并绘制细胞生长曲线,对比CRIP1-OE和Control组的细胞增殖速度。
1.3.5细胞顺铂耐药性的检测 用胰酶将CRIP1-OE和Control细胞从100 mm培养皿消化下来,以800 r/min离心5 min后收取细胞,用培养基重悬,使用血球计数法调整细胞浓度为20 000个/毫升,加入96孔板培养至贴壁牢固,设置3组平行实验;以0、40、80、120、160、200、240、320、400 μmol/L顺铂作用于细胞24 h,用CCK8计数,通过测量细胞的存活率研究CRIP1对细胞耐药性的影响。
1.3.6定量蛋白质组学分析 定量蛋白质组学分析包括以下四个步骤:第一步,蛋白提取。利用胰酶分离100 mm培养皿中贴壁的CRIP1-OE细胞和Control组细胞,分别收集于1.5 mL的EP管,用PBS洗2~3次,然后,用适量的8 mol/L尿素充分裂解,高速离心20 min,取上清用BCA试剂盒测量蛋白浓度,取100 μg蛋白,稀释至浓度1 g/L以下。
第二步,溶液内酶解。在室温下向蛋白溶液中加入适量的DTT,反应1 h;避光并加入适量的IAM,反应30 min;加入PBS稀释尿素;按照与蛋白1∶50比例加入质谱级胰酶,于37 ℃反应14 h,用10% TFA终止反应,调节pH为2左右。
第三步,样品除盐、TMT标记和鉴定。用Sep-Pak C18除盐柱,依次经活化、平衡、上样(3次过柱)、漂洗和洗脱5个步骤,使用的试剂依次是乙腈、0.1%TFA、50%ACN+0.1%TFA,除盐后离心,用100 mmol/L TEAB复溶,调至pH 8;向CRIP1-OE组中加入TMT6-128试剂,Control组中加入TMT6-131试剂,于室温反应1 h,用羟胺终止反应,合并两种样品,挥去TMT试剂中的乙腈,再次进行除盐、挥干,用0.1%FA复溶至100 μL,进行HPLC分离肽段,后续在蛋白质组学平台上进行二级质谱鉴定。
第四步,蛋白质组学数据分析。采用Protein Discoverer(Thermo)软件搜索数据库,找到鉴定蛋白。根据软件评分,选取大于4分的蛋白质进行分析。上调蛋白的标准是TMT比值大于1.5,下调同理。用Panther软件对上调和下调蛋白进行聚类分析。
2 实验结果
2.1CRIP1过表达稳转细胞系构建结果及鉴定
将构建成功的真核表达载体pcDNA3.1(+)-CRIP1酶切后,经琼脂糖凝胶电泳鉴定的目的条带符合预期大小,测序结果证实了目的条带已与载体连接。亚克隆的测序结果证实了pLVX-CRIP1-IRES-Zsgreen1质粒构建成功。用pLVX-CRIP1-IRES-Zsgreen1质粒包装病毒颗粒侵染A549/DDP细胞后,选取较亮的多克隆细胞进行qPCR分析,证明质粒成功转入细胞并表达CRIP1,示于图1a。对筛选出的过表达细胞系CRIP1-OE1、CRIP1-OE2和转入空载的Control细胞进行Western Blotting分析,发现在CRIP1过表达细胞中CRIP1表达量明显增加,证明过表达细胞系构建成功,示于图1b。
2.2CRIP1对细胞增殖和耐药性的影响
用CCK8试剂盒测量细胞数目,根据绘制的细胞生长曲线,发现与对照组细胞相比,CRIP1-OE细胞系生长速度明显加快,表明CRIP1能够促进细胞增殖。从第48 h开始,能够明显看到细胞数目的显著差异,到96 h时,差异更加明显,测量CRIP1-OE细胞吸光度为3.11,对照组为2.65,说明CRIP1-OE细胞的表型之一是增殖速度加快,示于图2a。通过1.3.5节的方法检测CRIP1-OE细胞的顺铂耐药性,比较实验组和对照组细胞存活率,发现实验组的细胞存活率大于对照组。实验结果表明,CRIP1过表达增强了A549/DDP细胞的耐药性,示于图2b。

注:a.CRIP1过表达细胞和Control细胞中CRIP1 mRNA的水平;b.CRIP1过表达细胞和Control细胞中CRIP1蛋白的表达水平图1 稳转细胞系中CRIP1过表达的验证Fig.1 Confirmation of CRIP1 overexpression in stably transfected cells
2.3CRIP1过表达稳转细胞系的蛋白质组学分析
为了理解CRIP1在细胞中的生物学功能,对CRIP1过表达的A549/DDP细胞和对应的Control细胞进行基于TMT标记的定量蛋白质组学分析。共鉴定到4 525个蛋白,其中上调蛋白质(比值>1.5)152个,下调蛋白质(比值<0.67)142个。用Panther软件对所有变化的蛋白质进行聚类分析,结果表明,CRIP1过表达能够影响代谢过程、细胞组成、生物学调控、细胞粘附、细胞增殖等细胞过程,尤其对细胞初级代谢中含氮化合物的代谢有较大影响,示于图3。
为了证实CRIP1的表达量变化,鉴定到了CRIP1蛋白胰酶降解后被TMT标记的一个肽段:TLTSGGHAEHEGKPYCNHPCYAAMFGPK。该肽段的二级质谱图示于图4a,其中碰撞碎裂后产生的b、y系列离子与肽段序列匹配,断裂形成的TMT报告基团质量分别为128.13 Da和131.11 Da,其离子强度比值为2.4∶1,说明该肽段在CRIP1过表达细胞的含量是对照细胞的2.4倍。对CRIP1的全部二级谱图的定量结果进行平均,计算出CRIP1在过表达细胞系中升高了4倍左右。同理,鉴定了烟酰胺磷酸核糖转移酶(NAMPT)降解后被TMT标记的一个肽段VLEILGK,其二级图谱显示该肽段相较于对照细胞升高了1.7倍,将NAMPT的全部二级图谱的定量结果进行平均,计算出NAMPT在CRIP1过表达细胞系中升高了1.7倍,示于图4b。同时,在定量蛋白质组学数据中,发现NAD依赖型氧化还原酶表达量增加,例如AKR1C1和AKR1B10分别上调2.3倍和2.1倍,示于图5。

注:a.CRIP1过表达细胞和Control细胞的生长曲线;b.顺铂刺激下CRIP1过表达细胞和Control细胞的存活率曲线图2 CRIP1过表达对细胞生长和耐药性的影响Fig.2 Effects of CRIP1 overexpression on cell growth and drug resistance

图3 CRIP1过表达细胞与对照细胞差异表达蛋白的聚类分析Fig.3 GO analysis of differentially expressed proteins between CRIP1 overexpressing cells and the control cells
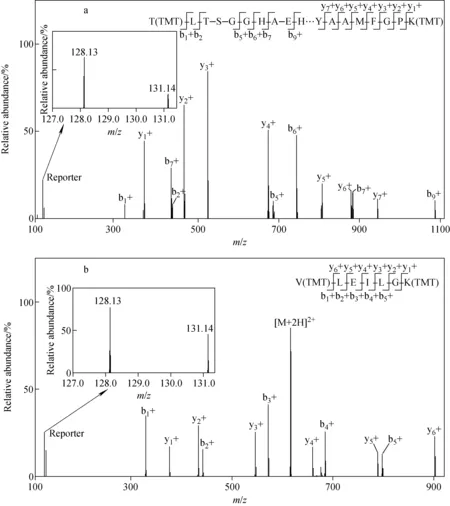
注:a.TMT标记过的CRIP1酶解肽段TLTSGGHAEHEGKPYCNHPCYAAMFGPK的二级质谱图;b.TMT标记过的NAMPT酶解肽段VLEILGK的二级质谱图图4 CRIP1过表达细胞和Control细胞中CRIP1和NAMPT表达的定量蛋白质组学分析Fig.4 Quantitative proteomics analysis of CRIP1 and NAMPT expression in CRIP1 overexpression and the control cells

图5 蛋白质组学中NAD依赖型氧化还原酶的表达Fig.5 Expression of oxidoreductase dependent on NAD in proteomics analysis
3 讨论
本研究构建了CRIP1过表达细胞系,发现CRIP1过表达可使细胞生长速度加快,耐药性增强。通过蛋白质组学分析,发现CRIP1过表达会影响细胞的代谢过程,尤其会导致NAMPT上调。NAMPT是细胞内NAD合成的限速酶,可以将烟酰胺转化为哺乳动物的烟酰胺单核苷酸,进而合成NAD[13],主要影响补救途径[14]。它在很多肿瘤中高表达,其中包括乳腺癌、卵巢癌、黑色素瘤等实体瘤[15]和血液瘤[16]。肿瘤细胞代谢与NAMPT的调节作用有关,可以通过影响NAD依赖的酶(如SIRT家族和PARP家族)[17-18],维持细胞NAD水平。另外,NAMPT在清除细胞内ROS方面,可以与GSH系统相互作用,从而减轻药物处理引发的ROS对细胞的杀伤作用[19]。也有一些研究认为,通过影响细胞内NAD含量的变化,NAMPT保障了糖酵解和线粒体呼吸链代谢需求[20],从而维持了线粒体稳态并增加了细胞的耐药性。NAMPT还与肿瘤预后有关[21-22]。其高表达可能导致细胞内NAD的增加,从而引起NAD依赖的氧化还原酶表达上调,促进肺癌细胞的生长。
[1] DAVIS B A, BLANCHARD R K, LANNINGHAM-FOSTER L, et al. Structural characterization of the rat cysteine-rich intestinal protein gene and overexpression of this LIM-only protein in transgenic mice[J]. DNA Cell Biol, 1998, 17(12): 1 057-1 064.
[2] KHOO C, BLANCHARD R K, SULLIVAN V K, et al. Human cysteine-rich intestinal protein: cDNA cloning and expression of recombinant protein and identification in human peripheral blood mononuclear cells[J]. Protein Expr Purif, 1997, 9(3): 379-387.
[3] LI A, PONTEN F, DOS R C. The interactome of LIM domain proteins: the contributions of LIM domain proteins to heart failure and heart development[J]. Proteomics, 2012, 12(2): 203-225.
[4] HEMPEL A, KUHL S J. Comparative expression analysis of cysteine-rich intestinal protein family members crip1, 2 and 3 during Xenopuslaevisembryogenesis[J]. Int J Dev Biol, 2014, 58(10/11/12): 841-849.
[5] HEMPE J M, COUSINS R J. Cysteine-rich intestinal protein binds zinc during transmucosal zinc transport[J]. Proc Natl Acad Sci USA, 1991, 88(21): 9 671-9 674.
[6] BAUMHOER D, ELSNER M, SMIDA J, et al. CRIP1 expression is correlated with a favorable outcome and less metastases in osteosarcoma patients[J]. Oncotarget, 2011, 2(12): 970-975.
[7] LUDYGA N, ENGLERT S, PFLIEGER K, et al. The impact of cysteine-rich intestinal protein 1 (CRIP1) in human breast cancer[J]. Mol Cancer, 2013, 12(1): 28.
[8] XIE H, LI H, HUANG Y, et al. Combining peptide and DNA for protein assay: CRIP1 detection for breast cancer staging[J]. ACS Appl Mater Interfaces, 2014, 6(1): 459-463.
[9] LAMBROPOULOU M, DEFTEREOU T E, KYNIGOPOULOS S, et al. Co-expression of galectin-3 and CRIP-1 in endometrial cancer: prognostic value and patient survival[J]. Med Oncol, 2016, 33(1): 8.
[10]WANG Q, WILLIAMSON M, BOTT S, et al. Hypomethylation of WNT5A, CRIP1 and S100P in prostate cancer[J]. Oncogene, 2007, 26(45): 6 560-6 565.
[11]HUO Y, ZHENG Z, CHEN Y, et al. Downregulation of vimentin expression increased drug resistance in ovarian cancer cells[J]. Oncotarget, 2016, 7(29): 45 876-45 888.
[12]TANG H, CHEN Y, LIU X, et al. Downregulation of HSP60 disrupts mitochondrial proteostasis to promote tumorigenesis and progression in clear cell renal cell carcinoma[J]. Oncotarget, 2016, 7(25): 38 822-38 834.
[13]REVOLLO J R, GRIMM A A, IMAI S. The regulation of nicotinamide adenine dinucleotide biosynthesis by Nampt/PBEF/visfatin in mammals[J]. Curr Opin Gastroenterol, 2007, 23(2): 164-170.
[14]REVOLLO J R, GRIMM A A, IMAI S. The NAD biosynthesis pathway mediated by nicotinamide phosphoribosyltransferase regulates Sir2 activity in mammalian cells[J]. J Biol Chem, 2004, 279(49): 50 754-50 763.
[15]SHACKELFORD R E, MAYHALL K, MAXWELL N M, et al. Nicotinamide phosphoribosyltransferase in malignancy: a review[J]. Genes Cancer, 2013, 4(11/12): 447-456.
[16]OLESEN U H, HASTRUP N, SEHESTED M. Expression patterns of nicotinamide phosphoribosyltransferase and nicotinic acid phosphoribosyltransferase in human malignant lymphomas[J]. APMIS, 2011, 119(4/5): 296-303.
[17]GUAN X, LIN P, KNOLL E, et al. Mechanism of inhibition of the human sirtuin enzyme SIRT3 by nicotinamide: computational and experimental studies[J]. PLoS One, 2014, 9(9): e107729.
[18]RAPPOU E, JUKARAINEN S, RINNANKOSKI-TUIKKA R, et al. Weight loss is associated with increased NAD(+)/SIRT1 expression but reduced PARP activity in white adipose tissue[J]. J Clin Endocrinol Metab, 2016, 101(3): 1 263-1 273.
[19]HONG S M, PARK C W, KIM S W, et al. NAMPT suppresses glucose deprivation-induced oxidative stress by increasing NADPH levels in breast cancer[J]. Oncogene, 2016, 35(27): 3 544-3 554.
[20]YING W. NAD+/NADH and NADP+/NADPH in cellular functions and cell death: regulation and biological consequences[J]. Antioxid Redox Signal, 2008, 10(2): 179-206.
[21]GARTEN A, SCHUSTER S, PENKE M, et al. Physiological and pathophysiological roles of NAMPT and NAD metabolism[J]. Nat Rev Endocrinol, 2015, 11(9): 535-546.
[22]GROLLA A A, TRAVELLI C, GENAZZANI A A, et al. Extracellular nicotinamide phosphoribosyltransferase, a new cancer metabokine[J]. Br J Pharmacol, 2016, 173(14): 2 182-2 194.
QuantitativeProteomicsAnalysisonBiologicalFunctionofCRIP1
YANG Chao, HUO Yi, DENG Hai-teng
(MOEKeyLaboratoryofBioinformatics,SchoolofLifeSciences,TsinghuaUniversity,Beijing100084,China)
The aim of the present study is to investigate the effects of cysteine-rich intestinal protein (CRIP1) over-expression on cellular processes using proteomics. CRIP1 is a protein that contains a double zinc finger domain, and is highly expressed in many tumor cells, but its roles in lung cancer cells remain elusive. In this study, the CRIP1 gene was cloned and used pLVX-IRES-Zsgreen1 lentiviral vector that encodes a GFP to transfect A549/DDP cells that was a cisplatin resistant cells as compared to A549 cells. The real-time quantitative PCR (qPCR) was used to confirm the transfection efficiency of CRIP1. Flow cytometry was used to isolate single cells, which was seeded into a 96-well plate and cultured to obtain monoclonal cell lines. The monoclonal cell lines with high brightness were screened by fluorescence microscopy and CRIP1 over-expression was confirmed by western blot analysis, indicating that a stable cell line was established successfully, in which CRIP1 was over-expressed. The growth curves of CRIP1 over-expression cells and the control cells were measured by the CCK8 assay. It showed that CRIP1 over-expression increased cell proliferation. CRIP1 over-expression cells and the control cells were also treated with cisplatin at different concentrations for 24 h and examined the viability of cisplatin-treated cells. Results showed that CRIP1 over-expression enhanced cells’ resistance to cisplatin.
To further explore the mechanism underlying CRIP1 over-expression mediated cellular processes, quantitative proteomics was applied to identify differentially expressed proteins between the control and CRIP1 over-expression cells. The quantitative proteomics was carried out using the TMTsixplexTMIsobaric Label Reagent, which modified the free amino group at the peptideN-terminus and lysine residues. Modified peptides generated reporter ions in MS/MS spectra to provide quantitative information about the proteins being analyzed. It was found that CRIP1 over-expression upregulated the expressions of nicotinamide phosphoribosyltransferase (NAMPT), which was the rate limiting enzyme in the scavenge pathway for production of the intracellular nicotinamide adenine dinucleotide (NAD). The CRIP1 over-expression mediated upregulation of NAMPT can increase the cellular NAD level. It was also revealed that CRIP1 over-expression upregulated expressions of NAD-dependent oxidoreductases, such as aldo-keto reductase family 1 member C1 (AKR1C1) and aldo-keto reductase family 1 member B10 (AKR1B10). Extensive studies have reported that NAD is essential for the cellular anti-oxidant systems and for maintaining mitochondrial integrity. The results suggest that the CRIP1 over-expression mediated NAMPT upregulation contributed to the enhanced cell growth and resistance to cisplatin.
cysteine-rich intestinal protein 1 (CRIP1); overexpression; growth rate; proteomics; nicotinamide phosphoribosyltransferase (NAMPT)
2017-04-17;
2017-05-25
科技部973项目(2014CBA02005)资助
杨 超(1991—),女(满族),河北承德人,硕士研究生,生物学专业。E-mail: hiyangchao@163.com
邓海腾(1964—),男(汉族),云南宣威人,博士生导师,从事蛋白质组学研究。E-mail: dht@mail.tsinghua.edu.cn
O657.3
:A
:1004-2997(2017)04-0486-08
10.7538/zpxb.2017.0077

