DNA修复基因XPD XPC XRCC4基因多态性与结直肠癌易感性的关联性研究*
张 莎 陈自平 杜文军 熊宏超 徐昌青
·临床研究与应用·
DNA修复基因XPD XPC XRCC4基因多态性与结直肠癌易感性的关联性研究*
张 莎①陈自平①杜文军①熊宏超②徐昌青①
目的:探讨DNA修复基因XPD rs13181(codon751A/C,Lys751Gln)、rs238406(codon156C/A,Arg156Arg)、XPC rs2279017(i11C/A)和XRCC4 rs3734091(codon247T/C,Ala247Ser)的单核苷酸多态性与结直肠癌易感性的关系。方法:采用TaqMan技术对2013年4月至2016年1月北京肿瘤医院收治的338例结直肠癌患者(病例组)和315例健康者(对照组)进行多态位点基因型的检测。结果:XPD rs13181基因型GT和等位基因G增加个体结直肠癌的发病风险(GT>TT,adjusted OR=1.69,95%CI:1.15~2.47,P= 0.007;G>T,adjusted OR=1.77,95%CI:1.19~2.64,P=0.005);XRCC4 rs3734091基因型GT和等位基因T增加个体结直肠癌的易感性(GT>GG,adjusted OR=9.02,95%CI:5.61~14.50,P<0.001;T>G,adjusted OR=4.06,95%CI:2.49~6.61,P<0.001);XPD rs13181和rs238406的单倍体型GT显著降低结直肠癌的发病风险(adjusted OR=0.39,95%CI:0.18~0.85,P=0.018)。XPCrs2279017等位基因G和XRCC4 rs3734091等位基因T的联合效应(adjusted OR=28.43,95%CI:6.85~117.95,P<0.001)以及XPD rs13181等位基因G和XRCC4 rs3734091等位基因T的联合效应(adjusted OR=10.24,95%CI:4.69~22.35,P<0.001)显著增加个体结直肠癌的易感性。结论:XPD rs13181和XRCC4 rs3734091位点的多态性与结直肠癌的易感性相关。
XPDXPCXRCC4单核苷酸多态性结直肠癌TaqMan技术
结直肠癌发病率在全球居第3位[1],在中国居第4位,且发病率和死亡率呈上升趋势[2]。多项研究表明,结直肠癌家族史患者的结直肠癌发病率较高[3-5],因此,基因方面的影响不容忽视。DNA修复基因对于防止DNA双链突变起着至关重要的作用。DNA修复基因的单核苷酸多态性可能会影响DNA修复基因的修复能力,使其不能及时修复在DNA复制过程中产生的突变,并最终导致癌症的发生。有研究表明,DNA的修复能力在多种癌症患者中降低,其中包括结直肠癌[6]。DNA的修复能力部分存在于DNA修复系统的管家基因中,包括XPD、XPC和XRCC4等。因此,DNA修复基因XPD、XPC、XRCC4的单核苷酸多态性可能是导致结直肠癌发生发展的原因之一。
XPD基因,也被称为ERCC2(excision repair crosscomplementation rodent repair deficiency group 2),位于第19号染色体的长臂上,包含23个外显子[7],参与核苷酸切除修复(NER)。XPD基因编码一种依赖ATP的5'→3'解螺旋酶,是基础转录因子IIH(TFIIH)的亚单位[8]。TFIIH复合体负责调控NER修复机制的精确性[9]。作为TFIIH复合体的一部分,XPD主要有核苷酸切除修复和基础转录两个作用[10]。XPC基因位于第3号染色体的长臂,包含16个外显子和15个内含子[11]。XPC与HR23B形成的XPC-HR23B复合体主要参与修复早期对DNA损伤的识别以及修复的启动,是NER通路的限速步骤[12]。XRCC4位于第5号染色体上,通过非同源末端连接(NHEJ)来修复受损的DNA双链。在NHEJ修复通路中,DNA损伤末端与被XRCC5和XRCC6蛋白异二聚体(KU70/80)激活的DNA依赖蛋白酶(DNA-PKcs)结合[13],随后LIG4-XRCC4异二聚体完成最终的末端再连接,XRCC4是连接LIG4和DNA-PKcs的核心磷蛋白,稳定并促进LIG4的活性[14]。
有研究报道XPD基因多态性与包括非小细胞肺癌、食管鳞癌、胃癌、膀胱癌等[15-20]的易感性关系尚无一致结论。多态性对肿瘤易感性的微弱影响和种族差异性可能对研究结果有所影响。因此,需要更多的研究加以证实。另外,与一个SNP位点的多态性产生的效应相比,多个位点多态性的联合效应对癌症易感性的影响更显著[21]。
本研究选取XPD rs13181、rs238406,XPC rs2279017和XRCC4 rs3734091四个位点,采用TaqMan技术检测上述位点的基因型,并分析其单核苷酸多态性及其联合效应对结直肠癌易感性的影响。
1 材料与方法
1.1 临床资料
选取2013年4月至2016年1月在北京肿瘤医院住院的结直肠癌患者338例(病例组),所有患者均经过病理组织学检查确诊为结直肠腺癌,并排除其他恶性肿瘤病史。临床资料不完整及已经过化疗和放疗的患者均已剔除。年龄、性别、吸烟及饮酒史等人口学资料均从病历资料中获得。对照组为315例于北京肿瘤医院同时期健康体检者。
1.2 方法
DNA提取和基因型检测抽取5 mL外周血于EDTA抗凝管中,4℃短期保存,采用DNeasy®Blood& Tissue Kit(250)(购自上海玉博生物科技有限公司)试剂盒提取全基因组DNA,-80℃冰箱长期保存。采用TaqMan基因型检测技术检测XPD rs13181、rs238406,XPC rs2279017及XRCC4 rs3734091多态性位点基因型。在Agilent Mx3005P QPCR系统中按照说明书(购自德国Agilent Technologies公司)进行检测。引物和探针均由Applied Biosystems设计。热循环条件为:95℃10 min、95℃30 s、60℃1 min共40个循环。采用Agilent Mx3005P QPCR系统进行荧光信号的实时探测。
1.3 统计学分析
运用Stata 11.0软件进行统计学分析。用拟合优度χ2检验检测每个位点的基因型频率是否符合Har⁃dy-Weinberg平衡定律。用PHASE软件构建单倍体型并计算单倍体型频率。运用Logistic回归模型计算比值比(crude ORs)和95%的可信区间(95%CIs)来分析基因型、等位基因及单倍体型与结直肠癌易感性的关联性。校正了年龄、性别、吸烟、饮酒等因素的调整比值比(adjusted ORs)以及95%的可信区间(95%CIs)采用1 000次取样(1 000 resamples)[22]的方法获得。所有的统计检验均为双侧检验,以P<0.05为差异具有统计学意义。
2 结果
2.1XPD、XPC、XRCC4基因多态性与结直肠癌易感性的关联性分析
XPD rs13181、rs238406,XPC rs2279017和XRCC4 rs3734091位点基因型分布符合Hardy-Weinberg平衡定律(P>0.05)。XPD rs13181位点基因型GT增加了结直肠癌易感性(GT>TT,adjusted OR=1.69,95%CI:1.159~2.47,P=0.007);携带等位基因G的个体患结直肠癌的风险约为非携带者的2倍(G>T,adjusted OR=1.77,95% CI:1.19~2.64,P=0.005)。XRCC4 rs3734091位点基因型GT和等位基因T明显增加个体患结直肠癌的风险(GT>GG,adjusted OR=9.02,95%CI:5.61~14.50,P<0.001;T>G,adjusted OR=4.06,95%CI:2.49~6.61,P<0.001)。XPD rs238406和XPC rs2279017位点基因多态性与结直肠癌易感性无显著相关性(表1)。
2.2 单倍体型与结直肠癌易感性的关联性分析
根据XPD rs238406 and rs13181位点基因型,共构建出四种单倍体型。携带单倍体型GT的个体患结直肠癌的风险降低(adjusted OR=0.39,95%CI:0.18~0.85,P=0.018,表2、3)。
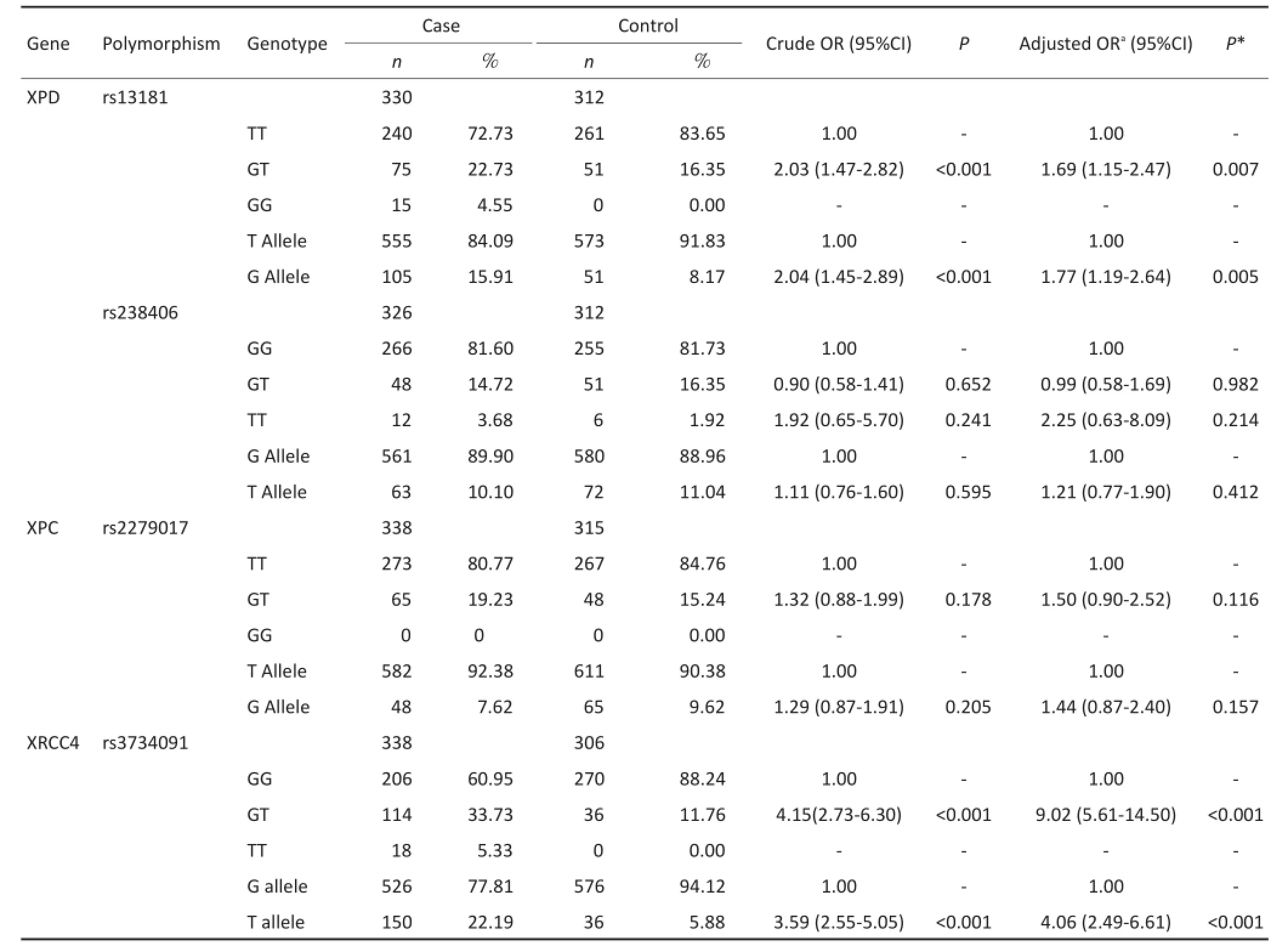
表1XPD、XPC、XRCC4基因多态性位点基因型与等位基因分布及其与结直肠癌易感性的关联性分析Table1 Distribution of XPD,XPC,and XRCC4 polymorphisms and CRC risk
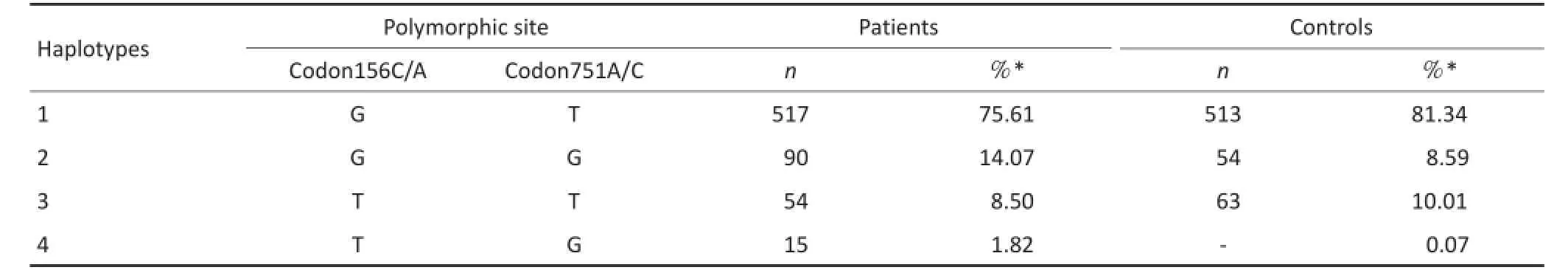
表2 XPDrs238406和rs13181位点单倍体型频率分布Table2 Haplotype frequencies of XPD in patients and controls
2.3 XPD、XPC、XRCC4基因的联合效应与结直肠癌易感性的关联性分析
根据基因型频率分布情况,将XPD rs13181和XPC rs2279017位点的等位基因T与XRCC4 rs3734091位点的等位基因G作为无风险参考对象,本研究中,由于没有研究对象在XPDrs13181、XPCrs2279017、XRCC 4rs3734091位点同时存在这三种等位基因,因此进行基因的两两关联比较。结果表明同时在XPC rs2279017位点含有等位基因G和在XRCC4 rs3734091位点含有等位基因T的个体患结直肠癌的风险明显增加(adjusted OR=28.43,95%CI:6.85~117.95,P<0.001)。同样,同时在XPD rs13181位点含有等位基因G和在XRCC4 rs3734091位点含有等位基因T的个体患结直肠癌的风险明显增加(adjusted OR=10.24,95%CI:4.69~22.35,P<0.001,表4)。
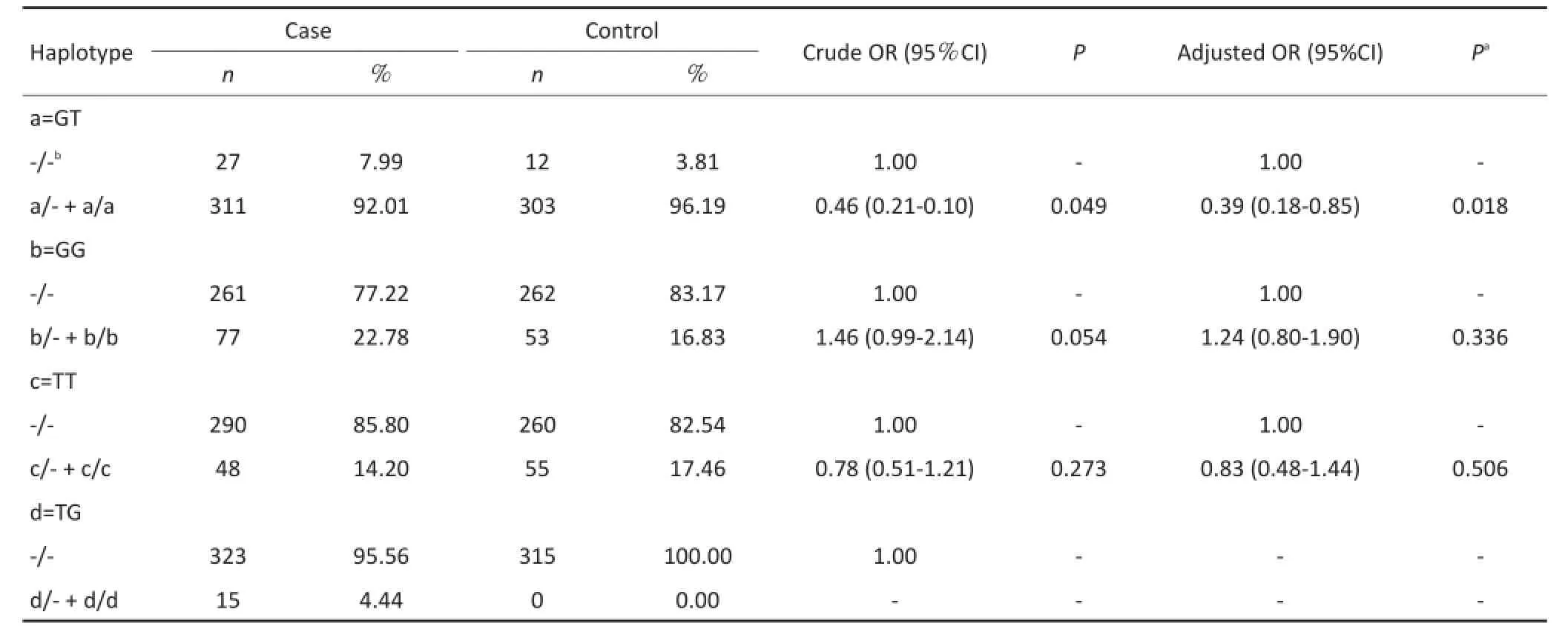
表3 XPD rs238406和rs13181单倍体型分布与结直肠癌易感性的关联性分析Table3 Association between XPD haplotypes and CRC risk
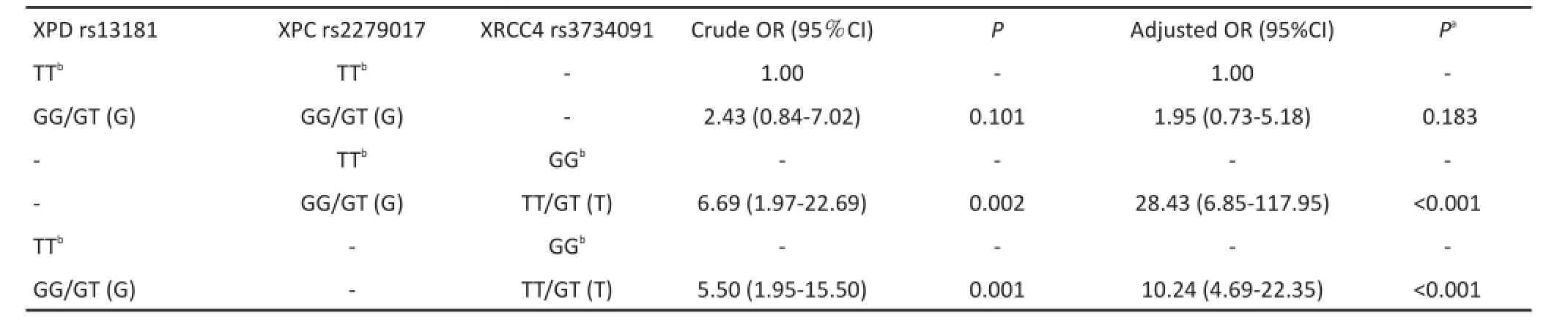
表4 XPD、XPC、XRCC4基因多态性的联合效应与结直肠癌易感性的关联性分析Table4 Combined effects of XPD,XPC,and XRCC 4 and CRC risk
3 讨论
全球结直肠癌的发病率在男性中居第3位,女性中居第2位,虽然发展中国家结直肠癌的发病率低于发达国家[23],但结直肠癌在中国的发病率和死亡率呈明显上升趋势[24]。结直肠癌的发生与饮食习惯、环境、遗传等因素有关,但最终取决于机体对肿瘤的易感性。DNA修复基因的基因多态性可能会影响其所编码的蛋白的结构和功能状态,从而无法及时识别并修复突变的DNA,进而导致结直肠癌的发生。
本研究结果表明,XPD rs13181位点的基因型GT及等位基因G增加个体结直肠癌的患病风险。XPD rs13181位点碱基T→G的颠换会导致氨基酸序列中赖氨酸被谷氨酸替换,引起氨基酸序列的改变,从而影响螺旋酶的活性[9]。这种改变可能会减弱XPD的修复能力,使其不能及时修复突变的DNA而导致癌症的发生。Chang等[25]的研究未发现XPD rs13181基因多态性与结直肠癌易感性相关,与本研究结果不一致。可能与以下因素有关:1)该研究以中国台湾人群为研究对象,本研究的研究对象为中国内地人群,不同人群在生活环境、饮食习惯、遗传因素等方面存在差异,基因-环境、基因-基因的交互作用共同影响着癌症的发生和发展。2)两个研究均为以医院为基础的病例-对照研究,存在选择偏倚也可能会导致结果的不一致。因此,需要多中心及更大样本量的研究加以证实。
本研究发现,XPC rs2279017位点基因多态性与结直肠癌易感性无显著相关性。XPC-HR23B主要参与早期损伤的探测及使NER修复通路中的其他功能分子聚集在受损的DNA部位[26]。XPC rs2279017位点基因多态性与结直肠癌易感性无显著相关,可能是因为XPC rs2279017位点位于内含子区域,碱基的改变不会直接引起氨基酸序列的变化,进而不会影响XPC蛋白的功能。Gil等[27]在一个相对较大的样本中研究XPC rs2279017位点基因多态性与结直肠癌易感性的关系,发现两者无显著相关性,与本研究得出的结论一致。
本研究表明,XRCC4 rs3734091位点的基因型GT能增加个体患结直肠癌的风险。携带等位基因T的个体患结直肠癌的风险为非携带者的4倍。有研究发现,细胞内XRCC4的损伤会引起机体对放射线高敏感、V(D)J基因重排的缺失以及染色体不稳定[28]。实验表明,小鼠XRCC4的缺失能引起大量的神经元细胞凋亡进而引起胚胎死亡,并且XRCC4与p53在调控细胞凋亡过程中相互影响,这些证据都表明XRCC4在保持基因组稳定及肿瘤抑制中起着至关重要的作用[29]。
本研究单倍体型的分析表明,XPD rs238406和rs13181位点的单倍体型GT可降低结直肠癌的发病风险。目前,关于XPD rs238406和rs13181位点的单倍体型与结直肠癌易感性的关系尚缺乏报道。Zhao等[30]在非西班牙裔白人健康人群中研究发现XPD rs13181,rs1799793 and rs238406三个多态性位点构建的单倍型能增加体外二羟环氧苯并芘(BPDE)所诱导的DNA加合物水平。DNA加合物被认为与患癌风险相关,因此,该研究与本研究所得结论不一致。可能与以下因素有关:1)该研究对象为健康非西班牙裔白人,不同人种在环境、生活习惯及遗传上差异较大,可能造成结果的不一致。2)该研究为三个多态位点构建的单倍型,而本研究为两个多态位点构建的单倍型。3)该研究探讨的是单倍型与体外化学物所诱导的DNA加合物水平的关系,并没有直接对单倍型与癌症易感性的关系进行研究,虽然DNA加合物被认为与患癌风险有关,但两者不能等同。
同时携带XPC rs2279017位点等位基因G和XRCC4 rs3734091位点等位基因T或XPD rs13181等位基因G和XRCC4 rs3734091位点等位基因T的个体结直肠癌的患病风险明显增加,表明基因-基因的联合效应在分析基因多态性与结直肠癌易感性的相关分析中具有显著作用。本研究将XPD、XPC和XRCC4的联合效应与结直肠癌的易感性关系进行分析,以期为结直肠癌的早期诊断提供新的标志物。
综上所述,XPD rs13181位点基因型GT和等位基因G与XRCC4 rs3734091位点基因型GT和等位基因T增加结直肠癌的发病风险,携带这些基因型和等位基因的个体应定期进行消化道内镜的检查,以实现对结直肠癌的早诊早治,提高患者的生存质量,延长患者的生存时间。
[1]Shike M,Winawer SJ,Greenwad PH,et al.Primary prevention of colorectal cancer.The WHO Collaborating Centre for the Prevention of Colorectal Cancer[J].Bull World Health Organ,1990,68(3):377‐385.
[2]Li S,Wang J,Lu Y,et al.Screening and early diagnosis of colorectal cancer inC hina:a 12 year retrospect(1994‐2006)[J].J Cancer Res Clin Oncol,2007,133(10):679‐686.
[3]Hsu L,Jeon J,Brenner H,et al.A model to determine colorectal cancer risk using common genetic susceptibility loci[J].Gastroenterology, 2015,148(7):1330‐1339.
[4]Johnson CM,Wei C,Ensor JE,et al.Meta‐analyses of colorectal cancer risk factors[J].Cancer Causes Control,2013,24(6):1207‐1222.
[5]Schoen RE,Razzak A,Yu KJ,et al.Incidence and mortality of colorectal cancer in individuals with a family history of colorectal cancer[J]. Gastroenterology,2015,149(6):1438‐1445.
[6]de Boer JG.Polymorphisms in DNA repair and environmental inter‐actions[J].Mutat Res,2002,509(1‐2):201‐210.
[7]Wu KG,He XF,Li YH,et al.Association between the XPD/ERCC2 Lys751Gln polymorphism and risk of cancer:evidence from 224 case‐control studies[J].Tumour Biol,2014,35(11):11243‐11259.
[8]Jiao L,Hassan MM,Bondy ML,et al.The XPD Asp312Asn and Lys751Gln polymorphisms,corresponding haplotype,and pancreatic cancer risk[J].Cancer Lett,2007,245(1‐2):61‐68.
[9]Wu HY,Ding LY.Comprehensive assessment of the association between XPD rs13181 polymorphism and lung cancer risk[J].Tumour Biol,2014, 35(8):8125‐8132.
[10]CoinF,Marinoni JC,RodolfoC,et al.Mutations intheXPDhelicasegene result in XP and TTD phenotypes,preventing interaction between XPD and the p44 subunit of TFIIH[J].Nat Genet,1998,20(2):184‐188.
[11]Zhu ML,Hua RX,Zheng L.Associations between polymorphisms of the XPC gene and lung cancer susceptibility:a meta‐analysis[J].Tumour Biol,2014,35(4):2931‐2939.
[12]Zhang Y,Wang X,Zhang W,et al.AnassociationbetweenXPCLys939Gln polymorphism and the risk of bladder cancer:a meta‐analysis[J]. Tumour Biol,2013,34(2):973‐982.
[13]Abe T,Ishiai M,Hosono Y,et al.KU70/80,DNA‐PKcs,and Artemis are essential for the rapid induction of apoptosis after massive DSB for‐mation[J].Cell Signal,2008,20(11):1978‐1985.
[14]Su Y,Qi S,Dou C,et al.Association of LIG4 and XRCC4 gene poly‐morphisms with the risk of human glioma in a Chinese population[J]. Int J Clin Exp Pathol,2015,8(2):2057‐2062.
[15]Huang CG,Liu T,Lv GD,et al.Analysis of XPD genetic polymorphisms of esophageal squamous cell carcinoma in a population of Yili Pre‐fecture,in Xinjiang,China[J].Mol Biol Rep,2012,39(1):709‐714.
[16]Ji HX,Chang WS,Tsai CW,et al.Contribution of DNA repair xeroder‐mapigmentosum group D genotype to gastric cancer risk in taiwan[J]. Anticancer Res,2015,35(9):4975‐4981.
[17]Malats N.Genetic epidemiology of bladder cancer:scaling up in the identification of low‐penetrance genetic markers of bladder cancer risk and progression[J].Scand J Urol Nephrol Suppl,2008(218):131‐140.
[18]Michalska MM,Samulak D,Jablonski F,et al.The R156R ERCC2 poly‐morphism as a risk factor of endometrial cancer[J].Tumour Biol,2016, 37(2):2171‐2176.
[19]Zhang L,Ruan Z,Hong Q,et al.Single nucleotide polymorphisms in DNA repair genes and risk of cervical cancer:A case‐control study[J]. Oncol Lett,2012,3(2):351‐362.
[20]Geredeli C,Artac M,Yildirim S,et al.Prognostic value of ERCC1,ERCC2, XRCC1,and TP53 single nucleotide polymorphisms in patients withearly‐stage non‐small cell lung cancer[J].Tumour Biol,2015,36(6): 4279‐4285.
[21]Farnebo L,Stjernstrom A,Fredrikson M,et al.DNA repair genes XPC, XPD,XRCC1,and XRCC3 are associated with risk and survival of squamous cell carcinoma of the head and neck[J].DNA Repair(Amst), 2015,31:64‐72.
[22]Westfall PH,Young SS.p‐Value adjustments for multiple tests in multivariate binomial models[J].J Am Stat Assoc,1989,84(84):780‐786.
[23]Torre LA,Bray F,Siegel RL,et al.Global cancer statistics,2012[J].CA Cancer J Clin,2015,65(2):87‐108.
[24]Zeuner A,Todaro M,Stassi G,et al.Colorectal cancer stem cells:from the crypt to the clinic[J].Cell stem cell,2014,15(6):692‐705.
[25]Chang WS,Yueh TC,Tsai CW,et al.Contribution of DNA repair xero‐derma pigmentosum group D genotypes to colorectal cancer risk in taiwan[J].Anticancer Res,2016,36(4):1657‐63.
[26]Zhang Y,Li Z,Zhong Q,et al.Polymorphisms of the XPC gene may contribute to the risk of head and neck cancer:a meta‐analysis[J]. Tumour Biol,2014,35:3917‐3931.
[27]Gil J,Gaj P,Misiak B,et al.CYP1A1 Ile462Val polymorphism and colorectal cancer risk in Polish patients[J].Med Oncol,2014,31(7):72.
[28]Barnes DE,Stamp G,Rosewell I,et al.Targeted disruption of the gene encoding DNA ligase IV leads to lethality in embryonic mice[J].Curr Biol,1998,8:1395‐1398.
[29]Gao Y,Ferguson DO,Xie W,et al.Interplay of p53 and DNA‐repair protein XRCC4 in tumorigenesis,genomic stability and development [J].Nature,2000,404:897‐900.
[30]Zhao H,Wang LE,Li D,et al.Genotypes and haplotypes of ERCC1 and ERCC2/XPD genes predict levels of benzo[a]pyrene diol epoxide‐in‐duced DNA adducts in cultured primary lymphocytes from healthy individuals:a genotype‐phenotype correlation analysis[J].Carcino‐genesis,2008,29:1560‐1566.
(2016‐12‐01收稿)
(2017‐03‐07修回)
(编辑:武斌校对:杨红欣)
Genetic polymorphisms of DNA repair genes XPD,XPC,and XRCC4 in relation to colorectal cancer susceptibility
Sha ZHANG1,Ziping CHEN1,Wenjun DU1,Hongchao XIONG2,Changqing XU1
1Department of Gastroenterology,Shandong Provincial Qianfoshan Hospital,Shandong University,Jinan 250014,China;2Department of Thoracic Surgery,Beijing Cancer Hospital,Beijing 100142,China
Ziping CHEN;E‐mail:chenzipingqy@163.com
Objective:To investigate the association of XPD rs13181(codon751A/C,Lys751Gln),rs238406(codon156C/A,Arg156Arg), XPC rs2279017(i11C/A),and XRCC4 rs3734091(codon247T/C,Ala247Ser)polymorphisms with colorectal cancer(CRC)susceptibility. Methods:A total of 338 patients with CRC who were treated at the Beijing Cancer Hospital from April 2013 to January 2016(case group)and 315 healthy controls(control group)were genotyped using TaqMan technology.Results:The genotype GT and G alleles of XPD rs13181 increased the risk of CRC(GT>TT,adjusted OR=1.69,95%CI=1.15‐2.47,P=0.007;G>T,adjusted OR=1.77,95%CI=1.19‐2.64, P=0.005).The genotype GT and T alleles of XRCC4 rs3734091 increased the susceptibility of CRC(GT>GG,adjusted OR=9.02,95%CI= 5.61‐14.50,P<0.001;T>G,adjusted OR=4.06,95%CI=2.49‐6.61,P<0.001).Analyses of XPD rs13181 and rs238406 indicated that the haplotype GT significantly decreased the risk of CRC(adjusted OR=0.39,95%CI=0.18‐0.85,P=0.018).Moreover,the combinations of the XPC rs2279017 G allele and the XRCC4 rs3734091 T allele(adjusted OR=28.43,95%CI=6.85‐117.95,P<0.001)and the XPD rs13181 G allele and the XRCC4 rs3734091 T allele(adjusted OR=10.24,95%CI=4.69‐22.35,P<0.001)exhibited significantly increased CRC risk. Conclusion:The polymorphisms of XPD rs13181 and XRCC4 rs3734091 increased the risk of CRC.
XPD,XPC,XRCC4,single‐nucleotide polymorphism,colorectal cancer,TaqMan technology

10.3969/j.issn.1000-8179.2017.08.385
张莎专业方向为消化系统肿瘤标志物的研究等。E-mail:zhangshagz123@126.com
①山东大学附属千佛山医院消化内科(济南市250014);②北京大学肿瘤医院胸外科*本文课题受山东省科技发展计划项目(编号:2013G0021823)资助
陈自平chenzipingqy@163.com
This work was supported by the Program of Shandong Province Science and Technology Development Plans(No.2013G0021823)

