应用微滴数字PCR技术快速检测食用菌中沙门氏菌
赵 新, 兰青阔, 陈 锐, 朱 珠, 刘 娜, 王 永
(天津市农业科学院 天津市农业质量标准与检测技术研究所,天津300381)
应用微滴数字PCR技术快速检测食用菌中沙门氏菌
赵 新, 兰青阔, 陈 锐, 朱 珠, 刘 娜, 王 永*
(天津市农业科学院 天津市农业质量标准与检测技术研究所,天津300381)
定量风险评估是整个风险评估体系的发展趋势和终极体现形式,而快速、准确、简便的定量技术则是保障整个体系顺利、有效完成的核心支撑。作者根据沙门氏菌invA毒力基因序列,在前人对引物研究结果基础上,合成特异性引物和探针。以沙门氏菌标准菌株为研究对象,建立沙门氏菌微滴数字PCR的快速定量检测方法,并对其特异性、灵敏度、样品实测等关键指标与传统培养法进行对照验证。结果表明,所建立的微滴数字PCR方法具有较好的特异性,检测灵敏度可达到102CFU/mL,样品实测与传统国标法对比,二者符合率100%。所建立的沙门氏菌微滴数字PCR定量检测方法,可以快速、准确、直接地分析沙门氏菌的含量,无需构建质粒和标准曲线,避免了因标准曲线量值偏差而影响样品定值的准确性,所建立的方法为食源性沙门氏菌污染的快速定量检测提供了技术支撑,为微生物风险评估体系的完善奠定了基础。
微滴数字PCR技术;沙门氏菌;定量检测
食用菌是一类有机、营养、保健的绿色食品,为人们广泛食用。质检总局将食品安全的风险监测列入了民生民计的重大解决问题,影响食品安全的因素有很多,而其中由微生物危害导致的食品安全问题达到了总量的40%[1-2]。当前中国在食源性病原菌微生物方面面临着严峻的挑战,急需对潜在危害做出合理的风险评估,将危害控制在可接受水平[3]。而目前微生物定量监测的技术手段主要为传统培养法,费时费力,辅助的实时荧光定量PCR方法,虽在时间上有了很大突破,但其定量结果的准确性要依赖于标准曲线构建的准确性和引物的扩增效率,而微滴数字PCR技术在食源性病原菌上的应用,弥补了这一缺陷。
微滴数字PCR技术作为第三代PCR技术,是在传统PCR方法基础上采用一种全新的方式进行核酸分子的定量,与实时荧光定量PCR相比,无需构建标准曲线,不受扩增效率的影响,其结果具有更高的精确度、准确性和灵敏度。微滴数字PCR的核心是能够将一份样品分成20 000个纳升级的微滴,其中每个微滴或不含待检核酸靶分子,或者含有一个至数个待检核酸靶分子,且每个微滴都作为一个独立的PCR反应器。经PCR扩增后,采用微滴分析仪逐个对每个微滴进行检测,有荧光信号的微滴判读为1,没有荧光信号的微滴判读为0,最终根据泊松分布原理以及阳性微滴的比例,分析软件可计算给出待检靶分子的浓度或拷贝数,以绝对定量的方式直接“数”出靶分子的个数[4]。
沙门氏菌是肠道杆菌中最重要的病原菌,人的沙门氏菌感染和带菌非常普遍。食用生前感染的动物或食品受到污染,均可使人发生食物中毒。在世界各地的食物中毒中,沙门氏菌食物中毒常占首位或第二位。传统培养法是目前常用检测方法,但其检测沙门氏菌的检验程序复杂繁琐、耗时费力,全过程需至少4~7 d,才能得出明确的诊断结果。因而,建立一种准确快速的检测沙门氏菌的方法一直是研究的热点[5],而微滴数字PCR的应用解决了这一难题,并填补了微滴数字PCR技术在食源性病原菌检测上的空白。
研究表明,沙门氏菌的侵袭蛋白(invasion protein,inv)决定细菌进入宿主上皮细胞的能力,与沙门氏菌的致病性密切相关,它是由invA、invB、invC、invD和invE等一组基因编码,其中invA为沙门氏菌的主要毒力因子,因此在前人研究的基础上根据invA毒力基因序列合成特异性引物和TaqMan探针,通过特异性、灵敏度等试验的优化摸索,建立微滴数字PCR沙门氏菌快速定量检测方法,以克服传统致病微生物检测方法的诸多不便,并与传统国家标准方法进行比较,为食源性病原菌检测开辟了新的技术领域。
1 材料与方法
1.1 材料与试剂
乙型副伤寒沙门氏菌CMCC(B)50094、甲型副伤寒沙门氏菌CMCC(B)50433、鼠伤寒沙门氏菌CMCC(B)50013、肠炎沙门氏菌CMCC(B)50041、金黄色葡萄球菌CMCC(B)26003、蜡样芽孢杆菌CMCC(B)63301、枯草芽孢杆菌CMCC(B)63501、大肠埃希氏菌CMCC(B)44102、单核增生李斯特氏菌CMCC(B)54002、痢疾志贺氏菌CMCC(B)51592、铜绿假单胞菌CMCC(B)10104、马红球菌ATCC6939、乙型溶血性链球菌CMCC(B)32210:作者所在实验室保藏标准菌株。
缓冲蛋白胨水(BPW)培养基、生化鉴定试剂盒:北京陆桥有限公司;法国科玛嘉沙门氏菌显色培养基:郑州博赛生物科技有限公司;微滴数字PCR试剂,包括ddPCR Supermix for probes、Droplet Generator Oil:美国 Bio-Rad公司;Chelex-100 sodium:Sigma公司。
1.2 仪器与设备
QX-l00微滴式数字PCR仪:美国Bio-Rad公司;Allegra 21R Centrifuge高速冷冻离心机:美国BECKMAN公司;PCR仪:德国耶拿;制冰机:日本三洋公司;电磁炉:天津中环电炉实验有限公司;全自动高压灭菌锅:日本三洋;生物安全柜、超净工作台、电热恒温培养箱、OLYMPUS CX31显微镜等。
1.3 方法
1.3.1 引物和探针的合成 根据沙门氏菌invA毒力基因序列,在前人研究的基础上[6],合成特异性引物和探针序列,由上海生工生物工程有限公司完成,上游引物5ˊ-CTCACCAGGAGATTACAACATG G-3ˊ,下游引物5ˊ-AGCTCAGACCAAAAGTGAC CATC-3ˊ,荧光探针5ˊ-CACCGACGGCGAGACC GACTTT-3ˊ,探针5ˊ端标记荧光报告基团FAM,3ˊ端标记荧光淬灭基团TAMRA。
1.3.2 传统培养法计数 取5种沙门氏菌标准菌株磁珠,分别接种于缓冲蛋白胨水中,于(36±1)℃培养箱增菌8 h,增菌液进行10倍梯度稀释制成不同浓度的菌悬液,参照传统国标培养法(GB 4789.4-2010)[7]选择适宜的梯度分别计数,并对典型菌落进行生化鉴定和血清学鉴定。
1.3.3 沙门氏菌DNA提取 分别取108~105CFU/mL 4个稀释度菌悬液1 mL离心取沉淀,沉淀中加入100 μL 13%Chelex-100溶液,振荡混匀,100℃水浴10 min后,立即冰浴10 min,13 200 r/min离心3 min,上清液即为DNA溶液,-20℃保存备用。
1.3.4 微滴数字PCR检测体系的建立 在20 μL的PCR反应体系中,包括2×ddPCR Supermix for probes预混液10 μL,上下游引物和探针各1 μL,模板1 μL,无菌超纯水补足20 μL,混匀后离心。将其小心转移至微卡发生器,切忌产生气泡,同时与微卡发生器相应位置加入重油70 μL,盖上垫片,放入微滴生成器中生成微滴。吸取40 μL生成的微滴,小心转移至数字PCR96孔反应板中,170℃热封后在PCR仪上开始循环,PCR仪爬坡速度设置为2℃/s,循环参数为:95℃,10 min;40cycles(94℃,30 s,60℃,1 min);98℃,10 min。
1.3.5 特异性实验 分别选取金黄色葡萄球菌、蜡样芽孢杆菌、枯草芽孢杆菌、大肠埃希氏菌、单核增生李斯特氏菌、痢疾志贺氏菌、铜绿假单胞菌、马红球菌、乙型溶血性链球菌适宜梯度浓度的DNA作为PCR反应的模板,各取1 μL,以乙型副伤寒沙门氏菌、甲型副伤寒沙门氏菌、鼠伤寒沙门氏菌、肠炎沙门氏菌DNA作为阳性对照模板,分别按1.3.4所建立的微滴数字PCR检测体系进行定量分析,以验证所设计引物、探针的特异性。
1.3.6 灵敏度实验 分别选取 106、105、104、103、102、101、100CFU/mL 7个梯度稀释度菌悬液进行DNA提取,各取1 μL作为PCR反应模板,以1.3.4所建立的微滴数字PCR检测体系进行定量分析,确定所建立方法的检测灵敏度。
1.3.7 模拟污染样品的检测 将沙门氏菌制备一定浓度的菌悬液,分别取1 mL加入10 g灭菌的金针菇样品中,制成模拟污染待测样,同时以灭菌金针菇样品作为空白本底对照。于模拟污染待测样中加入90 mL缓冲蛋白胨水,均质后取1 mL稀释液按照1.3.3的方法进行DNA提取,取1 μL作为PCR反应模板,以1.3.5所建立的微滴生成步骤和扩增程序进行数字PCR绝对定量分析,同时与传统国标法相验证。
1.3.8 样品实测 将120份不同食用菌样品(样品来自农业部风险监测项目山东、河南两地的市场抽样汇总)参照传统国标方法分别制备1∶10的稀释液,对稀释液进行DNA提取,采用所建立的微滴数字PCR方法进行检测,同时将样品进一步稀释与传统国标方法进行对照,验证所建立的微滴数字PCR方法检测沙门氏菌的适用性。
2 结果与分析
2.1 传统培养法测定结果
4种沙门氏菌磁珠经复苏活化后,参照GB 4789.4-2010进行传统培养及生化鉴定,对活化菌液浓度进行计数测定,整个过程要7 d,结果见表1。

表1 4种沙门氏菌活化菌液计数结果Table 1 Count results of four Salmonella spp.
2.2 沙门氏菌DNA提取结果
利用简易的Chelex-100法提取的细菌DNA完整性较好,同时Chelex-100中的螯合剂又对杂质具有一定的吸附能力,从而减少杂质对PCR扩增体系的抑制,保证DNA标本初始浓度。对4种沙门氏菌的4种梯度浓度的DNA提取结果见表2。
2.3 沙门氏菌微滴数字PCR分析结果
对4种沙门氏菌的4种浓度梯度进行微滴数字PCR定量检测,同时与传统培养法结果相验证,以确定所建立方法的准确性以及对沙门氏菌的适用性。由于数字PCR最终的拷贝数浓度是由阳性微滴数与总微滴数的泊松分布情况换算得来,因此要求阳性微滴数不能过于接近总微滴数,否则会影响换算结果的可信性。4种沙门氏菌4种浓度梯度的微滴分析结果见表3和图1。我们得出106CFU/mL的菌液浓度是数字PCR最高分析浓度,同时根据DNA提取菌量为1 mL,回溶量为100 μL,PCR总体系20 μL,上样模板1 μL,推算菌液浓度换算公式为CFU/mL=拷贝数/μL×20 μL×100 μL,经计算与传统培养法基本一致。

表2 4种沙门氏菌活化菌液DNA提取结果Table 2 DNA concentration of four Salmonella spp.
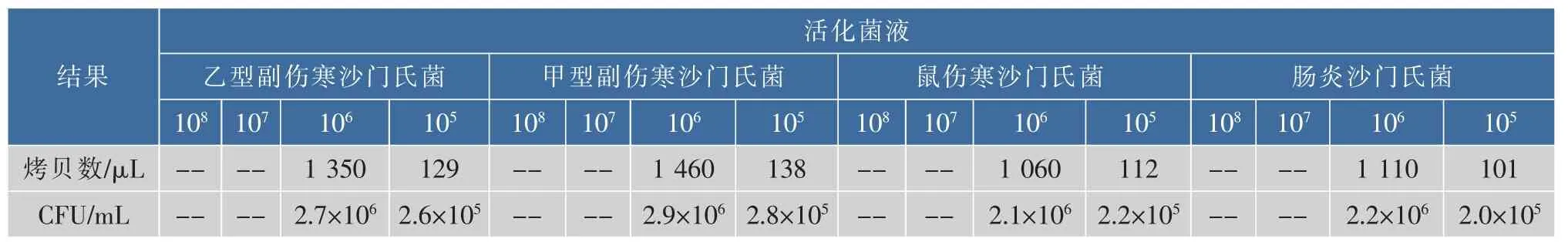
表3 4种沙门氏菌活微滴数字PCR分析结果Table 3 Results of four Salmonella spp.by droplet digital PCR
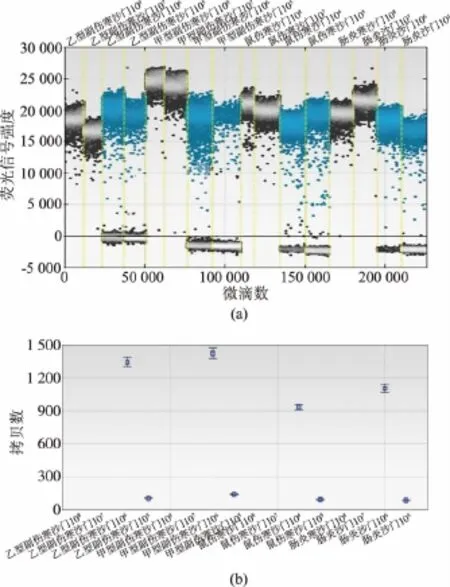
图1 4种沙门氏菌活微滴数字PCR分析结果Fig.1 Results of four Salmonella spp.by droplet digital PCR
2.4 特异性实验结果
以4种沙门氏菌和9种其它细菌制备的基因组 DNA为模板,进行微滴数字PCR分析,结果除沙门氏菌外,其他9种相关细菌均无微滴生成,见图2。证实所建立的方法具有较好的特异性。

图2 方法特异性微滴数字PCR实验结果Fig.2 Specificity results of the method by droplet digital PCR
2.5 灵敏度实验结果
以乙型副伤寒沙门氏菌为例,将其活化菌液作10倍梯度稀释,进行微滴数字PCR定量检测,结果显示为106~102CFU/mL,见表4、图3。均能检测到阳性微滴,表明本方法最低检测限为102CFU/mL菌液浓度。

表4 乙型副伤寒沙门氏菌灵敏度实验结果Table 4 Sensitivity results of Salmonella paratyphi b

图3 乙型副伤寒沙门氏菌灵敏度实验微滴数字PCR分析结果Fig.3 Sensitivity results of Salmonella paratyphi b by droplet digital PCR
2.6 模拟污染样品检测结果
取10份模拟污染沙门氏菌的金针菇样品,分别采用微滴数字PCR方法和传统培养法进行同步检测,结果两种方法均能对金针菇中的沙门氏菌进行有效检出,见表5、图4。二者符合率为100%。
2.7 样品实测结果
应用微滴数字PCR方法和传统培养法分别对120份不同食用菌样品进行对比检测,结果微滴数字PCR方法阳性10份,阳性检出率为8.3%,与传统培养法一致,见表6。结果显示,微滴数字PCR方法和传统培养法均能对食用菌样品中的沙门氏菌进行有效的检出,而微滴数字PCR方法整个实验可在1 d内完成,简便快捷,与传统培养法(7 d)相比时间大为缩短。
3 结语
据全球食源性致病菌最新数据统计,沙门氏菌一直是居高不下的食源性危害之首,对其在食品中的有效监测和控制将成为风险评估长期预警的关键环节[8-10]。随着GB29921-2013《食品中致病菌限量》标准的正式发布实施,预示着对于食品中致病菌的定量检测正在不断的规范和完善,该标准中明确规定了相关产品不同致病菌的定量判定方法。而微滴数字PCR技术开启了新一代PCR技术定量检测食源性致病菌的先河[11-14]。
试验结果证实微滴数字PCR方法对食品中沙门氏菌的检验具有快速、检出率高、方便等优点,且对被检细菌不需要进行纯培养,只要存在于增菌培养基中即可检出,整个实验可在1 d内完成,简便快捷,与传统培养法(7 d)相比时间大为缩短,且准确率高,具有较好的特异性。在120份样品中,微滴数字PCR方法检出10份阳性样品,与传统国标方法一致。

表5 10份模拟污染样品两种方法实测结果Table 5 Comparison of results detected by two methods of ten simulation samples

图4 10份模拟污染样品微滴数字PCR分析结果Fig.4 Results of ten simulation samples by droplet digital PCR
然而,数字PCR也是基于普通常规PCR的原理,因此常规PCR中假阳性问题的存在,导致了数字PCR假阳性的无法避免,因此要注意多方面的控制:如引物设计;引物探针的保存与使用,避免反复冻融;数字PCR微滴操作,防止气泡产生,但气泡造成的假阳性可以用经验排除,其产生的微滴是线性的,非散点式微滴,纵向分布于阳性微滴与阴性微滴的交接处;环境控制,避免污染。
微滴数字PCR方法简单、快捷、灵敏、特异,因此作为一种快速筛查方法用来检测沙门氏菌是完全可行的,符合快速检测的要求,且更适合于大批量应急预案样品的检测。在实际检测中也可以将微滴数字PCR方法和国标方法以及分子生物学方法结合应用,提高样品中病原菌的检出率,但由于数字PCR检测食源性致病菌前期需进行DNA提取,而该技术的可信性浓度范围的最低检出限为1个拷贝数,由于1 mL菌株中的带菌量经提取转化为20 μL上样体系中的拷贝数存在一定的浓度换算,因此要想提高菌株初始浓度的检出率,可在DNA提取步骤进行富集提取,该研究有待于进一步的试验验证。表6 本研究建立的数字PCR方法和传统国标方法的样品检测比对结果
Table 6 Comparison of results detected by real-time PCR and traditional national standard method

产品 本研究建立的数字PCR方法 国标方法GB 4789.4-2010样品数/个 检出数/个 检出率/% 样品数/个 检出数/个 检出率/%金针菇 21 3 14.3 21 3 14.3凤尾菇 36 5 13.9 36 5 13.9鲜香菇 23 0 0 23 0 0鸡腿菇 21 2 9.5 21 2 9.5猴头菇 18 0 0 18 0 0总计 120 10 8.3 120 10 8.3
[1]ROBERT L.Buchanan.Quantitative assessment of the relative risk to public health from foodborne Listeria monocytogenes among selected categories of ready-to-eat foods[J].Centers for Disease Control and Prevention,2003,9:1-27.
[2]HU Jieyun,OU Jie,LIBailin,etal.The role ofpredictive microbiologyin food safetyrisk assessment[J].Institute of Microbiology,2009,36(9):1397-1403.(in Chinese)
[3]WANG Yong,ZHAO Xin,LAN Qingkuo,et al.The microbiological hazard control and risk assessment of food[J].Tianjin Agricultural Sciences,2014,20(5):47-50.(in Chinese)
[4]CAI Yicun,LI Xiang,LV Rong,et al.Quantitative analysis of pork and chicken products by droplet digital PCR[J].BioMed Research International,2014,81:1-27.
[5]ZHU Zhu,WANG Yong,LAN Qingkuo,et al.Application on rapid detection of Salmonellla spp in food by pyrosequencing technology[J].Food Science,2013,34(2):182-186.(in Chinese)
[6]国家质量监督检验检疫总局.SNT 1059.7-2010进出口食品中沙门氏菌检测方法实时荧光PCR法[S].2010.
[7]中华人民共和国卫生部.GB 4789.4-2010食品安全国家标准食品微生物学检验沙门氏菌检验[S].2010.
[8]MAURISCHAT S1,BAUMANN B1,MARTIN A,et al.Rapid detection and specific differentiation of Salmonella enterica subsp. Enteritidis,Typhimurium and its monophasic variant 4,[5],12:i:-by real-time multiplex PCR[J].Int J Food Microbiol,2014,13(193):8-14.
[9]PARK SH1,RICKE S C.Development of multiplex PCR assay for simultaneous detection of Salmonella genus,Salmonella subspecies I,Salm Enteritidis,Salm Heidelberg and Salm Typhimurium[J].J Appl Microbiol,2014,30(13):118-124.
[10]ARNOLD ME1,GOSLING RJ2,MARTELLI F,et al.Evaluation of the sensitivity of faecal sampling for detection of monophasic Salmonella typhimurium and other Salmonella in cattle and pigs[J].Epidemiol Infect,2014,30(5):1-11.
[11]LIU Fangfang,ZHANG Ling,WANG Jing,et al.Quantification of DNA by digital polymerase chain reaction and uncertainty evaluation of determination result[J].Chemical Analysis and Meterage,2013,22(1):18-22.(in Chinese)
[12]PHILIPPE Corbisier,SOM anath Bhat,LINA Partis,et a1.Absolute quantification of genetically modified MON810 maize(Zea mays L.)by digital polymerase chain reaction[J].Anal Bioanal Chem,2009,396(6):2143-2150.(in Chinese)
[13]LI Liang,SUI Zhiwei,WANG Jing,et al.Based on digital PCR single molecule of DNA quantitative techniques is reviewed[J]. Progress in Biochemistry and Biophysics,2012,39(10):1017-1023.(in Chinese)
[14]KELLEY K1,COSMAN A,BELGRADER P,et al.Detection of methicillin-resistant Staphylococcus aureus by a duplex droplet digital PCR assay[J].J Clin Microbiol,2013,51(7):2033-2039.
Rapid Detection of Salmonellla spp.in Edible Fungi by Droplet Digital PCR
ZHAO Xin, LAN Qingkuo, CHEN Rui, ZHU Zhu, LIU Na, WANG Yong*
(Tianjin Institute of Agricultural Quality Standard and Testing Technology,Tianjin Academy of Agricultural Sciences,Tianjin,300192,China)
Quantitative Risk Assessment(QRA)is the future trend of the risk assessment system in which simple,rapid and accurate quantitative technologies made crucial contributions to the smooth and efficient accomplishment of QRA.In this study,a method for detecting the virulence-specific gene,invA in Salmonellla spp.was established by using droplet digital PCR technology.In contrast to the traditional method of national standard(GB 4789.4-2010)for Salmonellla detection,our developed method showed higher specificity and sensitivity as low as 102CFU/mL.The coincidence rate was 100%as compared with simulation specimens with culture strains.The establishment of this method based on droplet digital PCR for Salmonella detection provided a rapid and accurate approach for identification of food-borne pathogens without the use of standard curves or plasmids. These results demonstrated that this developed method offered a promising way for rapid quantitativedetection of Salmonella and provided technical support for the future studies of the microbial risk assessment system.
droplet digital PCR,Salmonellla,quantitative detection
TS 251.5
A
1673—1689(2017)03—0315—07
2015-03-02
天津市应用基础与前沿技术研究计划项目(14JCQNJC14800)。
赵 新(1983—),女,北京人,理学硕士,助理研究员,主要从事农产品分子检测技术方面的研究。E-mail:zhaoxin2008999@126.com
*通信作者:王 永(1977—),男,天津人,理学博士,研究员,主要从事农产品安全质量分子检测技术方面的研究。
E-mail:wyzl2007@yahoo.com.cn
赵新,兰青阔,陈锐,等.应用微滴数字PCR技术快速检测食用菌中沙门氏菌[J].食品与生物技术学报,2017,36(03):315-321.

