蚕豆酱传统酿造过程中细菌菌群的动态变化
蒯 辉, 朱林江, 刘春凤, 李 崎*
(1.江南大学 生物工程学院,江苏 无锡 214122;2.江南大学 食品安全与营养协同创新中心,江苏 无锡 214122)
蚕豆酱传统酿造过程中细菌菌群的动态变化
蒯 辉1,2, 朱林江1,2, 刘春凤1,2, 李 崎*1,2
(1.江南大学 生物工程学院,江苏 无锡 214122;2.江南大学 食品安全与营养协同创新中心,江苏 无锡 214122)
蚕豆酱传统酿造过程中微生物的动态变化对其产品的成熟、风味形成及食品安全具有重要影响。为了有效分析蚕豆酱的传统日光暴晒酿造过程中菌种的动态变化,作者采用菌株专一性的Rep-PCR指纹技术,分析可培养细菌菌种在酿造过程中的定量变化,并分离鉴定优势菌株。结果表明,日光暴晒的传统酿造过程中,好氧细菌总数均在较高水平,为107~108CFU/g,主要为芽孢杆菌属,包括枯草芽孢杆菌(Bacillus subtilis),淀粉液化芽孢杆菌(B.amyloliquefaciens)和甲基营养型芽孢杆菌(B.methylotrophicus)。兼性厌氧菌的数量在103~104CFU/g,主要是是蜡状芽孢杆菌(B.cereus)。发酵前期枯草芽孢杆菌为优势菌,占80%以上,到发酵后期,淀粉液化芽孢杆菌和甲基营养型芽孢杆菌含量增多。所以,该蚕豆酱传统酿造过程中,芽孢杆菌起到主要的作用;此外,蜡状芽孢杆菌含量较多,这与敞开式发酵相关,但与好氧菌相比,其数量保持较低水平,表明其生长受到一定抑制,从而能够保证这一传统发酵食品的安全性。
豆瓣酱;传统发酵;细菌;动态变化;Rep-PCR指纹图谱鉴定技术
传统发酵豆瓣酱是豆瓣、面粉等植物性原料,经过米曲霉接种和天然晒露发酵,再经调配加工而成,其营养丰富、易于消化、风味独特、口感细腻。具有生理功能,如抑制血清胆固醇上升、抑制肝脂肪积蓄、预防肝癌、降血压、去除放射性物质、防止胃溃疡、抗氧化作用等[1-2]。豆瓣酱的传统发酵过程中,有大量的微生物参与代谢,加上光照、温度变化等产生一系列非酶化学反应,这些复杂的生物和非生物反应,给豆瓣酱产品带来独特的风味特色[3-4]。此外,豆瓣酱制作工艺和风味特征具有典型的地域特征。豆瓣酱传统发酵历史虽悠久,但仍保持传统手工作坊方式,生产水平低、发酵周期长(约3个月)、规模化程度低、发酵条件不易控制、产品安全得不到保障等问题[5];同时其传统发酵方式的风味形成机制仍需大量的研究,包括微生物菌群的变化和特征微生物的分离鉴定。
目前国内外对传统发酵豆瓣酱微生物的研究有很多,例如刘福林等[6]研究多种菌种协同作用发酵豆瓣酱,提高原料蛋白质和淀粉的利用率,增强酱的风味。贡汉坤等[7]使用稀释涂布平板的方法从传统发酵的酱中分离了占优势的霉菌、酵母菌和乳酸菌等,其中乳酸菌包括片球菌 (Pediococcus spp.)、植物乳杆菌(Lactobacillus plantarum)。高秀芝等[8]采用变性梯度凝胶电泳(PCR-DGGE)的方法对山东传统发酵豆酱进行研究,发现成品酱中的优势细菌主要包括乳酸乳球菌 (Lactococcus lactis)和地衣芽孢杆菌的近缘种(B.licheniformis)等。Nam[9]等人采用高通量测序的方法研究韩国豆瓣酱doenjang细菌组成,发现韩国中部地区的豆瓣酱中主要的细菌为芽孢杆菌,而韩国其它地区的豆瓣酱中主要的细菌为乳酸菌。Kim等人使用PCR-DGGE的方法研究了日本豆瓣酱中的微生物,发现其中的优势菌为嗜盐四联球菌(Tetragenococcus halophilus)、鸡葡萄球菌 (Staphylococcus gallinarum) 和鲁氏酵母(Zygosaccharomyces rouxii)[10]。然而,对豆瓣酱的传统酿造过程中微生物菌种的定量变化研究较少。
针对豆瓣酱中微生物菌落分析方法主要包括基于培养基的细菌、酵母、霉菌总数分析和基于样品总DNA的PCR-DGGE方法或基于16SrDNA的高通量测序方法[9,11]。基于总DNA的分析方法,能够较全面的分析微生物组成,但难以定量分析,同时微生物的发酵活性难以确定。而基于培养基的分析方法,可实现定量分析,但难以鉴定平板分离的各个菌落,即菌种组成。为快速鉴定各个菌落,可采用菌株专一性的Rep-PCR指纹技术,快速区分各个菌落,从而定量分析菌种的组成,鉴定优势菌株[12-13]。Rep-PCR指纹技术是基于原核及真核生物体基因组中广泛分布着一类短重复序列,因短重复序列在属、种和菌株水平上有分布和拷贝数量的差异性,且具有高度保守性,从而用于对菌株进行分型和同源性分析,能够快速鉴定到菌株水平[14]。为了定量分析豆瓣酱传统酿造过程中菌种变化,作者针对安徽地区一著名蚕豆酱产品酿造过程,采用菌株专一性Rep-PCR技术,选取原核微生物通用引物M13进行指纹扩增[15],分析其酿造过程中各个菌种的定量变化。
1 材料与方法
1.1 材料
1.1.1 材料与试剂 蚕豆酱样品:传统发酵30、40、50、60、70、80 d样品,安徽安庆某酿造食品有限公司;溶菌酶:上海生工生物有限公司;PCR相关试剂、核酸电泳相关试剂、T载体相关试剂:TaKaRa公司;16S rDNA引物对:27F(上游)5'-AGA GTT TGA TCC TGG CTC AG-3’;1541R (下 游 )5’-AAGGAGGTGATCCAGCCGCA-3’。M13-PCR引物:5’-GAGGGTGGCGGTTCT-3’。
1.1.2 仪器 核酸电泳仪:北京市六一仪器厂;紫外照胶系统:美国UVP凝胶成像系统;PCR仪:美国Thermo公司;QYC 2102恒温摇床:上海福玛实验设备有限公司;GSP-9050MBE隔水式恒温培养箱:上海博迅实业有限公司医疗设备厂;HH-4型数显恒温水浴锅:金坛市富华电器有限公司;密封罐和吸氧袋:淮安九和生物科技有限公司。
1.1.3 培养基 好氧细菌的分离培养用LB培养基,厌氧细菌的分离培养用MRS培养基。LB培养基:蛋白胨 10 g,酵母粉 5 g,氯化钠 10 g,琼脂18 g,加水定容至1 000 mL,自然pH值,121℃下灭菌20 min;MRS培养基:牛肉膏10 g,蛋白胨10 g,酵母粉5 g,葡萄糖20 g,乙酸钠5 g,柠檬酸氢二铵2 g,磷酸氢二钾2 g,硫酸镁0.58 g,硫酸锰0.25 g,吐温-80 0.1 g,琼脂18 g,加水定容至1 000 mL,自然pH值,115℃下灭菌15 min。
1.2 实验方法
1.2.1 酱醅样品细菌的平板培养分离 取1 g传统发酵酱醅样品于小三角瓶(内置玻璃珠若干)内,加9 mL无菌水,打散混匀。取4个试管,编号1、2、3、4,各加9 mL无菌水,取1 mL样品稀释液于1号试管内,混匀;从1号试管中取1 mL于2号试管内,混匀,依次稀释至10-5稀释度。各取10-1、10-2、10-3、10-4、10-5的样品稀释液100 μL,分别均匀涂布于LB和MRS培养基平板上,每个稀释度涂两个平板。涂布好后,将LB平板于37℃倒置培养12 h,MRS平板放入密封罐中,迅速放入吸氧袋并盖紧盖子,于32℃倒置培养2~3 d。培养好后,取出平板,选择合适的稀释梯度进行菌落计数,并将平板上的若干单菌落分别接种至相应的新鲜培养基中,好氧细菌接种到透气管中,于摇床中37℃、200 r/min培养12 h,厌氧细菌接种到厌氧管中,于32℃静置培养24~36 h。培养好后各取500 μL菌液加上500 μL 30%的甘油,混匀后-20℃保藏。
1.2.2 酱醅样品中细菌DNA的提取 分离培养的好氧细菌和厌氧细菌培养液用于提取DNA,采用之前报道的十六烷基三甲基溴化铵(CTAB)法[16],其步骤简单介绍如下:收集细胞后用含有800 μL CTAB的 DNA提取缓冲液悬浮细胞,加入 100 μL、50 mg/mL的溶菌酶,37℃水浴至少30 min;加入80 μL 10%SDS和10 μL、20 mg/mL的蛋白酶K,混匀,55℃水浴1 h,期间每隔15 min上下颠倒混匀;取出后12 000 r/min离心5 min,小心吸取中间层于新的2 mL离心管中,加入等体积的酚/氯仿/异戊醇(25∶24∶1)和氯仿/异戊醇(24∶1)依次混匀,12 000 r/min离心,取上清液;加入等体积异丙醇,颠倒混匀,-20℃沉淀1 h;12 000 r/min离心10 min,弃上清液;加入1 mL 70%乙醇,12 000 r/min离心5 min,弃尽上清液,室温干燥10~15 min后用50 μL 1×TE Buffer溶解所提得DNA,供下一步实验或-20℃冷冻保藏。
1.2.3 细菌的 Rep-PCR指纹鉴定及 16S rDNA PCR测序鉴定 将所提基因组作为模板,进行M13-PCR指纹扩增,反应体系25 μL,组分如下:ddH2O 13.9 μL,10×Buffer 2.5 μL,dNTPs 2 μL,M13引物(浓度10 μmol/L)4 μL,rTaq DNA聚合酶 0.6 μL,DNA模板 2 μL。反应程序如下:94℃变性3 min后,42℃退火2 min,72℃延伸3 min,94℃变性45 s开始循环,42℃退火40 s,72℃延伸2 min,35个循环后,72℃延伸10 min,4℃恒温。PCR产物用1 g/dL核酸胶电泳,凝胶成像仪拍照得到菌株M13指纹图谱,将指纹图谱进行分类,随机选取每类指纹中的3株菌株的基因组作为16S rDNA PCR模板,进行PCR。反应体系25 μL,组分如下:ddH2O 17 μL,10×Buffer 2.5 μL,dNTPs 2 μL,27F引物和1541R引物(浓度10 μmol/L)各2 μL,rTaq DNA聚合酶0.5 μL,DNA模板1 μL。反应程序如下:94℃变性3 min后,从94℃变性40 s开始循环,56℃退火35 s,72℃延伸80 s,30个循环后,72℃延伸10 min,4℃恒温。该PCR产物用1 g/dL核酸胶电泳,经凝胶成像仪拍照验证正确后,寄送上海华大基因进行测序,测序结果在NCBI网站上BLAST进行比对,得到鉴定结果。
2 结果与讨论
2.1 好氧细菌和厌氧细菌总数分析
为了分析蚕豆酱样品中好氧菌总数,将传统酿造过程中不同发酵时间 (30、40、50、60、70、80 d)的酱醅样品,稀释涂布平板,在37℃培养12 h,计数结果见图1(a)。结果显示,不同发酵天数的各样品中好氧细菌总数均较高,维持在107~108CFU/g水平。在发酵前期即30 d,细菌总数已达到较高水平;而发酵过程中,细菌总数有一定的波动,这可能与取样时间的天气环境和工人翻捣酱醅的因素相关。
优化比较多种培养基,包括MRS培养基、M17培养基、MC培养基和TJA培养基,最终确定采用MRS培养基分析厌氧菌总数。将酱醅样品稀释涂布平板,在32℃经过2~3 d厌氧培养,选取合适稀释度的平板进行菌落计数,结果见图1(b)。结果显示,不同发酵酱醅中的厌氧菌总数保持相对稳定,在103~104CFU/g水平,约为好氧菌的0.01%水平。

图1 不同发酵天数酱醅好氧细菌和厌氧细菌总数及各菌比例分布Fig.1 Totalnumber of aerobic bacteria and anaerobic bacteria of paste samples from different fermentation stages and their bacteriaproportion
2.2 Rep-PCR指纹快速鉴定不同的菌落
为了有效分析蚕豆酱中细菌菌群的组成,需要对平板分离的菌落进行快速鉴定。首先形态观察表明菌落主要呈两种形态。一种菌落呈现较透明、扁平、光滑、边缘整齐的菌落形态,另一种菌落呈现乳白色、不透明、凸起、边缘不整齐,且经长时间培养后表面会形成明显褶皱的菌落形态。再次,为了快速区分这些菌落,采用菌株专一性的M13-PCR指纹技术。针对每一个样品,从平板上生长的菌落中,随机选取40个菌落进行纯培养和DNA提取;将每一个菌落的基因组进行M13-PCR;通过凝聚电泳得到菌落的M13-PCR指纹图谱。本次研究针对6个样品,每个样品分析40个好氧菌菌落和40个厌氧菌菌落。如图2为70 d样品中部分菌落的PCR指纹,根据这些指纹特征,可区分相似形态的菌落之间在基因型上的差异。
由于M13-PCR指纹的多样性并不复杂,根据其大小和亮度分布判断指纹之间的差异,确定特征指纹。如图2(a)中,17个好氧菌菌落,产生了6个特征指纹,即编号1~6。图2(b)为17个厌氧菌菌落的指纹,包含了5种指纹,即编号为7~10。所以,通过特征指纹的聚类分析,将菌落进行分类,从而减少菌落鉴定的数量。

图2 发酵70 d酱醅好氧细菌和厌氧细菌M13-PCR指纹图谱Fig.2 M3-PCR fingerprints of aerobic bacteria and anaerobic bacteria of 70 d paste sample
2.3 16S rDNA测序鉴定细菌组成
为了鉴定菌株组成,针对每种特征指纹随机挑选3株菌进行16S rDNA测序分析,经NCBI网站上BLAST比对后,鉴定菌种。测序比对结果的相似性均在99%以上。比对结果表明,同属于一种特征指纹的菌株,均被鉴定为同一种菌;而不同特征指纹的菌株,则可能属于同一种菌。所有鉴定的好氧菌主 要 由 3种 菌 组 成 , 即 Bacillus subtilis、B. amyloliquefaciens和B.methylotrophicus;而厌氧菌主要是兼性厌氧的B.cereus和B.drentensis。如表1所示,其中B.subtilis包括了5种指纹,B.amyloliquefaciens包含了4种指纹,B.methylotrophicus包含了2种指纹以及B.cereus的5种指纹。指纹对应的菌株的性能差异,目前在做进一步的分析。
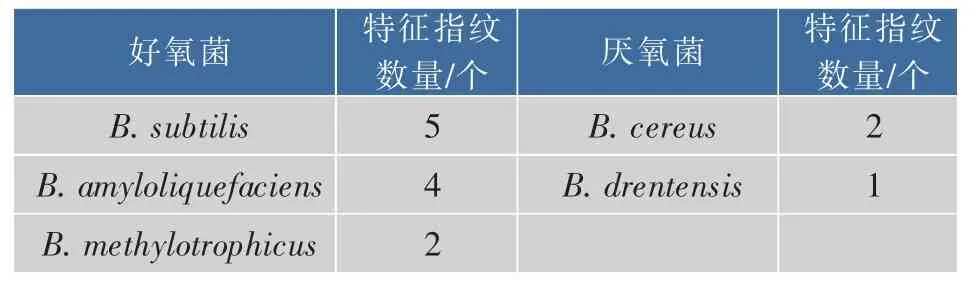
表1 好氧细菌和厌氧细菌M13-PCR特征指纹图谱数量Table 1 M13-PCR fingerprint numbers of aerobic bacteria and anaerobic bacteria
根据以上菌株鉴定结果,分析细菌菌群在蚕豆酱发酵过程中的变化。在发酵的前期和中期,B.subtilis在好氧菌中占有较大比例,都超过了75%;B.amyloliquefaciens和B.methylotrophicus的比例相当,占总菌数的比例约在10%~20%。随着发酵的进行,到发酵后期,B.amyloliquefaciens和B.methylotrophicus的数量开始增多,到发酵成熟时两者占总菌数的比例都均超过了25%。在30 d的样品中,好氧菌菌群中检测了少量的B.cereus。而厌氧菌在蚕豆酱传统发酵过程中变化不大,主要由兼性厌氧的B.cereus组成,只有在50 d的样品中检测到另外一种兼性厌氧菌B.drentensis。
根据以上研究结果,蚕豆酱传统酿造过程中,占优势的可培养细菌菌群并不丰富,主要由芽孢杆菌组成;发酵过程中,细菌菌群的变化主要表现为3种好氧芽孢杆菌比例的变化,其中B.amyloliquefaciens和B.methylotrophicus的比例在发酵后期略有增加,表明部分菌株具备一定的后期生长优势。B.cereus的存在在蚕豆酱这种敞开式发酵过程中是难以避免的,会对食品安全性构成一定的威胁。不过,B.cereus虽被鉴定为优势厌氧菌,但其数量较低,为好氧菌的万分之一,表明B.cereus的生长可能受到抑制。如从一个韩国酱中分离的一株B.subtilis HJ18-4,其生长能够有效抑制病原性的B.cereus生长[17]。所以,该蚕豆酱中,B.cereus保持在较低水平的原因有待进一步分析。另外,一些豆瓣酱中,分离的厌氧菌包括一些乳酸菌,但该蚕豆酱则不含乳酸菌。这可能与好氧芽孢杆菌的大量存在相关,或者是乳酸菌的生长在蚕豆酱发酵过程受到抑制。
3 结语
作者分析了安庆某蚕豆酱传统发酵过程中细菌菌群的动态变化。应用菌株专一性的Rep-PCR指纹技术,能够快速区分平板分离的细菌菌落,从而定量分析优势菌群的菌种组成。通过样品进行指纹图谱分类,对菌落进行快速分类,选取不同特征指纹类别的菌株进行16S rDNA测序鉴定,极大节约了时间和工作量,同时也减少了研究成本,表明Rep-PCR指纹技术可高效的定量分析混合微生物发酵过程菌种的变化。对蚕豆酱的优势菌群鉴定结果表明,发酵过程的优势菌群为芽孢杆菌,不同发酵阶段菌群变化主要表现为3种芽孢杆菌比例之间的变化。所以,该蚕豆酱的成熟应与芽孢杆菌的大量生长及发酵相关,进一步研究这三类菌在蚕豆酱发酵过程中的代谢和互作,对揭示豆瓣酱发酵的成熟和风味物质的形成机理有重要的作用。此外,该蚕豆酱酿造过程中,厌氧菌数量少,种类单一,不含乳酸菌,而且前期研究使用其它乳酸菌培养基包括M17培养基、MC培养基、TJA培养基等,均未能在各样品中检出乳酸菌。这种传统发酵的酱醅中不含乳酸菌的现象较少,表明乳酸菌生长受到一定抑制,可能是因为芽孢杆菌的大量生长,或者是因为周围环境和生产工艺 (比如原料、盐浓度和翻酱)等,这方面需要进一步研究。这一研究结果也证明酱中浓郁香味的形成,不一定需要乳酸菌参与,应与日光暴晒过程的物理化学反应更密切相关。
[1]BAO Qian.The function of bean paste[J].China Brewing,2002,3(119):1-6.(in Chinese)
[2]VILLARES A,ROSTAGNO M A,GARCIA L A,et al.Content and profile of isoflavones in soy-based foods as a function of the production process[J].Food and Bioprocess Technology,2010,4(1):27-38.
[3]SUN Changyan,MA Ying,LI Dehai,et al.The proteases formation and proteolysis during the incubation of naturally fermented soybean Koji[J].Food Technology,2007(8):188-192.(in Chinese)
[4]GAO Ling.Production process improvement of PI country douban[J].China Condiment,2006(5):34-39.(in Chinese)
[5]MELDRUM R J,LITTLE C L,SAGOO S,et al.Assessment of the microbiological safety of salad vegetables and sauces from kebab take-away restaurants in the United Kingdom[J].Food Microbiol,2009,26(6):573-577.
[6]LIU Fulin,YANG Wenxia,ZHU Lili,et al.The development of multi-strain fermentation bean sauce[J].China Brewing,1999(1):11-14.(in Chinese)
[7]GONG Hankun,YAO Qinghai.The microbiology analysis of spontaneously fermentation for traditional salted and fermented soybean paste[J].China Condiment,2003(10):9-12.(in Chinese)
[8]GAO Xiuzhi,WANG Xiaofen,LI Xianmei,et al.Changes of nutrition and bacterial diversity during traditional soypaste fermentation[J].Microbiology,2008,35(5):748-753.(in Chinese)
[9]NAM Y D,LEE S Y,LIM S I.Microbial community analysis of Korean soybean pastes by next-generation sequencing[J].Int J Food Microbiol,2012,155(1-2):36-42.
[10]KIM T,LEE J H,PARK M H,et al.Analysis of bacterial and fungal communities in Japanese-and Chinese-fermented soybean pastes using nested PCR-DGGE[J].Current Microbiology,2010,60(5):315-320.
[11]TANAKA Y,WATANABE J,MOGI Y.Monitoring of the microbial communities involved in the soy sauce manufacturing process by PCR-denaturing gradient gel electrophoresis[J].Food Microbiology,2012,31(1):1-7.
[12]ZHU Linjiang,ZHENG Feiyun,ZHAO Yazhou,et al.Study on application of rep-PCR fingerprint in rapid identification of beer-spoilager[J].Chinese Journal of Biotechnology,2006,22(6):1013-1020.(in Chinese)
[13]PASANEN T,KOSKEA S,MERO S,et al.Rapid molecular characterization of Acinetobacter baumannii clones with rep-PCR and evaluation of carbapenemase genes by new multiplex PCR in hospital district of helsinki and uusimaa[J].PLoS ONE,2014,9(1):1-8.
[14]MATSUMOTO M.Distribution analysis of population structures for Rhizoctonia solani AG-1 IA in Japanese paddy field,using rep-PCR assay[J].Archives of Phytopathology and Plant Protection,2013,47(9):1082-1088.
[15]DANESI P,FIRACATIVE C,COGLIATI M,et al.Multilocus sequence typing (MLST)and M13 PCR fingerprinting revealed heterogeneity amongst Cryptococcus species obtained from Italian veterinary isolates[J].FEMS Yeast Res,2014,14(6):897-909.
[16]ZHU L,XU H,ZHANG Y,et al.BOX-PCR and PCR-DGGE analysis for bacterial diversity of a naturally fermented functional food(Enzyme®)[J].Food Bioscience,2014(5):115-122.
[17]EOM J S,LEE S Y,CHOI H S.Bacillus subtilis HJ18-4 from traditional fermented soybean food inhibits Bacillus cereus growth and toxin-related genes[J].Journal of Food Science,2014,79(11):2279-2287.
Monitoring the Change in Bacterial Community during Traditional Fermentation of Broad Bean Sauce
KUAI Hui1,2, ZHU Linjiang1,2, LIU Chunfeng1,2, LI Qi*1,2
(1.School of Biotechnology,Jiangnan University,Wuxi 214122,China;2.Synergetic Innovation Center of Food Safety and Nutrition,Jiangnan University,Wuxi 214122,China)
Dynamic change of microorganismsin the process oftraditional fermentation of broad bean saucehas an important effecton the maturity,flavor formation and food safety of the product.To quantitatively analyze the dynamic changes of bacterial species during traditionalsunshineexposedfermentation of broad bean sauce,strain-specific Rep-PCR fingerprinttechnology was adopted.The dominant strains were identified.Results indicated that during the traditional fermentation process,the total number of aerobic bacteria remained at a higher level(107~108CFU/g),mainly including Bacillus subtilis,B.amyloliquefaciens and B.methylotrophicus.The total number of facultative anaerobe was 103~104CFU/g and composed of B.cereus.At the early stage ofthe fermentation,B.subtiliswas the dominant bacteria and accounted for more than 80%,while the content of B.amyloliquefaciens and B.methylotrophicus increasedat the later period of fermentation. Therefore,Bacilli played a major role in the traditional fermentation of the broad bean sauce.The identified B.cereuswas related to the open fermentation mode.By comparison to aerobic bacteria,The number of B.cereuswaskept at a low level,which guarantees the safety of the traditionally fermented food.
broad bean sauce,traditionalfermentation,bacteria,dynamic change,Rep-PCR fingerprintidentificationtechnology
TS 264
A
1673—1689(2017)03—0271—06
2015-01-22
国家863计划项目(2013AA102106);中央高校基本科研业务费项目(JUSRP51402A,JUDCF13008);江苏高校优势学科建设工程项目;江苏省111引智计划项目(111-2-06)。
*通信作者:李 崎(1971—),女,上海人,工学博士,教授,博士研究生导师,主要从事啤酒酿造及传统酿造方面的研究。
E-mail:liqi@jiangnan.edu.cn
蒯辉,朱林江,刘春凤,等.蚕豆酱传统酿造过程中细菌菌群的动态变化[J].食品与生物技术学报,2017,36(03):271-276.

