CRISPR/Cas9基因编辑技术在致瘤病毒感染研究中的应用
王玉,徐维祯,钟照华
哈尔滨医科大学微生物学教研室及伍连德研究所,哈尔滨 150081
·综述·
CRISPR/Cas9基因编辑技术在致瘤病毒感染研究中的应用
王玉,徐维祯,钟照华
哈尔滨医科大学微生物学教研室及伍连德研究所,哈尔滨 150081
成簇的规律间隔的短回文重复序列及其相关蛋白9〔clustered regularly interspaced short palindromic repeat (CRISPR)/CRISPR-associated protein 9 (Cas9),CRISPR/Cas9〕基因编辑技术的发现源于真细菌和古细菌中CRISPR/Cas系统介导的适应性免疫机制研究。该技术利用特异性向导RNA识别靶点基因,引导核酸内切酶Cas9对其切割,并通过同源重组或非同源末端连接完成对目的DNA的编辑。某些病毒感染机体后,可将其基因组整合到宿主细胞基因组中或潜伏于组织中而无法被彻底清除,从而引起持续性感染。本文参考2013年以来CRISPR/Cas9基因组编辑技术的最新相关研究报道,重点综述其在人类免疫缺陷病毒1型(human immunodeficiency virus type 1,HIV-1)、人乳头瘤病毒(human papillomavirus,HPV )、乙型肝炎病毒(hepatitis B virus, HBV)、 Epstein-Barr病毒(Epstein-Barr virus,EBV)等致瘤病毒感染相关疾病研究中的应用,并概括其作用于这些病毒的有效靶点。
成簇的规律间隔的短回文重复序列及其相关蛋白9;基因编辑;病毒
致瘤病毒感染相关疾病传染性强,危害大。如人类免疫缺陷病毒(human immunodeficiency virus,HIV)虽不直接引发肿瘤,但HIV感染者易并发卡波西肉瘤(Kaposi’s sarcoma,KS);乙型肝炎病毒(hepatitis B virus, HBV)与肝细胞肝癌的发生密切相关;人乳头瘤病毒(human papillomavirus,HPV) 可引起宫颈癌等鳞状上皮细胞癌;Epstein-Barr病毒(Epstein-Barr virus,EBV)感染机体后可引起伯基特淋巴瘤(Burkitt lymphoma)和鼻咽癌。这些病毒危害性大,但目前尚无有效的治疗方法。近年来,人们开发出一种新型基因编辑技术,称为成簇的规律间隔的短回文重复序列及其相关蛋白9〔clustered regularly interspaced short palindromic repeat (CRISPR)/CRISPR-associated protein 9 (Cas9),CRISPR/Cas9〕。该技术源于细菌获得性免疫系统,可切割外源性DNA[1]。基于CRISPR/Cas9系统的基因编辑技术因简单、高效、价廉、毒性小等优点而成为新一代基因组定向编辑的有力工具。本文主要综述CRISPR/Cas9基因编辑技术在致瘤病毒HIV、HPV、HBV和EBV研究中的应用;在其他致瘤病毒如卡波希肉瘤相关疱疹病毒(Kaposi’s sarcoma-associated herpesvirus, KSHV)、人类嗜T细胞病毒(human T cell lymphotropic virus,HTLV)、丙型肝炎病毒(hepatitis C virus,HCV)和传染性软疣病毒(molluscum contagiosum virus,MCV)研究中尚未见其应用。
1 CRISPR/Cas9基因编辑技术概述
CRISPR/Cas9源于细菌的Ⅱ型CRISPR/Cas系统。外源噬菌体或质粒入侵细菌后,细菌通过识别外源DNA的前间区序列邻近基序(protospacer adjacent motif,PAM),将外源DNA加工成间隔序列并插入CRISPR序列中。CRISPR转录产生前体crRNA(pre-CRISPR RNA,pre-crRNA)与反式激活crRNA(trans-activating crRNA,tracrRNA)配对,在RNase Ⅲ作用下加工形成成熟的crRNA,并与Cas9蛋白形成复合物。复合物中的crRNA通过识别位于PAM 5′端互补的DNA序列,与基因组稳定结合,并对目标DNA进行切割,导致基因组双链断裂。
2012年,Jinek等[2]根据crRNA∶tracrRNA复合体的结构特征,人工合成crRNA序列,被称为小向导RNA(small guide RNA,sgRNA)。sgRNA可被Cas9识别,形成sgRNA-Cas9复合物,并引导其结合于目标DNA。CRISPR/Cas9基因编辑技术可通过设计sgRNA,实现对含有PAM序列的任一目标DNA进行敲除、插入或定点突变(图1);同时具有易于构建、打靶效率高、应用范围广并可进行多基因组编辑等优点。自此,CRISPR/Cas9基因编辑技术广泛用于人类细胞及其他生物体研究[3-6]。
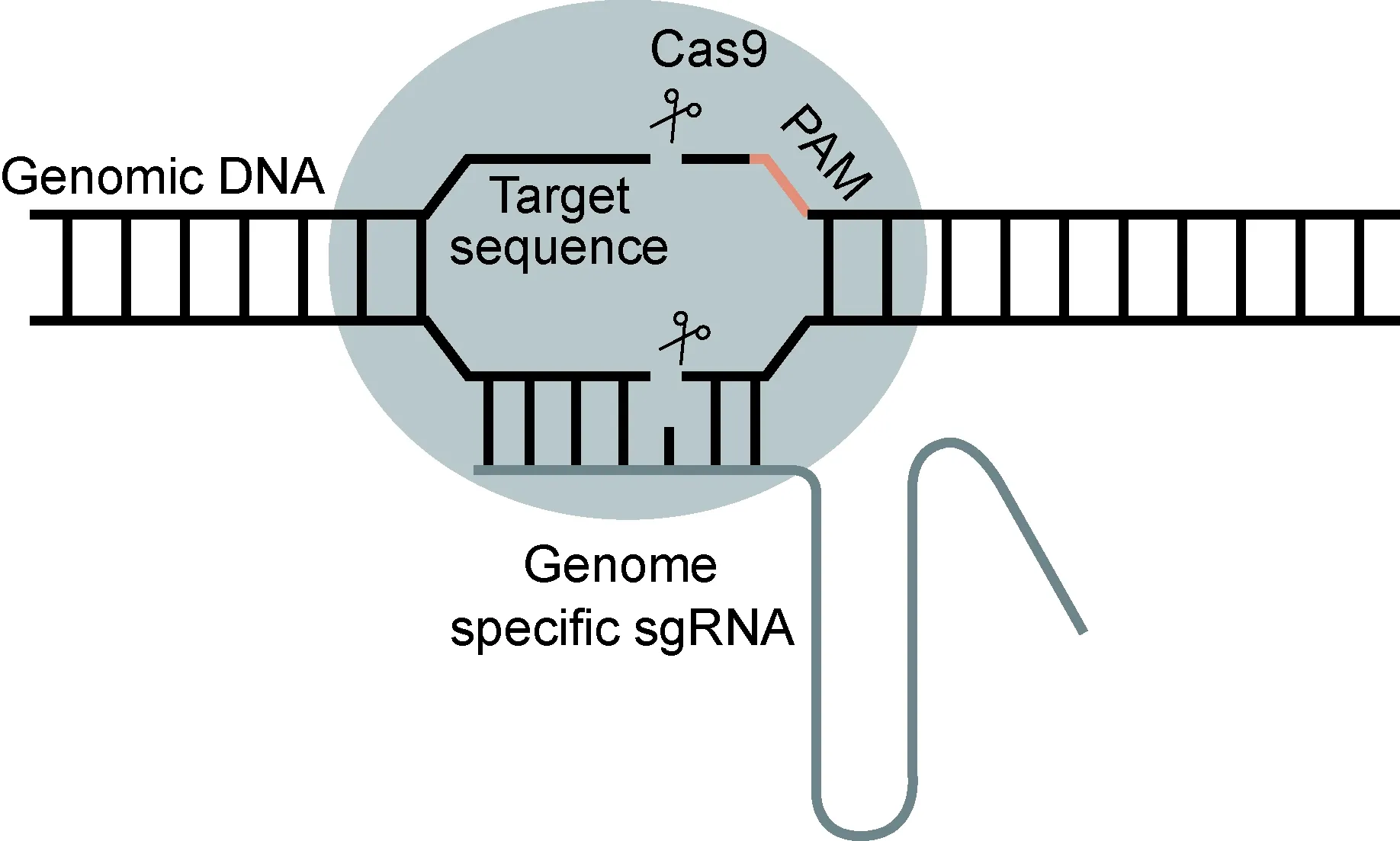
图1 sgRNA-Cas9复合物介导的靶向基因组编辑
Fig.1 Targeted genome editing by sgRNA-Cas9 complex
2 CRISPR/Cas9基因编辑技术在持续性病毒感染研究中的应用
病毒持续性感染一直困扰着人类。尽管一般的抗病毒治疗能抑制病毒复制,但不能彻底清除宿主体内的病毒。因此,对许多病毒持续性感染缺乏有效治疗手段。CRISPR/Cas9基因编辑技术的快速发展为抗病毒持续性感染研究提供了新的工具,目前主要应用于HIV、HPV、HBV和EBV的持续性感染研究(表1)。
2.1 HIV-1
HIV分为HIV-1型和HIV-2型。HIV-1型是引起艾滋病的主要病原体。目前,CRISPR/Cas9基因编辑技术应用于HIV-1研究,主要是通过靶向ccr5阻止HIV-1对CD4+T细胞侵染,以及通过靶向HIV-1前病毒终止其持续性感染。
HIV-1通过与细胞表面CD4+受体和CCR5/CXCR4共受体结合而侵染CD4+T细胞[7]。2014年,Wang等[8]构建3个靶向ccr5的sgRNA/Cas9慢病毒载体,并将其转导入HIV-1易感人类CD4+细胞中。流式细胞仪分析显示,绿色荧光蛋白(green fluorescent protein,GFP)阳性细胞比例从86.8%分别降至81.3%、78.2%和77.5%。2015年,Hou等[9]证明利用CRISPR/Cas9基因编辑技术靶向人类cxcr4基因可使CD4+T细胞抵御HIV-1入侵。
表1 已知CRISPR/Cas9抗病毒作用靶点
Tab.1 Known antiviral targets for CRISPR/Cas9

VirusTargetNucleotidepositionSequence(5'-3')ReferenceHIV-1LTRTAR171-193GTTAGACCAGATCTGAGCCTGGGEbina,etalCCR55889-5911GCTTGTGACACGGACTCAAGTGGWang,etal,20145446-5468GGTCCTGCCGCTGCTTGTCATGG5831-5853TAAACTGAGCTTGCTCGCTCGGGLTRU3162-184ATCAGATATCCACTGACCTTTGGHu,etal,20149291-9313CCGGAGTACTTCAAGAACTGCTGCXCR44753-4775GCTTCTACCCCAATGACTTGTGGHou,etal,20154784-4806GTTCCAGTTTCAGCACATCATGGHPV18E6/E7109-131CGCGCTTTGAGGATCCAACACGGKennedy,etal,2014608-630CGAGCAATTAAGCGACTCAGAGGHPV16E6/E77065-7084CCGACTAAGGGCGTAACCGAAATZhen,etal,20147247-7266CCGCAACAGTTACTGCGACGTG7727-7747CCGTCCGGTTCTGCTTGTCCAGCHPV16E77589-7611AACCCAGCTGTAATCATGCATGGHu,etal,20147619-7641CCTACATTGCATGAATATATGTT7652-7674CCAGAGACAACTGATCTCTACTG7724-7746CCAGCTGGACAAGCAGAACCGGAHPV6/11E7485-507CCCTGTAGGGTTACATTGCTATGLiu,etal,2016518-540AGACAGCTCAGAAGATGAGGTGGHBVP11292-1314GTTTTGCTCGCAGCCGGTCTGGGLin,etal,2014XCp1742-1764GGGGGAGGAGATTAGGTTAAAGGRT729-750TTTCAGTTATATGGATGATGTGGKennedy,etal,2015ENII-CP/X3006-2987CATTCGGTGGGCGTTCACGGSeegerandSohn,2014HBsAg354-376CAACTTGTCCTGGTTATCGCTGGZhen,etal,2015HBx4034-4053CCTCTGCACGTCGCATGGAGCCP234-256TACCGCAGAGTCTAGACTCGTGGLiu,etal,20156465-6487CCCTCGCCTCGCAGACGAAGGTCX1523-1542GGGGCGCACCTCTCTTTACGCGGDong,etal,20151681-1700AATGTCAACGACCGACCTTGAGGC2336-2355ACTACTGTTGTTAGACGACGWang,etal,2015EBVBART137842-137863TAATTGCAGTGGACCCCGGAGGYuen,etal,2015138397-138418AAGAAGCTCCTCAGCAACATGG
HIV-1前病毒可整合至宿主DNA并随之复制,这是HIV-1持续性感染的主要原因。其长末端重复序列(long terminal repeat,LTR)区含有顺式调控序列,可控制前病毒基因表达。因此,有研究通过CRISPR/Cas9技术靶向前病毒LTR区,流式细胞仪分析显示GFP阳性细胞比例从97.8%降至35.5%[10]。2014年,又有研究证实CRISPR/Cas9技术能高效靶向HIV-1前病毒LTR区U3启动子,从而使小神经胶质细胞和T细胞系中的HIV-1失活[11]。以上研究表明,CRISPR/Cas9技术可能成为抗HIV持续性感染的有力工具。
2.2 HPV
E6和E7是HPV的主要致癌基因。E6可与肿瘤抑制蛋白p53结合使其降解,E7则与肿瘤抑制蛋白pRb结合使其失活,引起细胞恶性增生。
2014年,Hu等[12]和Kennedy等[13]先后证明利用CRISPR/Cas9技术靶向E6或E7基因可诱导HPV基因组切割及E6、E7基因突变,进而导致肿瘤细胞周期阻滞并发生细胞凋亡。同年,Zhen等[14]将构建的靶向E6和E7的sgRNA和Cas9转染入人子宫颈鳞癌细胞SiHa,注射到小鼠体内,发现实验组小鼠的肿瘤体积显著减小,体重也显著低于对照组。2015年,Liu等[15]构建了针对HPV6/11靶向E7基因的CRISPR/Cas9系统,将其转染至能稳定表达HPV6E7或HPV11E7的角质细胞中,48 h后蛋白免疫印迹检测发现E7表达量减少,并引起细胞凋亡。以上研究显示,CRISPR/Cas9技术可通过靶向两种关键的原癌基因E6和E7,从而达到抑制细胞恶性增生的目的,使其成为研究治疗HPV所致宫颈癌的潜在工具。
2.3 HBV
HBV基因组包含4个开放读码框:S基因区、C基因区、P基因区和X基因区。这4个基因区分别负责编码病毒的结构蛋白和功能蛋白。理论上,靶向破坏任何一个基因区均可有效抑制病毒蛋白的表达和复制。
Lin等[16]首次构建了靶向HBV基因组的sgRNA/Cas9质粒,并将其与HBV表达质粒共转染至Huh7细胞中,结果发现转染了sgRNA/Cas9的细胞上清液中HBV表面抗原(HBV surface antigen,HBsAg)明显减少。随后,他们将这些质粒与HBV表达质粒共同注射入小鼠尾静脉,小鼠血清HBsAg水平和肝内HBV表达水平均明显下降。Kennedy等[17]利用慢病毒载体,将靶向HBV的sgRNA/Cas9转导入慢性HBV感染模型(HepAD38 细胞)和再次感染模型(HepaRG 细胞)中,发现HBV DNA复制和共价闭合环状DNA(covalently closed circular DNA,cccDNA)积聚被有效抑制。Seeger和Sohn也证明,靶向HBV的sgRNA可高效抑制HBV感染[18]。在另一项研究中,Liu等[19]提出靶向HBV的sgRNA/Cas9 能造成HBV DNA模板的突变和清除,体内和体外HBV复制减少,且适用于不同的HBV基因型。Zhen等[20]将靶向HBV S基因区的sgRNA/Cas9用于HepG2.2.15细胞和小鼠模型,发现实验组细胞培养上清液及小鼠血清中HBsAg表达明显降低。Dong等[21]证明,利用CRISPR/Cas9技术能在肝细胞及携带HBV cccDNA的一种新型小鼠模型中减少HBV cccDNA的复制。Ramanan等[22]设计了一系列靶向HBV保守序列的sgRNA,能在体内和体外实验中高效抑制病毒复制。Wang等[23]利用双gRNA使细胞培养上清液中的HBsAg、HBV e抗原(HBV e antigen,HBeAg)、HBV DNA和cccDNA均减少,且适用于HBV A~D基因型。以上研究表明,CRISPR/Cas9技术可在体内外实验中靶向破坏HBV cccDNA,有望在未来成为治疗HBV持续性感染的新策略。
2.4 EBV
EBV多表现为持续性感染,主要发生于病毒潜伏期。此阶段病毒基因组以游离附加体形式存在于宿主细胞中,迄今还没有任何治疗方法能将其完全清除。
2014年,有研究者构建了7个靶向EBV基因组的sgRNA/Cas9质粒,并将其转染入EBV慢性感染细胞(Raji细胞)。结果发现,EBV基因组的靶位点发生突变,同时转染了sgRNA/Cas9的细胞增殖效率降低。定量聚合酶链反应(polymerase chain reaction,PCR)显示,在sgRNA/Cas9转染细胞中,25%细胞中检测不到EBV基因组,50%细胞中EBV基因组拷贝数明显减少[24]。
2015年,有研究利用两个gRNA直接靶向删除人类EBV持续性感染上皮细胞中负责编码病毒微小RNA(microRNA,miRNA)BART(BamHI A rightward transcript)区启动子的558 bp序列,导致病毒不能表达BART miRNA,表明利用CRISPR/Cas9技术可有效靶向编辑EBV基因组[25]。
3 结语
CRISPR/Cas9作为一种新型基因编辑技术,具有简单、精确、经济等优点,但其仍存在脱靶问题。目前,减少CRISPR/Cas9技术脱靶的措施主要有3种:①使Cas9蛋白的HNH或RuvC核酸酶结构域失活,产生功能缺陷的Cas9(D10A),这种Cas9只能产生DNA单链断裂[26]。有研究者在靶位点设计了两条sgRNA,分别与正负链配对,使突变型Cas9对两条链分别切割,引发特定位点的双链断裂,实验证明该方法提高了CRISPR/Cas9的特异性[27]。②通过截短sgRNA长度,提高其识别靶序列的特异性[28]。③将无活性的Cas9蛋白与FokⅠ内切酶融合[29],构建sgRNA引导的FokⅠ基因编辑技术。以上3种方法均能有效降低CRISPR/Cas9技术的脱靶率。
虽然CRISPR/Cas9技术在应用中仍有许多需要解决的问题,但与其他基因编辑技术相比,其更简单、廉价、快速、灵活。最近,有报道称CRISPR/Cas9技术不仅可对DNA进行编辑,还可对RNA进行编辑[30]。总之,CRISPR/Cas9技术作为一种新型基因编辑工具,在病毒持续性感染研究中具有巨大潜能。
[1] Horvath P, Barrangou R. CRISPR/Cas, the immune system of bacteria and archaea [J]. Science, 2010, 327(5962): 167-170.
[2] Jinek M, Chylinski K, Fonfara I, Hauer M, Doudna JA, Charpentier E. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity [J]. Science, 2012, 337(6096): 816-821.
[3] Jiang W, Bikard D, Cox D, Zhang F, Marraffini LA. RNA-guided editing of bacterial genomes using CRISPR-Cas systems [J]. Nat Biotechnol, 2013, 31(3): 233-239.
[4] Shan Q, Wang Y, Li J, Zhang Y, Chen K, Liang Z, Zhang K, Liu J, Xi JJ, Qiu JL, Gao C. Targeted genome modification of crop plants using a CRISPR-Cas system [J]. Nat Biotechnol, 2013, 31(8): 686-688.
[5] Hwang WY, Fu Y, Reyon D, Maeder ML, Tsai SQ, Sander JD, Peterson RT, Yeh JR, Joung JK. Efficient genome editing in zebrafish using a CRISPR-Cas system [J]. Nat Biotechnol, 2013, 31(3): 227-229.
[6] Li D, Qiu Z, Shao Y, Chen Y, Guan Y, Liu M, Li Y, Gao N, Wang L, Lu X, Zhao Y, Liu M. Heritable gene targeting in the mouse and rat using a CRISPR-Cas system [J]. Nat Biotechnol, 2013, 31(8): 681-683.
[7] Cocchi F, DeVico AL, Garzino-Demo A, Arya SK, Gallo RC, Lusso P. Identification of RANTES, MIP-1α, and MIP-1β as the major HIV-suppressive factors produced by CD8+T cells [J]. Science, 1995, 270(5243): 1811-1815.
[8] Wang W, Ye C, Liu J, Zhang D, Kimata JT, Zhou P. CCR5 gene disruption via lentiviral vectors expressing Cas9 and single guided RNA renders cells resistant to HIV-1 infection [J]. PLoS One, 2014, 9(12): e115987.
[9] Hou P, Chen S, Wang S, Yu X, Chen Y, Jiang M, Zhuang K, Ho W, Hou W, Huang J, Guo D. Genome editing of CXCR4 by CRISPR/Cas9 confers cells resistant to HIV-1 infection [J]. Sci Rep, 2015, 5: 15577.
[10] Ebina H, Misawa N, Kanemura Y, Koyanagi Y. Harnessing the CRISPR/Cas9 system to disrupt latent HIV-1 provirus [J]. Sci Rep, 2013, 3: 2510.
[11] Hu W, Kaminski R, Yang F, Zhang Y, Cosentino L, Li F, Luo B, Alvarez-Carbonell D, Garcia-Mesa Y, Karn J, Mo X, Khalili K.RNA-directed gene editing specifically eradicates latent and prevents new HIV-1 infection [J]. Proc Natl Acad Sci USA, 2014, 111(31): 11461-11466.
[12] Hu Z, Yu L, Zhu D, Ding W, Wang X, Zhang C, Wang L, Jiang X, Shen H, He D, Li K, Xi L, Ma D, Wang H. Disruption of HPV16-E7 by CRISPR/Cas system induces apoptosis and growth inhibition in HPV16 positive human cervical cancer cells [J]. Biomed Res Int, 2014. doi:10.1155/20141612823.
[13] Kennedy EM, Kornepati AV, Goldstein M, Bogerd HP, Poling BC, Whisnant AW, Kastan MB, Cullen BR. Inactivation of the human papillomavirus E6 or E7 gene in cervical carcinoma cells by using a bacterial CRISPR/Cas RNA-guided endonuclease [J]. J Virol, 2014, 88(20): 11965-11972.
[14] Zhen S, Hua L, Takahashi Y, Narita S, Liu YH, Li Y. In vitro and in vivo growth suppression of human papillomavirus 16-positive cervical cancer cells by CRISPR/Cas9 [J]. Biochem Biophys Res Commun, 2014, 450(4): 1422-1426.
[15] Liu YC, Cai ZM, Zhang XJ. Reprogrammed CRISPR-Cas9 targeting the conserved regions of HPV6/11 E7 genes inhibits proliferation and induces apoptosis in E7-transformed keratinocytes [J]. Asian J Androl, 2016, 18(3):475-479.
[16] Lin SR, Yang HC, Kuo YT, Liu CJ, Yang TY, Sung KC, Lin YY, Wang HY, Wang CC, Shen YC, Wu FY, Kao JH, Chen DS, Chen PJ. The CRISPR/Cas9 system facilitates clearance of the intrahepatic HBV templates in vivo [J]. Mol Ther Nucleic Acids, 2014, 3(10): e186.
[17] Kennedy EM, Bassit LC, Mueller H, Kornepati AV, Bogerd HP, Nie T, Chatterjee P, Javanbakht H, Schinazi RF, Cullen BR. Suppression of hepatitis B virus DNA accumulation in chronically infected cells using a bacterial CRISPR/Cas RNA-guided DNA endonuclease [J]. Virology, 2015, 476: 196-205.
[18] Seeger C, Sohn JA. Targeting hepatitis B virus with CRISPR/Cas9 [J]. Mol Ther Nucleic Acids, 2014, 3(12): e216.
[19] Liu X, Hao R, Chen S, Guo D, Chen Y. Inhibition of hepatitis B virus by the CRISPR/Cas9 system via targeting the conserved regions of the viral genome [J]. J Gen Virol, 2015, 96(8): 2252-2261.
[20] Zhen S, Hua L, Liu YH, Gao LC, Fu J, Wan DY, Dong LH, Song HF, Gao X. Harnessing the clustered regularly interspaced short palindromic repeat (CRISPR)/CRISPR-associated Cas9 system to disrupt the hepatitis B virus [J]. Gene Ther, 2015, 22(5): 404-412.
[21] Dong C, Qu L, Wang H, Wei L, Dong Y, Xiong S. Targeting hepatitis B virus cccDNA by CRISPR/Cas9 nuclease efficiently inhibits viral replication [J]. Antiv Res, 2015, 118: 110-117.
[22] Ramanan V, Shlomai A, Cox DB, Schwartz RE, Michailidis E, Bhatta A, Scott DA, Zhang F, Rice CM, Bhatia SN. CRISPR/Cas9 cleavage of viral DNA efficiently suppresses hepatitis B virus [J]. Sci Rep, 2015, 5: 10833.
[23] Wang J, Xu ZW, Liu S, Zhang RY, Ding SL, Xie XM, Long L, Chen XM, Zhuang H, Lu FM. Dual gRNAs guided CRISPR/Cas9 system inhibits hepatitis B virus replication [J]. World J Gastroenterol, 2015, 21(32): 9554-9565.
[24] Wang J, Quake SR. RNA-guided endonuclease provides a therapeutic strategy to cure latent herpesviridae infection [J]. Proc Natl Acad Sci USA, 2014, 111(36): 13157-13162.
[25] Yuen KS, Chan CP, Wong NH, Ho CH, Ho TH, Lei T, Deng W, Tsao SW, Chen H, Kok KH, Jin DY. CRISPR/Cas9-mediated genome editing of Epstein-Barr virus in human cells [J]. J Gen Virol, 2015, 96(Pt3): 626-636.
[26] Ran FA, Hsu P, Lin CY, Gootenberg JS, Konermann S, Trevino A, Scott DA, Inoue A, Matoba S, Zhang Y, Zhang F. Double nicking by RNA-guided CRISPR Cas9 for enhanced genome editing specificity [J]. Cell, 2013, 154(6): 1380-1389.
[27] Dianov GL, Hübscher U. Mammalian base excision repair: the forgotten archangel [J]. Nucleic Acids Res, 2013, 41(6): 3483-3490.
[28] Fu Y, Sander JD, Reyon D, Cascio VM, Joung JK. Improving CRISPR-Cas nuclease specificity using truncated guide RNAs [J]. Nat Biotechnol, 2014, 32(3): 279-284.
[29] Guilinger JP, Thompson DB, Liu DR. Fusion of catalytically inactive Cas9 to FokI nuclease improves the specificity of genome modification [J]. Nat Biotechnol, 2014, 32(6): 577-582.
[30] O’Connell MR, Oakes BL, Sternberg SH, East-Seletsky A, Kaplan M, Doudna JA. Programmable RNA recognition and cleavage by CRISPR/Cas9 [J]. Nature, 2014, 516(7530): 263-266.
敬请关注《微生物与感染》微信公众号
. ZHONG Zhaohua, E-mail:zhongzh@hrbmu.edu.cn
Application of CRISPR/Cas9 gene editing technology in tumorigenic virus infection
WANG Yu, XU Weizhen, ZHONG Zhaohua
DepartmentofMicrobiologyandWuLien-TehInstitute,HarbinMedicalUniversity,Harbin150081,China
Clustered regularly interspaced short palindromic repeat (CRISPR)/CRISPR-associated protein 9 (Cas9) (CRISPR/Cas9) gene editing technology was originated from the study of microbial CRISPR adaptive immune system, by using a specific guide RNA to identify target genes and guide endonuclease of Cas9. Some viral genomes are integrated into the cellular genome or lurk in the organization resulting in persistent infection. This review refers to the latest research results since 2013, focusing on the applications of CRISPR/Cas9 technology in tumorigenic virus infection, such as human immunodeficiency virus type 1 (HIV-1), human papillomavirus (HPV), hepatitis B virus (HBV), and Epstein-Barr virus (EBV).
Clustered regularly interspaced short palindromic repeat (CRISPR)/CRISPR-associated protein 9 (Cas9); Gene editing; Virus
国家自然科学基金(81271825、81571999)
钟照华
2016-12-01)

