遮光性套袋对桃果实转录组的影响
何平,李林光,王海波,常源升,李慧峰
(山东省果树研究所,山东泰安 271000)
遮光性套袋对桃果实转录组的影响
何平,李林光,王海波,常源升,李慧峰
(山东省果树研究所,山东泰安 271000)
【目的】探明遮光性套袋在桃果实上的转录组差异,丰富桃转录组数据信息。【方法】选取遮光性套袋与对照的桃果实样品,利用Illumina HiSeqTM 2500进行高通量测序,构建桃果实转录组文库,并用测序评估、基因功能注释等生物信息学方法进行分析。【结果】经过测序获得16.62 Gb clean data测序数据,且碱基百分比(Q30)大于91%,遮光性套袋和对照2个桃果实样品分别获得65 300 730个reads和66 603 686个reads,分别有85.73%和84.60%的reads与桃参考基因组匹配。以无袋处理为参考,对转录组数据进行比较,遮光性套袋处理共获得1 963个差异表达基因,其中,下调基因708个,上调基因1 255个;在Nr数据库中对差异基因进一步进行注释,注释到1 957个基因,其中,下调基因有705个,上调基因有1 252个;COG功能注释分析发现这些差异表达基因共获得853个功能注释,涉及23个功能类别;在GO功能注释分析中,注释到1 609个基因,可以分为53个功能分类,这些分类主要涉及到分子结合、催化活性、细胞过程、生物调节等诸多生理生化过程;KEGG分析发现共有421个基因被注释到94个代谢通路中,其中,光合作用相关通路、类黄酮生物合成、核糖体生物合成等通路显著富集。光合作用通路和类黄酮生物合成通路在果实着色中发挥了重要作用,而核糖体生物合成代谢通路在果实成熟着色中的作用尚不明确。同时也进行了两组样品的果实品质检测,结果表明,遮光性套袋对桃果实的可溶性固形物及可溶性总糖产生显著性影响,可溶性固形物及可溶性总糖显著降低,而对于果实总酸的影响不大,在果实大小上几乎没有影响。【结论】在遮光性套袋处理状态下,获得一定数量的桃果实差异表达基因,光合作用通路和类黄酮生物合成通路基因在果实着色中发挥重要作用,遮光性套袋对桃果实的可溶性固形物及可溶性总糖产生显著性影响。
桃;遮光;套袋;转录组;高通量测序;功能分类
0 引言
【研究意义】桃(Prunus persica [L.] Batsch)起源于中国,属于蔷薇科(Rosaceae)李属(Prunus)植物,是世界重要果树树种之一,分布广泛,栽培历史悠久。中国是世界桃的第一生产大国,面积和产量居世界首位。在桃的种植中,存在病虫害严重、着色不良、外观品质差等问题,降低了果实的商品性。果实套袋已成为生产无公害、绿色果品的主要途径和技术措施之一,但套袋对桃果实生长发育及其内在品质也造成一定程度的负面影响[1-4]。【前人研究进展】郭宝林等[5]和陈海江等[6]对燕红、早露蟠桃、北京早艳等桃品种进行套袋试验,结果表明,套袋处理使果实单果重明显减轻。陈建军[7]对9个桃品种套袋,套袋果的可溶性固形物含量均低于对照,而且套袋处理后油桃的固形物含量比普通桃降低幅度更大。沈玉英等[8]和LI等[9]在桃果实套袋上也得到了类似的结果。WANG等[10]发现套袋导致桃果实风味降低,不套袋桃果实的总挥发性成分的含量及C6化合物和酯类要显著高于套袋果实,γ-癸内酯、δ-癸内酯的质量浓度要显著低于不套袋果实。套袋在改善果实外观的同时,对果实内在品质产生一定影响,因此,光照可直接和间接影响果实品质[11-12]。套袋减少了果实着色,降低了果实可溶性固形物含量,使得风味变淡[13]。【本研究切入点】目前,关于桃果实套袋对果实品质和色泽发育的影响,多数是从生理生化角度进行研究,从全基因组表达方面的研究仍鲜见报道。【拟解决的关键问题】本研究以易着色的晚熟桃品种‘秋雪’为材料,研究遮光性套袋与无袋栽培在桃果实转录组中基因表达上的差异,以期探讨遮光性套袋影响桃果实着色和品质的机制,为深入研究桃果实着色的基因调控机制提供理论依据,进一步为优质桃果品的生产提供参考。
1 材料与方法
1.1 试验材料
供试桃品种为山东省果树研究所选育的桃新品种‘秋雪’,定植于山东省果树研究所桃种质资源圃内,5年生树,株距为2 m,行距为4 m,主干形整枝,选择干周、冠径及树势等基本一致的作为试验材料。
供试果袋(双层外黄内红纸袋,规格为14 cm× 18 cm,外层为全木浆纸,内层为石蜡纸)选自青岛小林制袋有限公司。
试验设不套袋(对照)和盛花后 50 d套袋(2014年5月10日),单株小区,随机排列,8月18日沿树冠外围距地面1.5—2 m处随机采收30个果实,3次重复,用液氮迅速冷冻,-80℃保存备用(图1)。
1.2 样品的RNA提取和检测
采用天根 TRNzol Universal总 RNA提取试剂(DP424)提取桃果实RNA,步骤参考其说明书。采用Nanodrop、Qubit 2.0、Agilent 2100分别检测RNA样品的纯度、浓度和完整性。

图1 桃果实的形态特征Fig. 1 Morphological characteristics of peach fruits
1.3 cDNA文库构建和质控文库制备
样品检测合格后,进行文库构建,用带有Oligo(dT)的磁珠富集真核生物mRNA;加入Fragmentation Buffer将mRNA进行随机打断;以mRNA为模板,用六碱基随机引物(random hexamers)合成第一条cDNA链,然后加入缓冲液、dNTPs、RNase H和 DNA polymerase I合成第二条cDNA链,利用AMPure XP beads纯化cDNA;纯化的双链cDNA再进行末端修复、加 A尾并连接测序接头,然后用 AMPure XP beads进行片段大小选择;最后通过PCR富集得到cDNA文库。
文库构建完成后,分别使用 Qubit2.0和 Agilent 2100对文库的浓度和插入片段大小(Insert Size)进行检测,使用Q-PCR方法对文库的有效浓度进行准确定量,以保证文库质量。
1.4 测序和数据处理
利用Illumina HiSeqTM 2500平台进行高通量测序,测序读长为PE125。
通过对原始序列(Raw reads)去除接头、重复冗余、低质量序列来获得高质量序列(Clean reads),并采用TopHat2[14]软件将clean reads与桃参考基因组(ftp://ftp.bioinfo.wsu.edu/species/Prunus_persica/Prunu s_ persica-genome. v1.0/)进行序列比对(允许有2个碱基的错配)。
1.5 差异基因的筛选
采用FPKM(Fragments Per Kilobase of transcript per Million fragments mapped)值来反映基因的表达量[15],然后将不同样品的基因表达量使用IDEG6软件(http://telethon.bio.unipd.it/ bioinfo/IDEG6)进行卡方检验,通过多重假设检验(false discovery ratio,FDR)对P值进行校正;校正后取P<1%,并且样品间FPKM比值(Fold change≥2)≥2的基因,作为差异表达基因。
1.6 差异表达基因的COG分类、GO分类和KEGG富集分析
将差异表达基因和 Nr数据库进行比对,获得差异表达基因在 Nr数据库中的注释信息;与 COG(Clusters of orthologous groups of proteins)数据库进行比对分析,获得差异表达基因的 COG功能注释及其分类;利用Blast2GO[16]和WEGO软件[17]对差异表达基因进行GO(Gene ongology)功能注释及分类;与KEGG(Kyoto encyclopedia of genes and genomes)数据库进行比对,获得差异表达基因相对应的Pathway注释信息。
1.7 差异表达基因荧光定量PCR分析
采用TransScript II All-in-One First-Strand cDNA Synthesis SuperMix for qPCR(北京全式金)试剂盒对1.2中提取的RNA进行反转录;用SYBRGreen染料进行荧光定量PCR(qRT-PCR)分析,以2-ΔΔCt法计算基因的差异倍数。每个样品 3次重复。选用 PpN1(ppa009483m)为内参基因,引物序列参考文献[18](表1)。

表1 用于qRT-PCR分析的基因及其引物Table 1 The qRT-PCR primers for the 6 selected genes
1.8 果实品质检测
选用1.1部分采收的果实,每个处理随机选取30个果实测定平均单果质量,根据 GB/T12295-1990、GB/T12456-2008和 GB/T6194-1986标准委托农业部食品质量监督检验测试中心(济南)检测果实可溶性固形物、可溶性总糖和总酸含量的测定。
2 结果
2.1 测序数据质量分析
遮光性套袋和对照的测序原始数据碱基组成基本平衡,而且大部分reads的碱基质量值分布于20以上,说明测序所得的原始数据质量较好,可以满足后续的分析。
2个样品共得到16.62 Gb Clean Data测序数据,并且碱基百分比(Q30)大于 91%。充分说明测序得到的数据质量可靠,满足后续分析。遮光性套袋和对照分别获得65 300 730个reads和66 603 686个reads,与参考基因组匹配的reads分别有55 984 355个和56 344 011个,占总读数的85.73%和84.60%。表明 2个样品的 reads与参考基因组的比对效率较高。
2.2 差异表达基因的筛选
通过对桃果实转录组FPKM值的测定(表2),发现遮光性套袋处理的桃果实转录组 FPKM值<5的基因占 70.50%,>100的基因占 4.25%;对照组的FPKM值<5的基因占70.59%,>100的基因占4.34%,二者差异不显著。为了检测遮光性套袋对桃果实基因表达的影响,使用IDEG6软件,将差异倍数(fold change)>2,显著水平P<0.05作为显著差异的表达基因。共获得1 963个差异表达基因,其中,下调基因708个,上调基因1 255个,进一步将这些差异基因在Nr数据库中进行注释,得到1 957个注释基因,其中下调基因有 705个,上调基因有1 252个。
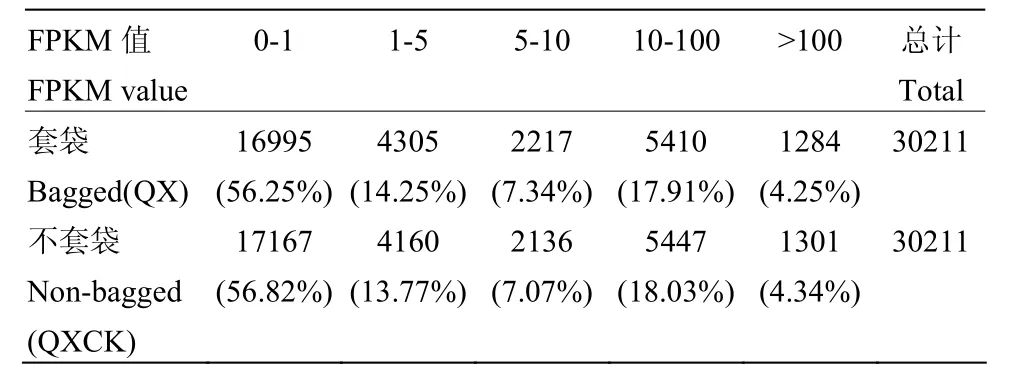
表2 桃果实FPKM值Table 2 FPKM values of peach fruits
2.3 差异表达基因COG注释
对Nr数据库中注释到的1 957个差异表达基因在COG数据库中进行比对和功能注释,结果表明,在COG功能分类体系中,共获得853个COG功能注释,涉及23个COG功能类别(图2)。其中,最大群体比例为R(一般功能基因),有242个基因;其次为K类(转录)、L类(复制、重组和修复)和T类(信号传导机制),分别有116、126和105个基因。
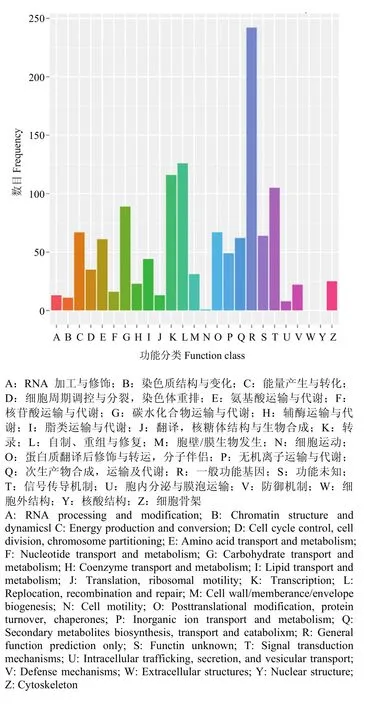
图2 差异表达基因COG功能分类图Fig. 2 COG function classification of differentially expressed genes
2.4 差异表达基因的GO的分类
利用Blast2GO软件对差异表达基因进行GO分类和功能注释。结果表明,共分为3大类别(细胞组分、分子功能和生物过程),53个小类(图 3),1 963个差异表达基因中有1 609个获得功能注释,其中有1 431个差异基因(占总差异基因的88.94%)分类到细胞组分中的16个类别,另外按照分子功能分成17个类别,有 1 342个差异基因(占总差异基因的83.41%),按照生物过程分成20个类别,有1 533个差异基因(占总差异基因的95.28%)。在细胞组分分类中,细胞部分(cell part)和细胞(cell)所占比例最多,分别占 97.83%和 96.09%,其次是细胞器(organelle),占82.50%;而细胞外基质(extracellular matrix)和细胞外基质部分(extracellular matrix part)比例最低,分别仅有2条序列。在分子功能分类中,结合(binding)和催化活性(catalytic activity)所占比例最多,分别占 68.18%和 68.48%,其次是转动活性(transporter activity)和核酸结合转录因子活性(nucleic acid binding transcription factor activity),分别占9.24%和8.42%;而营养库活性(nutrient reservoir activity)的比例最低,仅有4条序列。在生物学过程中,细胞过程(cellular process)、代谢过程(metabolic process)、单一生物过程(single organism process)和刺激反应(response to stimulus)所占比例最多,分别占91.45%、88.39%、87.21%和74.56%,而细胞杀伤(cell killing)和生物相(biological phase)比例最低,仅有2条和1条序列。
2.5 差异表达基因KEGG富集分析
差异表达基因的KEGG分析结果如图4所示,共有421个差异表达基因被注释到了94个通路中,其中显著富集通路(P<0.05)有5个,主要参与到光合作用通路、类黄酮生物合成、核糖体生物合成等通路中(表 3)。在各通路中注释到基因多数表达下调,其中光合作用(photosynthesis)注释到25个差异表达基因(22个下调,3个上调),光合作用-天线蛋白通路(photosynthesis-antenna proteins)注释到9个差异表达基因(8个下调,1个上调),类黄酮生物合成途径(flavonoid biosynthesis),注释到12个差异表达基因(11个下调,1个上调),DNA复制路径(DNA replication)注释到13个差异表达基因(11个下调,2个上调),光合生物碳固定作用通路(carbon fixation in photosynthetic organisms)注释到14个差异表达基因(13个下调,1个上调)。类黄酮生物合成途径KEGG数据库图谱如图 5所示,差异表达的基因大多下调(绿色),注释到的12个差异表达基因多数参与花色苷生物合成途径,如F3H(ppa007636m)、LDOX(ppa007738m)、DFR(ppa008069m)、ANR(ppa008295m)、CHI(ppa011276m)等功能基因,且均表现下调。
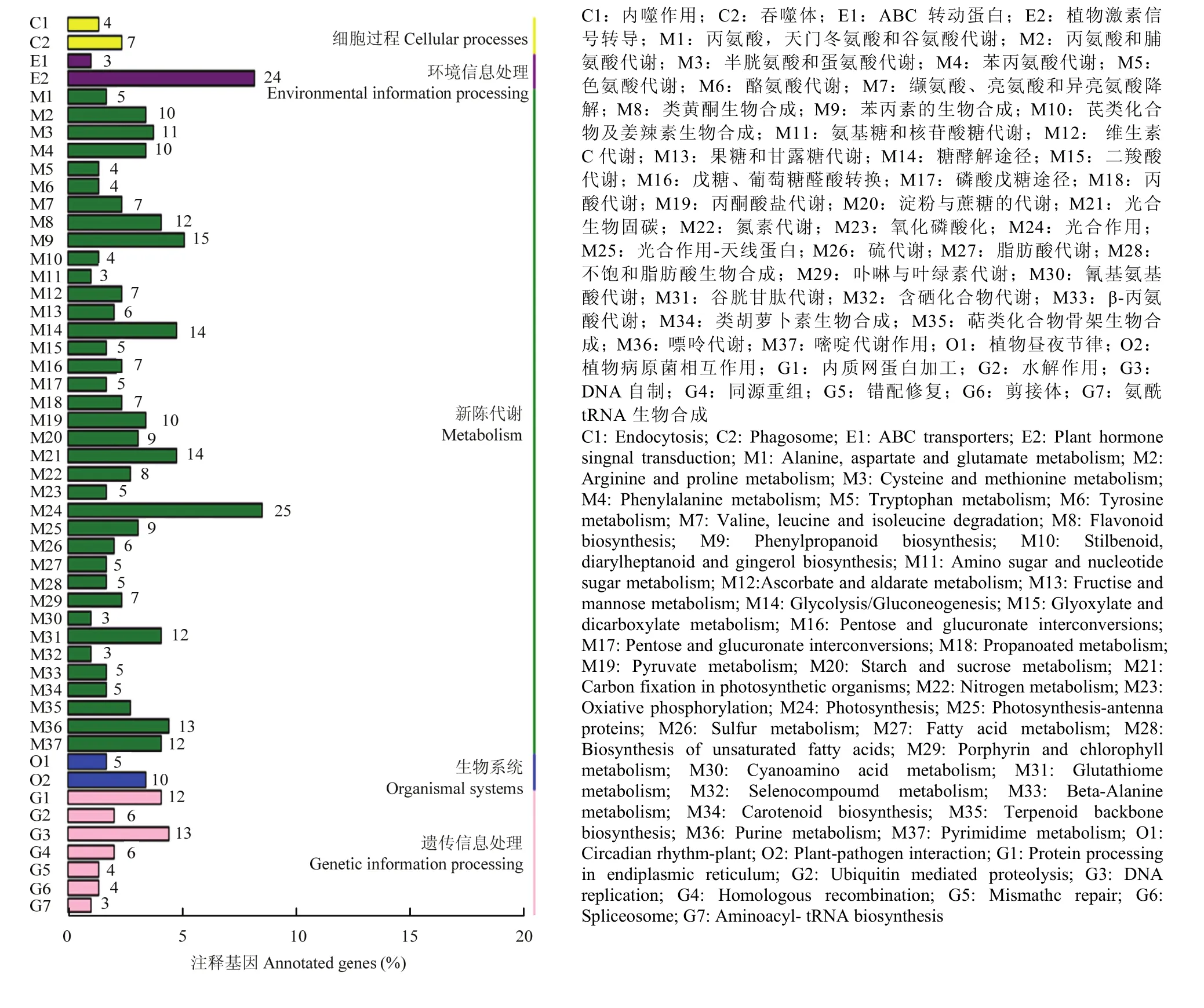
图4 差异表达基因KEGG功能分类图Fig. 4 KEGG function classification of differentially expressed genes
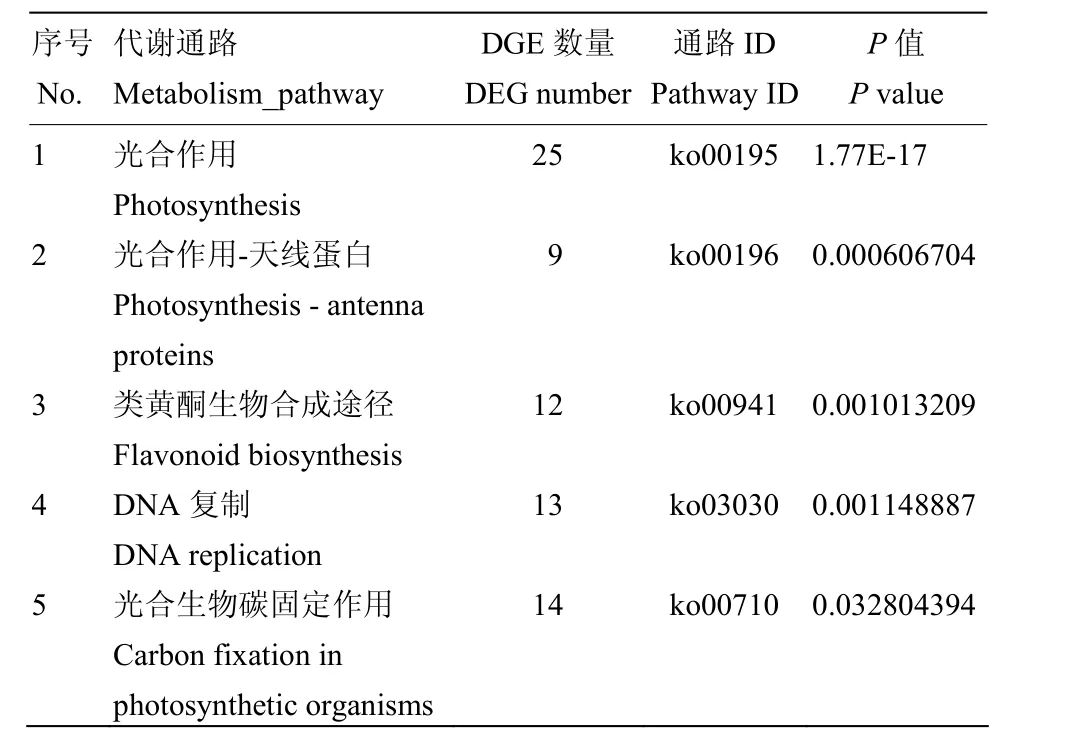
表3 差异表达基因KEGG代谢途径分类Table 3 KEGG classification of differentially expressed genes

图5 KEGG数据库中类黄酮生物合成途径Fig.5 Flavonoid biosynthesis pathway in KEGG
2.6 差异表达基因的qRT-PCR荧光定量分析
对类黄酮途径中的关键基因F3H(ppa007636m)、LDOX(ppa007738m)、DFR(ppa008069m)、ANR(ppa008295m)、CHI(ppa011276m)进行qRT-PCR荧光定量分析(表4),发现遮光性套袋处理的桃果实样品各基因的表达均与对照表现下调趋势,只是差异倍数上与转录组的数据有所区别。

表4 差异表达基因的RNA-Seq FPKM值、qRT-PCR相对表达水平及其比率Table 4 RNA-Seq FPKM value, qRT-PCR relative expressionlevel, ratio of CK and QX in the differentially expressed genes
2.7 果实品质的检测
由表5的测评结果可知桃果实遮光性套袋对果实的可溶性固形物及可溶性总糖产生显著性影响,可溶性固形物及可溶性总糖显著降低,而对于果实总酸的影响不大。在果实大小上几乎没有影响。
3 讨论
高通量转录组测序技术在挖掘差异表达基因,揭示生物基因表达及调控机理发挥了重要作用[19-22]。本研究通过Illumina HiSeqTM 2500高通量测序技术构建了遮光性套袋桃果实及其对照的转录组文库,获得大量的转录组信息。处理与对照共获得16.62 Gb Clean Data 数据量,比对到桃参考基因组上的reads在84%以上,说明本试验文库构建很成功,数据覆盖度较高。通过对文库分析,获得1 963个差异基因,其中1 957个基因在Nr数据库中得到功能注释,未得到注释的差异表达基因有6个;有1 609个基因获得GO功能注释,348个基因未得到GO功能注释;421个基因被注释到了KEGG的94个代谢通路中,153个基因未得到KEGG的注释。这种造成基因注释信息缺失的现象,前人认为转录组序列越短,获得注释信息就越少[23]。

表5 果实品质检测分析Table 5 Analysis of the fruit quality detection
COG功能分类表明,853个差异表达基因获得COG功能注释,包括23个功能类别。GO功能注释分类显示,差异表达基因主要涉及细胞相关类别(包括细胞部分、细胞、细胞器等)、分子结合、细胞过程、代谢过程等类型。深入对差异基因进行 Pathway分析,进一步对差异表达基因参与的主要代谢途径和信号传导途径进行研究,有利于明确差异表达基因在生物代谢周期中发辉的功能以及与其他基因的相互作用[22-23]。本研究KEGG Pathway富集分析表明,差异表达基因共涉及到94个通路,其中光合作用相关通路3个、类黄酮生物合成1个、核糖体生物合成1个等5个通路显著富集。果实色泽是果实商品品质的重要因素之一,果实成熟着色是由于叶绿素降解,同时形成显现类胡萝卜素或花青苷的结果。花青苷是一类类黄酮化合物,花青苷是果实的次生代谢产物之一,它是经过莽草酸途径合成的[24],花青苷的生物合成是依赖光照进行的反应过程[25]。花青苷是由花色素和糖组成,而花色素又是在糖代谢的基础上由丙酮酸和乙酸缩合而成的[26-27]。花青苷含量和着色程度与果肉中还原糖和可溶性糖含量呈显著相关,可溶性固形物含量与花青苷合成呈正相关[28-29]。套袋虽然在一定程度上降低果实的含糖量,但也能有效地促进果实着色[13,30]。这一事实说明果实着色在一定范围内受限于果实中含糖量,如果越过一定阈值则含糖量不再成为限制因素;再则充分表明光照对果实着色的重要性,果实套袋增加光有效性的同时也促使光照不足,以致有关基因不能活化[31-32]。前人研究认为光照影响花青苷合成和果实着色,首先光照影响光合作用,进而影响糖、苯丙氨酸等有机物合成;其次光照调解花青苷合成相关酶的活性,其中查尔酮合成酶(CHS)、查尔酮异构酶(CHI)、黄烷酮 3-羟化酶(F3H)、二羟基黄酮醇还原酶(DFR)、花色素合成酶(ANS)等基因的表达受到光照因子的诱导[33-37]。本研究显著富集的通路中有3个与光合作用相关,充分说明了光照在果实成熟期对着色的重要性,同时又显著富集到类黄酮生物合成通路,进一步说明光照调控花青苷代谢过程相关基因表达的事实。本研究筛选到了参与此通路上的F3H、LDOX、DFR、ANR、CHI等5个差异表达基因,且表达水平较对照低,说明套袋增加了光的有效性,但光照不足,以致有关基因不能活化或表达水平较低,此结果与王惠聪在荔枝[27]上和马瑞娟等[38]在桃上的研究相似。同时检测发现遮光性套袋显著降低了果实的可溶性固形物的含量,进一步证明套袋在一定程度上降低果实的含糖量的论点,说明可溶性固形物含量与花青苷合成呈正相关。此外,核酸生物合成(DNA复制)也得到了显著富集,这类代谢通路在果实着色上的作用尚不明确。差异表达基因 KEGG Pathway分析表明光照与类黄酮生物合成通路调控着果实着色,但具体的调控机制还需要对相关通路的基因进行更深入的研究。
4 结论
通过高通量测序技术获得一定数量在遮光性套袋处理状态下桃果实差异表达基因,光合作用通路和类黄酮生物合成通路基因在果实着色中发挥了重要作用,再次印证了遮光性套袋对桃果实的可溶性固形物及可溶性总糖产生显著性影响。
[1] 刘厚宇, 李源念, 刘国琴, 任艳芳. 套袋对艳红桃果实品质的影响.贵州农业科学, 2009, 37(5): 152-153.
LIU H Y, LI Y N, LIU G Q, REN Y F. The effect of bagging on fruit quality of yanhong peach. Guizhou Agricultural Sciences, 2009, 37(5): 152-153. (in Chinese)
[2] 张安宁, 刘伟, 李桂祥, 张毅, 王长君. 8种不同类型果袋对‘岱妃’桃果实品质的影响. 山东农业科学, 2014, 46(3): 33-36.
ZHANG A N, LIU W, LI G X, ZHANG Y, WANG C J. Effects of bagging with eight types of bags on fruit quality of Daifei peach. Shandong Agricultural Sciences, 2014, 46(3): 33-36. (in Chinese)
[3] 李林, 苏俊, 陈霞, 舒群. 不同套袋时期和果袋类型对云红梨 1号果实品质的影响. 园艺与种苗, 2011(5): 77-79.
LI L, SU J, CHEN X, SHU Q. Effect of bagging time and bag type onthe quality of Yunhongli 1. Horticulture & Seed, 2011(5): 77-79. (in Chinese)
[4] 罗华, 李敏, 宋红日, 张连忠. 套袋处理对肥城桃果实香气成分的影响. 湖北农业科学, 2012, 51(22): 5072-5075.
LUO H, LI M, SONG H R, ZHANG L Z. The effects of bagging on aroma compounds of Feicheng peach. Hubei Agricultural Sciences, 2012, 51(22): 5072-5075. (in Chinese)
[5] 郭宝林, 杨俊霞, 鲁韧强, 陈兆峰, 刘丽林. 套袋提高燕红桃果品质量的试验研究. 中国果树, 2000(3): 19-20.
GUO B L, YANG J X, LU R Q, CHEN Z F, LIU L L. The test on improving the fruits quality of ‘Yanhong’ peach by bagging. China Fruits, 2000(3) : 19-20. (in Chinese)
[6] 陈海江, 段红喜, 徐继忠, 皇甫中泗. 提高设施桃果实品质试验.山西果树, 2003(1): 4-5.
CHEN H J, DUAN H X, XU J Z, HUANGPU Z S. The test on improving the fruits quality of facilities peach. Shanxi Fruits, 2003(1): 4-5. (in Chinese)
[7] 陈建军. 几个桃品种不同时期套袋试验结果简报. 甘肃农业科技, 2004(6): 26-27.
CHEN J J. The study brief report on different period bagging in a few peach cultivars. Gansu Agriculture Science and Technology, 2004(6): 26-27. (in Chinese)
[8] 沈玉英, 李斌, 贾惠娟. 不同纸质果袋对湖景蜜露桃果实品质的影响. 果树学报, 2006, 23(2): 182-185.
SHEN Y Y, LI B, JIA H J. Effect of bagging on fruit quality of hujingmilu peach cultivar. Journal of Fruit Science, 2006, 23(2): 182-185. (in Chinese)
[9] LI B, JIA H J, ZHANG X M. Effects of fruit pre-harvest bagging on fruit quality of peach (Prunus persica Batsch cv. Hujingmilu). Journal of Plant Physiology and Molecular Biology, 2006, 32(3): 280-286.
[10] WANG Y, YANG C, LIU C, XU M, LI S, WANG Y. Effects of bagging on volatiles and polyphenols in “Wanmi” peaches during endocarp hardening and final fruit rapid growth stages. Journal of Food Science, 2010, 75(9): 455-460.
[11] 周君, 王红清. 铺设反光膜对桃树不同冠层叶片最大光合能力和果实品质的影响. 中国农业大学学报, 2009, 14(4): 59-64.
ZHOU J, WANG H Q. Effect of reflective film on the photosynthetic capacity of leaf and fruit quality at different canopies of peach. Journal of China Agricultural University, 2009, 14(4): 59-64. (in Chinese)
[12] 黄成思, 苏智先, 朱利君, 方志荣. 套袋对新都柚果实外观品质的影响. 安徽农业科学, 2011, 39(20): 12036-12037.
HUANG C S, SU Z X, ZHU L J, FANG Z R. Effect of bagging on outside quality of Citrus grandis var.. Journal of Anhui Agricultural Science, 2011, 39(20): 12036-12037. (in Chinese)
[13] 杜纪红. 桃果实花青苷和糖酸含量变化及其与套袋关系研究[D].南京: 南京农业大学, 2007.
DU J H. Variations of anthocyanin, sugars and acid contents in peach fruit and the influences affected by bagging measurement during different fruit development stages [D]. Nanjing: Nanjing Agricultural University, 2007. (in Chinese)
[14] TRAPNELL C, PACHTER L, SALZBERG S L. TopHat: Discovering splice junctions with RNA-Seq. Bioinformatics, 2009, 25(9): 1105-1111.
[15] MORTAZAVI A, WILLIAMS B A, MCCUE K, SCHAEFFER L, WOLD B. Mapping and quantifying mammalian transcriptomes by RNA-Seq. Nature Methods, 2008, 5(7): 621-628.
[16] CONESA A, GÖTZ S, GARCÍA-GÓMEZ J M, TEROL J, TALÓN M, ROBLES M. Blast2GO: A universal tool forannotation, visualization and analysis in functional genomics research. Bioinformatics, 2005, 21: 3674-3676.
[17] KOBAYASI K, MATSUI T, YOSHIMOTO M, HASEGAWA T. Effects of temperature, solar radiation, and vapor-pressure deficit on flower opening time in rice. Plant Production Science, 2010, 13(1): 21-28.
[18] CHENG J, WEI G, ZHOU H, GU C, VIMOLMANGKANG S, LIAO L, HAN Y. Unraveling the mechanism underlying the glycosylation and methylation of anthocyanins in peach. Plant Physiology, 2014, 166: 1044-1058.
[19] 杜琛, 付绍印, 高鸿雁, 郑竹清, 孟宪然, 阿娜, 萨仁, 张文广, 李金泉. 绒山羊肌内脂肪细胞成熟前后比较转录组分析. 畜牧兽医学报, 2014, 45(5): 714-721.
DU C, FU S Y, GAO H Y, ZHENG Z Q, MENG X R, A N, SA R, ZHANG W G, LI J Q. Transcriptome analysis of intramuscular preadipocytes and matureadipocyte in cashmere goats. Acta Veterinaria et Zootechnica Sinica, 2014, 45(4): 714-721. (in Chinese)
[20] 吴小波, 王子龙, 李淑云, 甘海燕, 刘浩, 颜伟玉, 曾志将. 羽化和性成熟中华蜜蜂蜂王和雄峰转录组分析. 昆虫学报, 2014, 57(8): 905-913.
WU X B, WANG Z L, LI S Y, GAN H Y, LIU H, YAN W Y, ZENG Z J. Transcriptomic analysis of apis cerana cerana (Hymenoptera: Apidae) queens and drones newly emerged and sexually matured. Acta Entomologica Sinica, 2014, 57(8): 905-913. (in Chinese)
[21] ZENG T, ZHANG L, LI J, WANG D, TIAN Y, LU L. De novo assembly and characterization of muscovy duck liver transcriptome and analysis of differentially regulated genes in response to heat stress.Cell Stress and Chaperones, 2015, 20: 483-493.
[22] CHUNQING O, SHULING J, FEN W, CHANGYONG T, NINGNING H. An RNR-Seq analysis of the pear (Pyrus communis L.) transcriptome, with a focus on genes associated with dwarf. Plant Gene, 2015, 4: 69-77.
[23] 魏利斌, 苗红梅, 张海洋. 芝麻发育转录组分析. 中国农业科学, 2012, 45(7): 1246-1256.
WEI L B, MIAO H M, ZHANG H Y. Transcriptomic analysis of sesame development. Scientia Agricultura Sinica, 2012, 45(7): 1246-1256. (in Chinese)
[24] SALISBURY F, ROSS C. Plant Physiology. California: Wads-worth publishing co., 1992: 323- 325.
[25] VIMOLMANGKANG S, ZHENG D, HAN Y, KNAN M A, SORIA-GUERRA R E, KORBAN S S. Transcriptome analysis of the exocarp of apple fruit identifies light-induced genes involved in red color pigmentation. Gene, 2014, 534: 78-87.
[26] ZHANG X, WANG L, LIU Y, CHEN X, YANG Y, ZHAO Z. Differential gene expression analysis of‘Granny Smith’apple(Malus domestica Borkh.)during fruit skin coloration. South African Journal of Botany, 2013, 88: 125-131.
[27] 王惠聪, 黄旭明, 胡桂兵, 黄辉白. 荔枝果皮花青苷合成与相关酶的关系研究. 中国农业科学, 2004, 37(12): 2028-2032.
WANG H C, HUANG X M, HU G B, HUANG H B. Studies on the relationship between anthocyanin biosynthesis and related enzymes in litchi pericarp. Scientia Agricultura Sinica, 2004, 37(12): 2028-2032. (in Chinese)
[28] SPRINGOB K, NAKAJIMA J, YAMAZAKI M, SAITO K. Recent advances in the biosynthesis and accumulation of anthocyanins. Natural Product Report, 2003, 20: 288-303.
[29] 齐秀娟, 李作轩, 徐善坤. 山楂果实中可溶性糖与果皮色素的关系. 果树学报, 2005, 22(1): 81-83.
QI X J, LI Z X, XU S K. Relationship between soluble sugars and peel pigments in hawthorn fruit. Journal of Fruit Science, 2005, 22(1): 81-83. (in Chinese)
[30] LAYNE D, JIANG Z, RUSHING J. Tree fruit reflective film improves red skin coloration and advances maturity in peach. Hort Technology, 2001, 11(2): 234-242.
[31] 陈文龙, 赵昶灵, 支伟特, 陈中坚, 缪康汝, 王文亚, 翁晨. 高等植物花色苷生物合成调控的研究进展. 中国农学通报, 2013, 29(33): 271-276.
CHEN W L, ZHAO C L, ZHI W T, CHEN Z J, MIAO K R, WANG W Y, WENG C. Progress of regulation of anthocyanin biosynthesis in higher plants. Chinese Agricultural Science Bulletin, 2013, 29(33): 271-276. (in Chinese)
[32] MASAKAZU H, KARIN O, YOKO H. Enhancement of anthocyanin biosynthesis by sugar in radish. Hypocotyl Plant Science, 2003, 164: 259-265.
[33] 张蕾, 朱立新, 徐川, 崔春梅, 盛宏亚, 李瑞, 王红清. 查尔酮合酶基因对桃果实花色苷代谢的影响. 园艺学报, 2015, 42(1): 31-37.
ZHANG L, ZHU L X, XU C, CUI C M, SHENG H Y, LI R, WANG H Q. The effect of silencing chalcone synthase on anthocyanin metabolism in peach. Acta Horticulturae Sinica, 2015, 42(1): 31-37. (in Chinese)
[34] TSUDA T, YAMAGUCHI M, HONDA C, MORIGUCHI T. Expression of anthocyanin biosynthesis genes in the skin of peach and nectarine fruit. Journal of the American Society for Horticultural Science, 2004, 129(6): 857-862.
[35] GUO J, WANG M. Ultraviolet a specific induction of anthocyanin biosynthesis and PAL expression in tomato. Plant Growth Regulation, 2010, 62(1): 1-8.
[36] UBI B, HONDA C, BESSHO H, KONDO S, WADA M, KOBAYASHI S, MORIGUCHI T. Expression analysis of anthocyanin biosynthetic genes in apple skin: Effect of UV-B and temperature. Plant Science, 2006, 3(170): 571-578.
[37] 周君, 陈宗玲, 张琼, 王红清. 桃果皮中羟基肉桂酸类、类黄酮类和花色苷类物质合成机理的研究. 园艺学报, 2009, 36(12): 1717-1724.
ZHOU J, CHEN Z L, ZHANG Q, WANG H Q. Effects of bagging on accumulation of phenolic acids and flavonoids in peach pericarp during fruit maturity. Acta Horticulturae Sinica, 2009, 36(12): 1717-1724. (in Chinese)
[38] 马瑞娟, 张斌斌, 张春华, 蔡志翔, 颜志梅. 采前除袋铺设反光膜对桃果实着色及相关基因表达的影响. 园艺学报, 2015, 42(11): 2123-2132.
MA R J, ZHANG B B, ZHANG C H, CAI Z X, YAN Z M. Effect of bag removing with reflective film mulching before harvest on fruit coloration and expression of anthocyanin related genes in peach. Acta Horticulturae Sinica, 2015, 42(11): 2123-2132. (in Chinese)
(责任编辑 李莉)
Effects of Shading Fruit with Opaque Paper Bag on Transcriptome in Peach
HE Ping, LI LinGuang, WANG HaiBo, CHANG YuanSheng, LI HuiFeng
(Shandong Institute of Pomology, Tai’an 271000, Shandong)
【Objective】 The objective of this study was to find the transcriptome differences between shading fruits with opaque paper bags and CK in peach, and enrich the peach transcriptome data. 【Method】 Selecting peach fruits as samples (shading fruits with opaque paper bags and CK, respectively), then the transcriptome libraries of peach fruits were constructed by using the Illumina HiSeqTM 2500 sequencing technique, and were analyzed by using the bioinformatics methods subsequently, such as sequencing assess and gene function annotation. 【Result】 The results showed that the 16.62 Gb transcriptome data were obtained from both the shading bagged fruits and CK , and the base ratios with quality values higher than 30 in reads (Q30) were more than 91% from both the shading fruits and CK. There were 65 300 730 reads and 66 603 686 reads transcriptome data were obtained from the peach fruits used bagging or not, and 85.73% and 84.60% reads could be compared to the peach reference genome sequence, respectively. Compared the transcriptome of shading bagged fruits with CK, 1 963 differentially expressed genes were found in theshading bagged fruits, including 1 255 up-regulated genes and 708 down-regulated genes. Annotation analysis indicated that 1 957 genes were annotated in Nr data, including 1 252 up-regulated genes and 705 down-regulated genes. Through COG analysis, there were 853 functional annotations of these differentially expressed genes, involving 23 functional classifications. With GO function annotation classifications, a total of 1 609 genes were divided into 53 function categories, in which many functional categories were mainly involved, such as molecular binding, catalytic activity, cell process, biological regulation. KEGG analysis showed that a total of 421 genes were annotated to 94 metabolic pathways, and photosynthesis signaling pathway, carbon fixation in photosynthetic organisms, photosynthesis-antenna proteins signaling pathway, flavonoid biosynthesis, ribosome biosynthesis (DNA replication) pathway were significantly enriched. The photosynthesis and flavonoid biosynthesis signaling pathway played an important role in fruit color, but the role of ribosome biosynthesis was not clear. The fruit quality also was detected from the shading bagged fruits and CK. The results showed that shading bagged peach fruits had a remarkable impact on soluble solids and total soluble sugar, which were significantly reduced. But the study also found, on average, little or no effect on total acid and per fruit weight from both the shading fruits and CK. 【Conclusion】 The number of differentially expressed genes under different physiological status of the fruit tissue in peach were obtained. The study found that the photosynthesis and flavonoid biosynthesis signaling pathway played an important role in fruit color and shading bagged peach fruits had a remarkable impact on soluble solids and total soluble sugar.
peach; shading; bagging; transcriptome; high-throughput sequencing; functional classification
2016-07-01;接受日期:2016-10-09
国家现代农业产业技术体系(CARS-31)、山东省农业重大应用技术创新课题(2014-38)、山东省农业科学院农业科技创新工程(CXGC2016B07)
联系方式:何平,E-mail:heping024@163.com。通信作者李林光,Tel:0538-8266675;E-mail:llg6536@163.com

