结核分枝杆菌H37Rv蛋白Rv2364c的表达纯化与生物信息学分析
徐宛玲,付陈增
结核分枝杆菌H37Rv蛋白Rv2364c的表达纯化与生物信息学分析
徐宛玲,付陈增
目的 在大肠杆菌中表达结核分枝杆菌H37Rv的Rv2364c蛋白,为研发抗结核分枝杆菌的药物提供材料。方法 PCR扩增目的基因Rv2364c。将PCR产物克隆至原核表达载体pET-28a中,构建重组原核表达质粒pET-28a-Rv2364c。将重组质粒转化大肠杆菌BL21(DE3)中,用异丙基-β-D-硫代半乳糖苷(IPTG)诱导表达融合蛋白。表达产物经镍亲和层析柱纯化,并对该蛋白做生物信息分析。结果 双酶切和基因测序结果表明,成功构建了含有Rv2364c基因的重组表达质粒。SDS-PAGE结果显示,目的蛋白在大肠杆菌中得到了大量表达,经镍亲和层析柱纯化后目的蛋白的纯度达到了90%以上。生物信息学分析表明Rv2364c为稳定酸性蛋白,α-螺旋和无规卷曲为其主要二级结构元件,主要结构域类型有Era结构域和KH结构域两种。结论 成功表达和纯化了Rv2364c蛋白,并利用生物信息学方法初步预测其结构域等,为进一步研究Rv2364c在结核病发生发展中的作用奠定了基础。
结核分枝杆菌H37Rv;Rv2364c;转染;生物信息分析
结核分枝杆菌(mycobacterium tuberculosis,Mtb)感染者约占全球总人口的1/3,其中约有5%~10%的患者会发展成活动性肺结核,从而导致每年约150万人的死亡,目前结核病已成为全球最致命的传染病之一[1]。耐药菌的出现及HIV合并感染使得控制这种疾病变得非常困难,据报道,2015年约有9.7%的患者表现出了对多种抗生素的耐受[2],因此迫切需要研发新型抗结核药物、疫苗和诊断方法。Mtb致病株H37Rv中Rv2364c发现较晚,目前对其研究较少。本研究克隆、表达和纯化Rv2364c蛋白,并对其理化性质等进行了分析预测,希望能为研发新的抗结核药物提供帮助。现报道如下。
1 材料与方法
1.1 材料 根据结核分枝杆菌H37Rv基因组(NC_000962.3)中Rv2364c的序列,在其上下游设计一对引物,引物由生工生物公司合成。载体pET-28a为本实验室保存,大肠杆菌菌株DH(5α)和BL21(DE3)感受态细胞购自北京全式金生物公司。EcoRⅠ,BamHⅠ和T4连接酶购自赛默飞世尔科技公司, DNA marker和Protein Marker购自北京全式金公司,质粒小提试剂盒和胶回收试剂盒购自北京莱科百奥公司。
1.2 方法
1.2.1 Rv2364c基因的扩增 利用primer 5.0设计引物。引物为正向-CGGGATCCATGACCGAATTCCA-TTCTGGCTTT,反向-CGGAATTCCTAAAACCCCAGT-CGGCCAA。以合成的Rv2364c序列为模板进行PCR扩增,参数为:94 ℃预变性10 min; 94 ℃ 30 s,60 ℃ 30 s,72 ℃ 1 min,30个循环;72 ℃终延伸10 min。扩增产物用1%琼脂糖凝胶电泳回收。
1.2.2 重组质粒的构建与鉴定 将回收的目的片段与pET-28a载体分别用EcoRⅠ和BamHⅠ双酶切,将酶切产物按比例16 ℃连接5 h。将连接产物转化到大肠杆菌DH(5α)感受态细胞,卡那霉素筛选阳性克隆,对阳性克隆做双酶切鉴定和测序鉴定。
1.2.3 目的基因在大肠杆菌中的融合表达 将测序正确的重组质粒转化到大肠杆菌BL21(DE3)感受态细胞中。挑取单克隆于含卡那霉素的LB培养基中培养过夜。再取过夜的菌液接种至1L LB液体培养基中,继续振荡培养。当菌液浓度OD600值达到0.6时,加入异丙基-β-D-硫代半乳糖苷(isopropyl-β-D-thiogalactoside, IPTG)至终浓度为0.1 mmol· L-1,16 ℃振荡培养8 h。
1.2.4 融合蛋白的纯化 收集菌体,用PBS重悬菌体沉淀,超声破碎后15 000 r· min-1,4 ℃离心30 min,将上清过Ni-NTA亲和柱纯化,并分别取上清、沉淀、穿过、50 mmol· L-1咪唑洗杂、500 mmol· L-1咪唑洗脱和洗脱后的柱料制样。SDS-PAGE电泳分析,收集500 mmol· L-1咪唑洗脱液并超滤浓缩。
1.2.5 Rv2364c的生物信息学分析 利用ProtParam对Rv2364c的氨基酸序列及理化性质进行分析,SOPMA对其二级结构进行预测,最后用CD-search预测Rv2364c的功能结构域。
2 结果与分析
2.1 目的蛋白的表达与纯化
2.1.1 重组质粒的构建及鉴定 构建的重组质粒用EcoRⅠ和BamHⅠ作双酶切鉴定(图1),获得5 369 bp的载体片段和约900 bp的目的基因片段,结果表明成功构建重组质粒。
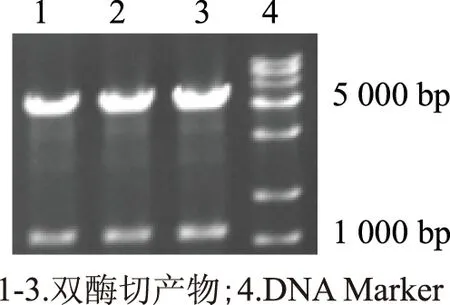
图1 重组质粒的双酶切鉴定
2.1.2 重组蛋白的表达及纯化 在0.1 mmol· L-1IPTG, 16 ℃条件下诱导8 h后收集菌体,超声裂解后,融合蛋白经镍柱纯化、SDS-PAGE电泳结果显示大部分蛋白位于沉淀中,少部分蛋白位于上清中,将上清中的蛋白过镍柱后得到纯度大于90%的蛋白(图2)。
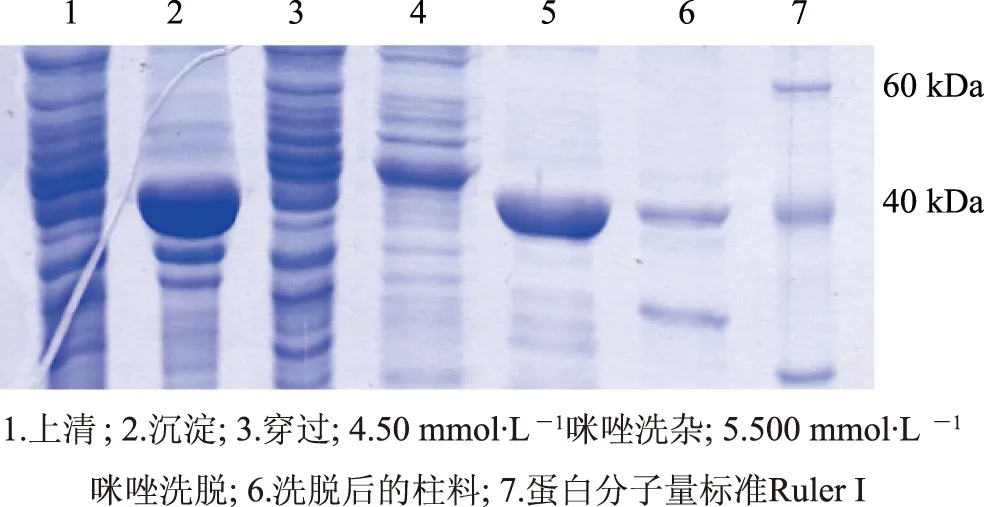
图2 融合蛋白亲和层析后的SDS-PAGE分析
2.2 Rv2364c的生物信息学分析
2.2.1 Rv2364c的氨基酸序列及理化性质分析 ProtParam分析表明Rv2364c分子量为32.5 kDa,有300个氨基酸残基,含量较高的是亮氨酸(11.0%)、缬氨酸(11.7%),含量较少的氨基酸有半胱氨酸(0.7%)、色氨酸(0.7%)。理论等电点5.76为酸性蛋白。在溶液中的不稳定指数为26.48,是稳定蛋白。总平均亲水性为0.003。
2.2.2 Rv2364c的二级结构预测 利用SOPMA预测二级结构,结果表明:α-螺旋的比例最高,达到42%,无规卷曲次之,为31.67%,延伸链和β-转角所占比例分别为20%和6.33%。因此α-螺旋和无规卷曲是该蛋白的主要二级结构元件。无规卷曲主要分布于氨基酸残基的13-20、36-42、92-100、177-190这4个区域。
2.2.3 Rv2364c的功能结构域分析 利用CD-search预测Rv2364c的功能结构域,得到两种结构域类型:①Era结构域,位于氨基酸残基4~169位。②KH结构域,位于氨基酸残基210~288位。
3 讨论
本研究以质粒pET-28a为表达载体,在大肠杆菌中融合表达Rv2364c蛋白。菌液浓度OD600为0.6时加入IPTG开始诱导,8 h后收集细菌。细菌经超声破碎后,上清中可见可溶性蛋白存在,但沉淀中也有大量蛋白。蛋白不溶解的原因可能是蛋白的表达量过高,pET系统的T7启动子是原核表达系统中最强的启动子,强启动容易引起目的蛋白没有足够时间正确折叠而聚集成没有生物活性的不溶结构,即包涵体[3]。目前实验室常用的增加蛋白溶解度的方法有:①降低蛋白合成速度,比如降低诱导温度和IPTG浓度;②在裂解缓冲液中加入非离子型去污剂(如Triton X-100)或双性去污剂;③选用带有亲水标签的载体,比如pGEX-6p-1等[4]。
经CD-search预测,Rv2364c含有Era结构域和KH结构域,其应属于Era GTP酶家族。Era(E.coli Ras-like protein)是一种存在于所有细菌中的多功能GTP酶,能将GTP水解成GDP[5],其在16 s rRNA的加工和核糖体30 s小亚基的合成中扮演了重要角色[6]。大肠杆菌中的Era蛋白可能还参与了细胞周期的调控[7]和能量代谢[8],Era不止存在于原核生物,其同源蛋白也存在于哺乳动物中,称之为ERA蛋白,目前对其功能知之甚少[9]。Era的N端为GTP酶结构域,具有结合鸟苷酸和水解GTP的活性,C端为KH结构域。Era不属于Ras家族,因其C端存在KH结构域,这个结构域使其成为一个新的G蛋白亚家族。KH结构域是一种在真核生物和原核生物中都存在的核酸识别结构域,大小约为70个氨基酸残基[10], 能和单链核酸特异性结合[11]。含KH结构域的蛋白一般具有多种生物学功能,研究表明一些癌症副肿瘤综合征及脆性x染色体的发生可能与KH结构域的功能缺失有关[12]。结核分枝杆菌H37Rv的Rv2364c蛋白可能也具备以上功能。
本研究成功构建pET-28a-Rv2364c重组原核表达载体并表达出融合蛋白,通过亲和层析获得了纯度较高的目的蛋白,并对其进行了生物信息学分析,为进一步探索该蛋白在结核病致病过程中的作用及研究新抗结核分枝杆菌的治疗方法奠定了基础。
[1] Yuen CM,Amanullah F,Dharmadhikari A,et al.Turning off the tap: stopping tuberculosis transmission through active case-finding and prompt effective treatment[J].Lancet,2015,386(10010):2334-2343.
[2] Moore DA.What can we offer to 3 million MDRTB household contacts in 2016[J].BMC Med,2016(14):64-68.
[3] Singh A,Panda AK.Solubilization and refolding of inclusion body proteins[J].Methods Mol Biol,2015(1258):283-291.
[4] Young CL,Britton ZT,Britton ZT,et al.Recombinant protein expression and purification: a comprehensive review of affinity tags and microbial applications[J].Biotechnol J,2012,7(5):620-634.
[5] Wu YM,Zhang XN,Xin XH,et al.Soluble expression, purification and bioactivity of recombinant human Era protein[J].Chin J Cell Mol Immunol,2008,24(6):560-563.
[6] Meier TI,Peery RB,McAllister KA,et al.Era GTPase of Escherichia coli: binding to 16S rRNA and modulation of GTPase activity by RNA and carbohydrates[J].Microbiology,2000,146 (5):1071-1083.
[7] Inoue K,Chen J,Tan Q,et al.Era and RbfA have overlapping function in ribosome biogenesis in Escherichia coli[J].J Mol Microbiol Biotechnol,2006,11(1-2):41-52.
[8] Tu C,Zhou X,Tropea JE,et al.Structure of ERA in complex with the 3′ end of 16S rRNA: implications for ribosome biogenesis[J].Proc Natl Acad Sci U S A,2009,106(35):14843-14848.
[9] Diaz-Moreno I,Hollingworth D,Frenkiel TA,et al.Phosphorylation-mediated unfolding of a KH domain regulates KSRP localization via 14-3-3 binding[J].Nat Struct Mol Biol,2009,16(3):238-246.
[10]Tripathi BK,Surabhi S,Bhaskar PK,et al.The RNA binding KH domain of Spoonbill depletes pathogenic non-coding spinocerebellar ataxia 8 transcripts and suppresses neurodegeneration in Drosophila[J].Biochim Biophys Acta,2016,1862(9):1732-1741.
[11]Chen T,Cui P,Chen H,et al.A KH-domain RNA-binding protein interacts with FIERY2/CTD phosphatase-like 1 and splicing factors and is important for pre-mRNA splicing in Arabidopsis[J].PLoS Genet,2013,9(10):e1003875.
[12]Lewis HA,Musunuru K,Jensen KB,et al.Sequence-specific RNA binding by a Nova KH domain:implications for paraneoplastic disease and the fragile X syndrome[J].Cell,2000,100(3):323-332.
Expression, Purification and Bioinformatics Analysis of Rv2364c fromMycobacteriumTuberculosisH37Rv
XU Wan-ling, FU Chen-zeng
(Luohe Medical College, Luohe 462000, China)
ObjectiveTo obtain Rv2364c ofMycobacteriumtuberculosisH37Rv expressed in E. coli and predict its bioinformatics characters for the further development of anti-Mycobacterium tuberculosis drugs.MethodsThe target DNA fragment of the Rv2364c gene was amplified by PCR and cloned into the prokaryotic expression vector pET-28a. Then the recombinant vector (pET-28a-Rv2364c) was transformed into E.coliBL21(DE3). The expression of the His-Tag fusion protein was induced with isopropyl-β-D-thiogalactoside(IPTG). Finally, we purified the protein through affinity chromatography. We also used bioinformatic methods to analyze and predict this protein.ResultsDouble restriction enzyme analysis and sequencing technique proved that recombinant plasmid was constructed correctly, and the expression of the fusion protein in E.coli BL21(DE3) was detectable by SDS-PAGE. The target protein was gained after purified (purity>90%) using the affinity chromatographic column. Bioinformatics analysis revealed that Rv2364c was a stable and acidic protein;α-helix and random coil were the main components of its secondary structure; The main function domain types were Era domain and KH domain.ConclusionRv2364c had been successfully cloned, expressed and purified, the domain was predicted by bioinformatics method,which would be useful for further research on Rv2364c in the genesis and development of tuberculosis.
mycobacterium tuberculosis H37Rv;Rv2364c;transfection;bioinformatics
1672-688X(2017)01-0022-03
10.15926/j.cnki.issn1672-688x.2017.01.007
漯河医学高等专科学校基金(2014-S-LMC07)
2016-09-13
漯河医学高等专科学校,河南漯河 462000
徐宛玲(1965—),女,河南漯河人,教授,主任医师,从事临床教学科研工作。
Q811.4;R378.91+1
A

