基于16S rDNA的醋酸菌筛选及其发酵特性
姚洪礼,李兴江,2,郑 志,2,宋 山,姜绍通,2,李 顺,邓永东,潘丽军,2,吴学凤,2,*
(1.合肥工业大学食品科学与工程学院,安徽 合肥 230009;2.安徽省农产品精深加工重点实验室,安徽 合肥 230009)
基于16S rDNA的醋酸菌筛选及其发酵特性
姚洪礼1,李兴江1,2,郑 志1,2,宋 山1,姜绍通1,2,李 顺1,邓永东1,潘丽军1,2,吴学凤1,2,*
(1.合肥工业大学食品科学与工程学院,安徽 合肥 230009;2.安徽省农产品精深加工重点实验室,安徽 合肥 230009)
醋酸菌是影响纯种液态酿醋效率的关键因素。为获得高质量浓度醋酸生产菌,本研究将常规筛选方法与分子生物学技术相结合进行优良醋酸菌的筛选。采用高质量浓度酒精和醋酸培养基富集,挑取醋酸菌膜初步分离目的菌;借助生理生化特征与16S rDNA保守序列分析方法鉴定并获得一株产酸高的巴氏醋酸杆菌JST-S(Acetobacter pasteurianus JST-S,BJST-S);在与沪酿1.01(Acetobacter pasteurianus HN 1.01)进行发酵特性对比研究时发现,BJST-S在生长速率、产酸速率及耐醇和耐酸等方面均优于沪酿1.01;半连续发酵3 批次BJST-S的产酸量维持在58.10~59.68 g/L,发酵强度维持在1.21~1.24 g/(L·h),说明该菌产酸特性稳定。研究结果可为醋酸工业化生产以及优良菌株的选育提供一定的理论参考。
巴氏醋酸杆菌;菌膜;16S rDNA;半连续发酵
醋是我国传统发酵酿造的调味品之一[1],食醋酿造大多是以醋酸菌为主的多种菌混合发酵。目前纯种液态发酵酿醋已成为大规模工业化生产的趋势,菌种是影响酿醋品质的重要因素之一,耐醇、耐酸醋酸菌的选育已成为该领域的研究热点。由于醋醅[2]是多种菌(醋酸杆菌属、葡萄糖醋杆菌属、肠杆菌属和乳酸杆菌属)的混合体[3],用单纯的产酸为主的分离方法很难分离醋酸菌。此外,醋酸杆菌各种属[4](醋酸杆菌属、葡萄糖醋杆菌属、肠杆菌属和乳酸杆菌属)之间细胞形态和生理生化特征区别不明显,因此采用菌体形态观察和生理特性鉴定方法不够准确。
微生物染色体上编码16S rRNA相对应基因组DNA上的一段基因序列称为16S rDNA[5-7]。16S rDNA可以作为细菌群落结构分析最常用的系统进化标记分子[8]。随着分子分类理论和方法的日趋成熟,16S rDNA已经广泛应用于系统发生学和菌种鉴定研究[9-10]。在醋酸菌的选育与同源性鉴定方面[11-14]也有一定的应用。
本研究根据巴氏醋酸杆菌形成菌膜的特性,将生理生化特征鉴定与16S rDNA保守序列分析方法相结合,试图获得一株耐酸、耐醇、产酸高的醋酸菌,并将其与沪酿1.01(Acetobacter pasteurianus HN 1.01)进行发酵特性对比分析,以评价筛选菌的产酸能力,拟为工业化醋酸生产和优良菌株的选取提供一定的理论参考。
1 材料与方法
1.1 材料与试剂
1.1.1 菌株
醋醅购自烟台帝伯仕自酿机有限公司;沪酿1.01、大肠杆菌、金黄色葡萄球菌购于中国工业微生物菌种保藏管理中心,保藏于合肥工业大学农产品加工研究院实验室。
1.1.2 培养基
种子培养基(YG1):葡萄糖10 g/L、酵母膏5 g/L、MgSO41.1 g/L、K2HPO43.3 g/L,于0.1 MPa、121 ℃条件下蒸汽灭菌20 min,取出冷却,在无菌操作台接种,并加入2%(体积分数,下同)无水乙醇。
发酵培养基(YG2):葡萄糖10 g/L、酵母膏5 g/L、MgSO41.1 g/L、K2HPO43.3 g/L,于0.1 MPa、121 ℃条件下蒸汽灭菌20 min,取出冷却,接种前根据具体情况加入无水乙醇。
斜面、平板培养基(YG3):酵母提取物10 g/L、葡萄糖10 g/L、琼脂20 g/L,pH 4.5,于0.1 MPa、121 ℃条件下蒸汽灭菌20 min,使用前加3%无水乙醇,摆成斜面制成斜面保存培养基。
YPG培养基:酵母提取物5 g/L、蛋白胨2 g/L、葡萄糖3 g/L。
分离培养基:酵母提取物10 g/L、葡萄糖10 g/L、碳酸钙10 g/L、琼脂20 g/L,pH 4.5,于0.1 MPa、121℃条件下蒸汽灭菌20 min,使用前加3%无水乙醇分装无菌培养皿,制成碳酸钙平板分离培养基。
淀粉琼脂平板、乙醇利用培养基、甘油利用培养基均参考文献[15]配制。
1.1.3 试剂
十二烷基硫酸钠(sodium dodecyl sulfate,S D S)、蛋白酶K、Tr i s-H C l、乙二胺四乙酸(ethylenediaminetetraacetic acid,EDTA) 上海生物工程科技服务有限公司;苯酚-氯仿-异戊醇(25∶24∶1,V/V) 北京索莱宝科技有限公司;胶回收试剂盒 宝生物工程(大连)有限公司;无水乙醇、Triton X-100、Dupanol溶液、Mcllvaine缓冲液 国药集团化学试剂有限公司。
1.2 仪器与设备
恒温振荡培养箱 上海品顿实验设备公司;721紫外-可见分光光度计 江苏天瑞仪器股份有限公司;酒精计 衢州艾普计量仪器有限公司;JY92-Ⅱ超声波细胞粉碎仪 宁波新芝生物科技股份有限公司;聚合酶链式反应(polymerase chain reaction,PCR)仪、凝胶成像系统 美国Bio-Rad公司。
1.3 方法
1.3.1 发酵醋醅中菌体富集
称取固体醋醅样品2 g于50 mL pH 4的YG2中,32 ℃、150 r/min振荡培养1 d。取1/3培养液接种在含6%无水乙醇和4%乙酸的50 mL的YG2(pH 4)中,32 ℃、150 r/min振荡培养1 d。如此循环重复6 d,获得初筛醋酸菌富集培养液。
1.3.2 醋酸菌分离
初筛的富集培养液于32 ℃静置培养3 d,表面会形成一层菌膜,用接种环在培养基溶液表面轻轻挑取菌膜,涂布在分离培养基上,32 ℃培养48 h,挑取透明圈直径与菌落直径比值较大的单菌落,于YG3中划线保存[16]。
1.3.3 醋酸菌理化性质鉴定
采用在YPG上培养36 h后的醋酸产生菌及对照菌沪酿1.01进行生理生化反应,具体鉴定方法和依据参照《伯杰细菌鉴定册》[17]。
1.3.4 醋酸菌的16S rDNA鉴定
醋酸菌基因组DNA的提取参照文献[18-20]。目的菌株的16S rDNA基因扩增参照文献[21-22],引物序列为27F:5’-AGAGTTTGATCCTGGCTCAG-3’和1942R:5’-TACGGCTACCTTGTTACGACTT-3’。
以筛选菌株基因组为模板,克隆得到目的基因。通过宝生物试剂盒对PCR扩增产物进行纯化收集,经胶回收试剂盒纯化后的16S rDNA基因送交生工生物工程(上海)股份有限公司进行测序,用DNAStar软件将测序结果进行拼接,提交到NCBI与GenBank数据库中收录的菌株16S rDNA进行BLAST对比[23]。下载与待检菌株同源性较高的典型醋酸菌的16S rDNA核苷酸序列,用MEGA 2.5软件采用邻位相连法(neighbor-joining)构建系统进化树,根据基本的进化树拓扑结构,采用1 000次Bootstrap抽样对待检菌株的分类进化地位进行分析评价[24]。
1.3.5 培养与发酵
1.3.5.1 种子培养
将BJST-S和沪酿1.01接取1 环于50 mL YG1中,30 ℃条件下170 r/min培养,每隔6 h取样检测生物量。
1.3.5.2 醋酸发酵
接种BJST-S和沪酿1.01种子液按体积分数2%接种于YG2中,30 ℃、170 r/min培养,每隔6 h取样检测指标。
1.3.5.3 半连续发酵
醋酸发酵达到48 h时将发酵液全部取出,无菌条件下8 000×g离心10 min,将收集的菌体全部接入灭菌后含6%无水乙醇的YG3中,继续按上述条件发酵,如此循环操作,实现半连续发酵。每隔6 h取样检测指标。
1.3.6 指标测定
生物量的测定参照文献[25];产酸量的测定参照GB/T 12456—2008《食品中总酸的测定》指示剂法;乙醇脱氢酶(alcohol dehydrogenase,ADH)和乙醛脱氢酶(acetaldehyde dehydrogenase,ALDH)活力的测定参考文献[26-27]及Wood氏法[28];乙醇质量浓度的测定参照文献[29]。
2 结果与分析
2.1 醋酸菌的筛选与鉴定
2.1.1 理化性质的鉴定
按照1.3.1、1.3.2节菌株富集培养和分离的方法,将产生的菌膜涂布于分离培养基中培养,选取透明圈较大的优势单菌落保存,分别编号为1~10,并进行淀粉水解实验、乙醇利用实验、甘油利用实验、革兰氏染色实验及产酸定性实验,结果如表1所示。

表1 醋酸菌理化性质鉴定结果Table1 Physiological and biochemical characteristics of acetic acid bacterial isolates
依据《伯杰细菌鉴定手册》[17]与《常见细菌系统鉴定手册》[30]进行菌株生理生化特征鉴定,3号菌株能够利用乙醇和甘油,革兰氏染色为阴性且能够产酸,如表1所示,结合巴氏醋酸杆菌产生菌膜的特性可以初步确定3号菌株是巴氏醋酸杆菌。
2.1.2 16S rDNA的鉴定
2.1.2.1 基因片段提取和PCR扩增结果

图1 3号醋酸菌的16S rDNA扩增基因片段Fig.1 Agarose gel electrophoresis analysis of PCR-amplif i ed 16S rDNA gene fragments from acetic acid bacterial strain No. 3
对筛选菌株16S rDNA扩增结果进行凝胶电泳检测,如图1所示,16S rDNA泳道显示扩增基因大小在1 500 bp左右,结合相关参考文献[31-32]可知,该基因片段符合实验预计结果。
2.1.2.2 核苷酸序列分析

图2 3号醋酸菌的16S rDNA核苷酸序列Fig.2 16S rDNA Nucleotide sequences of acetic acid bacterial strain No. 3
经过筛选和鉴定的3号菌株的16S rDNA序列(图2)上传到NCBI数据库中,对比结果如表2所示,发现3号菌株的16S rDNA序列与Acetobacter属中编码为IFO 3283-01的Acetobacter pasteurianus IFO 3283-01的16S rDNA序列有很高的相似度,达到97%。Weisburg等[21]研究表明:16S rDNA可变序列的相似度超过97%即为同一属,达到99%即为同一种。因此可以确定筛选所得菌株为巴氏醋酸杆菌。
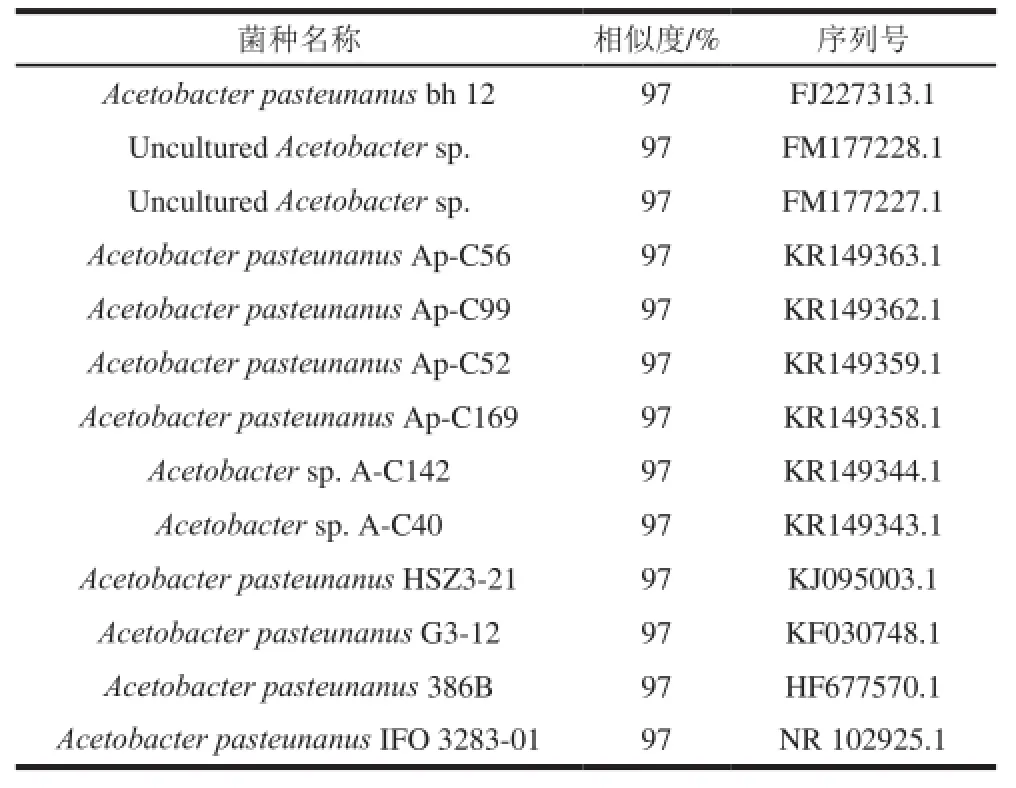
表2 3号醋酸菌的16S rDNA与NCBI的对比结果Table2 Results of 16S rDNA sequence alignment of acetic acid bacterial strain No. 3
2.1.2.3 系统进化树

图3 基于16S rDNA序列采用NJ法构建的系统进化树Fig.3 Phylogenetic tree based on 16S rDNA sequences constructed by the neighbor-joining method
如图3所示,由系统进化树显示所筛选的3号菌株与Acetobacter pasteurianus IFO 3283-01菌株处于同一分支,经过多次测试分析支持该分支;在NCBI数据库中,与从传统日本米醋发酵表面分离出的参考菌株Acetobacter pasteurianus IFO 3283-01有很高的相似度。综合菌落形态学特征、生理生化特征实验结果、16S rDNA序列同源性比较及系统进化树分析结果,鉴定筛选的3号菌株为醋酸杆菌属(Acetobacter),将该菌命名为巴氏醋酸杆菌JST-S(Acetobacter pasteurianus JST-S,BJST-S)。
2.2 BJST-S和沪酿1.01的发酵特性对比
2.2.1 BJST-S和沪酿1.01生长曲线
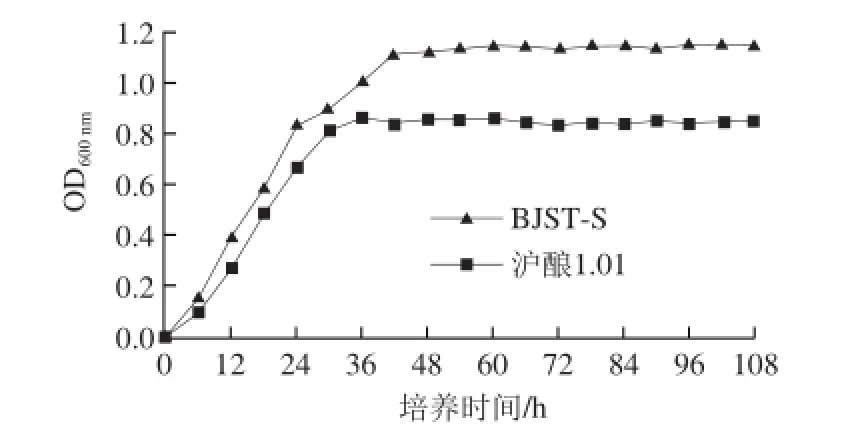
图4 BJST-S和沪酿1.01的生长曲线Fig.4 Growth curves of BJST-S and HN 1.01
由图4可知,BJST-S和沪酿1.01的生长曲线相似,在0~36 h为对数生长期,但是BJST-S菌种的OD600nm增长速率大于菌种沪酿1.01,说明在相同时间内BJST-S菌种生长速率高于沪酿1.01。36 h后则进入生长稳定期,且最终BJST-S菌种的OD600nm明显高于菌种沪酿1.01。处于对数生长期的菌种活性最高,对外界因素敏感,此时接种可使菌体活性最高,因此选取种子培养时间为24 h。
2.2.2 不同无水乙醇用量BJST-S和沪酿1.01产酸对比

图5 不同无水乙醇用量条件下沪酿1.01(A)和BJST-S(B)产酸对比Fig.5 Acid production of HN 1.01 (A) and BJST-S (B) at different initial alcohol concentrations
由图5可知,无水乙醇用量从0%增加到6%,沪酿1.01和BJST-S产酸量都有明显增加,且在6%乙醇体积分数时测得发酵液产酸量最高,沪酿1.01最高产酸量达到50.80 g/L,BJST-S的最高产酸量达到了53.88 g/L。当底物无水乙醇用量增加至8%时,测得发酵液中总酸量下降且二者差异明显,BJST-S的产酸达到24.57 g/L,而沪酿1.01只有15.04 g/L。这是由于酒精有抑制细菌生长的作用,在发酵初期,无水乙醇用量过高反而会抑制醋酸菌的繁殖代谢,使产酸量增长变缓甚至下降。通过比较可知,BJST-S对乙醇耐受性要高于沪酿1.01,而在最优无水乙醇用量条件下,BJST-S的产酸能力略高于沪酿1.01的产酸能力。
2.2.3 不同接种量BJST-S和沪酿1.01产酸对比

图6 不同接种量条件下沪酿1.01(A)和BJST-S(B)产酸对比Fig.6 Acid production of HN 1.01 (A) and BJST-S (B) under different inoculums sizes
采用6%无水乙醇的YG2培养基进行不同接种量BJST-S和沪酿1.01产酸对比研究,结果如图6所示。由图6可知,在所选接种量区间中,两种醋酸菌的产酸量均随接种量的升高有缓慢增加。当接种量为2%时,两者产酸量均在84 h达到峰值;接种量为10%时,沪酿1.01在48 h接近峰值,而BJST-S在60 h达到峰值,且测得沪酿1.01总酸为53.74 g/L,BJST-S总酸57.48 g/L。这是因为当接种量较少时,乙醇对菌种的生长起到一定的抑制作用,使得菌种生长和产酸速率缓慢,峰值出现迟缓;当接种量为10%时,环境含氧量为抑制菌体生长的主要影响因素,呼吸强度较高的BJST-S对环境含氧量较敏感,三角瓶中氧含量不足时,更不利于菌种BJST-S生长繁殖和代谢,导致BJST-S最高产酸量峰值迟于沪酿1.01。通过比较可知,在最优接种量条件下,BJST-S的产酸量高于沪酿1.01,沪酿1.01最高产酸量为53.74 g/L,BJST-S最高产酸量为57.48 g/L。
2.2.4 不同初始醋酸含量BJST-S和沪酿1.01产酸对比

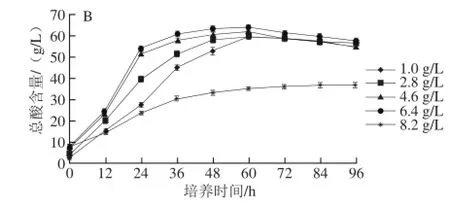
图7 不同初始醋酸含量下的沪酿1.01(A)和BJST-S(B)产酸对比Fig.7 Acid production of HN 1.01 (A) and BJST-S (B) under different initial acetic acid concentrations
分别向含6%无水乙醇的YG2培养基中接入10%种子培养液,进行不同初始醋酸含量BJST-S和沪酿1.01产酸对比研究结果如图7所示。由图7可知,初始醋酸含量从1.0 g/L增加到8.2 g/L的过程中,沪酿1.01的产酸速率和产酸量升高,当初始醋酸含量超过4.6 g/L时产酸速率和产酸量则有所下降;而BJST-S在初始醋酸含量从1.0 g/L增加到6.4 g/L的过程中产酸速率和产酸量升高,在6.4 g/L初始醋酸含量总酸含量最大;沪酿1.01的最大总酸含量为61.04 g/L,BJST-S的最大总酸含量为64.09 g/L;当初始醋酸含量超过6.4 g/L时产酸速率和总酸含量则有所下降。说明BJST-S和沪酿1.01在过酸、碱性环境下不宜生长繁殖和代谢,且机体中的大多数反应与酶有关,过酸、过碱都会导致酶活力降低,从而导致产酸速率和产酸量下降;同时,也说明了在相同发酵条件下BJST-S的生长繁殖和代谢速率快且对乙酸的耐受力强。
2.2.5 BJST-S、沪酿1.01的ADH和ALDH活力对比

图8 BJST-S和沪酿1.01的ADH/ALDH活性对比Fig.8 ADH/ALDH activities of BJST-S and HN 1.01
由图8可知,2 种菌种的ADH和ALDH活力存在差异性,但在不同无水乙醇用量条件下的变化趋势一致。在无乙醇情况下,2 种菌种的ADH和ALDH活力较低。无水乙醇用量在2%~4%间,随着培养基中无水乙醇用量的升高,ADH和ALDH活力也提高,并在4%时两种菌株酶活力达到最高,且沪酿1.01和BJST-S的ADH活力分别为3.50、3.72 U/mg,ALDH活力分别为2.25、2.48 U/mg,且BJST-S的ADH及ALDH活力明显高于沪酿1.01。当无水乙醇用量在4%~8%间,2 种菌种的ADH及ALDH活性水平均下降。这与上述菌体的生长代谢一致,在低无水乙醇用量条件下,菌体产酸速率相对较快,表现为最先达到最高产酸量峰值;但在高用量无水乙醇胁迫下,菌种需要一定的时间适应生长环境,而导致产酸速率相对较慢,表现为延迟发酵周期。
2.2.6 BJST-S半连续发酵稳定性
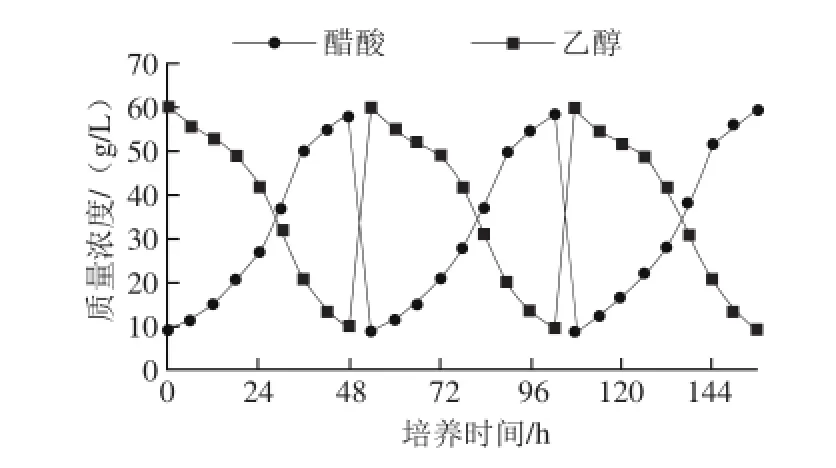
图9 BJST-S的批次摇瓶发酵过程中的变化曲线Fig.9 Time-course curves of batch fermentation of BJST-S in shaking fl asks
由图9可知,发酵在0~36 h阶段,产酸量呈线性上升,乙醇质量浓度降低;48 h时产酸量最大,为58.10 g/L,此时产酸强度为1.21 g/(L·h);第2批次发酵48 h,最大产酸量为58.60 g/L,此时产酸强度为1.22 g/(L·h);第3批次发酵48 h,最大产酸量为59.68 g/L,此时产酸强度为1.24 g/(L·h)。在半连续发酵3 批次中,BJST-S产酸量维持在58.10~59.68 g/L,发酵强度维持在1.21~1.24 g/(L·h),半连续发酵过程中产酸量和产酸强度变化趋势基本一致,说明BJST-S产酸稳定性良好。
3 结 论
本研究利用醋酸发酵过程中巴氏醋杆菌形成菌膜的特性,对醋醅中混合菌进行初步分离,采用生理生化特征与16S rDNA保守序列分析相结合的方法对分离出的醋酸产生菌进行鉴定,获得了BJST-S菌株;在与沪酿1.01进行发酵特性对比研究中,BJST-S菌株具有良好的生长特性和发酵产酸能力,且耐醇及耐酸特性优于沪酿1.01;在3批次半连续发酵中,BJST-S产酸量维持在58.10~59.68 g/L,发酵强度维持在1.21~1.24 g/(L·h),说明该菌产酸特性稳定,可进一步进行菌种选育,为纯种液态酿造食醋的工业化生产提供依据。
[1] 孙欣瑶. 麦芽醋醋酸菌株筛选及发酵工艺条件的优化[D]. 哈尔滨:东北农业大学, 2013: 1-2. DOI:10.7666/d.Y2295929.
[2] 郭明烨. 四川麸醋醋醅中一株乳酸菌的筛选及鉴定[J]. 中国调味品, 2016, 41(5): 23-29. DOI:10.3969/j.issn.1000-9973.2016.05.005.
[3] 邓红梅, 周如金, 王春, 等. 香蕉醋混菌发酵酿造及香气成分分析[J]. 中国调味品, 2013, 38(2): 96-99. DOI:10.3969/ j.issn.1000-9973.2013.02.027.
[4] 高鑫, 梅俊, 李博. 开菲尔产品微生态、成分和功能活性的研究进展[J]. 食品与发酵工业, 2016, 42(3): 243-250. DOI:10.13995/ j.cnki.11-1802/ts.201603043.
[5] FANG R S, DONG Y C, CHEN F, et al. Bacterial diversity analysis during the fermentation processing of traditional Chinese yellow rice wine revealed by 16S rDNA 454 pyrosequencing[J]. Journal of Food Science, 2015, 80(10): M2265-M2271. DOI:10.1111/1750-3841.13018.
[6] LOONG S K, KHOR C S, JAFAR F L, et al. Utility of 16S rDNA sequencing for identif i cation of rare pathogenic bacteria[J]. Journal of Clinical Laboratory Analysis, 2016, 30(6): 1056-1060. DOI:10.1002/ jcla.21980.
[7] YUAN Z S, LI F, ZHAN G F. Isolation of culturable endophytic bacteria from Moso bamboo (Phyllostachys edulis) and 16S rDNA diversity analysis[J]. Archives of Biological Sciences, 2015, 67(3): 1001-1008. DOI:10.2298/ABS141212063Y.
[8] POBLET M, ROZÈS N, GUILLAMÓN J M, et al. Identification of acetic acid bacteria by restriction fragment length polymorphism analysis of a PCR-amplified fragment of the gene coding for 16S rRNA[J]. Letters in Applied Microbiology, 2000, 31(1): 63-67. DOI:10.1046/j.1472-765x.2000.00765.x.
[9] 张朝正, 郭兰珍. 利用16S rDNA序列分析和Biolog快速鉴定方法鉴定产脂肪酶菌株[J]. 河北工业大学学报, 2009, 38(5): 52-55. DOI:10.3969/j.issn.1007-2373.2009.05.012.
[10] GREMION F, CHATZINOTAS A, HARMS H. Comparative 16S rDNA and 16S rRNA sequence analysis indicates that Actinobacteria might be a dominant part of the metabolically active bacteria in heavy metal-contaminated bulk and rhizosphere soil[J]. Environmental Microbiology, 2003, 5(5): 896-907. DOI:10.1046/ j.1462-2920.2003.00484.x.
[11] 陈洋, 汪超, 高冰, 等. 高耐受性醋酸菌的筛选及发酵特性研究[J]. 中国酿造, 2015, 35(12): 28-33. DOI:10.11882/ j.issn.0254-5071.2015.12.007.
[12] 邵向丽, 赵爽, 刘书亮, 等. 四川麸醋醋醅中优良醋酸菌的筛选及其产酸特性[J]. 食品工业科技, 2015, 36(6): 203-207. DOI:10.13386/ j.issn1002-0306.2015.06.037.
[13] CHEN Y, YE B, LI D, et al. Screening and characterization of ethanoltolerant and thermotolerant acetic acid bacteria from Chinese vinegar Pei[J]. World Journal of Microbiology & Biotechnology, 2016, 32(1): 1-9. DOI:10.11882/j.issn.0254-5071.2015.12.007.
[14] 李大为, 朱运平, 张雪, 等. 自然发酵的苹果醋中醋酸菌的分离鉴定[J]. 中国食品添加剂, 2015(8): 165-173. DOI:10.3969/ j.issn.1006-2513.2015.08.021.
[15] 张云茹. 乙醇脱氢酶菌株的选育及其动力学特性研究[D]. 重庆: 重庆大学, 2004: 12-17. DOI:10.7666/d.y703788.
[16] 钟浩, 王亮, 刘克营, 等. 开菲尔粒中主要组成菌的分离鉴定及微囊化纯培养混合发酵指标分析[J]. 食品工业科技, 2016, 37(5): 148-153. DOI:10.13386/j.issn1002-0306.2016.05.020.
[17] 布坎南. 伯杰细菌鉴定手册[M]. 北京: 科学出版社, 1984: 729-830.
[18] MAMLOUK D, HIDALGO C, TORIJA M J, et al. Evaluation and optimisation of bacterial genomic DNA extraction for no-culture techniques applied to vinegars[J]. Food Microbiology, 2011, 28(7): 1374-1379. DOI:10.1016/j.fm.2011.06.009.
[19] UNNO H, INADA M, NAKAMURA A, et al. Improved rapid and efficient method for Staphylococcus aureus DNA extraction from milk for identif i cation of mastitis pathogens[J]. Journal of Veterinary Medical Science, 2015, 77(8): 1007-1009. DOI:10.1292/jvms.14-0159.
[20] GAMBA C, HANGHØJ K, GAUNITZ C, et al. Comparing the performance of three ancient DNA extraction methods for highthroughput sequencing[J]. Molecular Ecology Resources, 2016, 16(2): 459-469. DOI:10.1111/1755-0998.12470.
[21] WEISBURG W G, BARNS S M, PELLETIER D A, et al. 16S ribosomal DNA amplification for phylogenetic study[J]. Journal of Bacteriology, 1991, 173(2): 697-703. DOI:10.1128/jb.173.2.697-703.
[22] BROSIUS J, NOLLER H F. Complete nucleotide sequence of a 16S ribosomal RNA gene from Escherichia coli[J]. Proceedings of the National Academy of Sciences of the United States of America, 1978, 75(10): 4801-4805. DOI:10.1073/pnas.75.10.4801.
[23] ALTSCHUL S F, MADDEN T L. Gapped BLAST and PSI-BLAST: a new generation of protein database search programs[J]. Nucleic Acids Research, 1997, 25(17): 3389-3402. DOI:10.1093/nar/25.17.3389.
[24] TAMURA K, PETERSON D, PETERSON N, et al. MEGA5: molecular, evolutionary, genetics, analysis, using maximum, likelihood, evolutionary, distance, and maximum, parsimony, methods[J]. Molecular Biology & Evolution, 2011, 28(10): 2731-2739. DOI:10.1093/molbev/msr121.
[25] 马勇, 樊永军. 用OD值监测产油酵母培养过程中的菌体生物量变化[J]. 安徽农业科学, 2011, 39(12): 7342-7343. DOI:10.3969/ j.issn.0517-6611.2011.12.154.
[26] 胡雪桃, 朱瑶迪, 邹小波, 等. 近红外光谱技术快速预测镇江香醋乙醇脱氢酶活性的研究[J]. 中国调味品, 2016, 41(2): 20-25. DOI:10.3969/j.issn.1000-9973.2016.02.004.
[27] 程书兰, 魏淼, 许琳, 等. 醇脱氢酶的纯化、酶学性质及其不对称催化[J]. 生物加工过程, 2016, 14(1): 8-13. DOI:10.3969/ j.issn.1672-3678.2016.01.002.
[28] 朱小明, 夏小乐, 杨海麟, 等. 巴氏醋酸杆菌沪酿1.01乙醇氧化产醋酸关键酶的研究[J]. 食品工业科技, 2013, 34(2): 167-170. DOI:10.13386/j.issn1002-0306.2013.02.092.
[29] 张琴. 工业甲醇中微量乙醇分析方法改进[J]. 中国化工贸易, 2014, 6(20): 164-164. DOI:10.3969/j.issn.1674-5167.2014.20.135.
[30] 蔡妙英, 东秀珠. 常见细菌系统鉴定手册[M]. 北京: 科学出版社, 2001: 30-36.
[31] 朱飞舟, 陈利玉, 陈汉春. 16S rRNA基因序列分析法鉴定病原细菌[J].中南大学学报(医学版), 2013, 38(10): 1035-1041. DOI:10.3969/ j.issn.1672-7347.2013.10.010.
[32] MIYAJIMA M, MATSUDA M, HAGA S, et al. Cloning and sequencing of 16S rDNA and 16S-23S rDNA internal spacer region (ISR) from urease-positive thermophilic Campylobacter (UPTC)[J]. Letters in Applied Microbiology, 2002, 34(4): 287-289. DOI:10.1046/ j.1472-765x.2002.01082.x.
Screening and Fermentation Characteristics of an Acetic Acid Bacterial Strain Based on 16S rDNA for Vinegar Production
YAO Hongli1, LI Xingjiang1,2, ZHENG Zhi1,2, SONG Shan1, JIANG Shaotong1,2, LI Shun1, DENG Yongdong1, PAN Lijun1,2, WU Xuefeng1,2,*
(1. School of Food Science and Engineering, Hefei University of Technology, Hefei 230009, China; 2. Key Laboratory for Agricultural Products Processing of Anhui Province, Hefei 230009, China)
Acetic acid bacteria are an important factor that inf l uences the eff i ciency of vinegar production by pure culture liquid-state fermentation. The goal of this study was to obtain an outstanding acetic acid bacterial strain for vinegar production by routine screening combined with molecular biological techniques. The target strain was isolated from the biof i lm formed in enrichment culture in the presence of high concentrations of alcohol and acetic acid. It was identif i ed as Acetobacter pasteurianus JST-S (BJST-S) based on physiological and biochemical characteristics combined with 16S rDNA sequence analysis. Comparative analysis of fermentation characteristics indicated that the BJST-S was better than Acetobacter pasteurianus CICC 20001 in term of growth rate, acid production rate, alcohol resistance and acid resistance. The concentration of acetic acid was maintained in the range of 58.10–59.68 g/L and the fermentation strength in the range of 1.21–1.24 g/(L·h) in three batches of semi-continuous fermentation, implicating that the fermentation characteristics of BJST-S were stable. Our fi ndings in this work may provide a valuable reference for strain breeding for the improvement of industrial vinegar production.
Acetobacter pasteurianus; bacterial biof i lm; 16S rDNA; semi-continuous fermentation
10.7506/spkx1002-6630-201704002
TS255.1
A
1002-6630(2017)04-0006-07
姚洪礼, 李兴江, 郑志, 等. 基于16S rDNA的醋酸菌筛选及其发酵特性[J]. 食品科学, 2017, 38(4): 6-12. DOI:10.7506/ spkx1002-6630-201704002. http://www.spkx.net.cn
YAO Hongli, LI Xingjiang, ZHENG Zhi, et al. Screening and fermentation characteristics of an acetic acid bacterial strain based on 16S rDNA for vinegar production[J]. Food Science, 2017, 38(4): 6-12. (in Chinese with English abstract)
DOI:10.7506/spkx1002-6630-201704002. http://www.spkx.net.cn
2016-06-28
国家自然科学基金青年科学基金项目(31601465);安徽省自然科学基金面上项目(1408085MKL17);安徽省科技重大专项(15CZZ03100);合肥工业大学青年创新项目(JZ2014HGQC0124)
姚洪礼(1992—),男,硕士,研究方向为食品微生物。E-mail:yaohongli-@hotmail.com
*通信作者:吴学凤(1981—),女,副教授,博士,研究方向为食品发酵工程。E-mail:applewuxf@hotmail.com

