2-型猪链球菌保护性抗原ESA的细胞定位及B细胞表位预测
刘丽娜,钟乃凤,王 赟,张 洁,周 静
(贵州医科大学生物与工程学院,贵阳 550025)
·简报·
2-型猪链球菌保护性抗原ESA的细胞定位及B细胞表位预测
通过生物信息学方法预测2-型猪链球菌(Streptococcus suis serotype 2,SS2)保护性抗原ESA(epidermal surface antigen, ESA)在SS2中的分布和B细胞表位,为研究SS2的多表位疫苗奠定基础。以ESA推定的氨基酸序列为基础,通过生物信息学软件对ESA进行信号肽、跨膜序列等分析,然后分别以Kyte-Doolittle法、Emini法和Jameson-Wolf法分析蛋白的亲水性、表面可能性以及抗原指数;辅以Garnier-Robson法、Chou-Fasman法和Karplus-Schulz法分析蛋白二级结构中柔性区域,进而预测ESA的B细胞表位。结果表明,ESA为跨膜蛋白,B细胞表位位于N-端第30~35、59~64、178~185、245~253区域。多参数或多个软件联合使用能预测SS2保护性抗原ESA的B细胞表位和细胞定位,为实验研究提供帮助,大大缩减实验时间。
2-型猪链球菌;ESA;细胞定位;B细胞表位
刘丽娜,钟乃凤,王 赟,张 洁,周 静
(贵州医科大学生物与工程学院,贵阳 550025)
多表位疫苗(multi-epitope vaccine)是近年发展起来的一种新型分子疫苗[1],是指同时携带多个目标抗原相关以及辅助性的表位的疫苗,也称鸡尾酒式疫苗。它采用的是一种独特的疫苗设计思路,其中只含有抗原中能有效刺激免疫应答的表位,无关部分均被去除。相比传统疫苗,多表位疫苗更安全、无毒、稳定,可以直接刺激机体产生特异性免疫反应。在疫苗设计时,从多个抗原分子的特异性保守氨基酸区域选择多个B细胞表位,构建多表位疫苗可以诱导更广泛且有效的免疫应答,以提供不同分离株之间的交叉免疫保护[2,3]。发展多表位疫苗有利于提供更完全更有针对性的免疫保护。另外,对抗原表位的研究,将深层次地揭示蛋白抗原诱导免疫应答的分子机制。在前期研究工作中,证实免疫小鼠2型猪链球菌(Streptococcus suis serotype 2,SS2)的表皮表面抗原(epidermal surface antigen, ESA)[4-8]能产生高效价抗血清,免疫小鼠能抵抗高致病力菌株的致死性攻击,是SS2新的保护性抗原[9]。
筛选表位的方法主要包括酶解法、化学法、串联重复多肽合成法、噬菌体展示肽库技术、蛋白质一级结构预测法和MHC单链分子结合法等[10]。其中,蛋白质结构预测法具有快速、直观等优点,广泛应用于表位疫苗研究[11,12]。本文对2型猪链球菌的新保护性抗原SS2的亲性、表面可能性、抗原些粒等蛋白质一级结构及亲性区域的二级结构进行了预测分析。
1 材料与方法
1.1 ESA的氨基酸序列 SS2全基因组序列测定和推定氨基酸序列参见文献[13],ESA含有489个氨基酸残基。
1.2 ESA在SS2中的细胞定位预测 利用跨膜螺旋结构预测软件TMHMM Server 2.0、信号肽预测软件SignalP 4.1 Server和非经典分泌蛋白预测软件SecretomeP对ESA的氨基酸序列进行分析。
1.3 ESA的二级结构柔性区域预测 用Garnier-Robson法[17]、Chou-Fasman法[18]和Karplus-Schulz法[19]预测ESA二级结构中的柔性区域。
1.4 ESA的B细胞表位预测 分别用Kyte-Doolittle法[14]、Emini法[15]和Jameson-Wolf法[16]分析蛋白的亲水性、表面可能性以及抗原性指数。综合以上各方法的预测结果,推测ESA的B细胞抗原表位。
2 结果与讨论
2.1 ESA的细胞定位预测 利用SignalP 4.1 Server分析ESA的氨基酸序列,发现其N端不存在信号肽序列(图1);使用SecretomeP对ESA的氨基酸序列进行分析,其SecP分值为0.100835(<0.5),ESA为非分泌蛋白;利用TMHMM Server 2.0分析ESA的氨基酸序列,发现ESA为跨膜蛋白,其氨基酸在1~6区域为胞内序列,7~29区域为跨膜序列,30~489区域为胞外序列(图2)。
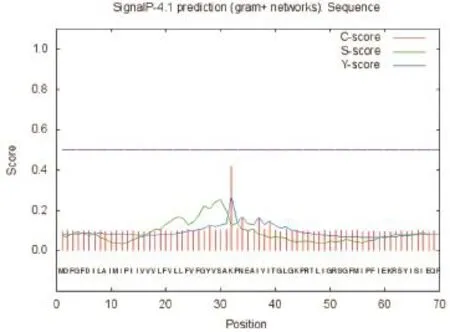
图1 ESA信号肽预测Fig. 1 Signal peptide prediction of ESA
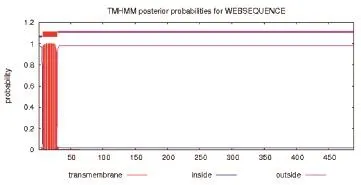
图2 ESA跨膜区域预测Fig. 2 Transmembrane region prediction of ESA
2.2 ESA蛋白预测的二级结构柔性区域 Garnier-Robson法、Chou-Fasman法和Karplus-Schulz法预测的结果见图3、图4和图5。综合分析图3、图4和图5,同时被三种方法预测为柔性结构的区域位于ESA蛋白N-端的第32~33、44~46、50-54、62~64、180~182、250~251。蛋白质的二级结构与蛋白质的表位分布有较大关系。蛋白抗原构象不是刚性不变的,其多肽骨架有一定程度的活动性;转角和无规则卷曲区域的结构比较松散,易于发生扭曲、盘旋,并展示在蛋白分子表面,成为抗原表位的可能性较大。

图3 Garnier-Robson法预测的ESA蛋白的二级结构柔性区域Fig. 3 Flexible regions of ESA predicted by the Garnier-Robson’s method

图4 Chou-Fasman法预测的ESA蛋白的二级结构柔性区域Fig. 4 Flexible regions of ESA predicted by the Chou-Fasman’s method

图5 Karplus-Schulz法预测的ESA蛋白的柔性区域Fig. 5 Flexible regions of ESA predicted by the Karplus-Schulz’s method
2.3 ESA蛋白的B细胞表位预测结果 蛋白抗原各氨基酸残基可分为亲水残基和疏水残基两类。在机体内,疏水性残基一般埋在蛋白内部,而亲水性残基位于表面,因此蛋白的亲水部位与蛋白的抗原位点有着密切的联系。表面可能性较高区域, 位于蛋白分子表面;表面可能性较低区域,即埋藏于蛋白分子内部区域。抗原性方案认为疏水性氨基酸残基也经常出现在表位中, 疏水性残基和亲水性残基在蛋白抗原位点中的相对出现率对抗原表位的定位预测有一定意义[20]。采用Kyte-Doolittle法分析ESA的亲水性,Emini法预测ESA的表面可能性,以及Jameson-Wolf法预测ESA的抗原性指数,结果见图6~8。经亲水性指数、表面可能性指数和抗原性指数筛选出的氨基酸区段中(亲水性指数≥0,表面可能性指数≥1,抗原性指数≥0),如果其内部或附近又有柔性结构,则很有可能为B细胞表位所在区域。综合各种预测结果,ESA蛋白N-端第30~35、59~64、178~185、245~253区段很有可能存在B细胞表位。
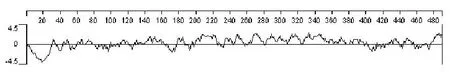
图6 ESA蛋白的亲水性分析Fig. 6 Hydrophilicity plot for ESA

图7 ESA蛋白的表面可能性区域Fig. 7 Surface probability plot for ESA

图8 ESA蛋白的抗原性指数Fig. 8 Antigenicity index for ESA
因此,本研究将蛋白亲水性、表面可能性以及抗原指数的预测结果与二级结构中柔性区域的分析相结合,综合判定ESA的B细胞表位所在区段。新的保护性抗原ESA B细胞表位的预测将为SS2多表位疫苗的研究奠定基础。
[1] Currier J R, Robb M L, Michael N L, et al. Defining epitope coverage requirements for T cell-based HIV vaccines: theoretical considerations and practical applications[J]. J Transl Med, 2011, 9: 212.
[2] Wang F, Feng X, Zheng Q, et al. Multiple linear epitopes (B-cell, CTL and Th) of JEV expressed in recombinant MVA as multiple epitope vaccine induces a protective immune response[J]. Virol J, 2012, 9: 204.
[3] Ye X, Ku Z, Liu Q, et al. Chimeric virus-like particle vaccines displaying conserved enterovirus 71 epitopes elicit protective neutralizing antibodies in mice through divergent mechanisms[J]. J Virol, 2014, 88(1): 72-81.
[4] Schroeder W T, Stewart-Galetka S, Mandavilli S, et al.Cloning and characterization of a novel epidermal cell surface antigen (ESA) [J]. J Biol Chem, 1994, 269(31): 19983-19991.
[5] Bickel P E, Scherer P E, Schnitzer J E, et al. Flotillin and epidermal surface antigen define a new family of caveolae-associated integral membrane proteins[J]. J Biol Chem, 1997, 272(21): 13793-13802.
[6] Schroeder W T, Siciliano M J, Stewart-Galetka S L, et al.The human gene for an epidermal surface antigen (M17S1)is located at 17q11-12[J]. Genomics, 1991, 11(2): 481-482.
[7] Hazarika P, Dham N, Patel P, et al. Flotillin 2 is distinct from epidermal surface antigen (ESA) and is associated with filopodia formation[J]. J Cell Biochem, 1999, 75(1): 147-159.
[8] Diaz LA, Marcelo CL. Pemphigoid and pemphigus antigens in cultured epidermal cells[J]. Br J Dermatol, 1978, 98(6): 631-637.
[9] Liu L, Cheng G, Wang C, et al. Identification and experimental verification of protective antigens against Streptococcus suis serotype 2 based on genome sequence analysis[J]. Curr Microbiol, 2009, 58(1): 11-17.
[10] 张中旺, 张永光. 口蹄疫表位疫苗的研究进展[J]. 中国人兽共患病学报, 2008, 24(6): 570-573.
[11] 云涛, 刘光清, 冷青文, 等. 口蹄疫病毒结构蛋白的二级结构及其B细胞抗原表位的预测[J]. 中国兽医科技, 2004, 34(7): 23-28.
[12] 吕燕波, 万瑛, 吴玉章. SARS病毒S蛋白的B细胞表位预测[J]. 第三军医大学学报, 2004, 26(2): 101-103.
[13] Chen C, Tang J, Dong W, et al. A glimpse of streptococcal toxic shock syndrome from comparative genomics of S. suis 2 Chinese isolates[J]. PloS ONE, 2007, 2(3): e315.
[14] Garnier J, Osguthorpe D J, Robson B. Analysis of the accuracy and implications of simple method for predicting the secondary structure of globular proteins[J]. J Mol Biol, 1978, 120(1): 97-120.
[15] Chou P Y. Prediction of protein structure and the principles of protein conformation[M]. New York: Plenum Press, 1990. 549-586.
[16] Karplus P A, Schulz G E. Prediction of chain flexibility in proteins[J]. Naturwissenschaften, 1985, 72(4): 212-213.
[17] Kyte J , Doolittle R F. A simple method for displaying the hydropathic character of a protein[J]. J Mol Biol, 1982, 157(1): 105-132.
[18] Emini E A, Hughes J V, Perlow D S, et al. Induction of hepatitis A virus-neutralizing antibody by a virus-specfic synthetic peptide[J]. J Virol, 1985, 55(3): 836-839.
[19] Jameson B A, Wolf H. The antigenic index: A novel algorithm for predicting antigenic determinants[J].Comput Appl Biosci, 1988, 4(1): 181-186.
[20] 杨清浩, 王详卫, 金燕, 等. MUC1抗原的B细胞表位预测[J]. 第三军医大学学报, 2005, 27(5): 406-409.
PREDICTION OF CELLULAR LOCALIZATION AND B CELL EPITOPES OF PROTECTIVE ANTIGEN ESA OF STREPTOCOCCUS SUIS SEROTYPE 2
LIU Li-na, ZHONG Nai-feng, WANG Yun, ZHANG Jie, ZHOU Jing
(School of Biology&Engineering, Guizhou Medical University, Guiyang 550025, China)
The cellular location and B cell epitopes of the protective antigen ESA (epidermal surface antigen, ESA) of Streptococcus suis serotype 2 (S. suis 2) were predicted for the development of a multi-epitope vaccine using bioinformatics. Putative amino sequence of ESA was analyzed using bioinformatics software containing signal peptide and transmembrane region prediction. The secondary structural fl exible regions of ESA were analyzed by using the Garnier-Robson's method, Chou-Fasman's method and Karplus-Schulz's method based on its putative amino sequence. The hydrophilicity plot, surface probability plot and antigenicity index were obtained by using the methods of Kyte-Doolittle, Emini and Jameson-Wolf, respectively. Combined with these results, ESA was predicted as a transmembrane protein and amino acids 30~35, 59~64, 178~185, 245~253 at the N-terminus might represent possible B cell epitopes for ESA. In conclusion, the cellular localization and B cell epitopes of ESA were predicted based on multiple parameters and softwares, which would benefi t design and future research of a multi-epitope vaccine.
Streptococcus suis serotype 2; epidermal surface antigen; localization; B cell epitope
S852.611
B
1674-6422(2016)05-0069-05
2016-04-26
贵州省科技合作计划(黔科合LH字[2015]7318)
刘丽娜,女,博士,主要从事微生物学方面研究
刘丽娜,E-mail:strawberry2@126.com

