陆地棉NF-YB基因家族的全基因组分析
刘 静,徐珍珍,袁 娜,郭 月,张保龙,杜建厂
(1.江苏省农业科学院 农业生物技术研究所,江苏省农业生物学重点实验室,江苏 南京 210014;2.江苏省农业科学院 经济作物研究所,农业部长江下游棉花和油菜重点实验室,江苏 南京 210014)
陆地棉NF-YB基因家族的全基因组分析
刘 静1,徐珍珍2,袁 娜1,郭 月1,张保龙1,杜建厂1
(1.江苏省农业科学院 农业生物技术研究所,江苏省农业生物学重点实验室,江苏 南京 210014;2.江苏省农业科学院 经济作物研究所,农业部长江下游棉花和油菜重点实验室,江苏 南京 210014)
为了进一步了解NF-YB基因家族结构的功能,利用生物信息学手段,系统研究了陆地棉标准系TM-1基因组中NF-YB基因家族的数目、亚细胞定位、染色体分布、进化关系、基序以及组织表达情况。结果表明:TM-1基因组中共有41个NF-YB基因家族成员,它们含有相同的CBFD_NFYB_HMF结构域,大部分定位到细胞核内;41个NF-YB基因家族成员分布在19条染色体上,其中有19组成员在A亚组和D亚组中表现为直系同源基因;进化树分为Ⅰ和Ⅱ 2个小组,每个小组成员间具有相似的基序类型和排列顺序;组织表达分析则发现,在这41个NF-YB基因家族成员中,至少有24个成员可以进行表达,且表现出一定的组织特异性。
NF-YB基因家族;陆地棉;生物信息
CCAAT-box是广泛存在于真核生物中的一种顺式作用元件。有数据显示,约30%基因的启动子中含有该元件,并在调控基因表达过程中起重要作用[1-4]。核因子Y(Nuclear factor-Y)简称NF-Y,是一个与CCAAT-box顺式作用元件结合进而调节靶基因表达的转录因子,普遍存在于酵母、植物、动物等真核生物中[5]。植物核转录因子由多个基因编码构成核转录因子A亚基家族(NF-YA)(CBF-B或HAP2),B亚基家族(NF-YB)(CBF-A或HAP3)和C亚基家族(NF-YC)(CBF-C或HAP5)[6]。在拟南芥和水稻中,NF-YA家族均包含10个成员,NF-YB家族分别包含13,11个成员,NF-YC家族分别包含13,14个成员[7-9],它们在调控胚发育、胚成熟、花发育过程以及非生物胁迫应答中发挥着重要作用[10-13]。
棉属包括大约46个二倍体种和5个四倍体种,是纤维产出及油料的重要来源。随着二倍体D亚组雷蒙德氏棉[14](Gossypiumraimondii)和A亚组亚洲棉石系亚1号[15](Gossypiumarboreum)测序工作的完成,陆地棉遗传标准系TM-1(GossypiumhirsutumL.)也完成了全基因组的测序工作[16]。但是,棉花全基因组水平上的NF-YB基因家族分析还未见报道。本研究利用公开发表的陆地棉遗传标准系TM-1基因组信息,对NF-YB家族基因进行了全基因组鉴定和生物信息分析,系统鉴定了棉花基因组中的NF-YB基因家族的数目、亚细胞定位、染色体分布、进化关系、基序以及组织表达情况等,旨在为后续深入探索NF-YB基因家族的功能提供一定的基础理论依据。
1 材料和方法
1.1 试验材料
拟南芥NF-YB基因家族的氨基酸序列来自TAIR(http://www.arabidopsis.org/)网站;陆地棉标准系TM-1(G.hirsutumL.)基因组氨基酸序列和CDS序列数据来自中国农业科学院棉花研究所(http://cgp.genomics.org.cn/page/species/index.jsp);陆地棉EST数据库来自NCBI(http://www.ncbi.nlm.nih.gov/)网站。
1.2 棉花NF-YB基因家族的鉴定及亚细胞定位
首先在SMART(http://smart.embl-heidelberg.de/)网站预测拟南芥NF-YB基因家族氨基酸序列的保守结构域,然后在Pfam(http://pfam.janelia.org/)网站下载其种子文件,用其在棉花基因组氨基酸序列中搜索同源序列,来鉴定棉花的NF-YB家族成员。其中,搜索工具使用HMMER3.1b1程序;亚细胞定位使用在线软件CELLO(http://cello.life.nctu.edu.tw/)预测。
1.3 棉花NF-YB基因家族的进化分析
为了明确棉花基因组中鉴定到的NF-YB基因家族成员的进化关系,将其与拟南芥NF-YB基因家族成员的氨基酸序列一起构建进化树,参考拟南芥的分组标准。进化树构建工具采用MEGA6,建树方法使用邻接法(Neighbor-Joining,NJ),并进行1 000次Boot strap抽样自检。
1.4 棉花NF-YB基因家族的染色体定位
利用Perl语言编程,根据gff3文件中标注的基因位置,将NF-YB基因家族成员在染色体上的位置进行提取和整理,然后利用MapInspect软件绘制NF-YB成员在染色体上的物理分布图,并标注相应的直系同源基因。
1.5 棉花NF-YB基因家族成员基序分析
利用MEME在线软件(http://meme-suite.org/tools/meme)对棉花NF-YB成员进行在线分析,输入其氨基酸序列,检测各自基序数目和类型。设置参数如下:基序最多显示10个,其他参数为默认值。
1.6 棉花NF-YB基因家族成员的组织表达分析
利用棉花NF-YB基因家族成员的Cds序列对搜集到的EST进行比对,采用BlastN程序,e-value设置为1e-10,根据比对结果,明确棉花NF-YB基因家族成员的组织表达模式。
2 结果与分析
2.1 棉花NF-YB基因家族的鉴定及亚细胞定位
通过SMART网站结构域预测,发现所有的拟南芥NF-YB转录因子家族成员都含有CBFD_NFYB_HMF结构域,pfam号为PF00808。在pfam网站下载此结构域的种子文件,用HMMER3.1b1程序在棉花基因组氨基酸序列搜索后,共鉴定出棉花41个NF-YB基因家族成员。根据一般蛋白的命名方法,对TM-1基因组中41条NF-YB基因家族成员进行了命名,从GhNF-YB1到GhNF-YB41,并统计了其相应的氨基酸ID号、长度以及对应的亚基因组(表1)。结果表明:该家族的氨基酸序列长度在200个氨基酸左右(GhNF-YB39除外,长度为746个氨基酸)。
亚细胞定位结果表明:棉花基因组中41条NF-YB基因家族成员中,除了GhNF-YB9定位在细胞核和叶绿体中,GhNF-YB34定位在细胞核和线粒体中,GhNF-YB18定位在细胞核和细胞外,其他成员都只定位在细胞核中(表1)。
2.2 棉花NF-YB基因家族的系统进化树分析
将拟南芥和棉花NF-YB基因家族成员的氨基酸序列进行多序列比对并构建了系统进化树。结果表明:与拟南芥类似,棉花NF-YB基因家族成员也分为3个小组(Ⅰ~Ⅲ)(图1)。小组Ⅰ中,棉花中有35个成员:GhNF-YB10、GhNF-YB31、GhNF-YB14、GhNF-YB35、GhNF-YB4、GhNF-YB25、GhNF-YB1、GhNF-YB22、GhNF-YB20、GhNF-YB41、GhNF-YB16、GhNF-YB37、GhNF-YB7、GhNF-YB28、GhNF-YB34、GhNF-YB13、GhNF-YB6、GhNF-YB27、GhNF-YB2、GhNF-YB23、GhNF-YB18、GhNF-YB39、GhNF-YB3、GhNF-YB24、GhNF-YB11、GhNF-YB32、GhNF-YB12、GhNF-YB33、GhNF-YB17、GhNF-YB38、GhNF-YB21、GhNF-YB9、GhNF-YB30、GhNF-YB15和GhNF-YB36,拟南芥有8个成员:AtNF-YB7、AtNF-YB3、AtNF-YB2、AtNF-YB1、AtNF-YB8、AtNF-YB10、AtNF-YB5和AtNF-YB4;小组Ⅱ中,棉花有6个成员:GhNF-YB8、GhNF-YB29、GhNF-YB19、GhNF-YB40、GhNF-YB5和

表1 陆地棉NF-YB家族基本信息
GhNF-YB26,拟南芥有2个成员:AtNF-YB9和AtNF-YB6;小组Ⅲ中,没有棉花NF-YB基因家族成员,拟南芥有3个成员:AtNF-YB12、AtNF-YB13和AtNF-YB11(图1)。
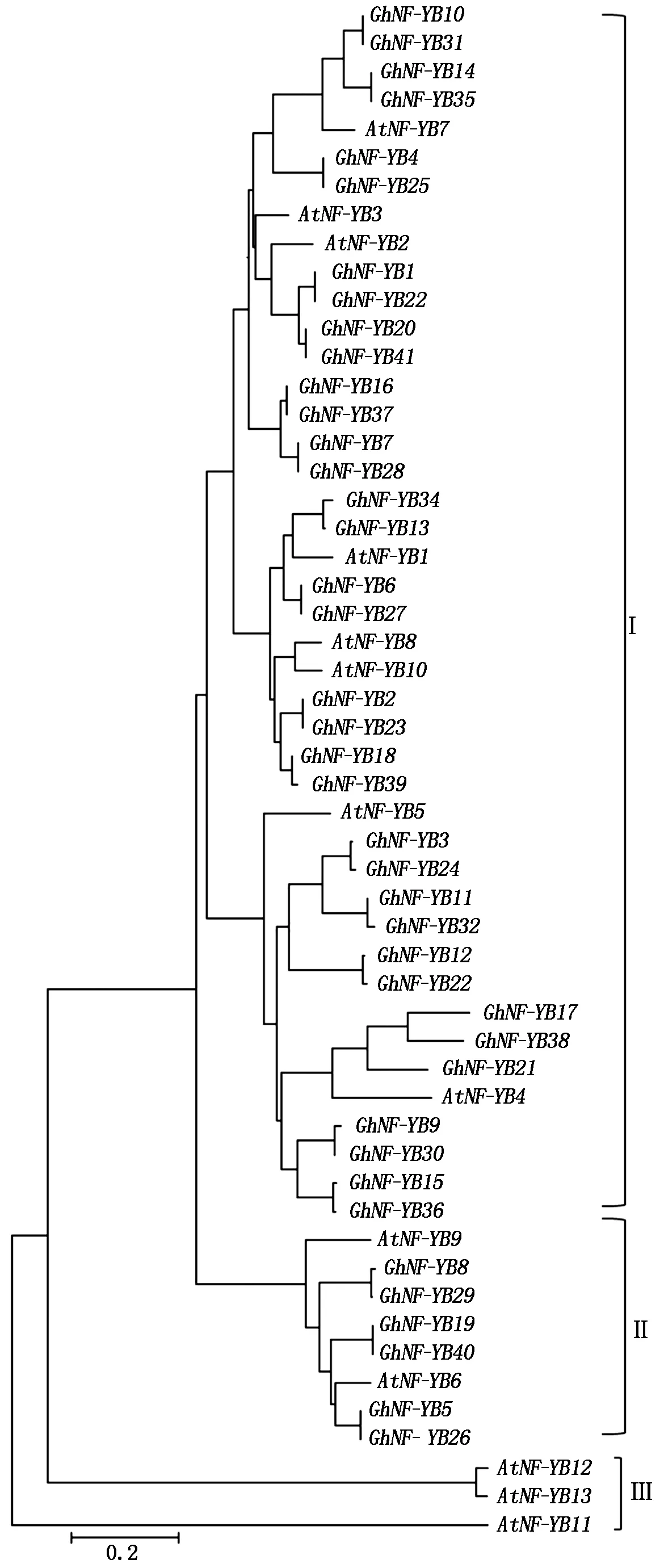
图1 陆地棉中NF-YB基因家族的系统进化树
2.3 棉花NF-YB基因家族的染色体定位
染色体定位结果表明:棉花41个NF-YB基因家族成员分布在2个亚基因组的19条染色体上。其中A11和D11染色体上分布最多,有5个成员。其他染色体上分布较为较少,一般为2个成员;另外,在这41个成员中,有19对成员(GhNF-YB1与GhNF-YB22、GhNF-YB2与GhNF-YB23、GhNF-YB3与GhNF-YB24、GhNF-YB5与GhNF-YB26、GhNF-YB6与GhNF-YB27、GhNF-YB7与GhNF-YB28、GhNF-YB8与GhNF-YB29、GhNF-YB9与GhNF-YB30、GhNF-YB10与GhNF-YB31、GhNF-YB11与GhNF-YB32、GhNF-YB12与GhNF-YB33、GhNF-YB13与GhNF-YB34、GhNF-YB14与GhNF-YB35、GhNF-YB15与GhNF-YB36、GhNF-YB16与GhNF-YB37、GhNF-YB17与GhNF-YB38、GhNF-YB18与GhNF-YB39、GhNF-YB19与GhNF-YB40、GhNF-YB20与GhNF-YB41)为直系同源基因(图2)。

灰线.直系同源基因。
2.4 棉花NF-YB基因家族的基序分析
基序分析结果表明:进化树上分支近的NF-YB基因家族成员具有相同或类似的基序类型和排列顺序。进化树中,棉花NF-YB基因家族成员分为2个小组(Ⅰ~Ⅱ)。小组Ⅰ:GhNF-YB10、GhNF-YB31、GhNF-YB14和GhNF-YB35具有完全相同的5个基序,且排列顺序完全相同。GhNF-YB4和GhNF-YB25具有完全相同的2个基序,且排列顺序完全相同。GhNF-YB1、GhNF-YB22、GhNF-YB20和GhNF-YB41具有完全相同的6个基序,且排列顺序完全相同。GhNF-YB16、GhNF-YB37、GhNF-YB7和GhNF-YB28具有相似的5~7个基序类型,排列顺序也基本相同。GhNF-YB34、GhNF-YB13、GhNF-YB6和GhNF-YB27具有相似的5~6个基序类型,排列顺序也基本相同。GhNF-YB2、GhNF-YB23、GhNF-YB18和GhNF-YB39具有相同的6个基序类型,排列顺序完全相同。GhNF-YB3、GhNF-YB24、GhNF-YB11、GhNF-YB32、GhNF-YB12、GhNF-YB33、GhNF-YB17、GhNF-YB38、GhNF-YB21、GhNF-YB9、GhNF-YB30、GhNF-YB15和GhNF-YB36具有相似的3~4基序类型,排列顺序也基本相同(图3);小组Ⅱ:GhNF-YB8和GhNF-YB29具有完全相同的6个基序类型和排列顺序。GhNF-YB19和GhNF-YB40具有6个相似的基序类型和排列顺序。GhNF-YB5和GhNF-YB26具有6个完全相同的基序类型和排列顺序(图3)。总体来说,小组Ⅰ的35个NF-YB成员具有相似的5~7个基序类型和排列顺序,小组Ⅱ的6个NF-YB成员具有6个相似的基序类型和排列顺序(图3)。
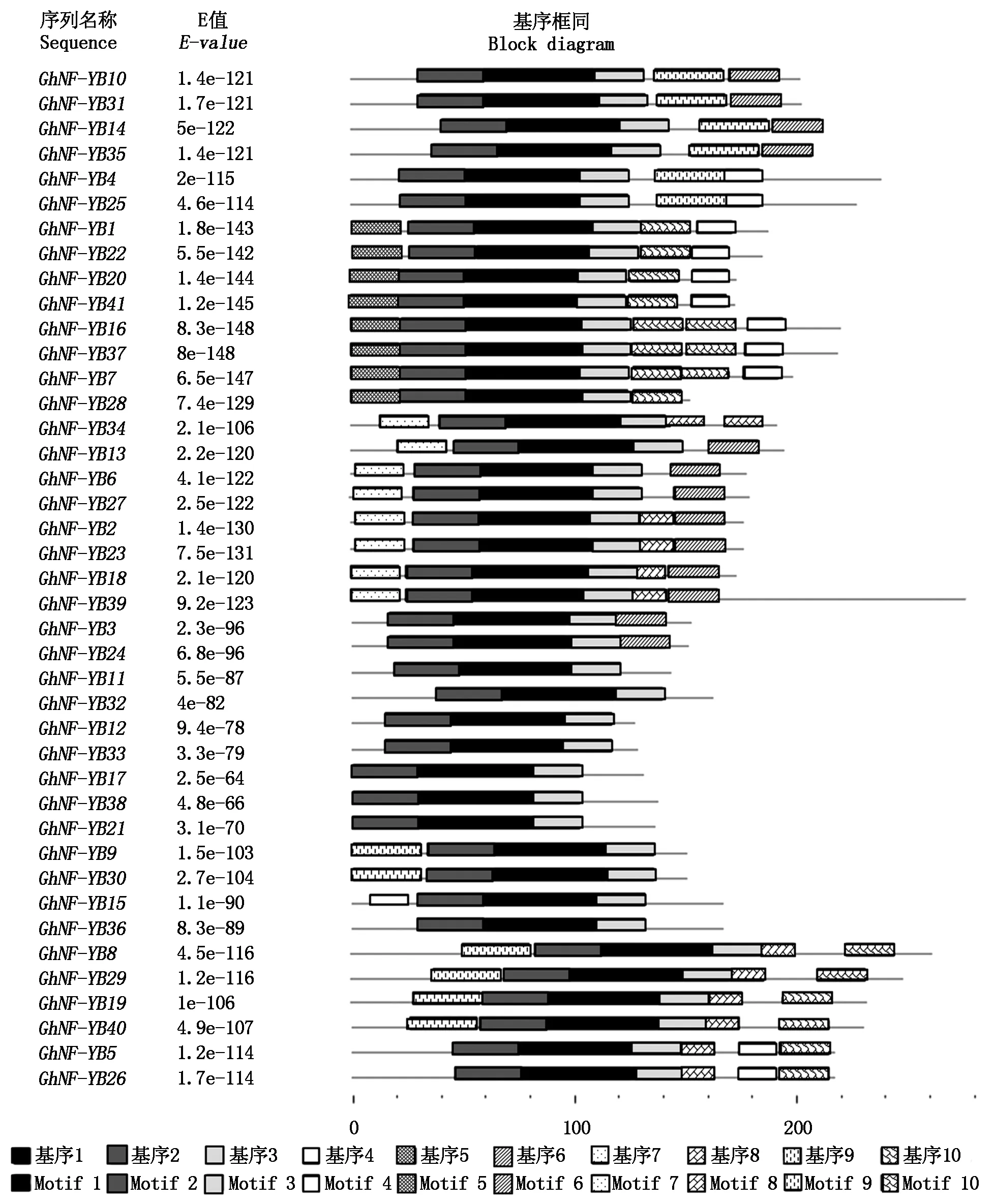
图3 陆地棉基因组中NF-YB基因家族的基序类型
2.5 棉花NF-YB基因家族的组织表达分析
组织表达分析发现,棉花41个NF-YB基因家族成员中,有24个成员在根、茎、叶、花、蕾、胚珠、纤维、铃、花药、胚性愈伤组织和分生组织中广泛表达(表2)。大部分基因在根、蕾、花、茎、纤维、胚珠和分生组织中表达。其中,在纤维组织中表达的NF-YB基因家族成员最多(20个基因),少数基因在叶、铃、花药和胚性愈伤组织中表达(表2)。

表2 陆地棉NF-YB基因家族成员的组织表达模式
注:Y.有表达;N.无表达。
Note:Y.Expression;N.No expression.
3 讨论
四倍体陆地棉标准系TM-1基因组属于AADD型,其A亚组和D亚组的基因之间存在较高的共线性[17]。本研究在陆地棉标准系TM-1基因组中共鉴定了41个NF-YB基因家族成员,其中有38个成员(19对)为直系同源基因,说明四倍体陆地棉在物种形成过程中没有经过基因组水平上大规模的基因重排现象。这与陆地棉基因组的研究结果相一致[16-18],也支持徐珍珍和倪万潮等在YABBY、ZIP和EPSPS基因家族上的分析结果[19-21]。此外,进一步研究发现,每对直系同源基因具有相同或相似的亚细胞定位、基序类型和排列顺序,在进化树上分布在1个小组,且具有相同或相似的组织表达类型,表明平行进化同源基因具有相同或相似的基因功能。
系统进化树分析结果表明,在拟南芥中,NF-YB基因家族成员分为Ⅰ、Ⅱ和Ⅲ,共3个小组[22]。在棉花中,含有Ⅰ和Ⅱ2个小组,这种分布情况跟拟南芥、水稻、大豆[22]和胡杨树[23]整体一致,可以作为NF-YB家族起源早于单、双子叶植物分化的1个证据。而在棉花中,没有小组Ⅲ的成员,推测在棉花中小组Ⅲ的成员可能在其进化过程中丢失了。另外,在进化上分支较近的NF-YB基因家族成员具有相似或相同的基序类型和排列顺序以及组织表达类型,可以推测每个小组成员之间具有相似的功能。
在我们鉴定到的41个NF-YB基因家族成员,有24个成员在根、茎、叶、花、蕾、胚珠、纤维、铃、花药、胚性愈伤组织和分生组织中广泛表达,推测其在这些组织的发育过程中起着重要的作用。大部分基因在根、蕾、花、茎、纤维、胚珠和分生组织中表达,其中在纤维组织中表达的NF-YB基因家族成员最多(20个),提示它们可能在纤维发育过程中发挥着重要的作用。由于棉花纤维是世界上重要的纺织原料之一,具有重要的经济价值,因此,本研究鉴定的在纤维组织表达的NF-YB基因家族成员将对深入了解棉纤维发生发育机制奠定一定的理论基础。
[1] Gelinas R,Endlich B,Pfeiffer C,et al.G to a substitution in the distal CCAAT box of the a gamma-globin gene in Greek hereditary persistence of fetal haemoglobin[J].Nature,1985,313(6000):323-325.
[2] Buchere P,Frifonov E N.CCAAT box revisited:bidirectionality,location and context[J].Journal of Biomolecular Structure & Dynamics,1988,5(6):1231-1236.
[3] Bucher P.Weight matrix descriptions of four eukaryotic RNA polymerase Ⅱ promoter elements derived from 502 unrelated promoter sequences[J].Journal of Molecular Biology,1990,212(4):563-578.
[4] Mantovani R.A survey of 178 NF-Y binding CCAAT boxes[J].Nucleic Acids Research,1998,26(5):1135-1143.
[5] Testa A,Donati G,Yan P,et al.Chromatin immunoprecipitation(ChIP) on chip experiments uncover a widespread distribution of NF-Y binding CCAAT sites outside of core promoters[J].The Journal of Biological Chemistry,2005,280(14):13606-13615.
[6] Romier C,Cocchiarella F,Mantovani R,et al.The NF-YB/NF-YC structure gives insight into DNA binding and transcription regulation by CCAAT factor NF-Y[J].Journal of Biological Chemistry,2003,278(2):1336-1345.
[7] Riechmann J L,Heard J,Martin G,et al.Arabidopsistranscription factors:genome-wide comparative analysis among eukaryotes[J].Science,2000,290(5499):2105-2110.
[8] Miyoshi K,Ito Y,Serizawa A,et al.OsHAP3 genes regulate chloroplast biogenesis in rice[J].Plant Journal,2003,36(4):532-540.
[9] Thirumurugan T,Ito Y,Kubo T A,et al.Identification,characterization and interaction of HAP family genes in rice[J].Molecular Genetics and Genomics,2008,279(3):279-289.
[10] Cai X,Ballif J,Endo S,et al.A putative CCAAT-binding transcription factor is a regulator of flowering timing inArabidopsis[J].Plant Physiology,2007,145(1):98-105.
[11] Chen N Z,Zhang X Q,Wei P C,et al.AtHAP3b plays a crucial role in the regulation of flowering time inArabidopsisduring osmotic stress[J].Journal of Biochemistry and Molecular Biology,2007,40(6):1083-1089.
[12] Li C,Dubcovsky J.Wheat FT protein regulates VRN1 transcription through interactions with FDL2[J].Plant Journal,2008,55(4):543-554.
[13] Nelson D E,Repetti P P,Adams T R,et al.Plant nuclear factor Y(NF-Y) B subunits confer drought tolerance and lead to improved corn yields on water-limited acres[J].Proceedings of the National Academy of Sciences of the United States of America,2007,104(42):16450-16455.
[14] Wang K,Wang Z,Li F,et al.The draft genome of a diploid cottonGossypiumraimondii[J].Nature Genetics,2012,44(10):1098-1103.
[15] Li F,Fan G,Wang K,et al.Genome sequence of the cultivated cottonGossypiumarboreum[J].Nature Genetics,2014,46(6):567-572.
[16] Li F G,Fan G Y,Lu C R,et al.Genome sequence of cultivated Upland cotton(GossypiumhirsutumTM-1) provides insights into genome evolution[J].Nature Biotechnology,2015,33(5):524-530.
[17] Zhang T,Hu Y,Jiang W,et al.Sequencing of allotetraploid cotton(GossypiumhirsutumL.acc.TM-1) provides a resource for fiber improvement[J].Nature Biotechnology,2015,33(5):531-537.
[18] Paterson A H,Wendel J F,Gundlach H,et al.Repeated polyploidization ofGossypiumgenomes and the evolution of spinnable cotton fibres[J].Nature,2012,492(7429):423-427.
[19] 徐珍珍,倪万潮,张香桂,等.棉花YABBY基因家族的全基因组分析[J].生物技术通报,2015,31(11):146-152.
[20] 倪万潮,巩元勇,徐珍珍,等.陆地棉GhZIP基因家族全基因组分析[J].华北农学报,2015,30(6):8-16.
[21] 巩元勇,徐珍珍,郭书巧,等.陆地棉EPSPS基因全基因组分析[J].华北农学报,2016,31(1):15-21.
[22] 徐兆师,郑炜君,冯志娟,等.大豆NF-YB家族全基因组鉴定,分类和表达[J].作物学报,2013,38(9):1570-1582.
[23] 严东辉.胡杨干旱响应转录组及NF-YB基因表达谱[D].北京:北京林业大学,2012.
Genome-wide Analysis ofNF-YBGene Family inGossypiumhirsutumL.
LIU Jing1,XU Zhenzhen2,YUAN Na1,GUO Yue1,ZHANG Baolong1,DU Jianchang1
(1.Institute of Biotechnology,Jiangsu Academy of Agricultural Sciences,Provincial Key Laboratory of Agrobiology,Nanjing 210014,China;2.Institute of Industrial Crops,Jiangsu Academy of Agricultural Sciences,Key Laboratory of Cotton and Rapeseed in the Lower Reaches of the Yangtze River,Ministry of Agriculture,Nanjing 210014,China)
In order to further understand the structure and function ofNF-YBgene family,we have systematically investigated the number,subcellular localization,chromosome distribution,evolutionary relationships,motif and tissue expression pattern of of family in the genome ofGossypiumhirsutumL.acc.TM-1 by bioinformatics method.41NF-YBgenes were identified in TM-1 genome;the 41NF-YBgenes contain the same CBFD_NFYB_HMF domain,and most of them were located in nucleus; they distributed on 19 chromosomes,and there were 19 pairs of the genes on the A subgroup and D subgroup were orthologous genes; 41NF-YBgenes can be divided into groupⅠand groupⅡ,and there were similar motif type and arrangement in each group;.24NF-YBgenes were expressed,but the expression pattern varied among different tissues.
NF-YBgene family;GossypiumhirsutumL.;Bioinformatics
2016-07-12
江苏省“333”人才工程项目(BRA2013267);江苏省农业科技自主创新基金项目(CX13(3059));棉花生物学国家重点实验室开放课题(CB2016B03)
刘 静(1986-),女,山东济南人,助理研究员,硕士,主要从事生物信息学研究。
Q78;S562.03
A
1000-7091(2016)05-0021-07
10.7668/hbnxb.2016.05.004

