白桦锌指蛋白基因的分离及表达分析
王宇航 秦琳琳 李 莉
(东北林业大学林木遗传育种国家重点实验室,哈尔滨 150040)
白桦锌指蛋白基因的分离及表达分析
王宇航 秦琳琳 李 莉*
(东北林业大学林木遗传育种国家重点实验室,哈尔滨 150040)
植物锌指蛋白是成员众多的转录因子家族,在植物的生长发育和对非生物胁迫响应等过程中具有重要作用。根据Cys和His残基的数目和位置将锌指转录因子分为C2H2、C8、C6、C3HC4、C2HC、C2HC5、C4、CCCH和C4HC3九种类型。本实验利用其他物种中已报导的植物逆境胁迫相关的锌指蛋白转录因子基因序列作为信息探针,在白桦基因组数据库中筛选出10个基因,分别是4个C2H2型、5个C3HC4-RING型和一个CCCH型。通过qRT-PCR技术对筛选的基因在根中的表达模式进行了分析,结果显示BpZFP2、BpZFP3、BpZFP4、BpZFP5和BpZFP8的表达受盐和干旱胁迫的显著诱导,表明这些基因参与了逆境胁迫的应答。
白桦;非生物胁迫;锌指蛋白转录因子;表达分析
转录因子是指能够与真核基因启动子区域中顺式作用元件特异结合并相互作用从而激活或抑制转录的DNA结合蛋白[1]。植物基因组中有相当一部分转录因子基因参与对环境变化的信号转导或转录调控,如CBFs、DREBs、WRKYs、NACs和ZFPs等[2]。锌指蛋白(ZFPs)是一类具有手指状结构域的转录因子,因其具有指状结构特征且能结合Zn2+而得名[3]。ZFPs的种类繁多,Berg和Shi根据半胱氨酸和组氨酸残基的数目和位置将ZFPs分为C2H2、C8、C6、C3HC4(RING型)、C2HC、C2HC5、C4、C4HC3和CCCH九种类型[4]。
已有研究表明,ZFPs在植物生长发育和耐逆性等多种生物学过程中发挥作用[5]。自从拟南芥(Arabidopsisthaliana)STZ/ZAT10第一个被证实是参与植物盐胁迫应答的转录因子以来[6],国内外研究者在多种植物中陆续发现和分离了多个抗逆相关的ZFPs基因,如C2H2型的拟南芥AZF1,AZF2[7],ZAT6[8],ZAT7[9]和ZAT12[10];水稻(Oryzasativa)ZFP182[11],ZFP245[12],ZFP252[13]和ZFP179[14]等;RING型的拟南芥AtAIRP4[15]和水稻OSRHC14[16]等。近年来,随着拟南芥等植物基因组测序计划的完成,一些CCCH型的ZFPs被发现参与调控植物应答逆境胁迫响应,如拟南芥AtSZF1,AtSZF2[17],AtTZF1和AtTZF2等[18]。
迄今为止,对ZFPs调控植物应答非生物胁迫的功能研究大都集中在拟南芥、水稻等草本植物,而在林木中的研究极少。白桦(BetulaplatyphyllaSuk.)是北温带的一个广布种,生长迅速,适应性和抗逆性较强。本研究以白桦作为实验材料,通过qRT-PCR分析了十个白桦BpZFP基因在NaCl(高盐胁迫)、PEG6000(聚乙二醇干旱诱导胁迫)以及ABA(脱落酸)胁迫不同时间后的表达模式,为系统研究白桦BpZFP基因的功能和通过基因工程手段将该基因用于林木抗性育种奠定了理论基础和提供优良基因。
1 材料与方法
1.1 植物材料
白桦组培苗茎段接种到WPM+6-BA 1 mg·L-1的分化培养基上,培养20 d后将展出的新枝切下,接种到1/2MS+NAA 0.2 mg·L-1的生根培养基中继续培养20 d,生根的组培苗移栽到直径11.5 cm、高10 cm的花盆中,基质是按3∶1∶1的比例混合的黑土∶蛭石∶珍珠岩,培养在温度为24±1℃,相对湿度为65%~75%,光周期为16 h光照/8 h黑暗,光强为400 μmol·m-2·s-1的温室中,待生长至2个月龄,选择生长状态良好、长势一致的白桦幼苗用于胁迫处理。
1.2 非生物胁迫处理
将配置浓度为200 mmol·L-1NaCl、15%(w/v) PEG6000和100 μmol·L-1ABA溶液分别浇灌盆中白桦幼苗,同时用蒸馏水同步浇灌作为对照,在胁迫处理0、6、12、24、48和72 h后,收集根组织并将其放入液氮速冻,储存于-80℃备用。每个时间点均设3个生物学重复,每个生物学重复均用10株幼苗混样。
1.3 总RNA的提取及反转录
总RNA的抽提采用BioTeKe公司生产的通用植物总RNA提取试剂盒,总RNA的反转录反应采用TaKaRa公司生产的PrimeScriptTMRT Reagent Kit with gDNA Eraser反转录试剂盒,按照说明书进行。
1.4 白桦BpZFPs的鉴定和生物信息学分析
以已经报导的拟南芥、水稻等植物中参与调控非生物逆境胁迫应答的锌指蛋白基因序列为信息探针,在白桦转录组以及部分已完成的白桦全基因组测序数据库(未发表数据)中比对并获取具有完整开放读码框的锌指蛋白基因的序列全长。氨基酸的保守性结构域、同源性和理化性质分别利用PFAM(http://pfam.sanger.ac.uk/)、NCBI(http://blast.ncbi.nlm.nih.gov/Blast.cgi)和PROTPARAM在线软件(http://www.expasy.org/tools/protparam.html)进行分析预测;氨基酸的多序列比对采用ClustalX1.83软件实现;使用MEGA5.1软件采用遗传距离Neighbor-Joining建树法构建无根进化树,并对生成的进化树进行Bootstrap校正,其中重复次数设定为1 000。

表1 实时荧光定量PCR引物序列
1.5 实时定量PCR
根据白桦数据库中获得的基因全长,并根据实时定量PCR引物要求用PrimerPremier5软件分别设计基因特异引物(表1)。实时定量PCR反应在ABI7500 realtime PCR荧光检测系统中进行,操作的过程按照TaRaKa SYBR Premix Ex TaqTM试剂盒使用说明完成。以白桦Tubulin(GenBank登录号:FG067376)为内参,其扩增引物为BpTubulin-F/R。为保证结果的可重复性,分别设有3个技术重复和3个生物学重复。基因的相对表达量用F(2-△△Ct)表示[19],当0
2 结果与分析
2.1 编码BpZFP蛋白基因的获得及序列分析
通过与其他物种中与抗逆相关锌指蛋白基因的序列比对,在白桦转录组以及部分完成的全基因组数据库中获得十个具有完整开放读码框(ORFs)的BpZFP基因,命名为BpZFP1~BpZFP10,这些基因的ORFs编码蛋白推测的氨基酸残基数为176~683(aa), 预测的相对分子质量为19.71~77.36(kDa),理论等电点为6.08~9.69(pI)(表2)。PFAM分析结果表明,这十个BpZFP蛋白都至少具有一个典型的Znf结构域,其中BpZFP1~BpZFP4基因具有保守的CX(2-4)CX12HX(2-6)H基序和QALGGH序列,属于C2H2型;BpZFP5~BpZFP9基因具有C-X2-C-X(9-39)-C-X(1-3)-H-X(2-3)-(N/C/H)-X2-C-X(4-48)C-X2-C基序,属于C3HC4-RING型;BpZFP10基因具有CX(4-15)CX(4-6)CX3H保守基序属于CCCH型。利用MEGA5.1软件绘制了三组系统进化树,结果表明:C2H2型的BpZFP2和BpZFP4基因和拟南芥ZAT7和ZAT12亲缘关系较近,其中BpZFP2与ZAT7和ZAT12的相似性分别为43%和46%,而BpZFP4与ZAT7和ZAT12的同源性分别为43%和47%;BpZFP3与矮牵牛的ZPT2-3亲缘关系较近,相似性为57%(图1);C3HC4-RING型的BpZFP6和BpZFP7基因与水稻的OsRHC22亲缘关系较近,相似性分别为43%和31%(图2);而在CCCH型这组中BpZFP10与玉米中的ZmC3H43相似性为42%(图3)。
2.2 逆境胁迫下白桦BpZFP基因的表达分析
通过qRT-PCR对十个BpZFP基因经不同胁迫处理(NaCl、PEG、ABA)不同时间点(0、6、12、24、48、72 h)后,在根中的相对表达量进行了分析,结果表明:在NaCl胁迫后,BpZFP6、BpZFP7和BpZFP10在胁迫的各个时间点的表达量变化不明显,其余七个基因的表达量都至少在一个时间点显著上调,其中BpZFP3和BpZFP4显著受到高盐胁迫诱导,在胁迫的各个时间点的表达量均超过对照值,最高F值分别是对照值的12.78和26.56倍;BpZFP2、BpZFP5、BpZFP8和BpZFP9基因在胁迫后短时间内变化不明显,但随着胁迫时间的延长开始上调表达,其中BpZFP2、BpZFP8和BpZFP9表达量均在胁迫72 h时达到最高,分别为对照的4.55、14.46和18.67倍,而BpZFP5基因的表达量则是在处理48 h时达到顶峰,约为对照的2.44倍(表3)。经PEG处理后:BpZFP1和BpZFP9的表达量变化不明显,而其余八个基因的表达量都至少在一个时间点上发生改变,其中BpZFP2、BpZFP5、BpZFP7和BpZFP8均在胁迫24 h时表达量达到最高,分别为对照的4.04、3.51、2.39和3.36倍,而BpZFP3、BpZFP4、BpZFP6和BpZFP10则在胁迫初期6 h时表达量达到最高,分别是对照的19.79、13.43、2.51和3.38倍,随着胁迫时间的延长,表达量均下降,其中BpZFP6和BpZFP10的表达量在48 h后基本接近于对照水平,而BpZFP3和BpZFP4基因转为下调模式(表4)。经ABA处理后:BpZFP2、BpZFP3和BpZFP4基因在处理6 h后稍有上调,随后又转为下调模式,其余七个基因的表达量在各个处理时间点的变化均不明显(表5)。综上所述:BpZFP2、BpZFP3、BpZFP4、BpZFP5和BpZFP8在高盐和干旱胁迫条件下产生了表达水平的显著变化,其中BpZFP2、BpZFP3和BpZFP4基因还受ABA的调节,暗示它们在植物不同逆境应答途径的交叉现象中发挥作用。
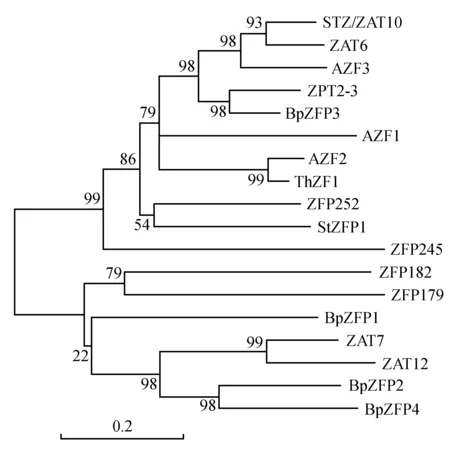
图1 白桦与其他物种C2H2型锌指蛋白系统进化树Fig.1 The phylogenetic relationship of the deduced amino acid sequence of C2H2 type BpZFP to ZFP proteins from other organisms Arabidopsis thaliana: AZF1(BAA85108),AZF2(BAB02542),AZF3(AB030732),ZAT6(NP_196054),ZAT7(NP_190195),ZAT12(AAM65582),STZ/ZAT10(NP_174094); Oryza sativa: ZFP179(AAL76091),ZFP182(NP001051718),ZFP245(AAQ95583),ZFP252(AAO46041); Petunia x hybrida: ZPT2-3(BAA05079); Thellungiella halophila: ThZF1(ABI74621); Solanum tuberosum: StZFP1(ABK78777)

基因名GenecDNA长度cDNAlength(bp)推导蛋白氨基酸残基数aa相对分子质量MW(kDa)理论等电点PIBpZFP169923225.516.41BpZFP254318019.719.25BpZFP374424726.248.66BpZFP453117619.759.69BpZFP5111036941.106.42BpZFP6147048953.328.11BpZFP7205268377.366.79BpZFP8120340043.928.40BpZFP972023926.956.08BpZFP10104134637.319.07

图2 白桦与其他物种中C3HC4-RING型锌指蛋白系统进化树Fig.2 The phylogenetic relationship of the deduced amino acid sequence of C3HC4 RING type BpZFP to ZFP proteins from other organisms A.thaliana: AtRING(NP_177517),RHG1a(AAF97276),ATL6(NM_111393),AtAIRP4(NP_568885.1); O.sativa: OsRHC13(BAG92291),OsRHC14(BAG92287),OsRHC16(NP_001054387),OsRHC22(BAG99939),OsRHC24(NP_001061386),OsRHC28(BAG92311); Artemisia desertorum: AdZFP1(AAY17949); Brassica rapa: BrRZFP1(ADK63390)
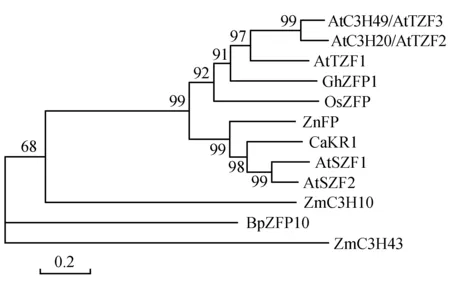
图3 白桦与其他物种中CCCH型锌指蛋白的系统进化树Fig.3 The phylogenetic relationship of the deduced amino acid sequence of CCCH type BpZFP to ZFP proteins from other organisms A.thaliana: AtC3H20/AtTZF2(NP_179571),AtC3H49/AtTZF3(NP_194648); AtTZF1(AEC07769),AtSZF1(NP_567030),AtSZF2(NP_181543); O.sativa: OsZFP(ACJ74073); Gossypium hirsutum: GhZFP1(AAX20386); Zea mays: ZmC3H10(AIB04474),ZmC3H43(AFU81639); Capsicum annuum: CaKR1(ABI30334); Triticum aestivum: ZnFP(AFS49947)
表3BpZFPs基因经NaCl胁迫后在白桦根中的表达模式分析
Table3ExpressionanalysisofBpZFPsunderNaClstressinroot

基因名称Gene时间Time(h)612244872BpZFP11.01±0.161.54±0.261.69±0.202.30±0.401.45±0.31BpZFP21.74±0.351.20±0.352.02±0.421.30±0.024.55±0.59BpZFP33.75±0.1112.78±0.472.50±0.183.16±0.393.16±0.23BpZFP45.05±0.239.05±0.4716.23±0.313.37±0.2126.56±0.29BpZFP50.39±0.041.18±0.172.24±0.062.44±0.192.38±0.02BpZFP61.15±0.061.19±0.061.47±0.071.00±0.081.11±0.40BpZFP71.10±0.031.00±0.021.35±0.081.32±0.091.05±0.15BpZFP81.38±0.051.67±0.281.39±0.341.38±0.3514.46±0.43BpZFP91.49±0.062.08±0.188.27±0.202.11±0.1218.67±0.02BpZFP100.99±0.011.22±0.010.81±0.061.81±0.041.45±0.03
表4BpZFPs基因经PEG胁迫后在白桦根中的表达模式分析
Table4ExpressionpatternsofBpZFPsunderPEGstressinroot

基因名称Gene时间Time(h)612244872BpZFP11.19±0.101.18±0.070.70±0.041.14±0.141.21±0.09BpZFP23.33±0.171.86±0.124.07±0.761.36±0.235.16±0.26BpZFP319.79±0.680.80±0.0413.53±0.271.13±0.040.79±0.04BpZFP413.43±0.470.34±0.015.60±0.430.29±0.010.22±0.02BpZFP51.51±0.081.11±0.063.51±0.111.17±0.261.16±0.07BpZFP62.51±0.171.09±0.032.04±0.161.12±0.041.17±0.07BpZFP71.82±0.041.07±0.062.39±0.191.01±0.1610.1±0.08BpZFP82.15±0.071.03±0.023.36±0.031.02±0.261.20±0.05BpZFP90.74±0.081.03±0.100.81±0.031.14±0.071.10±0.03BpZFP103.38±0.041.11±0.053.08±0.131.20±0.041.18±0.01
表5BpZFPs基因经ABA胁迫后在白桦根中的表达模式分析
Table5ExpressionanalysisofBpZFPsunderABAtreatmentinroot
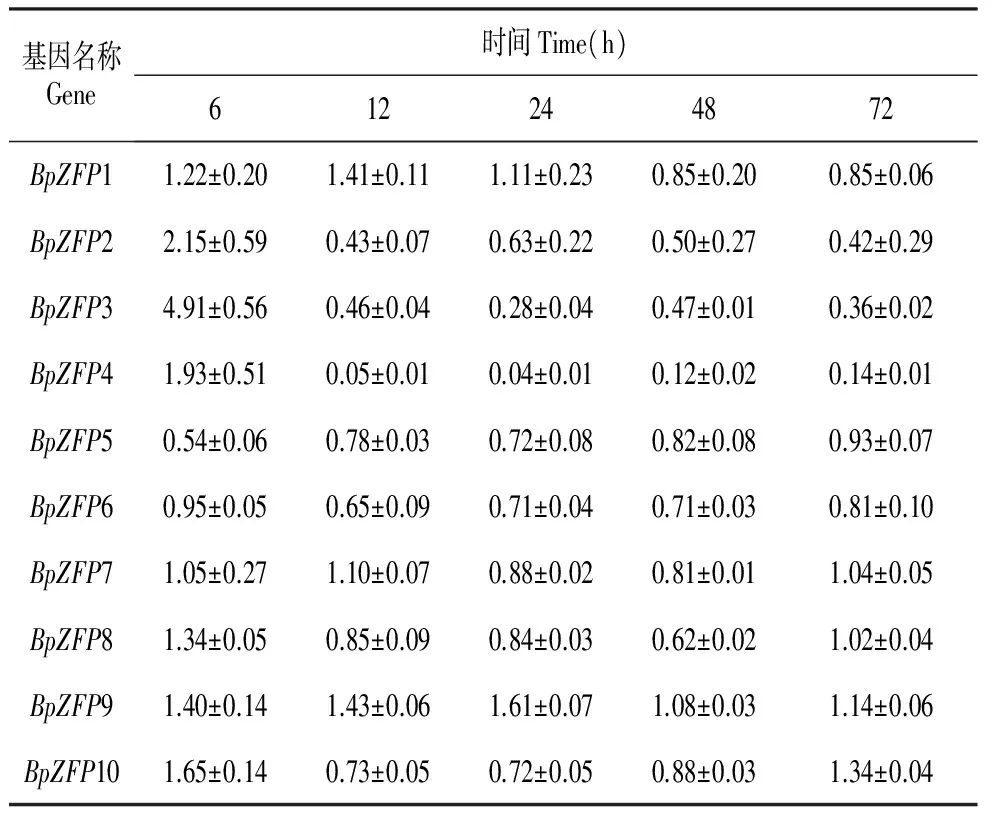
基因名称Gene时间Time(h)612244872BpZFP11.22±0.201.41±0.111.11±0.230.85±0.200.85±0.06BpZFP22.15±0.590.43±0.070.63±0.220.50±0.270.42±0.29BpZFP34.91±0.560.46±0.040.28±0.040.47±0.010.36±0.02BpZFP41.93±0.510.05±0.010.04±0.010.12±0.020.14±0.01BpZFP50.54±0.060.78±0.030.72±0.080.82±0.080.93±0.07BpZFP60.95±0.050.65±0.090.71±0.040.71±0.030.81±0.10BpZFP71.05±0.271.10±0.070.88±0.020.81±0.011.04±0.05BpZFP81.34±0.050.85±0.090.84±0.030.62±0.021.02±0.04BpZFP91.40±0.141.43±0.061.61±0.071.08±0.031.14±0.06BpZFP101.65±0.140.73±0.050.72±0.050.88±0.031.34±0.04
3 讨论
本研究利用白桦转录组以及部分已完成的白桦全基因组数据库,鉴定出10个具有完整开放读码框(ORFs)的BpZFP基因,其中4个C2H2型基因所编码蛋白推测的氨基酸残基数为176~247aa;五个C3HC4-RING型基因为239~683aa。虽然同一种类型的BpZFP基因所编码蛋白推测的氨基酸残基数变化很大,但都具有各自保守的锌指蛋白结构域和基序。同一类型基因编码的蛋白长度的变化较大,说明BpZFP基因在基因起源和基因结构进化上的复杂性,同时也说明它们可能参与不同的代谢调控途径,具有不同的功能。
系统进化分析表明,BpZFP基因与同类型来自草本植物的锌指蛋白基因的相似性均小于80%,说明了尽管这些基因具有相当的保守性,但在一定程度上仍具有种属特异性,尤其对于一些木本植物来说。序列相似性比对发现BpZFP2、BpZFP3、BpZFP4和BpZFP10分别与ZAT7、ZAT12、ZPT2-3和ZmC3H43的同源性在42%~57%(图1,3)。在前人的研究中已经发现,ZAT7在抑制生长发育、应答高温、高盐以及H2O2的胁迫中具有多种不同的功能[9];ZAT12的表达受包括低温、高盐、干旱、高温、伤害、过氧化氢、光照等多种胁迫的诱导,过量表达ZAT12的拟南芥转基因植株提高了耐盐性、耐旱性和耐冷性[10];Sugano等研究表明ZPT2-3的表达受低温、伤害、茉莉酸甲酯的快速诱导,转基因矮牵牛植株表现了更强的耐旱性[20];玉米ZmC3H43在ABA和干旱胁迫下的表达水平显著提高[21]。本研究结果表明,BpZFP2、BpZFP3和BpZFP4的表达受高盐和干旱胁迫的诱导,它们可能参与白桦耐盐和耐旱的应答;BpZFP10的表达在盐胁迫下变化不大,但在干旱胁迫下发生显著的提高。对于以上这些基因的功能以及参与白桦的耐盐、耐旱应答调控途径有待进一步研究。
泛素化是一种真核生物翻译后的蛋白修饰,这种过程是由E1、E2和E3泛素(ub)连接酶的级联反应介导的,并且还参与调节大量的细胞功能[22]。含有RING基序的蛋白大都具有E3泛素连接酶的作用,并且在应对环境刺激时起到关键作用[23],如拟南芥的AtAIRP4[15]和沙蒿的ZFP1[24]等。本研究中的BpZFP5和BpZFP8是C3HC4-RING型,它们的表达受盐和干旱胁迫的诱导,表明参与了白桦耐盐和耐旱的应答,但BpZFP5和BpZFP8蛋白是否发挥了E3泛素连接酶的功能以及作用的机制还需要继续进行研究。
致谢对林木遗传育种国家重点实验室陈肃博士在白桦数据库使用方面的帮助表示衷心的感谢!
1.Singh K,Foley R C,Onate-Sanchez L.Transcription factors in plant defense and stress responses[J].Curr Opin Plant Biol.2002,5(5):430-436.
2.Golldack D,Lüking I,Yang O.Plant tolerance to drought and salinity:stress regulating transcription factors and their functional significance in the cellular transcriptional network[J].Plant Cell Rep,2011,30(8):1383-1391.
3.Miller J,McLachlan A D,Klug A.Repetitive zinc-binding domains in the protein transcription factor Ⅲ A fromXenopusoocytes[J].EMBO J,1985,4(6):1609-1614.
4.Berg J M,Shi Y.The galvanization of biology:A growing appreciation for the roles of zinc[J].Science,1996,271(5252):1081-1085.
5.Kielbowicz-Matuk A.Involvement of plant C2H2-type zinc finger transcription factors in stress responses[J].Plant Sci,2012,185-186:78-85.
6.Lippuner V,Cyert M S,Gasser C S.Two classes of plant cDNA clones differentially complement yeast calcineurin mutants and increase salt tolerance of wild-type yeast[J].J Biol Chem,1996,271:12859-12866.
7.Kodaira K S,Qin F,Tran L S,et al.Arabidopsis Cys2/His2 zinc-finger proteins AZF1 and AZF2 negatively regulate abscisic acid-repressive and auxin-inducible genes under abiotic stress conditions[J].Plant Physiol,2011,157(2):742-56.
8.Liu X M,Nguyen X C,Kim K E,et al.Phosphorylation of the zinc finger transcriptional regulator ZAT6 by MPK6 regulatesArabidopsisseed germination under salt and osmotic stress[J].Biochem Biophys Res Commun,2013d,430:1054-1059.
9.Ciftci-Yilmaz S,Morsy M R,Song L,et al.The EAR-motif of the Cys2/His2-type zinc finger protein Zat7 plays a key role in the defense response ofArabidopsisto salinity stress[J].J Biol Chem,2007,282(12):9260-9268.
10.Davletova S,Schlauch K,Coutu J,et al.The zinc-finger protein Zat12 plays a central role in reactive oxygen and abiotic stress signaling inArabidopsis[J].Plant Physiol,2005,139(2):847-856.
11.Zhang C,Zhang F,Zhou J,et al.Overexpression of a phytochrome-regulated tandem zinc finger protein gene,OsTZF1,confers hypersensitivity to ABA and hyposensitivity to red light and far-red light in rice seedlings[J].Plant cell reports,2012,31(7):1333-1343.
12.Huang J,Sun S J,Xu D Q,et al.Increased tolerance of rice to cold,drought and oxidative stresses mediated by the overexpression of a gene that encodes the zinc finger protein ZFP245[J].Biochemical and Biophysical Research Communications,2009,389(3):556-561.
13.Xu D Q,Huang J,Guo S Q,et al.Overexpression of a TFIIIA-type zinc finger protein gene ZFP252 enhances drought and salt tolerance in rice(OryzasativaL.)[J].FEBS Letter,2008,582(7):1037-1043.
14.Sun S J,Guo S Q,Yang X,et al.Functional analysis of a novel Cys2/His2-type zinc finger protein involved in salt tolerance in rice[J].J Exp Bot,2010,61(10):2807-2818.
15.Yang L,Liu Q,Liu Z,et al.Arabidopsis C3HC4-RING finger E3 ubiquitin ligase AtAIRP4 positively regulates stress-responsive abscisic acid signaling[J].J Integr Plant Biol,2015,Apr24,doi:10.1111/jipb.12364.
16.Ma K,Xiao J,Li X,Zhang Q,et al.Sequence and expression analysis of the C3HC4-type RING finger gene family in rice[J].Gene,2009,444:33-45.
17.Sun J,Jiang H,Xu Y,et al.The CCCH--type zinc finger proteins AtSZF1 and AtSZF2 regulate salt stress responses inArabidopsis[J].Plant and Cell Physiology,2007,48(8):1148-1158.
18.Lin P C,Pomeranz M C,Jikumaru Y,et al.TheArabidopsistandem zinc finger protein AtTZF1 affects ABA- and GA-mediated growth,stress and gene expression responses[J].Plant J,2011,65:253-268.
19.Regier N,Frey B.Experimental comparison of relative RTqPCR quantification approaches for gene expression studies in poplar[J].BMC Molecular Biology,2010,11:57.
20.Sugano S,Kaminaka H,Rybka Z,et al.Stress-responsive zinc finger gene ZPT2-3 plays a role in drought tolerance in petunia[J].Plant J,2003,36:830-841.
21.Peng X,Zhao Y,Cao J,et al.CCCH-type zinc finger family in Maize:Genome-Wide identification,classification and expression profiling under abscisic acid and drought treatments[J].PLoS ONE.2012,7(7):e40120.doi:10.1371/journal.pone.0040120.
22.Vierstra R D.The expanding universe of ubiquitin and ubiquitin-like modifiers[J].Plant Physiol,2012,160(1):2-14.
23.Lyzenga W J,Stone S L.Abiotic stress tolerance mediated by protein ubiquitination[J].Journal of Experimental Botany,2011,63:599-616.
24.Yang X,Sun C,Hu Y,et al.Molecular cloning and characterization of a gene encoding RING zinc finger ankyrin protein from drought-tolerantArtemisiadesertorum[J].Journal of Bioscience,2008,33:103-112.
This work was funded by the National Natural Science Foundation of China(Grant No.31470676) and by the Ministry of Science and Technology of China under the “863” program(Grant No.2013AA102704-0104)
introduction:WANG Yuhang(1990—),female,Master,student is specialized in plant molecular biology.
date:2015-12-15
IdentificationandExpressionAnalysisofGenesEncodingZincFingerProteinsinBetulaplatyphylla
WANG Yu-Hang QIN Lin-Lin LI Li*
(State Key Laboratory of Tree Genetics and Breeding,Northeast Forestry University,Harbin 150040)
Zinc finger proteins constitute a large family of transcription factors and numerous members of them play diverse roles in many biological processes, including plant growth, development and stress responses. Based on their structural and functional diversities they were classified into nine types including C2H2, C8, C6, C3HC4, C2HC, C2HC5, C4, CCCH and C4HC3. We studied the genes inBetulaplatyphylladatabase by the sequences and homology comparison of genes encoding zinc finger proteins involving in stress responses from other organisms. A total of ten genes (designated asBpZFP1 toBpZFP10), of which four were C2H2type, five were C3HC4-RING type, one was CCCH type were identified. By quantitative RT-PCR assay, we analyzed the expression patterns ofBpZFPsin roots.BpZFP2,BpZFP3,BpZFP4,BpZFP5 andBpZFP8 were induced under high-salt and drought treatment significantly indicating their involvement in stress response in Birch.
Betulaplatyphylla;abiotic stress;zinc finger transcription factor;expression analysis
国家自然科学基金(31470676)和科技部“863”课题(2013AA102704-0104)
王宇航(1990—),女,硕士研究生,主要从事植物分子生物学研究。
* 通信作者:E-mail:lili@nefu.edu.cn
2015-12-15
* Corresponding author:E-mail:lili@nefu.edu.cn
S792.153
A
10.7525/j.issn.1673-5102.2016.03.012

