刚毛柽柳Na+/H+逆向转运蛋白基因的克隆与表达分析
贾园园 张春蕊 王玉成 杨传平 王 超
(东北林业大学林木遗传育种国家重点实验室,哈尔滨 150040)
刚毛柽柳Na+/H+逆向转运蛋白基因的克隆与表达分析
贾园园 张春蕊 王玉成 杨传平 王 超*
(东北林业大学林木遗传育种国家重点实验室,哈尔滨 150040)
通过对刚毛柽柳(Tamarixhispida)转录组分析,鉴定获得一个Na+/H+逆向转运蛋白基因,命名为ThSOS1。ThSOS1基因cDNA全长3 917 bp,开放阅读框长3 498 bp,编码1 165个氨基酸,编码蛋白相对分子质量128.8 kDa,理论等电点(PI)为6.42。疏水性分析预测ThSOS1基因编码蛋白N端具有10个跨膜结构域,C端具有一个较长的亲水尾部。ThSOS1氨基酸序列与长叶红砂、藜麦、盐地碱蓬等植物的SOS1序列同源性较高,分别可达92%,73%,72%。系统发育分析表明ThSOS1为质膜型Na+/H+逆向转运蛋白,与液泡膜型Na+/H+逆向转运蛋白属于不同分支。实时荧光定量RT-PCR分析显示,该基因受高盐、干旱诱导上调表达,暗示ThSOS1可能在刚毛柽柳抗旱耐盐过程中发挥重要作用。
刚毛柽柳;Na+/H+逆向转运蛋白;胁迫响应;基因表达
土壤盐渍化是影响农业生产和生态环境的重要因素,盐害能造成植物体离子失衡和渗透胁迫,是植物最重要的非生物胁迫之一[1]。其中的离子毒害主要是指高浓度的Na+对植物的毒害作用[2]。Na+/H+逆向转运蛋白是植物耐盐的关键因子[3],它利用质膜或液泡膜H+-ATPase和H+-PPase建立的跨膜质子梯度为驱动力,将细胞内过多的Na+从质膜向胞外排放或将Na+富集在液泡内区隔化,使细胞质维持着较低的Na+水平,避免了过多的Na+对细胞的毒害作用,同时也维持了细胞的渗透平衡[4~5]。
植物Na+/H+逆向转运蛋白主要分为两类:质膜型和液泡膜型。质膜型Na+/H+逆向转运蛋白参与植物的Na+外排,液泡膜Na+/H+逆向转运蛋白功能是将Na+运入液泡区隔化以维持细胞质内正常的Na+浓度。植物质膜Na+/H+逆向转运蛋白首次于1976年在大麦中发现[6]。1985年Blumwald和Poole在红甜菜的根部贮藏组织中发现了液泡膜Na+/H+逆向转运蛋白[7]。到目前为止,已经在拟南芥(Arabidopsisthaliana)[8]、水稻(Oryzasativa)[9]、番茄(Lycopersiconesculentum)[10]和油菜(Brassicanapus)[11]等多种植物中分离得到了Na+/H+逆向转运蛋白基因。Shi等在拟南芥中克隆出了一个细胞质膜Na+/H+逆向转运蛋白SOS1,这个基因过量表达后能降低芽中的Na+含量,增强愈伤组织的耐盐性[12]。随后的研究又证实了SOS1基因过量表达,可以显著提高拟南芥对盐胁迫的耐性[13]。棉花(Gossypiumhirsutum)Na+/H+逆向转运蛋白基因GhNHX1过量表达能提高转基因烟草的耐盐性[14]。这些研究都表明Na+/H+逆向转运蛋白在植物耐盐过程中具有重要作用。
目前,对植物中Na+/H+逆向转运蛋白的研究主要集中在一些模式植物中,对木本植物的研究较少,对盐生木本植物的研究更少。刚毛柽柳(Tamarixhispida)属柽柳科(Tamaricaceae)柽柳属(Tamarix),灌木,作为优良的防风固沙植物,其耐干旱、盐碱、贫瘠、风蚀和沙埋[15],广泛分布于砾石戈壁、粘土、砂土、流沙及各种不同程度的盐渍化土壤。这表明刚毛柽柳体内有着一套非常有效的抗逆系统,是研究木本植物耐盐分子机理、分离重要耐盐功能基因的理想物种之一。本实验通过对刚毛柽柳转录组测序数据进行分析,克隆获得了一条Na+/H+逆向转运蛋白基因(ThSOS1)的全长cDNA序列,并对该序列进行了生物信息学分析。同时,利用实时定量RT-PCR技术分析了其在盐和干旱胁迫下不同时间点的表达模式,为进一步了解ThSOS1在柽柳非生物胁迫应答中的功能奠定基础。
1 材料与方法
1.1 试验材料及处理方法
将刚毛柽柳的种子播种于V泥炭土∶V沙=2∶1的混合土壤中,在平均温度24℃,光照时间14 h·d-1,相对湿度为70%~75%的温室中培养。待幼苗生长至苗高3~4 cm后,选取生长正常且大小一致的幼苗分别用水(对照组)、0.4 mol·L-1的NaCl溶液和20%的PEG6000溶液浇灌,在胁迫处理3、6、9、12、24、48、72、96 h后,取刚毛柽柳的根部组织和地上部组织,用液氮速冻后保存在-80℃冰箱,用于实时荧光定量RT-PCR分析,每个处理重复3次。
1.2 ThSOS1基因的克隆与序列分析
通过对刚毛柽柳转录组测序数据的分析,根据Unigenes功能注释结果查找获得一条Na+/H+逆向转运蛋白基因(ThSOS1),对其全长序列设计引物进行PCR,目的条带经胶回收纯化后,连接到pMD18-T载体上,进一步测序确定所获得的序列的准确性。
用在线工具ORF Finder程序确定ThSOS1基因序列的开放读码框(http://www.ncbi.nlm.nih.gov/gorf.html);利用序列处理在线工具包(SMS)将其基因序列翻译成氨基酸;利用BioEdit软件进行氨基酸序列的同源性分析;运用MEGA5.1软件构建系统进化树;用ProtParam程序进行蛋白质分子质量和等电点的预测(http://www.ncbi.n1m.nih.gov/gorf.htm1);利用Protscal程序进行蛋白质的疏水性预测分析(http://web.expasy.org/protscale/);用SOPMA软件预测ThSOS1的蛋白二级结构;使用在线工具TMpred(http://www.ch.embnet.org/software/TMPRED-form.html)和TMHMM server 2.0软件(http://www.cbs.dtu.dk/services/TMHMM/)预测ThSOS1的编码蛋白跨膜结构;利用SMART软件(http://smart.embl-heidelberg.de/smart/set_mode.cgi?NORMAL=1)进行蛋白结构域的分析;应用Psort软件(http://psort.hgc.jp/form.html)进行亚细胞定位预测。
1.3 实时荧光定量RT-PCR表达分析
用CTAB法提取刚毛柽柳根部组织和地上部组织的总RNA,经DNaseⅠ(Promega)消化除去DNA,用Prime ScriptTMRT reagent Kit(TaKaRa)反转录合成cDNA,将反应产物稀释10倍,并作为定量RT-PCR的模板。
根据ThSOS1全长cDNA序列设计定量引物,内参基因分别是β-actin(FJ618517)、α-tubulin(FJ618518)、β-tubulin(FJ618519),引物序列见表1。利用MJ OpticonTM2实时荧光定量PCR仪(Bio-Rad,Hercules,CA)对基因的表达模式进行分析,实时荧光定量RT-PCR使用的试剂盒是Trans Start Top Green qPCR Super Mix(Trans Gen),反应体系是:2×TransStart Top Green qPCR SuperMix 10 μL,上游引物和下游引物(10 μmol·L-1)各1 μL,稀释后的模板cDNA 2 μL,加灭菌去离子水补足体积至20 μL。反应程序为:94℃预变性30 s;94℃变性12 s,58℃退火30 s,72℃延伸45 s,79℃读板1 s,45个循环。待PCR反应结束后,将反应温度以0.5℃·s-1的速度从55℃升到99℃。每个样品重复3次,用2-△△Ct方法进行基因的相对定量分析[16]。
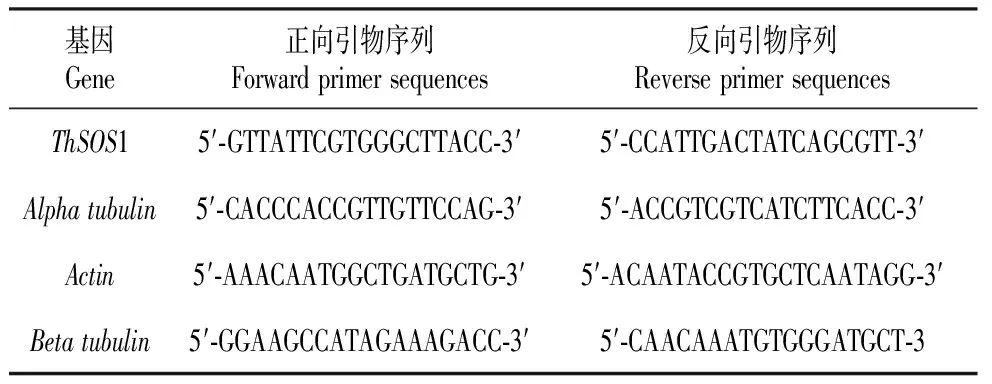
表1 实时定量RT-PCR引物序列
2 结果与分析
2.1 ThSOS1基因全长cDNA的获得及序列分析
通过对刚毛柽柳转录组数据的分析,根据基因功能注释结果查找获得一条编码Na+/H+逆向转运蛋白基因序列,对其进行重新测序,最终获得刚毛柽柳Na+/H+逆向转运蛋白基因全长cDNA序列,命名为ThSOS1。将该基因序列用ORF founder程序进行分析发现它的开放阅读框长3 498 bp,编码1 165个氨基酸。5′非编码区(UTR)长185 bp,3′非编码区(UTR)长234 bp(图1)。
ProtParam程序预测ThSOS1编码蛋白的相对分子质量128.8 kDa,理论等电点(PI)为6.42。疏水性分析发现,ThSOS1的N末端部分有一个很强的疏水性结构,而C末端的亲水性较强。按照氨基酸分值越高疏水性越强,分值越低亲水性越强的原则,发现整个肽链中疏水性氨基酸多于亲水性氨基酸,整个多肽链表现为疏水性(图2:A),因此ThSOS1属于疏水性蛋白,符合膜蛋白的特性。二级结构预测表明ThSOS1蛋白含有479个α-螺旋(占41.12%)、348个随意卷曲(占29.87%)、234个延伸链(占20.09%)和104个β-转角(占8.93%),其中二级结构主要由α-螺旋、随意卷曲和延伸链交错构成(图2:C)。跨膜结构预测表明ThSOS1包含有10个完全跨过液泡膜的跨膜结构域(图2:B),这与已报道的动植物Na+/H+逆向转运蛋白的跨膜区域数(10~12)一致。经SMART软件分析,发现ThSOS1蛋白在第32~448个氨基酸位点处存在典型的“Na+/H+Exchanger”蛋白结构域,E-值1.6e-64,表明ThSOS1属于Na+/H+逆向转运蛋白家族成员。
2.2 ThSOS1蛋白的亚细胞定位分析
应用Psort软件(http://psort.hgc.jp/form.html)对ThSOS1蛋白的亚细胞定位进行预测,结果显示其可能定位于质膜、内质网、液泡和高尔基体(表2)。ThSOS1定位于质膜的概率最高,为69.6%,定位于内质网、液泡和高尔基体的概率都相对较小,表明ThSOS1可能定位于细胞质膜上,在调控离子跨质膜转运过程中发挥重要作用。

表2 ThSOS1蛋白亚细胞定位
2.3 同源比对与系统树构建
将ThSOS1的氨基酸序列与其他植物Na+/H+逆向转运蛋白的氨基酸序列进行同源性比较,结果显示ThSOS1与长叶红砂RtSOS1(Reaumuriatrigyna,AGW30208.1)、藜麦CqSOS1B(Chenopodiumquinoa,ACN66494.1)、盐地碱蓬SsSOS1(Suaedasalsa,AHJ14584.1)、冰叶日中花McSOS1(Mesembryanthemumcrystallinum,ABN04858.1)、菠菜SoSOS1(Spinaciaoleracea,CDL70805.1)和大叶补血草LgSOS1(Limoniumgmelinii,ACF05808.1)的氨基酸序列都具有较高同源性,与同一科的长叶红沙Na+/H+逆向转运蛋白RtSOS1的同源性最高,达92%,和其他植物的质膜Na+/H+逆向转运蛋白也有较高的同源性,同源性在70%~73%(图3)。
对不同植物的Na+/H+逆向转运蛋白之间的遗传关系进行了系统发育分析(图4),结果显示ThSOS1与质膜型Na+/H+逆向转运蛋白的亲缘关系较近,而与液泡膜Na+/H+逆向转运蛋白亲缘关系较远,表明ThSOS1为刚毛柽柳的一个质膜型Na+/H+逆向转运蛋白。

图1 刚毛柽柳ThSOS1基因cDNA序列及其推测的氨基酸序列Fig.1 ThSOS1 nucleotide sequence and deduced amino acid sequence in T.hispida
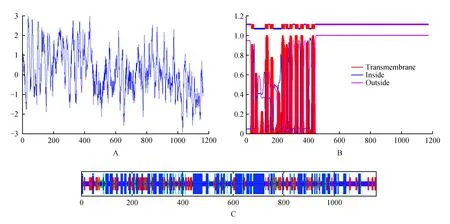
图2 刚毛柽柳ThSOS1蛋白的生物信息学分析 A.疏水性分析;B.跨膜结构分析;C.二级结构预测Fig.2 Bioinformatics analysis of ThSOS1 protein in T.hispida A. Hydrophobicity analysis; B. Transmembrane structure prediction; C. Secondary structure prediction
2.4 ThSOS1基因在胁迫条件下的表达分析
为了进一步了解ThSOS1对逆境胁迫的响应情况,利用实时荧光定量RT-PCR技术对NaCl和PEG胁迫下柽柳ThSOS1的表达模式进行了分析。
在NaCl胁迫下,ThSOS1在根部和地上部组织中的表达模式相似。随着胁迫时间的延长,ThSOS1的相对表达量都逐渐上升,并在胁迫24 h后达到最高峰,48 h后逐渐下降。其中,ThSOS1在地上部组织中的相对表达量变化较大,在24 h时相对表达量是正常生长状态下的22倍。
与NaCl胁迫下的表达模式相似,PEG胁迫下ThSOS1在柽柳的根部和地上部都被诱导上调表达。其中,根中的相对表达量在胁迫12 h后达到一个小的峰值,之后略有下降;在胁迫48 h时又逐渐增加,并在72 h时表达量上升到最高水平。ThSOS1在地上部的表达模式与根部相似,在胁迫9 h达到一个小高峰,之后下降,24 h迅速上升,在48 h达到峰值。另外,从图2中还可以明显看出高盐和干旱胁迫下ThSOS1在柽柳的根部和地上部均是上调表达,且在地上部的相对表达量显著高于根。这些结果表明了ThSOS1可能在刚毛柽柳的耐盐抗旱机制中具有重要作用,并且主要在叶和茎中发挥功能。
3 讨论
植物质膜Na+/H+逆向转运蛋白能够将细胞质中过多的Na+排出胞外,从而减轻Na+对细胞代谢活动及细胞器的破坏,使植物更好地适应盐渍生境[17~18]。因此,质膜Na+/H+逆向转运蛋白在植物耐盐性方面具有重要作用。已有研究表明,绝大多数植物质膜Na+/H+逆向转运蛋白基因的开放阅读框(ORF)长度为3 410~3 500 bp,编码1 129~1 169个氨基酸残基,推测分子量均为127 kDa左右[19~21]。本研究首次从刚毛柽柳中克隆到了Na+/H+逆向转运蛋白基因ThSOS1,预测分析表明其开放阅读框长3 498 bp,编码1 165个氨基酸,分子量为128.8 kDa,氨基酸序列包含典型的“Na+/H+Exchanger”结构域,ThSOS1符合Na+/H+逆向转运蛋白家族成员的序列特征。亚细胞定位分析显示,ThSOS1蛋白定位于质膜的概率最大,系统进化树分析也发现ThSOS1与质膜Na+/H+逆向转运蛋白的亲缘关系最近,而与液泡膜Na+/H+逆向转运蛋白亲缘关系较远,表明ThSOS1为质膜型Na+/H+逆向转运蛋白,在刚毛柽柳的耐盐反应中介导Na+的外排来维持细胞内的离子平衡。跨膜结构预测表明ThSOS1包含有10个完全跨膜结构域,与已报道的动植物和微生物的Na+/H+逆向转运蛋白的跨膜区域数(10~12)一致,跨膜区氨基酸序列与其他植物的Na+/H+逆向转运蛋白之间存在很高的相似性,表明跨膜区是高度保守的。ThSOS1蛋白的C末端有一个较长的亲水性区域,使SOS1能够与其它逆境相关调控因子发生互作,从而适应盐渍环境[22]。

图3 刚毛柽柳ThSOS1与其他植物SOS1同源序列的比较Fig.3 Comparison of ThSOS1 in T.hispida with other plant SOS1 genes

图4 几种植物Na+/H+逆向转运蛋白的系统进化关系分析 CqSOS1B.藜麦(ACN66494.1);SoSOS1.菠菜(CDL70805.1);SsSOS1.盐地碱蓬(AHJ14584.1);McSOS1.冰叶日中花(ABN04858.1);SpSOS1.海马齿(AFX68848.1);RtSOS1.长叶红沙(AGW30208.1);LgSOS1.大叶补血草(ACF05808.1);AtNHX1.拟南芥(NP_198067.1);SeNHX1.盐角草(AAN08157.1);ZxNHX1.霸王(ABU92562.1);PeNHX1.胡杨(ABY86892.1)Fig.4 Phylogenetic tree analysis of Na+/H+ antiporters from various plant species CqSOS1B. Chenopodium quinoa(ACN66494.1);SoSOS1. Spinacia oleracea(CDL70805.1);SsSOS1. Suaeda salsa(AHJ14584.1);McSOS1. Mesembryanthemum crystallinum(ABN04858.1);SpSOS1. Sesuvium portulacastrum(AFX68848.1);RtSOS1. Reaumuria trigyna(AGW30208.1);LgSOS1. Limonium gmelinii(ACF05808.1);AtNHX1. Arabidopsis thaliana(NP_198067.1);SeNHX1. Salicornia europaea(AAN08157.1);ZxNHX1. Zygophyllum xanthoxylum(ABU92562.1);PeNHX1. Populus euphratica(ABY86892.1)

图5 ThSOS1基因在不同胁迫下的表达模式分析Fig.5 Expression analysis of ThSOS1 gene under several abiotic stresses
不同胁迫处理下,植物中的SOS1基因的表达模式有所差异。Shi等发现拟南芥AtSOS1在盐胁迫下表达量明显提高,而在冷胁迫和外源ABA处理下表达却没有显著变化[11];与对照组相比,水稻OsNHA1受盐胁迫诱导显著表达,而干旱处理则没有明显变化[23];NaCl胁迫处理下,小麦TaSOS1的表达水平显著增加,但PEG处理无诱导作用[24]。Song等研究发现盐胁迫可以诱导大岛野路菊CcSOS1上调表达,干旱、低温和ABA处理也对CcSOS1的表达产生了不同程度的影响[25]。本研究中,在NaCl和PEG胁迫处理后,ThSOS1基因在柽柳的根部和地上部的表达量均明显增加,特别是在地上部组织中,ThSOS1基因在胁迫12 h后持续保持高丰度的表达。ThSOS1基因能对干旱和高盐胁迫做出响应,表明其可能参与柽柳的抗旱耐盐过程。ThSOS1在柽柳地上部的表达量明显高于根部,推测其可能参与柽柳叶片中的Na+通过韧皮部向老叶的转移,或者能够促进Na+从其叶片盐腺中的分泌,但该基因在柽柳抗逆反应中的具体功能及其参与抗逆的调控机制还需要进一步的研究。植物的耐盐性是一个复杂数量性状,是多基因相互作用的结果。然而有研究表明,转单一的Na+/H+逆向转运蛋白基因能够明显地提高作物的耐盐能力。拟南芥AtSOS基因转化拟南芥后使其过表达,转基因株系内的Na+含量明显降低,耐盐能力也显著增强[12]。Yue等发现拟南芥AtSOS1基因转入番茄后,提高了转基因植株的耐盐能力[26]。将星星草基因PtNHA1转入拟南芥中,PtNHA1过表达后能增强转基因拟南芥的耐盐性,还能消除活性氧的毒害作用,提高抗氧化能力[27]。这些研究表明利用转化SOS1基因提高植物的耐盐能力,是利用基因工程方法培育耐盐植物新品种的一种有效途径[26~28]。迄今为止,对植物中质膜Na+/H+逆向转运蛋白的研究主要集中在草本植物中,对木本植物的研究较少。木本植物与草本植物在形态结构和生理特性上都有一定的区别[29],木本植物体与草本植物的质膜Na+/H+逆向转运蛋白是否具有相似功能尚不清楚。因此,了解ThSOS1的功能将为木本植物的耐盐机理的研究提供有价值的参考,同时为林木抗逆育种提供有效的基因资源和理论依据。目前,ThSOS1的植物表达载体构建正在进行中,后续将对拟南芥和刚毛柽柳进行遗传转化,对ThSOS1的功能及其活性调控机制进行深入研究。
1.Tester M,Davenport R.Na+tolerance and Na+transport in higher plants[J].Annals of Botany,2003,91(5):503-527.
2.杨月红,孙庆艳,沈浩.植物的盐害和抗盐性[J].生物学教学,2002,27(11):1-2.
3.Wang Q,Li X,Zhang H,et al.Overexpression of Na+/H+antiporter gene(nhaA) improves salt tolerance in soybean[J].Molecular Plant Breeding,2010,8(4):701-707.
4.Blumwald E,Aharon G S,Apse M P.Sodium transport in plant cells[J].Biochim Biophys Acta,2000,1465(1-2):140-151.
5.张桦,张富春,张雨良,等.新牧一号杂花苜蓿MvNHX1基因的表达分析和提高烟草耐盐性的研究[J].植物研究, 2011,31(5):550-557.
6.Ratner A,Jacoby B.Effect of K+,its counter anion,and pH on sodium efflux from Barley roots[J].Exp Physiol,1976,148:425-433.
7.Blumwald E,Poole R.Na+/H+antiport in isolated tonoplast vesicles from storage tissue ofBetavulgaris[J].Plant Physiol,1985,78(1):163-167.
8.Apse M,Aharon G,Snedden W,et al.Salt tolerance conferred by overexpression of a vacuolar Na+/H+antiporter inArabidopsis[J].Science,1999(285):1256-1258.
9.Martínez-Atienza J1,Jiang X,Garciadeblas B,et al.Conservation of the salt overly sensitive pathway in rice[J].Plant Physiol,2007,143(2):1001-12.
10.Zhang H,Blumwald E.Transgenic salt-tolerant tomato plants accumulate salt in foliage but not in fruit[J].Nature Biotechnology,2001,19:765-768.
11.Zhang H X,Hodson J N,Williams J P,et al.Engineering salt-tolerantBrassicaplants:characterization of yield and seed oil quality in transgenic plants with increased vacuolar sodium accumulation[J].Proc Natl Acad Sci,2001,98:12832-12836.
12.Shi H,Ishitani M,Kim C,et al.TheArabidopsisthalianasalt tolerance gene SOS1 encodes a putative Na+/H+antiporter[J].Proc Natl Acad Sci,2000,97(12):6896-6901.
13.Shi H,Lee B,Wu S,et al.Overexpression of a plasma membrane Na+/H+antiporter gene improves salt tolerance inArabidopsisthaliana[J].Nature Biotechnology,2003,21(1):81-85.
14.Wu C A,Yang G D,Meng Q W.The cottonGhNHX1 gene encoding a novel putative tonoplast Na+/H+antiporter plays an important role in salt stress[J].Plant and Cell Physiol,2004,45:600-607.
15.杨维康,张道远,张立运,等.新疆主要柽柳属植物(TamarixL.)的生态类型划分与生境相似性研究[J].干旱区地理,2004,27(2):186-192.
16.Livak K J,Schmittgen T D.Analysis of relative gene expression data using real-time quantitative PCR and the 2(-Delta Delta C(T)) Method[J].Methods,2001,25(4):402-408.
17.Zhu J.Salt and drought stress signal transduction in plants[J].Annu Rev Plant Biol,2002,53:247-273.
18.Munns R,Tester M.Mechanisms of salinity tolerance[J].Annu Rev Plant Biol,2008,59:651-681.
19.马清,包爱科,伍国强,等.质膜Na+/H+逆向转运蛋白与植物耐盐性[J].植物学报,2011,46(2):206-215.
20.Pardo J M,Quintero F J.Plants and sodium ions:keeping company with the enemy[J].Genome Biol,2002,3(6):1-4.
21.Cosentino C,Fischer-Schliebs E,Bertl A,et al.Na+/H+antiporters are differentially regulated in response to NaCl stress in leaves and roots ofMesembryanthemumcrystallinum[J].New Phytol,2010,186(3):669-680.
22.Zhu J.Genetic analysis of plant salt tolerance usingArabidopsis[J].Plant Physiol,2000,124:941-948.
23.Martinez-Atienza J,Jiang X,Garciadeblas B,et al.Conservation of the Salt Overly Sensitive Pathway in Rice[J].Plant Physiol,2007,143(2):1001-1012.
24.Xu H,Jiang X,Zhan K,et al.Functional characterization of a wheat plasma membrane Na+/H+antiporter in yeast[J].Arch Biochem Biophys,2008,473(1):8-15.
25.Song A,Lu J,Jiang J,et al.Isolation and characterisation ofChrysanthemumcrassumSOS1,encoding a putative plasma membrane Na+/H+antiporter[J].Plant Biol,2012,14:706-713.
26.Yue Y,Zhang M,Zhang J,et al.SOS1 gene overexpression increased salt tolerance in transgenic tobacco by maintaining a higher K+/Na+ratio[J].J Plant Physiol,2012,169(3):255-261.
27.Wang X,Yang R,Wang B,et al.Functional characterization of a plasma membrane Na+/H+antiporter from alkali grass(Puccinelliatenuiflora)[J].Mol Biol Rep,2011,38(7):4813-4822.
28.郑琳琳,张慧荣,贺龙梅,等.唐古特白刺质膜Na+/H+逆向转运蛋白基因的克隆与表达分析[J].草业学报,2013,22(4):179-186.
29.宋鑫,董京祥,李开隆,等.柽柳中一种新型硫氧还蛋白H基因的克隆与鉴定[J].植物研究,2015,35(3):340-346.
CloningandExpressionAnalysisofaPlasmaMembraneNa+/H+AntiporterGeneinTamarixhispida
JIA Yuan-Yuan ZHANG Chun-Rui WANG Yu-Cheng YANG Chuan-Ping WANG Chao*
(State Key Laboratory of Tree Genetics and Breeding,Northeast Forestry University,Harbin 150040)
A full length cDNA of a Na+/H+antiporter gene (namedThSOS1) gene was isolated from the transcriptome cDNA librarys ofTamarixhispida.ThSOS1 was 3 917 bp in length, including an open reading frame of 3 498 bp which was predicted to encode a polypeptide of 1 165 amino acids. The estimated molecular weight and isoelectric point of the putative protein were 128.8 kDa and 6.42, respectively. By hydrophobic cluster analysis,ThSOS1 contained 10 potential transmembrane domains within the N- terminal protion and a long hydrophilic cytoplasmic tail in the C-terminal protion. By multiple sequence alignment,ThSOS1 show 92%, 73%, and 72% identities in amino acid sequence to plasma membrane Na+/H+antiporter genes fromReaumuriatrigyna,ChenopodiumquinoaandSuaedasalsa. By phylogenetic analysis,ThSOS1 was more related to the plasma membrane-type Na+/H+antiporter and clustered distantly with the vacuolar-typed Na+/H+antiporter. By quantitative real-time PCR assay, the mRNA levels ofThSOS1 was significantly up-regulated inT.hispidaunder NaCl and PEG treatments. Therefore,ThSOS1 might play an important role in salt and drought tolerance ofT.hispida.
Tamarixhispida;Na+/H+antiporter;stress responses;gene expression
国家自然科学基金项目(31300571);教育部博士点基金资助项目(20130062120012)
贾园园(1988—),女,硕士研究生,主要从事林木抗逆机理研究。
* 通信作者:E-mail:wzyrgm@163.com
2015-11-13
S793.5
A
10.7525/j.issn.1673-5102.2016.03.010

